一種用于構建表達載體的合成生物學數據庫
方 剛
(西安文理學院,生物與環境工程學院, 西安 710065)
?
一種用于構建表達載體的合成生物學數據庫
方剛
(西安文理學院,生物與環境工程學院, 西安 710065)
摘要:由于基因測序及DNA合成技術與工具的突破性進展,生物工程正在加速發展,導致合成生物學的出現。本文介紹了一種用于構建表達載體的合成生物學數據庫。闡述了如何利用MySQL數據庫管理系統(DBMS)對合成生物學數據庫gene_bank進行查詢,并借助BioEdit軟件對其中的多克隆位點(MCS)進行序列分析,通過查詢與分析找出這一合成生物學數據庫的特點。
關鍵詞:合成生物學;數據庫;MySQL查詢
由Science雜志數據庫搜索查詢,該刊最早于1911年33卷有兩篇文章出現“合成生物學”一詞。由Scirus搜索引擎搜索,合成生物學一詞最早出現于1911年7月8日著名醫學刊物《柳葉刀》發表的一篇書評中。后來雖然斷斷續續出現過多次,但在1980年第一次以“基因外科術:合成生物學的開始”為題出現在德文刊物的一篇長篇論文[1]。隨著人類基因組計劃的完成,2000年以后,合成生物學一詞在學術刊物及互聯網上逐漸大量出現。對于近幾年合成生物學的突然變熱,不同的人對其有不同的解釋。著名科普刊物 The Scientist為此專門采訪了一些合成生物學領域的參與者[2],其中加州大學伯克利分校(UCB)的化學工程教授Keasling說:合成生物學正在用“生物學”進行工程化,就像用“物理學”進行“電子工程”,用“化學”進行“化學工程”一樣。目前合成生物學與傳統的重組DNA技術之間的界限仍然是模糊的。從根本上說,合成生物學正在利用獲得的生物“零件”進行下一層次的工作——對細胞進行實際的工程化。是利用我們所確信的一些標準“零件”構造新生物系統的工程。“合成生物學組織”網站上公布的合成生物學的定義包括兩條路線:(1)新的生物零件、組件和系統的設計與建造;(2)對現有的、天然的生物系統的重新設計[3]。
合成生物學建立在“標準生物組件(BioBrick)”的基礎上[4-5],所謂的標準生物構件就是一些具有生物學意義的DNA分子。合成生物學就是在活細胞中使用這些可互換的標準生物組件重新組合構造新的生物系統,并加以操縱來實現某種特定的生理功能。隨著生物學的工程化和現代分子生物學的迅猛發展,這些所謂的“BioBrick”是以海量的形式出現的,對這些標準生物組件信息的組織、存儲和操控必然依賴現代的數據庫技術。本文就是通過使用現代數據庫技術從常用的質粒表達載體中提取“生物組件”信息,將這些載體拆解成“零件”,提取信息加以組織、存儲,然后期望使用這些零件構建新的載體。
1常用質粒載體數據庫gene_bank
數據庫是信息系統的核心,在信息社會中占據著舉足輕重的地位。數據庫技術主要研究如何科學地組織、存儲和管理數據庫中的數據。簡單地說,數據庫是存儲、管理數據的容器:嚴格地說,數據庫是“按照某種數據結構對數據進行組織,存儲和管理的容器”[6]。合成生物學信息的組織、存儲、管理以及操控就是依賴于現代的數據庫技術。
Gene_bank數據庫是源于常用質粒載體的數據庫,這些質粒可以用來傳染12種不同的宿主細胞(包括大腸桿菌、釀酒酵母、植物細胞、哺乳動物細胞、昆蟲細胞等),這一信息在數據庫中已予以存儲。這是一個“生物組件”數據庫,我們可以使用它來構造設計新的載體。每一個質粒載體的genbank文檔中的FEATURES區域包含具有生物學意義的序列,可以用作開發標準生物組件(BioBrick)[7]。
通過Perl語言編寫程序,可以提取FEATURES區域的信息。提取的信息包括features名,所有的注釋信息即note,重要的是提取各個features的序列信息,需要按照各features的起止號碼,根據ORIGIN區域的序列信息提取各個features的準確序列。將提取的信息輸入MySQL數據庫。輸入時產生兩個表,其中plasmids表包括了每個質粒的總體信息(包括完整的genbank文檔)。Features表中包含了從質粒genbank文檔FEATURES區域提取的信息,其中FEATURES名被定義為feature_qualifier,而第一個注釋即note在數據庫中被定義為feature_name (FEATURES名),第二個note被定義為description即FEATURES的描述,表中還包含各FEATURES的起止號碼和相對應的準確序列信息。genebank_feature表對各個feature_qualifier的含義進行了描述。snapgene_qualifier表和genocad_qualifier表是對各個feature_qualifier在兩種流行的合成生物學軟件Snapgene[8]和GenoCAD[8]中的含義的描述,表結構與genebank_feature的表結構基本一致。Gene_bank這一關系型數據庫中最重要的是features表,這個表里包含就是從質粒載體“拆解”下來的合成生物學“零件”信息,可以使用這些信息來開發BioBrick。該數據庫源于常用質粒載體,與標準生物組件(BioBrick)數據庫的組織與結構有所不同[5-6],其中最大的不同在于它源于成熟商業化的質粒可用來開發新的商用載體,而標準生物組件數據庫主要支持iGEM(International Genetically Engineered Machine)競賽。
2gene_bank數據庫的SQL查詢
利用MySQL查詢gene_bank數據庫的操作如下。
2.1打開MySQL
Windows+R——>cmd(進入DOS)——>mysql -u root -p——>password
2.2顯示庫表
show databases;
use gene_bank;
show tables;
經過查詢可知,gene_bank數據庫中共有5張表,分別是features,genebank_features,genocad_qualifier,plasmids,snapgene_qualifier。
2.3查詢表結構
2.3.1Desc plasmids
Plasmid(質粒)表中共有7個字段,如圖1所示。其中plasmid_id即質粒號;plasmid_name即質粒名;definition是對質粒的基本描述;sequence是質粒的序列信息;complete_genbank_text區存儲完整的質粒genbank文檔;is_circular表示如果該質粒是環形閉合的該區域值為1否則為0;origin表示質粒來源的數據集。
2.3.2Desc features
Features(特性)表中共有10個字段,如圖2所示。其中feature_id即features號;feature_name即features名稱;description是對該features的基本描述;feature_qualifier表示該features是屬于哪一類;complement表示該features是否是反向互補序列,如果是該區域值取1否則取0;start表示該features在它所屬質粒中序列的起始號;end表示該features在質粒中序列的終止號;sequence表示該features的序列信息;plasmid_id表示該features所屬質粒的號;flag區域表示該features的序列是否含有除a,g,c,t之外的特殊字符,如果含有則予以標出。
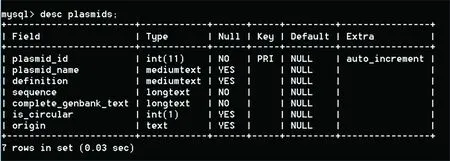
圖1 表plasmids的查詢結果Fig.1 The result of querying table plasmids

圖2 表features 的查詢結果Fig.2 The result of querying table features
2.3.3Desc genebank_features
Genebank_features表中共有3個字段,如圖3所示。其中qualifier_id表示feature_qualifier的號碼;feature_qualifier就是各個feature_qualifier的名稱;description是對各個feature_qualifier含義的解釋。
2.4查詢feature表中的總記錄
Select count(*) as totalItem from features; 17 760 結果features表中總共有17 760條記錄
Select count(*) from features where sequence is NULL; 0
Select count(distinct sequence) as totalsequence from features; 2 137sequence字段沒有NULL值,完全不同的sequence只有2 137個,因此有大量sequence是冗余的,冗余的序列信息被標識并保留。
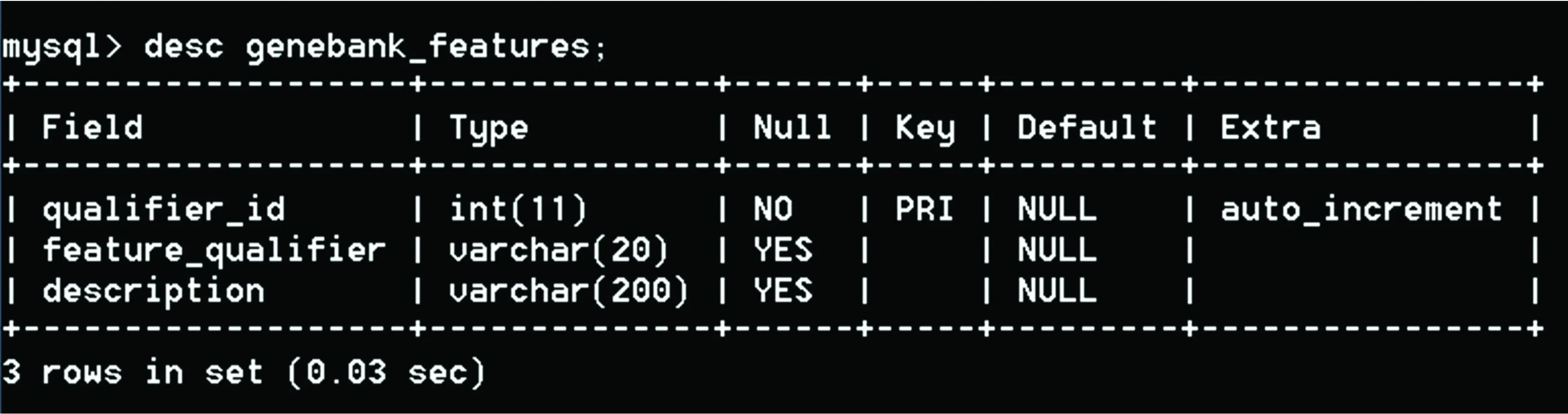
圖3 表genebank_features 的查詢結果Fig.3 The result of querying table genebank_features
2.5綜合查詢
Select feature_name,sequence,description,feature_qualifier,count(feature_id) from features group by feature_name,sequence,description,feature_qualifier having count(feature_id)>1 order by count(feature_id) desc;
通過這個語句,可以查詢到feature_name、feature_qualifier、description、sequence四者均相同的features有哪些,通過查詢可知AmpR promoter,AmpR,ori,T7 promoter是使用最多的四種features(分別是781次、516次、454次、452次)。這一查詢的意義在于知曉哪些序列使用最為普遍頻繁,為下一步開發BioBrick做準備。
Select feature_name,sequence,description,feature_qualifier, count(feature_id) from features group by feature_name,sequence, description,feature_qualifier having feature_name='MCS' order by count(feature_id) desc;
通過這個語句,可以查詢到當features是MCS(多克隆位點)時,所用序列的統計次數,可以得到使用次數最多的序列,并且feature_qualifier是misc_feature, 對于這些序列用BioEdit做了分析,可以顯示其中詳細的多克隆位點。這一查詢的意義在于知曉哪些多克隆位點使用最為普遍頻繁,可以用來提取較為有效的多克隆位點構建克隆或表達載體。
3gene_bank數據庫的意義
關于標準生物構件數據庫,最著名的莫過于麻省理工學院(Massachusetts Institute of Technology, MIT)倡導的Standard Biological Parts[9]。但是之前還少有基于成熟并常用的克隆、表達載體的數據庫[10]。Gene_bank數據庫就是源于常用質粒載體的數據庫,我們可以使用它構造新的載體。
Gene_bank數據庫源于成熟常用的商業化質粒載體,可以用來開發用作BioBrick。
Gene_bank數據庫便于合成生物學家查詢合成生物學研究所需要的數據,了解各個組件的具體信息,組合成新的生物系統。
4前景與展望
合成生物學將催生下一次生物技術革命。目前,科學家們已經不局限于非常辛苦地進行基因剪接,而是開始構建遺傳密碼,以期利用合成的遺傳因子構建新的生物體。合成生物學在未來幾年有望取得迅速進展。據估計,合成生物學在很多領域將具有極好的應用前景,這些領域包括更有效的疫苗的生產、新藥和藥物的改進、以生物學為基礎的制造、可再生能源利用、生產可持續能源、環境污染的生物治理、可以檢測有毒化學物質的生物傳感器。本文通過從常用的質粒載體中獲取序列信息,將完整的質粒序列拆成“零件”構建成數據庫,提供給合成生物學家使用。以期從這些零件中提取元素構建新的表達載體。
參考文獻
[1]HOBOM B. Gene surgery:on the threshold of synthetic biology[J]. Medizinische Klinik,1980,75(24):834-841.
[2]LUCENTINI, L. Just what is synthetic biology[J].The Scientist,2006,20(1):36.
[3]趙學明,王慶昭. 合成生物學:學科基礎、研究進展與前景展望[J]. 前沿科學, 2007,(3):56-66.
ZHAO Xueming, WANG Qingzhao. Synthetic biology: fundamentals, advances and prospect[J].Frontier Science, 2007,(3):56-66.
[4]SHETTY R P , ENDY D. Knight T F Jr. Engineering BioBrick vectors from BioBrick parts[J].Journal of Biological Engineering, 2008,2(1):5.
[5]孔祥盛. MySQL核心技術與最佳實踐(第一版)[M]. 北京: 人民郵電出版社, 2012.
KONG Xiangsheng. MySQL core technology & best practice(1sted.) [M].Beijing:Posts & Telecom Press, 2012.
[6]ADAMES N R , WILSON M L , FANG G , et al. GenoLIB:A database of standard biogical parts derived from a library of common plasmid features[J].Nucleic Acids Research, 2015 ,43(10):4823.
[7]COOLING M T , ROUILLY V , MISIRLI G , et al. Standard virtual biological parts: a repository of modular modeling components for synthetic biology[J].Bioinformatics, 2010, 26(7): 925-931.
[8]CZAR M J, CAI Y, PECCOUD J. Writing DNA with GenoCAD[J]. Nucleic Acids Research, 2009, 37(Web Server issue):W40-W47 .
[9]SMOLKE C D. Building outside of the box: iGEM and the BioBricks Foundation[J]. Nature Biotechnology, 2009, 27(12):1099-1102.
[10]CAI Y, WILSON M L, PECCOUD J. GenoCAD for iGEM: a grammatical approach to the design of standard-compliant constructs[J].Nucleic Acids Research, 2010,38(8):2637-2644.
A synthetic biology database for constructing expression vector
FANG Gang
(SchoolofBiologicalandEnvironmentalEngineering,Xi’anUniversity,Xi’an710065,China)
Abstract:Due to the breakthrough in the Gene Sequencing and DNA Synthesis Technology. Biological and genetic Engineering developed rapidly and resulted in the emergence of Synthetic Biology. A database of synthetic biology,which aims at constructing expression vector, was introduced in this paper. By using MYSQL database management system (DBMS), the Synthetic Biology database of gene_bank were queried. The sequences of multiple clone sites (MCS) were analyzed. In order to figure out some of the characteristic of this database, comprehensive analysis was carried out.
Keywords:Synthetic biology;Database;MySQL query
中圖分類號:K826.15
文獻標志碼:A
文章編號:1672-5565(2016)01-039-04
doi:10.3969/j.issn.1672-5565.2016.01.07
作者簡介:方剛,男,副教授,研究方向:合成生物信息學;E-mail : yuxiangqd@163.com.
基金項目:國家自然科學基金資助項目(61173113)。
收稿日期:2015-09-06;修回日期:2015-11-15.

