基于Miseq的好氧反硝化菌源水脫氮的種群演變
周石磊,黃廷林,張春華,白士遠,何秀秀 (西安建筑科技大學環境與市政工程學院,西北水資源與環境生態教育部重點實驗室,陜西 西安 710055)
?
基于Miseq的好氧反硝化菌源水脫氮的種群演變
周石磊,黃廷林*,張春華,白士遠,何秀秀 (西安建筑科技大學環境與市政工程學院,西北水資源與環境生態教育部重點實驗室,陜西 西安 710055)
摘要:為了研究好氧反硝化菌源水脫氮過程中水體微生物群落的演變,利用Miseq高通量測序法對投菌和對照兩系統水體樣本的微生物信息進行統計,并對兩組樣品進行了優化序列統計,OTU分布統計和分類學分析的基礎分析;以及細菌群落結構,PCA,Rank-Abundance, Hcluster, Specaccum和OTU分布的高級分析.結果顯示,投加貧營養好氧反硝化菌的源水系統的氮素得到有效去除,脫氮效果明顯;層次聚類和主成分分析顯示兩系統內的群落結構發生變化,投菌系統與對照系統主要表現為變形菌和擬桿菌門;細菌主要門類和水質參數的相關性分析得出,水質指標對兩系統群落變化作用明顯;與此同時,投菌系統中有關氮循環的細菌有上升的變化過程. Miseq高通量測序研究源水脫氮過程的微生物種群演變可行,為研究原位生物脫氮過程的水體微生物群落演變提供技術支撐.
關鍵詞:源水脫氮;好氧反硝化;Miseq測序;生物信息分析;微生物群落
? 責任作者, 教授, huangtinglin@xauat.edu.cn
20世紀80年代,Robertson等[1]在除硫和反硝化處理系統中首次分離了好氧反硝化菌(Thiosphaera pantotropha),這種新型脫氮生物的發現使人們對氮循環又有了新的認識.好氧反硝化菌[2]是在有氧條件下,利用好氧反硝化酶進行反硝化作用的一類脫氮菌.它的發現打破了反硝化只能在厭氧缺氧條件下進行的傳統反硝化的觀念,為新型生物脫氮提供了新思路.目前所分離的好氧反硝化菌主要分布于假單胞菌屬(Pseudomonas)[3]、芽孢桿菌屬(Bacillus)[4-5]、產堿菌屬(Alcaligenes)[6-7]、微枝桿菌屬(Microvirgula)[8-9]和叢毛單胞菌屬(Comamonas)[10-11].目前大多數關于好氧反硝化菌的研究集中在實驗室富營養配水培養條件下以及污廢水條件下的脫氮特性分析.如, 張培玉等[12]在富營養條件下(初始氮素109mg/L,COD 約2000mg/L)研究好氧反硝化菌假單胞菌qy37的脫氮特性;于愛茸等[13]分離的芽孢桿菌W2具有獨特的好氧反硝化作用,0.067hm2魚塘投入1Kg的該菌就能完全解決除氮問題;Joo等[14]使用Alcaligenes faecalis 處理豬場廢水,總氮去除率高于65%;Bouchez等[15]將Microvirgula aerodenitrificans包埋于藻朊酸鹽中,投加于傳統硝化反應器中,用于市政廢水處理,HN-AD脫氮率達26.8%;汪蘋等[16]在高濃度硝氮的富營養培養基條件下研究叢毛單胞菌WXZ-17的反硝化特性,初始硝氮為92mg/L培養4d后硝氮去除率達到61%.
然而,針對微污染水源水體的貧營養好氧反硝化菌的分離鑒定及其脫氮特性的研究很少.對于好氧反硝化菌進行源水脫氮過程中,微生物種群的演變更鮮有報道.1979年Kuznetsov等[17]提出寡營養細菌的概念,并定義第一次培養時能在含碳1~15mg/L培養基中生長的細菌稱為寡營養細菌,能同時在貧營養和富營養基中生長的稱為兼性寡營養細菌.Wainwright等[18]和Button等[19]研究表明,貧營養細菌對營養物質碳的親和力較大,在低營養環境中,具有較大的營養競爭優勢.結合本課題組之前關于貧營養好氧反硝化菌的研究[20-23],通過從微污染水體水庫底層沉積物中富集、馴化、分離出一批貧營養好氧反硝化菌,并通過對這些貧營養好氧反硝化菌進行源水梯度馴化,使其適應貧營養環境. 源水馴化完成后接種到微污染源水環境下,考察其在低氮微污染水體下的脫氮特性.
近年來,應用分子生物學手段來研究微生物群落結構變化成為熱點,如白玉濤等[24]應用末端限制性片段技術對反硝化和甲烷氧化細菌的群落進行分析;黨巖等[25]應用克隆文庫對應用EGSB 處理垃圾焚燒滲瀝液的微生物群落變化進行研究;郭建寧等[26]應用HiSeq 2000建Illumina測序文庫對臭氧/陶瓷膜對生物活性炭工藝的微生物群落結構進行分析.第2代高通量測序技術Illumina MiSeq以Illumina的邊合成邊測序技術為基礎,具有快速準確的分析特點,被廣泛應用于微生物群落結構及多樣性分析[27-28].結合該源水投菌實驗已發表的關于氮素以及有機物變化特性[29],本文主要針對在靜態源水條件下,應用Miseq高通量測序技術對兩系統的起始、過程以及結束3個階段的水樣進行分析,來考察投菌系統在源水脫氮過程中的微生物群落演變過程.為以后分析原位生物強化水源水體過程中對水體微生物群落影響提供技術支撐.
1 材料與方法
1.1 實驗裝置
實驗裝置為20L的玻璃瓶,外壁附有黑色塑料袋來模擬水庫的黑暗環境.取山東周村水庫源水(2013年8月)并通過充氧泵的間歇曝氣來控制反應器的溶解氧,實驗的溫度為室溫.經梯度馴化好的三株貧營養好氧反硝化菌Zoogloea sp. N299 (GenBank登錄號,KP717093)[29], Acinetobacter sp. G107 (GenBank登錄號, KP717096)[29]和Acinetobacter sp. 81Y (GenBank登錄號,KP717097)[29]分別以體積比2mL/20L.活化后菌液的好氧反硝化菌的菌密度達到106cfu/mL.通過測定反應器水樣中NO3--N, NO2--N,TN,COD和好氧反硝化菌菌落數來反應投菌的源水實驗效果. NO3--N,NO2--N和TN每5d一測;菌落數為10d一測.實驗裝置示意如圖1所示.

圖1 源水實驗裝置示意Fig.1 The schematic diagram of experiment system
1.2 微生物多樣性分析
1.2.1 DNA提取與PCR擴增 在進行投加貧營養好氧反硝化菌源水生物脫氮實驗的初始階段(0d),中間階段(30d),結束階段(60d).共3次從投菌系統和對照系統分別取1L的水樣經0.22μm醋酸纖維濾膜過濾,以備提取DNA.將投菌系統和對照系統的水樣濾膜利用水樣DNA提取試劑盒完成基因組DNA的抽提(美吉生物,上海).抽提DNA的含量和質量采用分光光度法進行檢驗.PCR擴增區域選擇樣本16S rRNA基因的V1~V3區域,合成帶有barcode的特異引物.全部樣本按照正式實驗條件進行,每個樣本3個重復,將同一樣本的PCR產物混合后用2%瓊脂糖凝膠電泳檢測,使用AxyPrepDNA凝膠回收試劑盒(AXYGEN公司)切膠回收PCR產物,Tris_HCl洗脫; 2%瓊脂糖電泳檢測.參照電泳初步定量結果, 將PCR產物用QuantiFluorTM-ST藍色熒光定量系統(Promega公司)進行檢測定量,之后按照每個樣本的測序量要求,進行相應比例的混合.
1.2.2 Miseq文庫構建和測序 應用新一代高通量測序平臺Miseq對擴增產物進行測序.Miseq文庫構建: (1)連接“Y”字形接頭; (2)使用磁珠篩選去除接頭自連片段; (3)利用PCR擴增進行文庫模板的富集; (4)氫氧化鈉變性,產生單鏈DNA片段. Miseq測序:(1)DNA片段的一端與引物堿基互補,固定在芯片上; (2)另一端隨機與附近的另外一個引物互補,也被固定住,形成“橋 (bridge)” ; (3)PCR擴增,產生DNA簇; (4)DNA擴增子線性化成為單鏈; (5)加入改造過的DNA聚合酶和帶有4種熒光標記的dNTP,每次循環只合成一個堿基; (6)用激光掃描反應板表面,讀取每條模板序列第一輪反應所聚合上去的核苷酸種類; (7)將“熒光基團”和“終止基團”化學切割,恢復3'端粘性,繼續聚合第二個核苷酸; 統計每輪收集到的熒光信號結果,獲知模板DNA片段的序列.
1.2.3 生物多樣性與分類學分析 將Miseq測序得到的PE reads首先根據overlap關系進行拼接,同時對序列質量進行質控和過濾,區分樣本后進行OTU聚類分析和物種分類學分析,基于OTU可以進行多種多樣性指數分析[30],基于OTU聚類分析結果,可以對OTU進行多種多樣性指數分析,以及對測序深度的檢測;基于分類學信息,可以在各個分類水平上進行群落結構的統計分析[31].在上述分析的基礎上,可以進行一系列群落結構和系統發育等深入的統計學和可視化分析[30,32-33].
1.3 水質分析方法
水質指標NO3--N,NO2--N和TN采用分光光度計測定[34].具體如下, NO3--N采用紫外分光光度法, NO2--N為N-(1-奈基)-乙二胺光度法, TN為過硫酸鉀氧化-紫外分光光度法.好氧反硝化菌菌落數采用平板計數法[29,35].NO3--N, NO2--N的水樣預處理為進過0.45μm醋酸纖維濾膜過濾.醋酸纖維濾膜經過超純水的3次煮沸,每次沸騰半小時去除雜質.
2 結果與討論
2.1 源水脫氮效果分析
結合之前研究結果[29]如下圖2所示,在溫度為20~27℃,DO在3~7mg/L條件下,通過60d的源水實驗,投菌系統的NO3--N從初始的(1.57± 0.02)mg/L下降到(0.42±0.01)mg/L,對照系統的NO3--N由(1.63±0.02)mg/L下降到(1.30± 0.01)mg/L;兩系統都沒有NO2--N的積累;投菌系統的TN從初始(2.31±0.12)mg/L下降到(1.09± 0.01)mg/L,對照系統的TN從(2.51±0.13)mg/L下降到(1.72±0.06) mg/L;投菌系統的好氧反硝化菌菌落數從初始的2.8×104上升到2×107cfu/mL,對照系統從7.75×103到5.5×105cfu/mL.結果表明投加好氧反硝化菌可以實現源水脫氮.
2.2 微生物多樣性分析
通過對投菌和對照兩系統3個時期取樣的樣品16S rRNA基因進行測序,在實驗初始階段投菌系統樣品和對照系統樣品各獲得了22412 和19776條有效序列,序列平均長度315bp;中間過渡階段投菌系統樣品和對照系統樣品各獲得了21433和27171條有效序列,序列平均長度318bp;結束階段投菌系統樣品和對照系統樣品各獲得了25475和16483條有效序列,序列平均長度317bp.通過Rank-abundance曲線(等級-多度曲線)[36],考察源水投菌實驗過程中細菌的物種豐度和物種均勻度.

圖2 源水實驗中氮素及好氧反硝化菌的變化情況Fig.2 The changes of nitrogen concentrations and aerobic denitrification bacteria in source water experiment
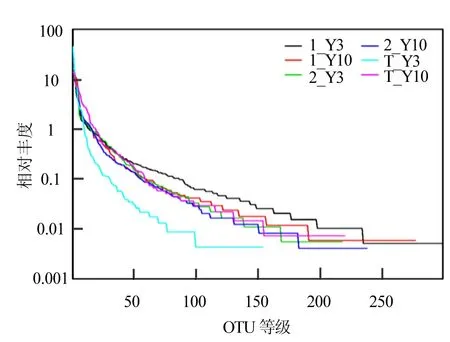
圖3 源水實驗的等級-多度曲線Fig.3 The rank-abundance curve of source water experiment
圖3中展示的為源水實驗過程中投菌系統和對照系統的Rank-abundance的變化情況.在水平方向,曲線的寬度來反映物種的豐富度,寬度越大表示豐富度越高;曲線形狀的平滑程度反映樣本中物種的均度,曲線越平緩說明物種分布越均勻.投菌系統和對照系統從源水實驗的開始階段到結束階段,細菌種群的豐富度逐漸減小.然而,源水投菌系統的Rank-abundance逐漸變陡峭,表示物種均度隨時間變得不均勻;對照系統的物種均度變化不明顯.
2.3 細菌群落結構組分
將源水實驗的投菌系統和對照系統共6個點的樣本,共116572個OTU進行注釋,然后統計在屬類別上的構成形成柱狀圖(圖4),同時分析在各個水平上的菌群結構.結果顯示這116572個 OTU主要屬于7個門類44個屬,具體結果如圖4和表1所示.

圖4 源水實驗的細菌群落結構組分Fig.4 The bacterial community during the water of bacteria and control experimental systems
兩系統的OUT主要屬于7個門類,分別是變形菌門(Protebacterice)、擬桿菌門(Bacteroidetes)、放線菌門(Actinobacteria)、藍藻門(Cyanobacteria)、酸桿菌門(Acidobacteria)、裝甲菌門(Armatimonadetes)和其他少數細菌門類,其中主要的細菌門類為變形菌門.對照系統和投菌系統的變形菌門從開始的32.83%和39.02%上升到實驗中間階段的87.55%和86.24%,到實驗結束時變為93.05%和94.97%.結果顯示,在源水脫氮過程中,對照和投菌兩系統的細菌菌落在門的類別上變化相差不大.在變形菌門類中對照和投菌兩系統在α-變形菌和β-變形菌變化較大.其中,對照系統中α-變形菌呈現上升趨勢,從開始階段的12.48%到中間階段的18.27%,最后到31.05%;投菌系統中α-變形菌呈現下降趨勢,從開始階段的19.12%到中間階段的11.16%,最后到7.56%.而兩系統中β-變形菌都是上升趨勢,對照和投菌系統從開始的19.20%和18.53%上升到實驗中間階段的49.80%和86.39%.其他類的變形菌變化不大.
在源水脫氮實驗過程中,對照和投菌兩系統的細菌群落分析得出44個屬. 投菌源水實驗系統的開始階段主要類別如下, SubsectionIII_ FamilyI_unclassified (32.74%)、LD12_freshwater_ group_norank (14.52%)、Azospira (8.43%)、CL500-29_marine_group(5.85%)、Polynucleobacter (3.81%)、hgcI_clade (2.52%)、CL500-3 (2.23%);中間階段時期主要類別如下, Hydrogenophaga (42.70%)、Betaproteobacteria_unclassified (10.56%)、Comamonadaceae_unclassified (8.26%)、NS11-12_marine_group_norank (7.53%)、Limnohabitans (4.20%)、Novosphingobium (3.35%);結束階段時期主要類別如下, Limnohabitans (59.62%)、Aquincola (19.78%)、Reyranella (5.92%)、Comamonadaceae_ unclassified (4.49%).
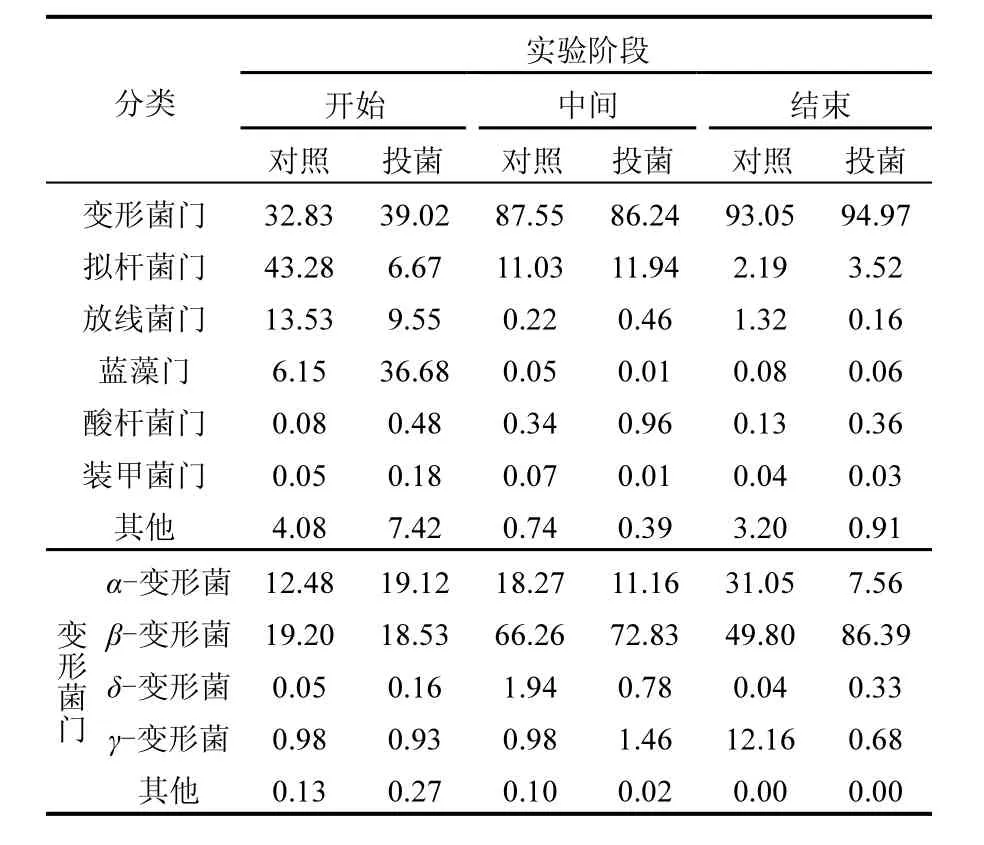
表1 源水實驗中細菌菌落結構變化情況(%)Table 1 The bacterial community during the source water experiment (%)
對照系統開始階段主要類別如下, Flavobacterium (40.36%)、Azospira (11.23%)、LD12_freshwater_group_norank (10.31%)、CL500-29_marine_group (9.40%)、SubsectionIII_ FamilyI_unclassified (5.03%)、hgcI_clade (2.96%)、Polynucleobacter (2.43%);中間階段時期主要類別如下, Comamonadaceae_unclassified (40.89%)、Aquincola (17.06%)、Reyranella (8.59%)、Candidatus_Aquirestis (4.22%)、 Sphingomonadaceae_unclassified (3.54%)、Betaproteobacteria_unclassified (3.36%);結束階段時期主要類別如下, Limnohabitans (22.71%)、Methyloversatilis (12.63%)、Sphingomonadaceae_ unclassified (10.38%)、Aquabacter (6.76%)、Reyranella (5.71%)、Acinetobacter (4.74%)、Stenotrophomonas (4.69%)、Betaproteobacteria_ unclassified (4.66%)、Comamonadaceae_ unclassified (3.34%)、Hydrogenophaga (2.76%).
結果顯示,與氮循環相關的β變形菌(Betaproteobacteria,如Limnohabitans和Acidovorax)[37]:投菌系統中從開始階段的0.02%,上升到中間階段的4.32%,到最后結束階段的60.19%;與此同時,對照系統的Limnohabitans從開始階段的0.05%,上升到中間階段的0.84%,到最后階段的23.53%.投菌系統的Acidovorax從開始的0.11%上升到實驗結束的0.22%;對照系統基本不存在.另一種菌為代爾夫特菌 (Delftia)與我們課題組之前從原水中分離的一株貧營養好氧反硝化菌SF9屬于同一種屬:投菌系統中從開始階段的0.65%,上升到中間階段的8.50%,最后到結束階段的4.53%;對照系統的從開始的0.38%,上升到中間階段的42.02%,最后到結束階段的3.46%.另外一β變形菌 (Aquincola)[38]:投菌系統中從開始階段的0.01%,上升到中間階段的1.55%,到最后結束階段的19.97%;與此同時,對照系統的從開始階段的0%,上升到中間階段的17.53%,到最后階段的1.34%.趙文莉等[28]研究復合填料反硝化脫氮過程的微生物群落特征時,氫噬胞菌(Hydrogenophaga)具有反硝化功能,本實驗中:投菌系統中從開始階段的0.02%,上升到中間階段的43.94%,到最后結束階段的0.73%;與此同時,對照系統的從開始階段的0.01%,上升到中間階段的2.03%,到最后階段的2.86%.不動桿菌 (Acinetobacter)與源水所投加的81Y和G107所屬種類一致:投菌系統中從開始階段的0.10%,上升到中間階段0.59%,到最后結束階段的0.00%;與此同時, 對照系統的從開始階段的0.01%,上升到中間階段的0.15%,到最后階段的4.91%.與此同時,主要門類細菌與水質參數的相關性分析如表2所示,結果顯示,投菌和對照系統在變形菌門的變化與系統的氮素參數呈現明顯的負相關,但是在擬桿菌門細菌與水質參數相關關系存在明顯差異,對照系統的正相關性更強.體現出兩系統細菌群落變化的差異性.相關性顯示,氮素去除主要表現為硝氮的下降.雖然所投加的好氧反硝化菌并沒有在細菌群落的heatmap圖發生顯著的增加,可能是由于反硝化菌的投加與水體中存在的氮循環菌發生協同作用,促進了其他好氧反硝化菌的脫氮進行.
本實驗中對照和投菌兩系統的變形菌是重要變化的細菌門類,并且變化趨勢一致.與此同時,投菌系統水體中的反硝化菌菌落數目在增加.通過Heatmap得出,具有反硝化功能的細菌,其中β變形菌(Limnohabitans, Aquincola和Acidovorax)、代爾夫特菌(Delftia)、氫噬胞菌(Hydrogenophaga)以及不動桿菌(Acinetobacter)的豐度隨實驗進行獲得提高.

表2 源水實驗中主要細菌群落與水質參數的相關關系Table 2 The correlation analysis between main bacterial community and water parameters in bacteria and control systems
Meta-群落假說[39-40]認為,細菌多樣性是因擴散作用而聯系在一起的一系列本地群落的集合,它強調不同時空尺度上群落的結構和動態過程.在meta-群落假說中,有兩個影響因子影響細菌群落組成:內部環境因子和外部環境因子.其中內部環境因子指食物網中生物間的相互作用及物種的隨機動態等物種本身之間的影響因子,而外部環境因子指環境溫度、主要離子濃度等外在因素.目前一般認為在水力停留時間長的水體(如湖泊和水庫)中細菌群落主要受其內部環境因子的影響[40].但是,本文僅僅考察了在靜態源水系統中投加貧營養好氧反硝化菌,脫氮過程中水體的細菌群落以及多樣性的演變過程,存在一定的局限性.考慮以后增加源水動態過程以及底泥存在條件下脫氮變化中的細菌群落變化特征的考察,以期為微污染水源水庫的生物修復過程中對水體細菌群落的影響提供技術支持.
2.4 源水實驗的細菌群落主成分分析
將源水實驗投菌和對照兩系統的開始時期、中間時期和結束時期共6個樣點的OTU (97%相似性)進行主成分分析,來反映樣本間的差異和距離.樣本間的組成越相似,反映在PCA圖中的距離越近.通過在PCA圖中的樣本間的分散和聚集分布反映樣本間細菌群落的相似程度.圖5的結果展示,6個樣點的主成分分析得出PC1+PC2達到68.38%.在源水實驗開始階段,投菌系統和對照系統的細菌群落組成相似性很高;隨著源水實驗的進行,各個系統的細菌群落組成發生變化,同時期的投菌系統與對照系統的細菌群落差異很大.與兩系統的OTU分布的Veen圖特征和樣本間聚類特征相一致.

圖5 源水實驗的細菌主成分分析Fig.5 Principal component analysis (PCA) of bacteria and control source experimental systems
2.5 源水實驗的OTU分布Venn圖特征
Venn圖可用于統計多個樣本中所共有和獨有的OTU數目,可以比較直觀的表現環境樣本的OTU數目組成相似性及重疊情況. 本實驗選用相似水平為97%的OTU樣本表.
投菌系統和對照系統的結果如圖6所示.源水實驗投菌系統結果顯示為,開始階段與中間階段共有85個相同的OTU樣本;中間階段與結束階段共有102個相同的OTU樣本;開始階段與結束階段共有54個相同的OTU樣本;開始階段、中間階段與結束階段共有45個相同的OTU樣本.源水實驗對照系統結果顯示為,開始階段與中間階段共有70個相同的OTU樣本;中間階段與結束階段共有143個相同的OTU樣本;開始階段與結束階段共有61個相同的OTU樣本;開始階段、中間階段與結束階段共有49個相同的OTU樣本,對照系統三階段共有的OTUs要高于投菌系統.

圖6 源水實驗兩系統的OUT分布Fig.6 OUT distribution of bacteria and control systems in source water experiment
然而,投菌系統和對照系統間的菌群間相似性如圖7所示,開始階段時投菌系統 對照系統共有212個相同的OTU樣本;中間階段時投菌系統對照系統共有158個相同的OTU樣本;結束階段時投菌系統和對照系統共有95個相同的OTU樣本. 隨著源水實驗的進行同一系統中從開始到結束階段,投菌系統內共有的OTUs要小于對照系統,并且同一時期投菌系統與對照系統共有的OTUs也隨著試驗的進行逐漸減少.從源水實驗開始到結束整個實驗過程中兩系統共有的OTUs明顯越來越少,說明投菌系統和對照系統間的細菌群落差異性逐漸顯現,這點與6個樣本間的聚類分析相一致.

圖7 源水實驗不同時期的OUT分布Fig.7 OUT distribution of different periods in source experiment
2.6 源水實驗的樣本間聚類特征
利用樹枝結構描述和比較多個樣本間的相似性和差異關系.首先使用描述群落組成關系和結構的算法計算樣本間的距離,即根據beta多樣性距離矩陣進行層次聚類分析,使用非加權組平均法UPGMA(Unweighted pair group method with arithmetic mean)算法構建樹狀結構,得到樹狀關系形式用于可視化分析.通過bray curtis算法使用qiime計算beta多樣性距離矩陣,算法如下:

式中:SA,i= 表示A樣本中第i個OTU所含的序列數; SB,i= 表示B樣本中第i個OTU所含的序列數.
本源水實驗的投菌系統和對照系統從實驗開始階段、中間階段和結束階段取的6個樣本,通過樣本的OTUs進行層次聚類分析得到結果如圖8所示.
結果顯示,實驗初始階段投菌系統和對照系統在同一分支下,樣本間相似性很高、差異不明顯;隨著源水實驗的進行,投菌系統和對照系統內樣本間的OTUs被分離,兩系統內的細菌群落結構發生變化.在貧營養好氧反硝化菌作用下的投菌系統中,隨著實驗的進行系統內水體的氮素在逐漸減小.因此水體的微生物生存受到限制,其物種豐富度下降,與細菌門類與對照和投菌系統水質參數相關性分析結果相一致.
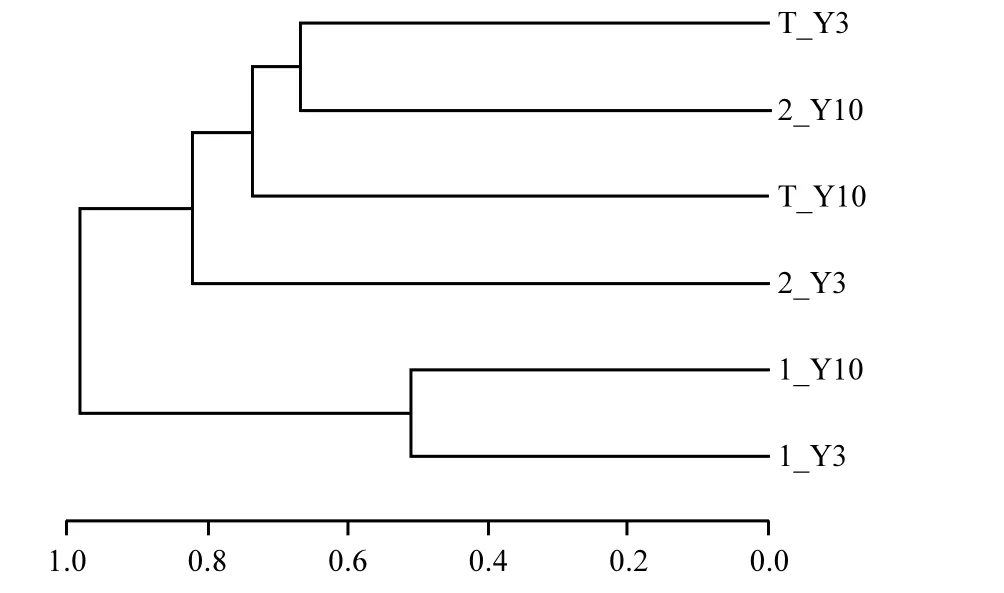
圖8 源水實驗細菌多樣本相似度樹狀圖Fig.8 The hcluster tree of source water experimental system
3 結論
基于Miseq高通量測序法研究源水脫氮過程的微生物群落的演變.結果顯示投菌系統與對照系統的水體中的微生物群落結構隨著實驗進行都發生了變化,兩系統不同時期差異性明顯.其中兩系統中變形菌的群落結構與氮素指標存在明顯的負相關.與此同時投菌系統中與氮循化相關的菌種明顯增加.考慮到靜態源水投菌實驗的局限性,以后增加底泥存在下流動源水投菌實驗的細菌群落變化,以期為研究微污染水源水庫的投加貧營養好氧反硝化菌進行生物修復過程中對水體細菌群落的影響提供技術支持.
參考文獻:
[1] Robertson L A, Kuenen J G. Thiosphaera pantotropha gen. nov. sp. nov., a facultatively anaerobic, facultatively autotrophic sulphur bacterium [J]. Journal of General Microbiology, 1983, 129(9):2847-2855.
[2] Gao H, Schreiber F, Collins G, et al. Aerobic denitrification in permeable Wadden Sea sediments [J]. The ISME journal, 2010, 4(3):417-426.
[3] Zhang J B, Wu P X, Hao B, et al. Heterotrophic nitrification and aerobic denitrification by the bacterium Pseudomonas stutzeri YZN-001 [J]. Bioresource Technology, 2011,102(21):9866-9869.
[4] Yang X P, Wang S M, Zhang D W, et al. Isolation and nitrogen removal characteristics of an aerobic heterotrophic nitrifying–denitrifying bacterium, Bacillus subtilis A1 [J]. Bioresource Technology, 2011,102(2):854-862.
[5] Yu A, Li Y, Yu J. Denitrification of a newly isolated Bacillus strain W2and its application in aquaculture [J]. Journal of Microbiology, 2004,25(3):77-81.
[6] Zhao B, An Q, He Y L, et al.N2O and N2production during heterotrophic nitrification by Alcaligenes faecalis strain NR [J]. Bioresource Technology, 2012,116(0):379-385.
[7] Joo H S, Hirai M, Shoda M. Characteristics of ammonium removal by heterotrophic nitrification-aerobic denitrification by Alcaligenes faecalis No. 4 [J]. Journal of Bioscience and Bioengineering, 2005,100(2):184-191.
[8] Cleenwerck I, De Wachter M, Hoste B, et al. Aquaspirillum dispar Hylemon et al. 1973 and Microvirgula aerodenitrificans Patureau et al. 1998 are subjective synonyms [J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53:(1457-1459).
[9] Patureau D, Helloin E, Rustrian E, et al. Combined phosphate and nitrogen removal in a sequencing batch reactor using the aerobic denitrifier, Microvirgula aerodenitrificans [J]. Water Research, 2001,35(1):189-197.
[10] Chen Q, Ni J R. Heterotrophic nitrification–aerobic denitrification by novel isolated bacteria [J]. J. Ind. Microbiol. Biotechnol., 2011, 38(9):1305-1310.
[11] Patureau D, Bernet N, Moletta R. Combined nitrification and denitrification in a single aerated reactor using the aerobic denitrifier Commonas sp. strain SGLY2 [J]. Water Research, 1997, 31(6):1363-1370.
[12] 張培玉,曲 洋,于德爽,等.菌株qy37的異養硝化/好氧反硝化機制比較及氨氮加速降解特性研究 [J]. 環境科學, 2010,31(8): 1819-1826.
[13] 于愛茸,李 尤,俞吉安.一株耐氧反硝化細菌的篩選及脫氮特性研究 [J]. 微生物學雜志, 2005,25(3):77-81.
[14] Joo H-S, Hirai M, Shoda M. Piggery wastewater treatment using Alcaligenes faecalis strain No. 4with heterotrophic nitrification and aerobic denitrification [J]. Water Research, 2006,40(16): 3029-3036.
[15] Bouchez T, Patureau D, Delgen S J, et al. Successful bacterial incorporation into activated sludge flocs using alginate [J]. Bioresource Technology, 2009,100(2):1031-1032.
[16] Wang P, Li X, Xiang M, et al. Characterization of efficient aerobic denitrifiers isolated from two different sequencing batch reactors by 16S-rRNA analysis [J]. Journal of Bioscience and Bioengineering, 2007,103(6):563-567.
[17] Kuznetsov S, Dubinina G, Lapteva N. Biology of oligotrophic bacteria [J]. Annual Reviews in Microbiology, 1979,33(1):377-387.
[18] Wainaright M, Barakah F, Al-Turk I, et al. Oligotrophic micro-organisms in industry, medicine and the environment [J]. Science Progress, 1990,75(298Pt 3-4):313-322.
[19] Button D, Schut F, Quang P, et al. Viability and isolation of marine bacteria by dilution culture: theory, procedures, and initial results [J]. Applied and Environmental Microbiology, 1993,59(3): 881-891.
[20] 黃廷林,周 娜,張海涵,等.3株貧營養好氧反硝化細菌的分離鑒定及反硝化特性 [J]. 環境工程學報, 2014,8(12):5507-5513.
[21] 黃廷林,魏 巍,王春燕,等.揚水曝氣-貧營養生物膜組合技術凈化微污染原水中試研究 [J]. 重慶大學學報, 2012,(1):125-131,146.
[22] 魏 巍,黃廷林,蘇俊峰,等.1株貧營養好氧反硝化菌的分離鑒定及其脫氮特性 [J]. 生態環境學報, 2010,19(9):2166-2171.
[23] 黃廷林,蘇俊峰,李 倩.好氧反硝化菌株的篩選培養及其反硝化性能研究 [J]. 西安建筑科技大學學報:自然科學版, 2009,(5):704-707.
[24] 白玉濤,周 玉,趙 吉.內蒙古高原干涸湖泊反硝化及甲烷氧化細菌的群落分析 [J]. 中國環境科學, 2012,32(7):1293-1301. [25] 黨 巖,張 瑞,葉杰旭,等.EGSB處理垃圾焚燒滲瀝液及其微生物群落變化 [J]. 中國環境科學, 2013,33(6):999-1004.
[26] 郭建寧,陳 磊,張錫輝,等.臭氧/陶瓷膜對生物活性炭工藝性能和微生物群落結構影響 [J]. 中國環境科學, 2014,34(3):697-704.
[27] 鄭林雪,李 軍,胡家瑋,等.同步硝化反硝化系統中反硝化細菌多樣性研究 [J]. 中國環境科學, 2015,35(1):116-21.
[28] 趙文莉,郝瑞霞,王潤眾,等.復合碳源填料反硝化脫氮及微生物群落特性 [J]. 中國環境科學, 2015,35(10):3003-3009.
[29] Huang T L, Zhou S L, Zhang H H, et al. Nitrogen removal from micro-polluted reservoir water by indigenous aerobic denitrifiers [J]. International Journal of Molecular Sciences, 2015,16(4): 8008-8026.
[30] Wang Y, Sheng H F, He Y, et al. Comparison of the levels of bacterial diversity in freshwater, intertidal wetland, and marine sediments by using millions of illumina tags [J]. Applied and environmental microbiology, 2012,78(23):8264-8271.
[31] Oberauner L, Zachow C, Lackner S, et al. The ignored diversity: complex bacterial communities in intensive care units revealed by 16S pyrosequencing [J]. Scientific Reports, 2013,3:1413.
[32] Fouts D E, Szpakowski S, Purushe J, et al. Next generation sequencing to define prokaryotic and fungal diversity in the bovine rumen [J]. PLOS One, 2012,7(11):e48289.
[33] Jami E, Israel A, Kotser A, et al. Exploring the bovine rumen bacterial community from birth to adulthood [J]. The ISME journal, 2013,7(6):1069-1079.
[34] 國家環境保護總局.《水和廢水監測分析方法》.第4版 [M]. 北京:中國環境科學出版社, 2002.
[35] Huang T L, Wei W, Su J F, et al. Denitrification performance and microbial community structure of a combined WLA–OBCO system [J]. PlOS One, 2012,7(11):e48339.
[36] Bates S T, Clemente J C, Flores G E, et al. Global biogeography of highly diverse protistan communities in soil [J]. The ISME journal, 2013,7(3):652-659.
[37] Yan Q, Bi Y, Deng Y, et al. Impacts of the Three Gorges Dam on microbial structure and potential function [J]. Scientific Reports, 2015,5:8605.
[38] Yang X, Xie P, Ma Z, et al. Decrease of NH4+-N by bacterioplankton accelerated the removal of cyanobacterial blooms in aerated aquatic ecosystem [J]. Journal of Environmental Sciences, 2013,25(11):2223-2228.
[39] Leibold M A, Holyoak M, Mouquet N, et al. The metacommunity concept: a framework for mult-scale community ecology [J]. Ecology Letters, 2004,7(7):601-613.
[40] Brendan Logue J, Lindstr?m E S. Biogeography of bacterioplankton in inland waters [J]. Freshwater Reviews, 2008,1(1):99-114.
Bacterial community structures of aerobic denitrification bacteria nitrogen removal process in source water experiment by using Miseq high-throughput sequencing technique.
ZHOU Shi-lei, HUANG Ting-lin*, ZHANG Chun-hua, BAI Shi-yuan, HE Xiu-xiu (Key Laboratory of Northwest Water Resource, Environment and Ecology, Ministry of Education, School of Environmental and Municipal Engineering, Xi’an University of Architecture and Technology, Xi’an 710055, China). China Environmental Science, 2016,36(4):1125~1135
Abstract:Miseq high-throughput sequencing technique was used to investage the bacterial community structure of nitrogen removal process in source water experiment systems added aerobic denitrification bacteria. Bioinformatics analysis were carried on the for the samples of bacteria and control systems. Then, the fundamental analysis, Optimized sequence statistics, OTU distribution, and Taxonomic analysis; the Advanced analysis, the microbial community, PCA, Rank-Abundance, Hcluster, Specaccum, and Venn of OTU distribution. As a result, the nitrogen of bacteria system was removed obviously, and Hierarchical cluatering and PCA showed that the microbial community structure of bacteria and control systems has changed, and the Protebacterice and Bacteroidetes were the main phylum in experiment systems. Meanwhile, the N-functional microbial of bacteria system had an increase process, obviously, which was consistent with the changes of aerobic denitrification bacteria. From all the results, the Miseq high-throughput sequencing technique was an effective tool to explore the changes of bacterial community structure of nitrogen removal process, which could supply a reference to study the changes of community structure of remediation the source water in situ.
Key words:nitrogen removal;aerobic denitrification;Miseq sequencing;bioinformatics analysis;bacterial community structure
作者簡介:周石磊(1987-),男,河北石家莊人,西安建筑科技大學博士研究生,主要從事水環境修復與水源水質微污染控制的相關研究.
基金項目:國家科技支撐計劃(2012BAC04B02)
收稿日期:2015-09-15
中圖分類號:X172
文獻標識碼:A
文章編號:1000-6923(2016)04-1125-11

