大腸癌表觀遺傳修飾在東西方人口中的異同研究
胡 強,熊 華,房靜遠上海交通大學醫學院附屬仁濟醫院消化科,上海市消化疾病研究所,上海 200001
?
大腸癌表觀遺傳修飾在東西方人口中的異同研究
胡 強,熊 華,房靜遠
上海交通大學醫學院附屬仁濟醫院消化科,上海市消化疾病研究所,上海 200001
[摘要]大腸癌對全世界人口的危害仍尤為重大,在其研究領域,大腸癌表觀遺傳學的研究越來越受到研究者重視。從大腸癌表觀遺傳學中的DNA甲基化和組蛋白修飾兩個主要方面,對比分析了東西方人口中大腸癌患者之間存在的異同。接著,初步介紹了東西方大腸癌患者用作生物標識和治療靶點的表觀遺傳修飾基因的異同點。分析表明,雖然大腸癌在世界范圍內有一些共同的特征,但其在不同人種中確實存在某些明顯的表觀遺傳的不同,例如特異性基因甲基化以及組蛋白修飾位點和甲基化,乙酰化的程度不同在各國的研究中都已得到證實,這就為不同人種中大腸癌特異性的診斷、治療及預后提供了依據。在進一步的規范研究方法和增加樣本量的情況下,相信更多的大腸癌組織特異性的分子靶點將會被發現,繼而針對單個患者的個體化治療將有望實現。
[關鍵詞]結直腸腫瘤;后成說;遺傳學;DNA甲基化
Correspondence to: XIONG hua E-mail: huaxong88@126.com
在全球范圍內,大腸癌是威脅人類健康的主要疾病之一。現在一般認為,大腸癌的發生是遺傳學改變(包括基因突變和基因缺失)與表觀遺傳學改變兩者逐漸累積,共同作用的結果[1]。自Feinberg和Vogelstein發現表觀遺傳修飾在大腸癌發生、發展中可能發揮重要作用以來,表觀遺傳修飾在大腸癌中的地位越來越受到研究者重視。表觀遺傳學的改變具有可逆性和可調控性,可以作為大腸癌預防、診斷、治療及預后的靶點,因而這一領域已經取得了長足的發展。令人遺憾的是,決定大腸癌進展和臨床過程因素的確切可用知識主要由西方國家構建。與西方國家相比,東方和中東國家的大腸癌可能出現新的臨床和分子分型,即不同的發病率、隱含的家族聚集性和發病年輕化等[2]。本文就東西方大腸癌患病人群中表觀遺傳修飾的異同研究作一綜述。
表觀遺傳學是指在基因核酸不發生改變的情況下發生基因表達可遺傳的變化,它包含的現象很多。我們將重點從DNA甲基化和組蛋白修飾兩個方面來進行本課題的探討,并就相關的分子標志及治療作簡明闡述。
1 DNA甲基化
DNA甲基化是研究得最為深入的表觀遺傳學現象,它指在DNA甲基轉移酶的催化下,DNA的CG核苷酸上的胞嘧啶添加甲基而形成5-甲基胞嘧啶。正常哺乳動物基因組中60%~90% 的CpG都被甲基化,未甲基化的CpG成簇組成CpG島,主要位于基因啟動子的核心序列和轉錄起始點。大腸癌最重要的表觀遺傳學特征就是全基因組的低甲基化和局部區域的高甲基化,兩者共同促進腫瘤的形成。一般認為,全基因組低甲基化激活原癌基因;而超甲基化主要發生在抑癌基因,導致抑癌基因失活。在大腸癌基因型中,成百上千的基因可以出現異常甲基化,但是,只有其中部分對大腸癌的發病機制具有重要作用,它們可能在大腸癌中表現為超甲基化或沉默(表1)[3]。
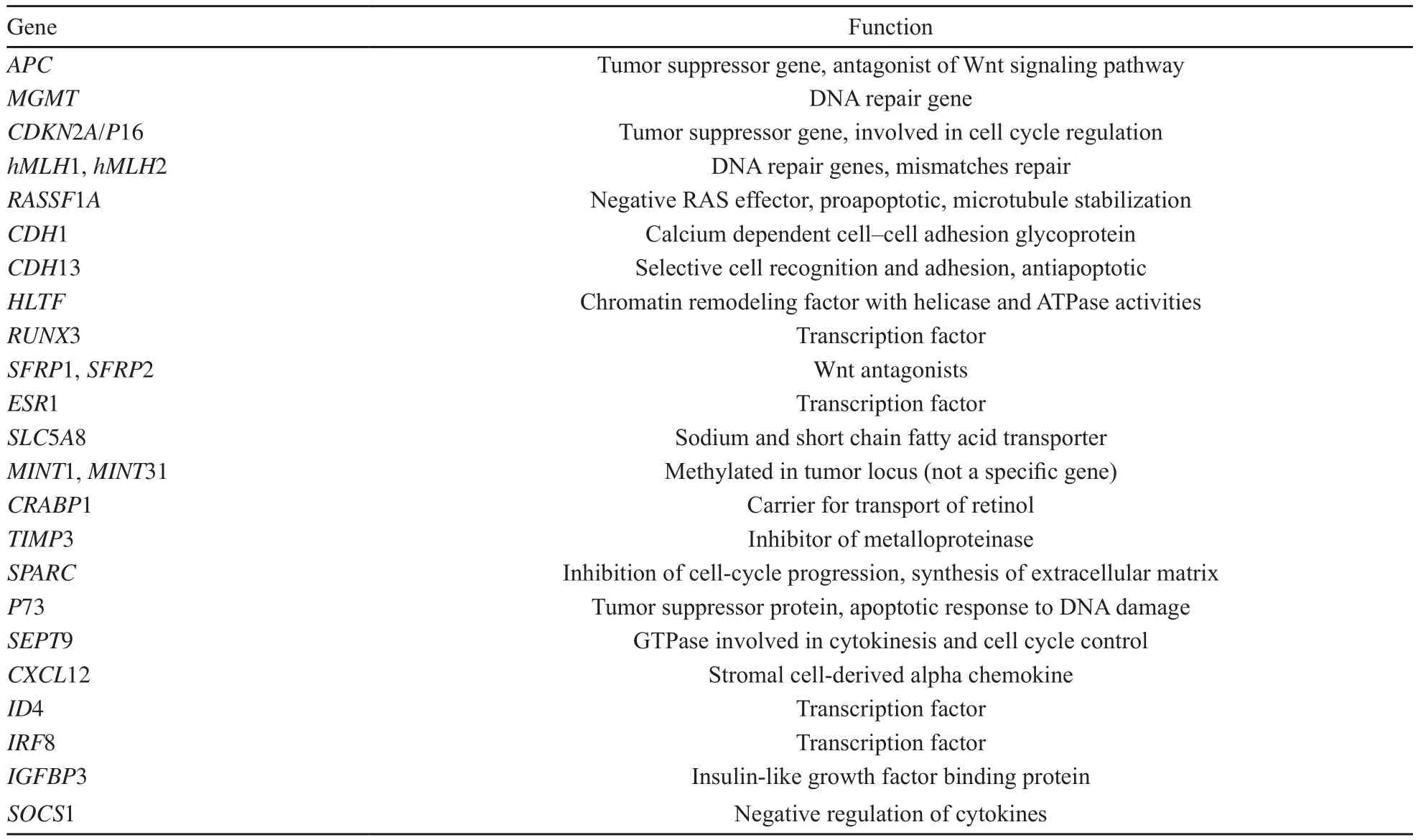
表1 大腸癌中常見有代表性的超甲基化和沉默基因Tab.1 Some examples of genes frequently methylated and silenced in colorectal cancer
大腸癌作為一種人類疾病,在各人種中具有某些共同的特征。例如,組織因子通路抑制劑2是一種潛在的腫瘤抑制基因,它被發現在超過97%的大腸腺瘤和大腸癌中甲基化。在對一些獨立族群的研究中,它在所有的大腸癌中均異常甲基化[4-6]。同樣,在一項對數個獨立族群大腸癌的研究中,TWIST1基因啟動子甲基化被確認為新近發現的基因沉默[7]。盡管如此,越來越多的研究證實,大腸癌中DNA甲基化在不同人種中存在差異。一項研究表明,非裔美國人有著比伊朗人更高的GPNMB、ICAM5和CHD5基因啟動子甲基化水平。西班牙人和其他族群有著比非裔美國人和高加索人更高水平的廣泛甲基化(表2)。在大腸癌中觀察到的種族分離,可能部分解釋其在不同種群中發病率和進展性的不同[8]。
2 組蛋白修飾
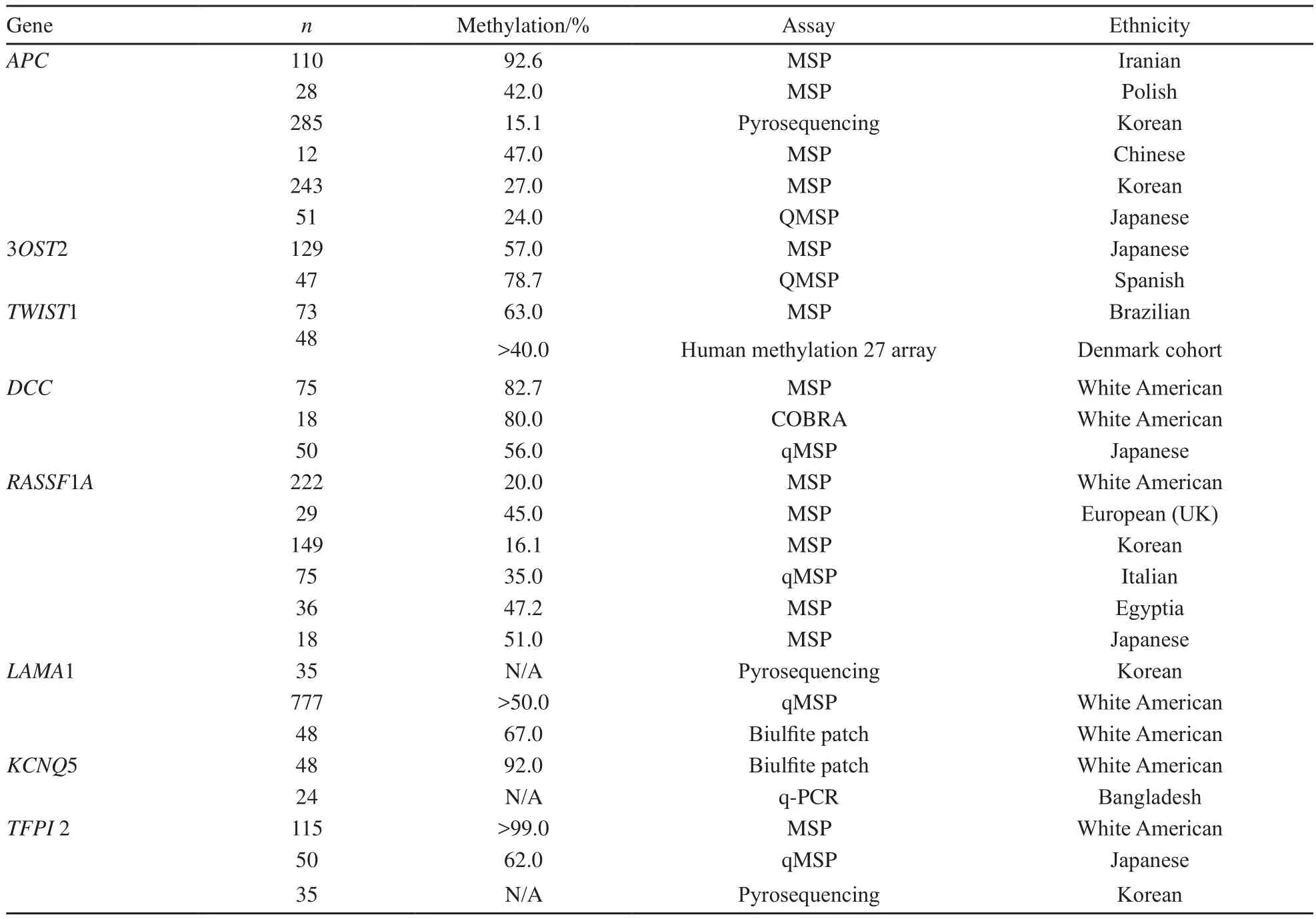
表2 不同基因甲基化在不同人種大腸癌中的比較Tab.2 Summary of literature review for nonCIMP-panes genes
組蛋白作為核小體的重要組成部分,其N-末端氨基酸(主要為精氨酸和賴氨酸)殘基可發生甲基化、乙酰化、磷酸化、泛素化和多聚ADP糖基化等多種共價修飾作用。組蛋白修飾是激活或抑制基因表達的關鍵可逆性表觀遺傳學事件[9],它對目的基因表達的影響由組蛋白修飾的類型和參與的特定氨基酸的種類兩個因素決定。目前,組蛋白甲基化和乙酰化是大腸癌中研究得最為廣泛的組蛋白譜異常。常見的組蛋白甲基化修飾有組蛋白H3第4位賴氨酸的二甲基化或三甲基化,H3第9和第27位賴氨酸的三甲基化。前兩者與基因的轉錄激活有關,后兩者則抑制甚至沉默基因的表達。大腸癌產生過程中最重要的組蛋白修飾有:H3K9的去乙酰化和甲基化,H4K16單乙酰化和H4K20的三甲基化。Kondo等[10]對結腸癌細胞株的研究表明,H3K9的低乙酰化、H3K4的低甲基化和H3K9的過度甲基化與P16、hMLH1和MGMT這3個基因啟動子過度甲基化引起的基因表達沉默相關,提示大腸癌DNA甲基化和組蛋白的修飾密切相關,共同作用于基因的調控進程。東西方人群中大腸癌的組蛋白修飾有較大的區別,具體結果見表3。
3 東西方人群中用作生物標志的甲基化基因的異同
眾所周知,大腸癌的早期診斷對患者的預后至關重要,傳統上用于大腸癌的早期篩查有多種方法,如電子直腸檢查、大便隱血試驗、乙狀結腸鏡檢查和結腸鏡檢查。然而,這些方法具有侵入性,也缺乏必要的靈敏度,臨床上迫切需要更敏感和更溫和的手段來檢測大腸癌。新穎的生物標志可以起到補充作用[17]。在大腸癌的形成過程中,我們相信腫瘤抑制基因的遺傳學和表觀遺傳學改變起到了重要作用。因為其啟動子區域CPG島的超甲基化而引起基因沉默,這在大腸癌發展的早期階段發生率較高。這樣的異常現象可以被用來作為早期大腸癌的診斷和風險評估的分子標志(表4)。
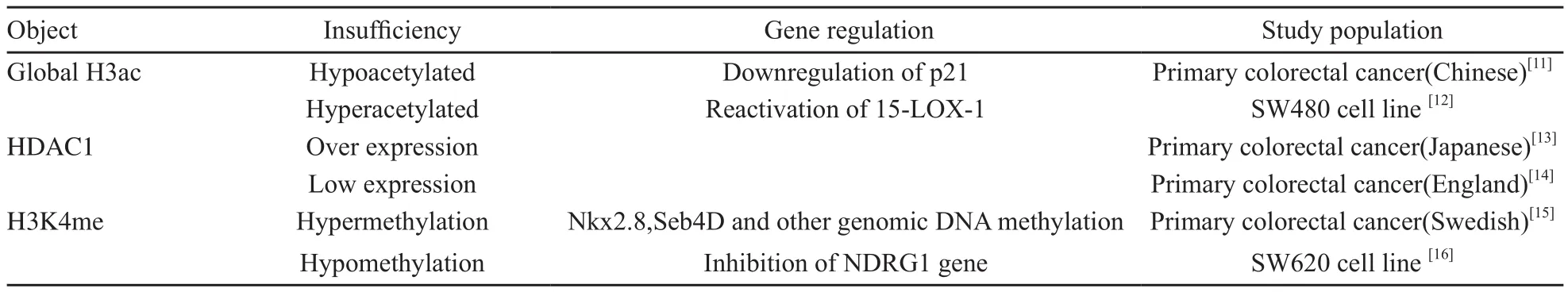
表3 大腸癌中組蛋白修飾的比較Tab.3 Comparison of histone modifications in colorectal cancer
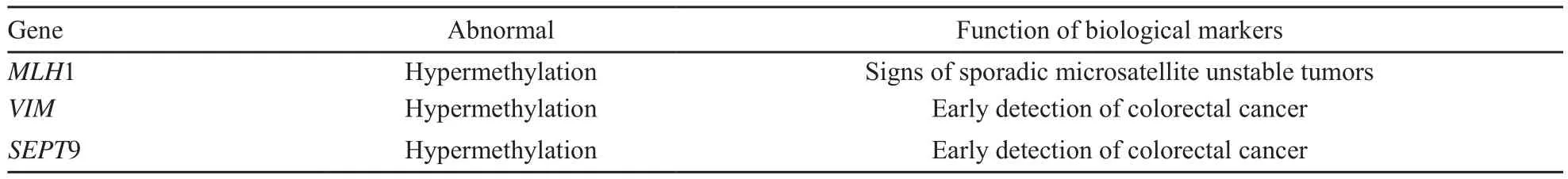
表4 在大腸癌中常見的有潛力的甲基化基因生物標志Fig.4 Common potential biomarks from methylation of genes in colorectal cancer
最近,新的甲基化生物標志芯片通過了臨床試驗,正逐步走向商業化。Colovantage是一項血標志測試,用于檢測甲基化sepT9的存在[18]。
候選生物標志在實驗室及臨床經過了大量的測試,而且取得了令人鼓舞的效果。更多的生物標志檢測見表5。
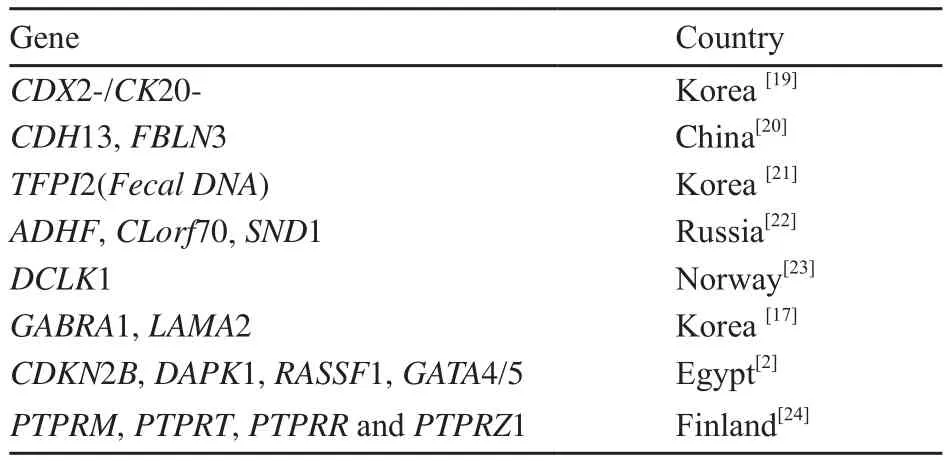
表5 不同國家新近發現可作為生物標志的基因甲基化Fig.5 Newly discovered gene methylation can be used as biomarkers in different countries
4 東西方人群中用作治療靶點的表觀遺傳修飾基因的異同
由于表觀遺傳修飾的改變具有可逆性,為大腸癌的治療提供了一個新的趨勢。相比于組蛋白甲基化,組蛋白乙酰轉移酶、染色質重塑和miRNA的干預劑的應用仍處于早期階段,DNMTi(DNA甲基轉移酶抑制劑)和HDACi(組蛋白去乙酰化酶)的使用已日趨成熟[18]。DNA甲基化抑制劑分為核苷類似物、非核苷類似物和反義寡核苷酸。其主要作用機制為:與DNA混合后,同DNMTS形成共價結合,阻礙酶的活性。其中,5-阿扎胞苷和5-脫氧胞苷已被美國食品和藥物管理局(Food and Drug Administration,FDA)批準用于治療骨髓異常增生綜合征,這提供了表觀遺傳學治療的證據原則[25]。由于HDACi多方面參與腫瘤的產生,吸引研究者用于腫瘤治療。然而,兩者在臨床的應用極其有限,究其原因是不能作為選擇性的抑制劑,在治療的同時干預了正常的生理。是否能根據甲基化或乙酰化的不同來選擇特異性的抑制劑,現在很多實驗室正進行探索(表6)。
表觀遺傳學的發展很快,對大腸癌的研究顯得越來越重要。目前來看,歐美國家大腸癌患者在3OST2、LAMA1、KCNQ5、TFPI2、EYA4和ADHEF1D等基因的甲基化上明顯高于亞洲人種,而RASSF1A等基因甲基化則在亞洲大腸癌患者中多發。一大批新的作為生物標志的甲基化基因的發現也說明大腸癌表觀遺傳學在東西方存在差異,例如,韓國發現的CDX2-/ CK20-、GABRA和LAMA2基因,中國發現的CDH13和FBLN3基因,挪威發現的DCLK1基因以及芬蘭發現的PTPR家族基因等。隨著更多的甲基化位點被各國研究者證實,我們需仔細分析其中隱藏的信息[29]。首先,重亞硫酸鹽測序是如今腫瘤甲基化分析的金標準,但在一些研究中沒有采用此方法。其次,很多研究存在樣本容量不足的現象。因此,研究者應該改進實驗方案和增加樣本統計量[8]。而且,最重要的一點,新發現的甲基化位點是否是因為人種差異而產生的呢?具有普遍性的甲基化位點可以為疾病的診斷提供依據,而具有種族特異性的位點在各自種族中的用處則更加重要。
利用高通量的DNA甲基化芯片技術與信息學方法進行更加深入的研究可加速特異性大腸癌抑癌基因的發現,為尋找大腸癌的組織特異性的分子靶點提供快捷的途徑。

表6 大腸癌中不同用作治療靶點的表觀遺傳修飾基因的比較Fig.6 Comparison of different epigenetic modifications that can be used as gene therapy targets in colorectal cancer
[參 考 文 獻]
[1]LAO V V, GRADY W M.Epigenetics and colorectal cancer [J].Nat Rev Gastroenterol Hepatol, 2011, 8(12): 686-700.
[2]NIEMINEN T T, SHOMAN S, EISSA S, et al.Distinct genetic and epigenetic signatures of colorectal cancers according to ethnic origin[J].Cancer Epidemiol Biomarkers Prev, 2012,21(1): 202-211.
[3]COPPEDè F.Epigenetic biomarkers of colorectal cancer:Focus on DNA methylation[J].Cancer Lett, 2014, 342(2):238-247.
[4]HIBI K, MIZUKAMI H, SHIRAHATA A, et al.Aberrant methylation of the netrin-1 receptor genes UNC5C and DCC detected in advanced colorectal cancer[J].World J Surg,2009, 33(5): 1053-1057.
[5]GL?CKNER S C, DHIR M, YI J M, et al.Methylation of TFPI2 in stool DNA: a potential novel biomarker for the detection of colorectal cancer[J].Cancer Res, 2009, 69(11):4691-4699.
[6]SCHNEKENBURGER M, DIEDERICH M.Epigenetics offer new horizons for colorectal cancer prevention[J].Curr Colorectal Cancer Rep, 2012, 8(1): 66-81.
[7]ASHKTORAB H, RAHI H, WANSLEY D, et al.Toward a comprehensive and systematic methylome signature in colorectal cancers[J].Epigenetics, 2013, 8(8): 807-815.
[8]XIA Y Y, DING Y B, LIU X Q, et al.Racial/ethnic disparities in human DNA methylation[J].Biochim Biophys Acta,2014, 1846(1): 258-262.
[9]ZORATTO F, ROSSI L, VERRICO M, et al.Focus on genetic and epigenetic events of colorectal cancer pathogenesis:implications for molecular diagnosis[J].Tumour Biol, 2014,35(7): 6195-6206.
[10]KONDO Y, SHEN L, ISSA J P.Critical role of histone methylation in tumor suppressor gene silencing in colorectal cancer[J].Mol Cell Biol, 2003, 23(1): 206-215.
[11]GARGALIONIS A N, PIPERI C, ADAMOPOULOS C, et al.Histone modifications as a pathogenic mechanism of colorectal tumorigenesis[J].Int J Biochem Cell Biol, 2012, 44(8):1276-1289.
[12]ZUO X, MORRIS J S, SHUREIQI I.Chromatin modification requirements for 15-lipoxygenase-1 transcriptional reactivation in colon cancer cells[J].J Biol Chem, 2008,283(46): 31341-31347.
[13]HIGASHIJIMA J, KURITA N, MIYATANI T, et al.Expression of histone deacetylase 1 and metastasis-associated protein 1 as prognostic factors in colon cancer[J].Oncol Rep, 2011,26(2): 343-348.
[14]OZDA? H, TESCHENDORFF A E, AHMED A A, et al.Differential expression of selected histone modifier genes in human solid cancers[J].BMC Genomics, 2006, 7(2): 90-104.
[15]ENROTH S, RADA-IGLESISAS A, ANDERSSON R, et al.Cancer associated epigenetic transitions identified by genome-wide histone methylation binding profiles in human colorectal cancer samples and paired normal mucosa[J].BMC Cancer, 2011, 11(5): 450-462.
[16]LI Q, CHEN H.Transcriptional silencing of N-Myc downstream-regulated gene 1 (NDRG1) in metastatic colon cancer cell line SW620[J].Clin Exp Metastasis, 2011,28(2): 127-135.
[17]LEE S, OH T, CHUNG H, et al.Identification of GABRA1 and LAMA2 as new DNA methylation markers in colorectal cancer [J].Int J Oncol, 2012, 40(3): 889-898.
[18]VAIOPOULOS A G, ATHANASOULA K, PAPAVASSILIOU A G.Epigenetic modifications in colorectal cancer: molecularinsights and therapeutic challenges[J].Biochim Biophys Acta, 2014, 1842(7): 971-980.
[19]KIM J H, RHEE Y Y, BAE J M, et al.Loss of CDX2/CK20 expression is associated with poorly differentiated carcinoma,the CpG island methylator phenotype, and adverse prognosis in microsatellite-unstable colorectal cancer[J].Am J Surg Pathol, 2013, 37(10): 1532-1541.
[20]WANG Z, YUAN X, JIAO N, et al.CDH13 and FLBN3 gene methylation are associated with poor prognosis in colorectal cancer[J].Pathol Oncol Res, 2012, 18(2): 263-270.
[21]KIM Y H, LEE H C, KIM S Y, et al.Epigenomic analysis of aberrantly methylated genes in colorectal cancer identifies genes commonly affected by epigenetic alterations[J].Ann Surg Oncol, 2011, 18(8): 2338-2347.
[22]NAUMOV V A, GENEROZOV E V, ZAHARJEVSKAYA N B,et al.Genome-scale analysis of DNA methylation in colorectal cancer using infinium human methylation 450 bead chips [J].Epigenetics, 2013, 8(9): 921-934.
[23]VEDELD H M, SKOTHEIM R I, LOTHE R A, et al.The recently suggested intestinal cancer stem cell marker DCLK1 is an epigenetic biomarker for colorectal cancer[J].Epigenetics, 2014, 9(3): 346-350.
[24]LACZMANSKA I, KARPINSKI P, BEBENEK M, et al.Protein tyrosine phosphatase receptor-like genes are frequently hypermethylated in sporadic colorectal cancer[J].J Hum Genet, 2013, 58(1): 11-15
[25]PATAI A V, MOLNáR B, KALMAR A, et al.Role of DNA methylation in colorectal carcinogenesis[J].Dig Dis, 2012,30(3): 310-315.
[26]CREA F, NOBILI S, PAOLICCHI E, et al.Epigenetics and chemoresistance in colorectal cancer: an opportunity for treatment tailoring and novel therapeutic strategies[J].Drug Resist Update, 2011, 14(6): 280-296.
[27]XIONG H, DU W, ZHANG Y J, et al.Trichostatin A, a histone deacetylase inhibitor, suppresses JAK2/STAT3 signaling via inducing the promoter-associated histone acetylation of SOCS1 and SOCS3 in human colorectal cancer cells[J].Mol Carcinog, 2012, 51(2): 174-184.
[28]BELANGER A S, TOJCIC J, HARVEY M, et al.Regulation of UGT1A1 and HNF1 transcription factor gene expression by DNA methylation in colon cancer cells[J].BMC Mol Biol,2010, 11(3): 9-20.
[29]RAJENDRAN P, KIDANE A I, YU T W, et al.HDAC turnover, CtIP acetylation and dysregulated DNA damage signaling in colon cancer cells treated with sulforaphane and related dietary isothiocyanates[J].Epigenetics, 2013, 8(6):612-623.
Research on similarities and differences of colorectal cancer epigenetic modifications in the Eastern and Western population
HU Qiang, XIONG Hua, FANG Jingyuan (Department of Gastroenterology,Renji Hospital, Shanghai Jiao Tong University School of Medicine, Shanghai Institute of Digestive Diseases, Shanghai 200001, China)
[Key words]Colorectal neoplasms; Epigenesis; Genetics; DNA methylation
[Abstract]Colorectal cancer remains a major threat to people’s health around the world.Researchers have paid more and more attention to colorectal cancer epigenetics.From two main aspects of colorectal cancer epigenetics: DNA methylation and histone modification, this article analyzes the similarities and differences between patients with colorectal cancer in Eastern and Western countries.This review briefly introduces epigenetic modification of genes that were used to be biomarkers and therapeutic targets.Although there are some common features of colorectal cancer in the world, analysis has showed that some obvious epigenetic differences do exist in different races.For example, it had been confirmed in the studies that there are differences in specific gene methylation, histone modification sites and the degree of methylation and acetylation among countries, which provide the basis for specific diagnosis, treatment and prognosis of colorectal cancer in different ethnic groups.With improved research methods and increased sample size,more and more special molecular targets of colorectal cancer tissues will be found, and then personalized therapy for colorectal cancer can be achieved.
DOI:10.3969/j.issn.1007-3969.2016.02.011
中圖分類號:R735.3+4
文獻標志碼:A
文章編號:1007-3639(2016)02-0182-06
通信作者:熊華 E-mail:huaxong88@126.com
收稿日期:(2015-02-03 修回日期:2016-01-11)

