母豬殺嬰行為的全基因組關聯分析
楊 慧,方紹明,黃曉暢,陳從英,張志燕
(省部共建豬遺傳改良與養殖技術國家重點實驗室,南昌 330045)
?
母豬殺嬰行為的全基因組關聯分析
楊慧,方紹明,黃曉暢,陳從英*,張志燕*
(省部共建豬遺傳改良與養殖技術國家重點實驗室,南昌 330045)
摘要:本研究測定了288頭來自白色杜洛克×二花臉F2群體的母豬殺嬰行為表型,旨在定位出影響母豬殺嬰行為的QTL。基于整個家系及已經掃描個體的60K SNP芯片信息推測出本試驗288頭個體60K的基因型。隨后,我們利用R語言GenABEL軟件包的廣義混合線性模型對母豬殺嬰行為表型進行全基因組關聯分析,定位影響母豬殺嬰行為的顯著關聯SNP位點。結果,在SSC2和SSC8上共發現50個與母豬殺嬰顯著關聯的SNPs位點,其中有21個SNPs位于SSC8:35.05~47.54 Mb區域達到基因組顯著水平,在SSC2上只有1個SNP位點達到染色體顯著水平。通過搜尋顯著相關SNP所在區域內的注釋基因,發現了12個可能影響母豬殺嬰行為的重要位置候選基因。該研究為進一步鑒別影響母豬殺嬰行為的主效基因和因果突變位點提供了研究基礎。
關鍵詞:豬;全基因組關聯分析;母豬殺嬰;候選基因
母豬母性性能的好壞是影響養豬業經濟效益的重要因素。母豬殺嬰行為即母豬在分娩期間或產后24 h之內咬傷甚至咬死新生仔豬的行為,給養豬業造成巨大經濟損失。M.J.Harris等[1]通過對大規模的商業母豬群體調查發現約5.3%的初產母豬表現出不同程度的殺嬰行為。在歐洲商業群體中調查發現母豬殺嬰發生頻率達到7%~12%,初產母豬發生頻率更高達10%~13%[2]。本研究前期在288頭白色杜洛克×二花臉F2母豬中觀察發現初產母豬殺嬰行為發生率達12.8%,分娩前發生殺嬰行為的母豬會比母性好的母豬表現得更活躍[1,3-4],且在產前爬欄行為及產后仔豬照料行為等方面也與正常母豬存在顯著差異[3]。
許多國內外專家致力于母性行為的研究,但是對母豬殺嬰行為的產生機制和影響因素還不是很清楚。除了受環境、激素水平和產仔經驗等因素影響外,遺傳因素也起很大作用[2]。P.W.Knap等[5]計算出了母豬殺嬰行為的遺傳力,母女遺傳力為0.4~0.9。H.A.van der Steen等[2]計算得到相似的母豬殺嬰行為遺傳力,母女遺傳力為0.5~0.9,且具有加性效應。E.F.Knol等[6]研究發現,選擇母性好的母豬能增加仔豬的成活率,并對仔豬的生長性狀沒有負影響。因此,通過對母豬母性行為的遺傳背景研究,找出影響母豬殺嬰行為的主效基因或相關聯的遺傳標記,從而選育出母性優良的母豬對當前養豬業具有重要意義。但目前從豬QTL 數據庫 (http://www.animalgenome.org/cgi-bin/QTLdb/index/) 可知,定位的與母豬殺嬰相關的數量性狀位點(QTL)較少,不到20個。C.Y.Chen等[7]通過非參數連鎖分析(NPL)在白色杜洛克×二花臉F2資源群體中將母豬殺嬰行為QTL定位于2、6、14、15、16和 X號染色體上,其中X染色體上QTL解釋的表型變異最大,且X染色體上的10個微衛星標記中有7個達顯著水平(P<0.01)。此外,在老鼠2號和7號染色體上也發現了與母性殺嬰行為相關的QTL,且通過比較基因組可知,鼠2號染色體QTL區域正好與A.C.Peripato等定位到的6號染色體QTL區域一致[8]。C.R.Quilter等[9]通過80個微衛星標記的全基因組掃描法,利用來自11個不同商業品系的119個殺嬰行為全同胞個體,定位到4個與母豬殺嬰行為顯著相關的QTLs,分別位于豬 2、10 和 X 號染色體的4個區域。隨后C.R.Quilter等[10]利用豬下丘腦和垂體構建的cDNA芯片檢測殺嬰行為的母豬下丘腦基因表達差異,發現了52個差異表達基因,而這些基因大部分位于之前定位到的QTL置信區間內。但是,基于低密度的微衛星標記構成連鎖圖譜在QTL定位上有著很大的不足,它只能鑒別出一些表型效應較大的數量性狀,且發現QTL的置信區間往往有數十個厘摩之大[11]。近年來,由于高通量基因分型平臺的技術迅速發展,利用高通量SNP芯片進行QTL定位在人類、動物和植物研究中得以廣泛應用[12]。自從豬60K SNP芯片推出以來,已大量采用此項技術在豬的血液性狀[13-14]、血脂性狀[15]、脂肪酸性狀[16]、胴體性狀[17]、繁殖性狀[18]等進行了全基因組關聯性研究。
本研究基于整個家系信息及60K SNP芯片信息推測出來自83個全同胞家系的288頭F2母豬的基因型進行母豬殺嬰行為的全基因組關聯性分析,為進一步鑒別影響母豬殺嬰行為的主效基因和因果突變位點提供研究基礎。
1材料與方法
1.1試驗動物
本研究試驗動物為白色杜洛克×二花臉F2資源家系[19],二花臉是產仔數高和母性性能好的中國地方豬種,而杜洛克是母性性能較差的西方豬種。該資源群體以2頭白色杜洛克豬為父本,17頭二花臉豬為母本,雜交產生F1代,隨機選取9頭F1公豬和59頭F1母豬自群繁育產生1 912頭F2個體。本試驗的研究對象是來自83個全同胞家系的288頭F2母豬。所有動物自由采食飲水,并按統一條件飼養。每千克日糧營養水平為:粗蛋白16%,消化能13.1 MJ,賴氨酸 0.78%,鈣0.6%,磷0.5%。
1.2殺嬰行為表型測定
從妊娠后期開始,每個欄位只養1頭母豬,母豬出現分娩跡象后提供適量的稻草,觀察母豬從分娩開始到分娩結束24 h內咬死或咬傷仔豬的行為。采用0/1法進行記錄,在分娩開始后24 h內發生殺嬰行為則記錄為1。
1.3DNA提取及基因型預測
采集來自F0、F1和F2個體的耳組織或尾尖組織,采用常規的酚/氯仿方法提取DNA。用NANODROP 1000 (Thermo Scientific,USA)測定DNA濃度和質量,質檢合格的樣品于-20 ℃冰箱保存。
F0、F1及F2共1 020個樣本進行了60K SNP芯片掃描,但本試驗288個樣品沒有做60K SNP芯片掃描,所以基于整個家系及掃描個體的SNP芯片信息推測出這288個個體缺失基因型。定義掃描了60K SNP芯片加微衛星的990個個體為參考群體,只有微衛星的288個個體為目標群體。首先利用Link PHASE軟件[20]構建參考個體和目標個體192個微衛星標記的單倍型;其次再利用Beagle[21]和 DAGPHASE[20]軟件構建出參考個體微衛星和60K SNP的單倍型;最后,基于微衛星間目標群體和參考群體之間的IBD,這些缺失個體的基因型通過CHROMIBD軟件推斷出來。CHROMIBD是基于連鎖和隱馬爾可夫模型預測后代和祖先染色體之間IBD的概率[22],利用祖先的高密度單倍型和后代的IBD概率可推測出后代缺失個體的基因型。再運用PLINK軟件對推測出的基因型結果進行質控,剔除哈迪溫伯格平衡檢驗P≤10-6和次等位基因型頻率MAF<0.05的SNP位點。
1.4統計分析
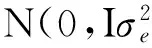
本研究采用Bonferroni校正多重檢驗確定關聯顯著性閾值,染色體顯著性閾值和基因組顯著性閾值分別為1/有效SNP位點數量和0.05/有效SNP位點數量。單倍型框的構建是用 Haploview version 4.2軟件分析顯著性SNP位點所在的QTL區域[27]。
1.5位置候選基因搜尋
結合生理生化及遺傳信息,利用NCBI (http://www.ncbi.nlm.nih.gov/) 或Ensembl (http://pre.ensembl.org/Susscrofa/Info/Index/)豬參考基因組數據庫搜尋顯著SNP左右5 Mb范圍內的候選基因。DAVID (http://david.abcc.ncifcrf.gov/)和GenCards (http://www.genecards.org/)網站用來查找候選基因功能。
2結果
2.1表型測定
本研究共觀察了288頭F2母豬的母性行為,其中47頭母豬從分娩開始到分娩結束24 h內有咬死或咬傷仔豬的行為,另241頭母豬有正常母性。
2.2全基因組關聯分析
本試驗基于整個家系及60K SNP芯片信息推測出目標個體的基因型,進行豬的全基因組關聯性分析,經過質控后有效的SNPs數是34 591。在8號染色體共檢測到49個SNPs位點與母豬殺嬰行為顯著相關(P<2.89E-05,圖1A),其中達到基因組顯著水平的SNPs有21個,位于染色體35.05~47.54 Mb內(P<1.45E-06)。針對SSC8顯著區域,構建了2 Mb (35~37 Mb)大小的單倍型框,結果,11個達到基因組顯著水平的位點落于1.25 Mb大小的一個單倍型框中(圖2)。此外在2號染色體上發現了1個SNP (ASGA0010781) 位點達到染色體顯著水平(P=1.19E-05),位于該染色體89.5 Mb區域(圖1A)。當固定8號染色體顯著性位點ALGA0047534做關聯性分析時,發現8號染色體QTL效應全部消失(圖1B),說明這些顯著SNP位于1個QTL區域。
2.3位置候選基因
通過NCBI或Ensembl網站,搜尋顯著 SNP左右5 Mb范圍內的候選基因,利用生物信息學和比較基因組學等分析手段挑選候選基因。在2號染色體區域找到2個候選基因分別是OTP和COX7C。在8號染色體上35~109 Mb區域搜尋到10個位置候選基因分別是SLC30A9、ATP8A1、GABRG1、GABRA2、GABRA4、GABRB1、TDO2、AREG、DCLK2和FGF2 (表1)。
3討論
自從R.J.Klein 等[28]在Science上報道了關于視網膜黃斑變性的GWAS研究以來,一系列復雜疾病的GWAS研究相繼被報道。豬高密度60K SNP芯片的應用為母豬殺嬰行為等復雜性狀分子機理的探索提供了重要的方法和途徑。
本研究基于整個家系信息及60K SNP芯片信息推測出288頭來自83個全同胞家系的F2母豬的基因型進行母豬殺嬰行為的全基因組關聯分析,在SSC2和SSC8上共發現50個SNPs與母豬殺嬰行為顯著關聯。本研究前期C.Y.Chen等[7]利用194個微衛星標記對該288頭F2資源家系母豬微衛星掃描時也在SSC2上定位到顯著區域,但是兩者的位置相差很遠(>5 Mb),并且在SSC8上也未定位到顯著區域。在同一個群體中不能夠相互驗證的原因可能有幾點:其一,分析模型不同,QTL定位分析是基于QTL基因型在兩個始祖品系中固定且基因型相異這個假設基礎上進行的,而實際群體中QTL基因型可能不固定,導致QTL定位無法檢測到顯著性位點,全基因組關聯分析無此假設,因此GWAS分析能檢測到QTL基因型在始祖品系中未固定的位點;其二,分析方法不同,GWAS分析中僅考慮的是加性效應,而在QTL定位中同時考慮了加性效應和顯性效應;其三,采用的標記類型不同,QTL采用的是數量少、密度低的微衛星標記,而本研究采用的是高密度豬60K SNP 芯片;最后,本研究是利用整個家系信息及60K SNP芯片信息推測出這288頭F2母豬的基因型,雖然準確率可達95%[29],但仍然存在一定的錯誤率。
8號染色體QTL區域是首次被報導與母豬殺嬰顯著相關,達到基因組顯著水平的有21個SNPs。針對SSC8顯著區域,構建了2 Mb大小的單倍型框,在此2 Mb區域搜尋到兩個位置候選基因分別是SLC30A9和ATP8A1(表1)。SLC30A9基因是鋅轉運體蛋白編碼基因,通過GenCards了解到它會與ESR1基因作用于雌激素受體信號通路中。ESR1編碼雌激素受體蛋白,在性發育和生殖功能中起到重要作用。并由于女性雌激素水平比男性更高,這可能也是導致女性焦慮頻率增加的原因[30]。ATP8A1是ATP酶家族成員之一,能夠水解ATP產生能量驅動離子跨膜運輸,并且在神經細胞的氨離子轉運活性中也有重要作用,能夠降低緊張感[31]。在SSC8:38.7~39.5 Mb區域,發現了GABRG1-GABRA2-GABRA4-GABRB1基因簇(表1)。它們編碼的蛋白是氨基丁酸的不同類型受體,都屬于配體門控離子通道家族,在抑制神經傳遞過程中起重要作用。氨基丁酸的受體基因發生變異會引起情感性分裂癥和躁郁癥[32-34]。在8號染色體47 Mb區域附近,TDO2是一個強的候選基因(表1)。TDO是色氨酸參與的犬尿氨酸代謝途徑中的限速酶,這種酶能夠控制血漿中色氨酸的濃度,且色氨酸又是5-羥色胺的前體,5-羥色胺是一種神經遞質,會參與行為、情緒和沖動等過程[35]。如果5-羥色胺發生機能障礙會導致血清素的生物合成發生變化,這種改變會引起消極情緒甚至沖動行為[36-37]。我們在8號染色體75和83 Mb區域附近分別找到兩個候選基因AREG和DCLK2,但是在SSC8:91.8~95.6 Mb區域沒有找到有關母性殺嬰行為的候選基因。在8號染色體109 Mb區域,FGF2基因可能與母豬殺嬰行為有關。FGF2(纖維母細胞生長因子2)能夠調節緊張感,在神經細胞再生和膠質細胞再生過程中起到重要作用,并且FGF2也已被作為治療抑郁癥和焦慮癥潛在的新目標[38-39]。
表1F2資源家系母豬殺嬰行為的全基因組關聯分析結果
Table 1The genome-wide association study result of maternal infanticide behavior in F2resource population
此外,2號染色體顯著區域與C.R.Quilter等[9]報導的QTL區域是一致的,在此區域發現OTP和COX7C是強的候選基因。OTP是一個蛋白編碼基因,參與下丘腦神經內分泌細胞的分化[40]。下丘腦能有效地調節控制垂體前葉各種激素的合成和分泌,如調節促甲狀腺激素、促腎上腺皮質激素、促性腺激素、生長激素、催乳素等蛋白質和肽類激素的生成和釋放。COX7C(細胞色素C氧化酶)是COX的第二個細胞核內基因,受轉錄因子YY1的調控。在神經元變化過程中,YY1是一個與壓力相關的信號轉導通路中的核內靶分子[41]。此外,肌肉中細胞色素C的缺乏也已被證明是與精神分裂癥有關[42]。
4結論
本試驗記錄了288頭來自全同胞家系F2母豬的殺嬰行為,利用整個家系信息及60K SNP芯片信息推測出這些F2母豬的基因型并進行全基因組關聯分析,在SSC2和SSC8上共發現50個與母豬殺嬰顯著關聯的SNPs位點,其中有21個SNPs達到基因組顯著水平。通過搜尋顯著SNP區域內的候選基因及其功能注釋,發現SLC30A9、ATP8A1、COX7C和OTP等基因是影響母豬殺嬰行為的重要候選基因。
參考文獻(References):
[1]HARRIS M J,LI Y Z,GONYOU H W.Savaging behaviour in gilts and sows[J].CanJAnimSci,2003,83(4):819-821.
[2]van der STEEN H A,SCHAEFFER L R,de JONG H,et al.Aggressive behavior of sows at parturition[J].JAnimSci,1988,66(2):271-279.
[3]CHEN C Y,GILBERT C L,YANG G C.Maternal infanticide in sows:Incidence and behavioural comparisons between savaging and non-savaging sows at parturition[J].ApplAnimBehavSci,2008,109(2-4):238-248.
[4]APPLEYARD S J,HALL A D,LAWRENCE A B.Prefarrowing behaviour distinguishes piglet-savaging gilts from non-savaging gilts[C].Proceedings of the 34th international congress of the ISAE,2000:62.
[5]KNAP P W,MERKS J W.A note on the genetics of aggressiveness of primiparous sows towards their piglets[J].LivestProSci,1987,17(2):161-167.
[6]KNOL E F,VERHEIJEN C.Genetic and biological aspects of mothering ability in sows[C].Proceedings of the 7th WCGALP,2002:1-4.
[7]CHEN C Y,GUO Y M,YANG G C,et al.A genome wide detection of quantitative trait loci on pig maternal infanticide behavior in a large scale White Duroc × Erhualian resource population[J].BehavGenet,2009,39(2):213-219.
[8]PERIPATO A C,DE BRITO R A,VAUGHN T T,et al.Quantitative trait loci for maternal performance for offspring survival in mice[J].Genetics,2002,162(3):1341-1353.
[9]QUILTER C R,BLOTT S C,WILSON A E,et al.Porcine maternal infanticide as a model for puerperal psychosis[J].AmJMedGenetBNeuropsychiatrGenet,2007,144B(7):862-868.
[10]QUILTER C R,GILBERT C L,OLIVER G L,et al.Gene expression profiling in porcine maternal infanticide:a model for puerperal psychosis[J].AmJMedGenetBNeuropsychiatrGenet,2008,147B(7):1126-1137.
[11]PEARSON T A,MANOLIO T A.How to interpret a genome-wide association study[J].JAMA,2008,299(11):1335-1344.
[12]PASAM R K,SHARMA R,MALOSETTI M,et al.Genome-wide association studies for agronomical traits in a world wide spring barley collection[J].BMCPlantBiol,2012,12:16.
[13]WANG J Y,LUO Y R,FU W X,et al.Genome-wide association studies for hematological traits in swine[J].AnimGenet,2013,44(1):34-43.
[14]LUO W Z,CHEN S K,CHENG D X,et al.Genome-wide association study of porcine hematological parameters in a Large White×Minzhu F2 resource population[J].IntJBiolSci,2012,8(6):870-881.
[15]CHEN C,YANG B,ZENG Z,et al.Genetic dissection of blood lipid traits by integrating genome-wide association study and gene expression profiling in a porcine model[J].BMCGenomics,2013,14:848.
[16]YANG B,ZHANG W,ZHANG Z,et al.Genome-wide association analyses for fatty acid composition in porcine muscle and abdominal fat tissues[J].PLoSOne,2013,8(6):e65554.
[17]FAN B,ONTERU S K,DU Z Q,et al.Genome-wide association study identifies Loci for body composition and structural soundness traits in pigs[J].PLoSOne,2011,6(2):e14726.
[18]SCHNEIDER J F,REMPEL L A,SNELLING W M,et al.Genome-wide association study of swine farrowing traits.Part II:Bayesian analysis of marker data[J].JAnimSci,2012,90(10):3360-3367.
[19]CHEN R,REN J,LI W,et al.A genome-wide scan for quantitative trait loci affecting serum glucose and lipids in a White Duroc×Erhualian intercross F(2) population[J].MammGenome,2009,20(6):386-392.
[20]DRUET T,GEORGES M.A hidden markov model combining linkage and linkage disequilibrium information for haplotype reconstruction and quantitative trait locus fine mapping[J].Genetics,2010,184(3):789-798.
[21]BROWNING S R,BROWNING B L.Rapid and accurate haplotype phasing and missing-data inference for whole-genome association studies by use of localized haplotype clustering[J].AmJHumGenet,2007,81(5):1084-1097.
[22]DRUET T,FARNIR F P.Modeling of identity-by-descent processes along a chromosome between haplotypes and their genotyped ancestors[J].Genetics,2011,188(2):409-419.
[23]AULCHENKO Y S,RIPKE S,ISAACS A,et al.GenABEL:an R library for genome-wide association analysis[J].Bioinformatics,2007,23(10):1294-1296.[24]HAYES B J,GODDARD M E.Technical note:prediction of breeding values using marker-derived relationship matrices[J].JAnimSci,2008,86(9):2089-2092.
[25]MATEUS J C,EDING H,PENEDO M C,et al.Contributions of Portuguese cattle breeds to genetic diversity using marker-estimated kinships[J].AnimGenet,2004,35(4):305-313.
[26]YANG J,FERREIRA T,MORRIS A P,et al.Conditional and joint multiple-SNP analysis of GWAS summary statistics identifies additional variants influencing complex traits[J].NatGenet,2012,44(4):369-375.
[27]BARRETT J C,FRY B,MALLER J,et al.Haploview:analysis and visualization of LD and haplotype maps[J].Bioinformatics,2005,21(2):263-265.
[28]KLEIN R J,ZEISS C,CHEW E Y,et al.Complement factor H polymorphism in age-related macular degeneration[J].Science,2005,308(5720):385-389.
[29]ZHANG Z,DRUET T.Marker imputation with low-density marker panels in Dutch Holstein cattle[J].JDairySci,2010,93(11):5487-5494.
[30]COMINGS D E,MUHLEMAN D,JOHNSON P,et al.Potential role of the estrogen receptor gene (ESR1) in anxiety[J].MolPsychiatry,1999,4(4):374-377.
[31]LEVANO K,PUNIA V,RAGHUNATH M,et al.Atp8a1 deficiency is associated with phosphatidylserine externalization in hippocampus and delayed hippocampus-dependent learning[J].JNeurochem,2012,120(2):302-313.
[32]GREEN E K,GROZEVA D,MOSKVINA V,et al.Variation at the GABAA receptor gene,Rho 1 (GABRR1) associated with susceptibility to bipolar schizoaffective disorder[J].AmJMedGenetBNeuropsychiatrGenet,2010,153B(7):1347-1349.
[33]CRADDOCK N,JONES L,JONES I R,et al.Strong genetic evidence for a selective influence of GABAA receptors on a component of the bipolar disorder phenotype[J].MolPsychiatry,2010,15(2):146-153.
[34]PINHEIRO A P,BULIK C M,THORNTON L M,et al.Association study of 182 candidate genes in anorexia nervosa[J].AmJMedGenetBNeuropsychiatrGenet,2010,153B(5):1070-1080.
[35]SOICHOT M,VAAST A,VIGNAU J,et al.Characterization of functional polymorphisms and glucocorticoid-responsive elements in the promoter of TDO2,a candidate gene for ethanol-induced behavioural disorders[J].AlcoholAlcohol,2013,48(4):415-425.
[36]VASILIEV G V,MERKULOV V M,KOBZEV V F,et al.Point mutations within 663-666 bp of intron 6 of the human TDO2 gene,associated with a number of psychiatric disorders,damage the YY-1 transcription factor binding site[J].FEBSLett,1999,462(1-2):85-88.
[37]MILLER C L,MURAKAMI P,RUCZINSKI I,et al.Two complex genotypes relevant to the kynurenine pathway and melanotropin function show association with schizophrenia and bipolar disorder[J].SchizophrRes,2009,113(2-3):259-267.
[38]PEREZ J A,CLINTON S M,TURNER C A,et al.A new role for FGF2 as an endogenous inhibitor of anxiety[J].JNeurosci,2009,29(19):6379-6387.
[39]TURNER C A,CLINTON S M,THOMPSON R C,et al.Fibroblast growth factor-2 (FGF2) augmentation early in life alters hippocampal development and rescues the anxiety phenotype in vulnerable animals[J].ProcNatlAcadSciUSA,2011,108(19):8021-8025.
[40]GOSHU E,JIN H,LOVEJOY J,et al.Sim2 contributes to neuroendocrine hormone gene expression in the anterior hypothalamus[J].MolEndocrinol,2004,18(5):1251-1262.
[41]KORHONEN P,HUOTARI V,SOININEN H,et al.Glutamate-induced changes in the DNA-binding complexes of transcription factor YY1 in cultured hippocampal and cerebellar granule cells[J].BrainResMolBrainRes,1997,52(2):330-333.
[42]YAMAZAKI M,IGARASHI H,HAMAMOTO M,et al.A case of mitochondrial encephalomyopathy with schizophrenic psychosis,dementia and neuroleptic malignant syndrome[J].RinshoShinkeigaku,1991,31(11):1219-1223.
(編輯郭云雁)
Genome-Wide Association Study on Maternal Infanticide Behavior in Pigs
YANG Hui,FANG Shao-ming,HUANG Xiao-chang,CHEN Cong-ying*,ZHANG Zhi-yan*
(TheStateKeyLaboratoryforPigGeneticImprovementandProductionTechnology,Nanchang330045,China)
Key words:pig;genome-wide association study;maternal infanticide;candidate gene
Abstract:Maternal infanticide behavior in 288 sows from a White Duroc × Erhualian F2resource population was measured.The aim of the study was to identify the QTL affecting maternal infanticide behavior.According to pedigree information and genotyped samples with 60K chip,we speculated the 60K genotypes of the 288 tested individuals.Furthermore,a genome-wide association study was carried out to identify the significant genomic loci associated with maternal infanticide behavior using GenABEL packages in R software.We detected a total of 50 significant SNPs associated with maternal infanticide behavior on SSC2 and SSC8.Totally 21 SNPs on SSC8:35.05-47.54 Mb reached the genome-wide significant level.Only 1 SNP on SSC2:89.5 Mb reached the suggestive significance.The candidate genes around significant SNPs were searched based on its functional annotations.A total of 12 genes were found to be the most important candidate genes for maternal infanticide behavior.This study provided a foundation for identification of major gene or causative mutation influencing the maternal infanticide behavior.
doi:10.11843/j.issn.0366-6964.2016.02.005
收稿日期:2015-03-06
基金項目:國家自然科學基金(30760164);江西省自然科學基金(2008GQN0031)
作者簡介:楊慧(1989-),女,江西彭澤人,博士生,主要從事動物遺傳育種研究,E-mail:389846652@qq.com *通信作者:陳從英,研究員,E-mail:chcy75@hotmail.com;張志燕,博士,Tel/Fax:0791-83813080,E-mail:bioducklily@hotmail.com
中圖分類號:S828;S813.3
文獻標志碼:A
文章編號:0366-6964(2016)02-0241-08

