馬鈴薯X病毒熒光定量PCR檢測體系的建立及應用
尚曉楠, 吳蓓蕾
(中國農業科學院植物保護研究所,植物病蟲害生物學國家重點實驗室,北京 100193)
?
馬鈴薯X病毒熒光定量PCR檢測體系的建立及應用
尚曉楠,吳蓓蕾*
(中國農業科學院植物保護研究所,植物病蟲害生物學國家重點實驗室,北京100193)
馬鈴薯X病毒(Potato virus X,PVX)是危害茄科作物的一種重要病毒,為了建立特異性檢測PVX的實時熒光定量PCR體系,本研究以PVX-1985分離物中外殼蛋白(coatprotein,CP)基因序列為模板,設計引物構建重組質粒并選擇擴增效率高、特異性強的引物成功構建出標準曲線。利用建立的體系,成功檢測到以含pCaPVX440侵染性克隆載體的農桿菌C58C1接種后的本氏煙(Nicotiana benthamiana)中PVX病毒RNA的拷貝數。
馬鈴薯X病毒;熒光定量PCR;本氏煙
馬鈴薯X病毒(Potato virus X,PVX)屬于甲型線形病毒科(Alphaflexiviridae)馬鈴薯X病毒屬(Potexvirus)[1],主要侵染茄科作物,如馬鈴薯、煙草和番茄等。PVX主要通過汁液摩擦傳播[2],單獨侵染引起癥狀較輕,而與馬鈴薯Y病毒(Potato virus Y,PVY)、馬鈴薯S病毒(Potato virus S,PVS)、煙草蝕紋病毒(Tobacco etch virus,TEV)和煙草脈斑病毒(Tobacco vein mottling virus,TVMV)等馬鈴薯Y病毒屬成員復合侵染時造成更嚴重的危害[35]。目前,用于檢測PVX病毒的方法主要有血清學檢測[68]、生物學檢測和分子生物學檢測如核酸分子雜交(nucleicacidhybridization)技術、反轉錄聚合酶鏈式反應(reversetranscriptionpolymerasechainreaction)技術和實時熒光定量PCR(real-timefluorescentquantitativePCR)技術等[910]。與實時熒光定量PCR技術相比,其他方法都存在耗時長、靈敏度低和不能對病毒進行定量檢測等缺點。實時熒光定量PCR技術具有特異性強、靈敏度高,同時可以對樣本進行實時快速定量檢測等優點[11]。本研究建立了PVX實時熒光定量PCR檢測體系,有助于檢測以侵染性克隆為基礎的PVX在單一寄主或多個寄主中連續接種多代后病毒RNA拷貝數的變化趨勢,從而有助于分析以侵染性克隆為基礎的PVX在單一寄主或多寄主侵染循環中的進化特點。另一方面,為監測預警PVX引起的病害提供技術手段。
1 材料與方法
1.1材料
供試材料:質粒pCaPVX440含有完整PVX外殼蛋白(coatprotein,CP)基因,來自PVX-1985分離物,由山東農業大學李向東教授饋贈,用于構建實時熒光定量PCR的標準品,同時由本實驗室轉化至農桿菌C58C1感受態中持續活化保存;本氏煙(Nicotiana benthamiana)由本實驗室長期種植保存,經含質粒pCaPVX440的農桿菌C58C1接種后,用于驗證所構建體系的可行性。
供試試劑:Wizard?SVGelandPCRClean-UpSystem和pGEM-TEasy試劑盒購于Promega公司;感受態Trans 109 E. coli購于Transgene公司;AxyPrepPlasmidMiniprepKit購于Axygen公司;SuperReal熒光定量預混試劑增強版(SYBRGreen)和FastQuantRTKit(withgDase)購自天根生化科技有限公司;TRIzol購于Invitrogen公司;TaKaRaTaqTM,dNTPs,10×PCRbuffer(Mg2+plus),DL2000DNAmarker,DL500DNAmarker購自TaKaRa公司。
1.2引物設計與合成
根據GenBank中的PVX-1985分離物中PVXCP基因的序列(登錄號:EU571480),集合世界已公布的63個PVX-cp基因,利用MEGA5.0分析該分離物CP核酸序列與NCBI中PVX其他分離物的CP序列的相似性,結果顯示與亞洲、澳洲、部分歐洲分離物的核酸序列相似性為94.1%~100%,與美洲、部分歐洲分離物的核酸相似性為74.7%~76.1%。利用PrimerPremier3.0軟件設計引物PVX-F和PVX-R,用于普通PCR檢測。同時,設計1對用于構建陽性質粒(實時熒光定量PCR的標準品)的引物BZP5和BZP3,在2個引物之間設計引物PVX-F1與PVX-R1用于熒光定量檢測PVX。引物均由上海生工生物工程(股份)有限公司合成。引物序列和擴增片段長度見表1。
1.3實時熒光定量方法的建立
1.3.1PVX片段的克隆和測序及重組質粒制備
以pCaPVX440質粒為模板,BZP5和BZP3為引物,擴增PVX部分片段。目的片段長度為653bp。在梯度PCR儀(Eppendorf)上設定程序為:94℃ 5min;94℃ 45s, 62℃ 90s, 72℃ 90s,30個循環;最后72℃ 10min。經1%瓊脂糖電泳檢測后利用Wizard?SVGelandPCRClean-UpSystem(Promega)試劑盒回收PCR產物并與pGEM-TEasy(Promega)載體連接,將連接產物轉化到感受態Trans 109 E. coli(Transgene),經測序確定陽性克隆,用AxyPrepPlasmidMiniprepKit(Axygen)提取陽性重組質粒。
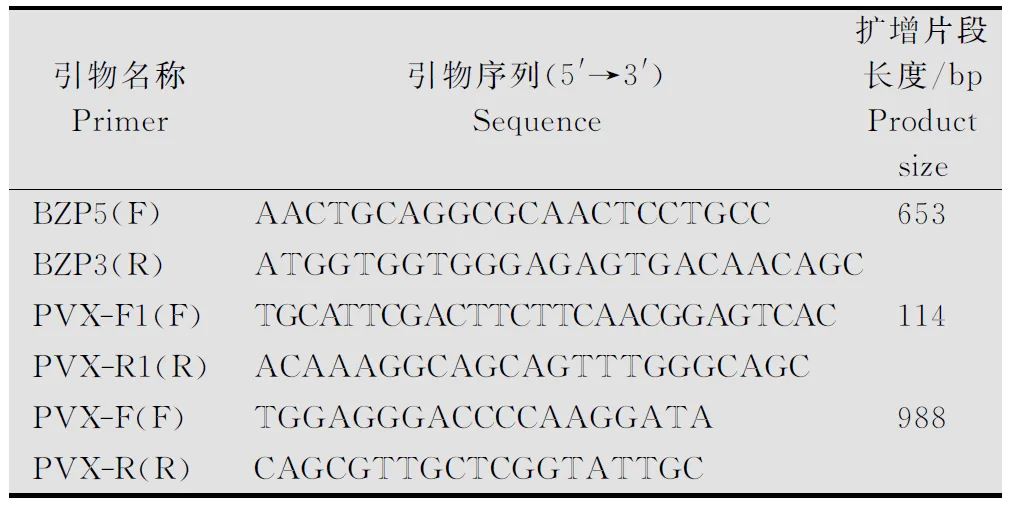
表1 檢測PVX的引物1)
1)F:上游引物;R:下游引物。
F:Forwardprimer;R:Reverseprimer.
1.3.2PVX 實時熒光定量PCR標準曲線的制作
經NanoDrop-2000分光光度計測定,陽性質粒濃度為140ng/μL,該重組質粒的大小為3 668bp,根據公式:A=(B×6.022×1023)/(C×650×109)[其中A為拷貝數/μL,B為質粒濃度,C為重組質粒長度]計算出拷貝數為3.54×1010/μL,將計算出的拷貝數轉換成2×108拷貝/3μL,再進行10倍梯度稀釋,得到2×104~2×108拷貝/3μL的重組質粒,保存在-70℃,作為標準品使用。
以2×104~2×108拷貝/3μL的重組質粒DNA為模板,利用SuperReal熒光定量預混試劑增強版(SYBRGreen)試劑盒進行PCR反應,共5個梯度,每個梯度3個重復。以RNase-free超純水為空白對照。總反應體系20μL:DNA/cDNA模板3μL,正向引物(10μmol/L)0.6μL,反向引物(10μmol/L)0.6μL,2×SuperRealPremixPlus10μL,50×ROXReferenceDye0.4μL,RNase-free超純水 5.4μL。體系配好之后在7500RealTimePCR儀器(AppliedBiosystems)上運行。反應程序:95℃預變性15min; 95℃ 10s,60℃ 32s,72℃ 32s,40個循環;之后進入溶解曲線階段95℃ 15s,60℃ 1min,95℃ 30s,60℃ 15s。反應結束后,系統會自動生成標準曲線,計算出標準曲線的斜率(slope)和引物的擴增效率。Efficiency(%)=(10(-1/slope)-1)×100。
1.4利用實時熒光定量PCR體系定量檢測本氏煙草中的PVX的 RNA拷貝數
用懸浮溶液(10mmol/LMgCl2,10mmol/LMES(pH=5.6),0.1mmol/L乙酰丁香酮)懸浮含pCaPVX440侵染性克隆載體的農桿菌C58C1菌體,調整濃度使其A600為0.5左右,28℃靜置3h。選取本氏煙植株(5~6周齡)的第4~6片真葉,取不含針頭的5mL一次性注射器吸取菌液進行注射接種15株本氏煙(N. benthamiana)[12],同時,吸取懸浮液進行注射作為陰性對照,觀察植株發病癥狀,采集發病癥狀明顯的植株、健康植株和陰性對照植株提取RNA(參照TRIzol試劑的說明書)。提取的RNA用1%瓊脂糖電泳檢測其完整性,NanoDrop-2000分光光度計檢測其純度。
利用FastQuantRTKit(withgDase)反轉錄1μgRNA得到cDNA第一條鏈。以PVX-F1與PVX-R1為引物,以2×104~2×108拷貝/3μL的重組質粒DNA為模板,利用建立的PVX實時熒光定量PCR體系定量檢測本氏煙草中的PVX的RNA拷貝數。
2 結果與分析
2.1PVX片段的克隆和重組質粒測序
以pCaPVX440質粒為模板,BZP5和BZP3為引物擴增,1%瓊脂糖凝膠電泳結果顯示,特異的擴增片段約為653bp(圖1a)。回收產物與pGEM-TEasy載體的連接產物轉入大腸桿菌感受態Trans 109 E. coli,PCR檢測得到陽性克隆(圖1b),最終選取4個陽性克隆搖菌提取質粒,經測序鑒定插入序列,通過VectorNT10.0軟件將測序結果與PVX-1985分離物CP序列(EU571480)比對,序列一致性達到100%,說明PVXCP基因的部分片段已經成功插入克隆載體。選取質粒濃度為140ng/μL,A260/A280為1.80的重組質粒作為標準質粒。
2.2PVX實時熒光定量PCR標準曲線的建立
分別以2×104~2×108拷貝/3μL的重組質粒為模板,采用引物PVX-F1與PVX-R1擴增,每個模板3個重復,建立PVX熒光定量PCR的標準曲線。圖2a為建立的熒光定量PCR標準曲線,擴增效率為109.39%,決定系數R2為0.999,Ct值與標準品濃度的對數之間存在很好的線性關系。圖2b是2%瓊脂糖凝膠電泳檢測熒光定量PCR的產物,目的條帶單一,即引物擴增特異性好。
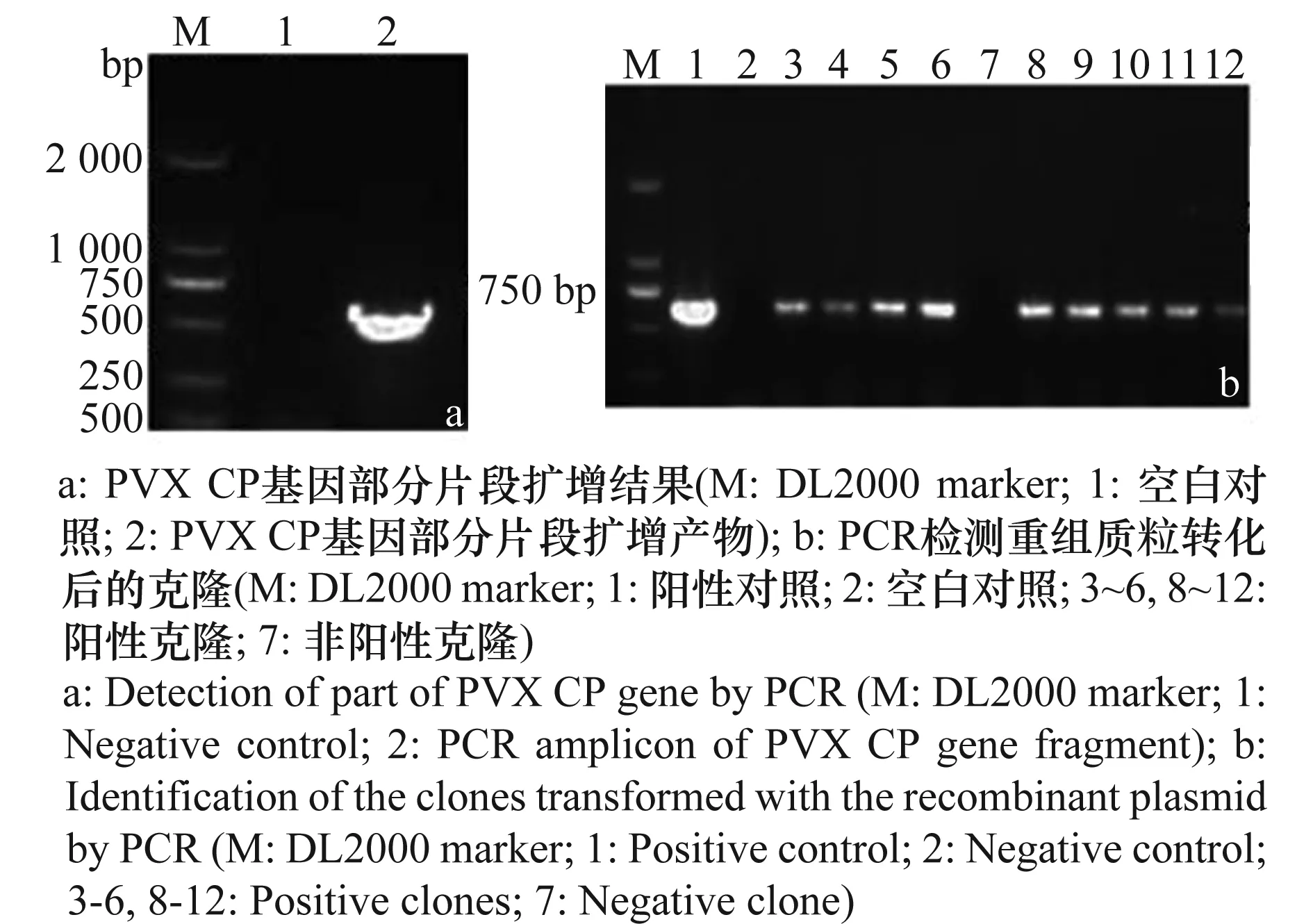
圖1 重組質粒的檢測結果Fig.1 Detection of the recombinant plasmid
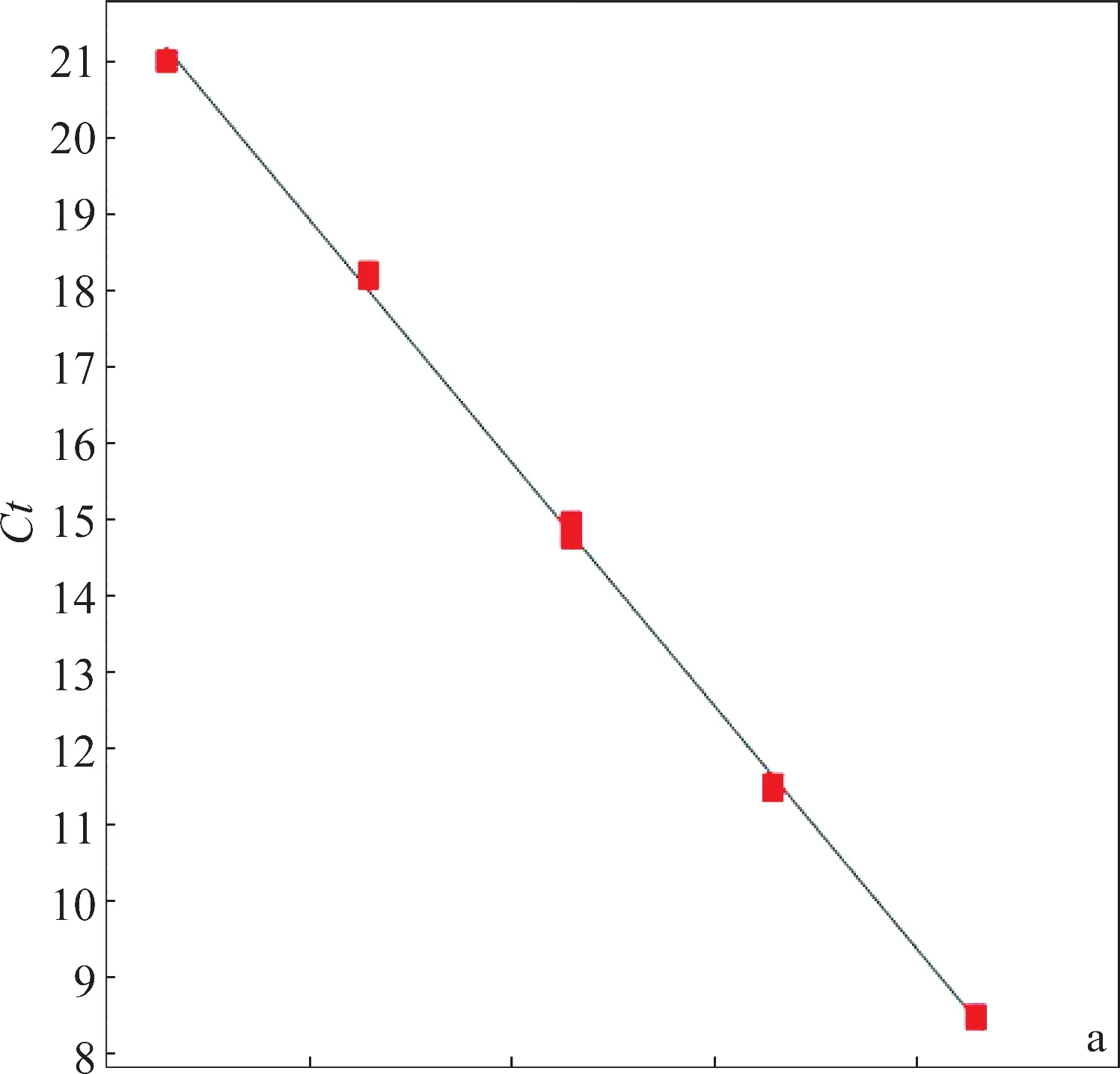
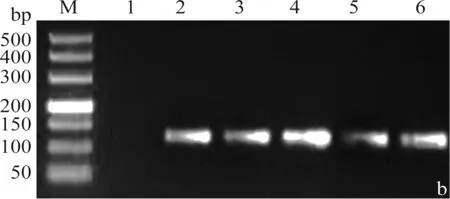
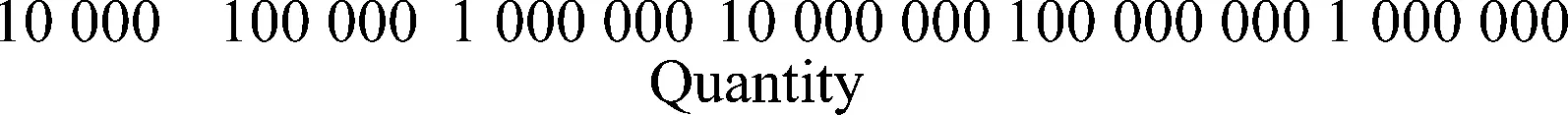

圖2 PVX熒光定量PCR標準曲線的建立Fig.2 Establishment of the real time quantitative PCR standard curve of PVX
2.3PVX實時熒光定量PCR定量方法的應用
2.3.1經農桿菌接種的本氏煙中PVX的普通RT-PCR檢測
接種14d后,在本氏煙植株頂端葉片出現花葉癥狀,如圖3a。選取發病癥狀明顯的植株6株、健康植株和陰性對照植株共計8株提取RNA,圖3b為經農桿菌接種的本氏煙RNA電泳圖。供試的每個樣品的RNA均為3條帶即28S、18S和5S,說明RNA完整性很好。利用FastQuantRTKit反轉錄1μgRNA得到cDNA第一條鏈。以cDNA為模板,使用引物PVX-F和PVX-R進行PCR擴增,擴增結果見圖3c。由圖可知,每株發病植株均檢測到PVX。
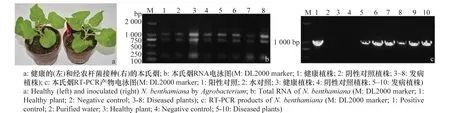
圖3 經農桿菌接種的本氏煙中PVX的普通RT-PCR檢測Fig.3 Detection of PVX in Nicotiana benthamiana inoculated by Agrobacterium by RT-PCR
2.3.2檢測發病本氏煙中PVX的RNA拷貝數
以拷貝數為2×104~2×108的標準質粒和6株發病明顯植株的cDNA為模板,使用引物PVX-F1與PVX-R1,在同一反應條件下進行熒光定量PCR。系統會自動生成標準曲線和計算出每個待測樣品(RNA含量為1μg)中PVX的RNA拷貝數(表2),擴增效率為106.359%,決定系數R2為0.999,標準曲線方程為:y=-3.178x+34.848。由表中數據看出,每個樣品都落在了標準曲線范圍內。
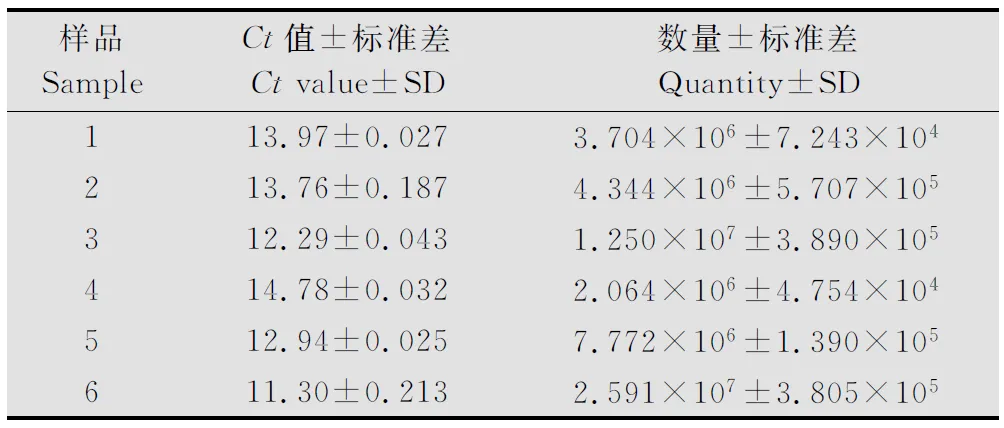
表2 RNA含量為1 μg時本氏煙中PVX的RNA拷貝數
3 討論
目前已有超過40種病毒可以侵染馬鈴薯,其中危害嚴重而且常見的主要有PVX、PVS、PVY、PLRV等[13]。檢測PVX的分子生物學技術大多為RT-PCR[9]和核酸斑點雜交(nucleicacidspothybridization,NASH)[14]等。這些方法只是定性檢測PVX,而且存在假陽性現象,為了可以定量檢測PVX,本試驗以PVXCP基因保守區域序列設計引物,建立了熒光定量PCR體系。由于PVX-1985與亞洲、澳洲、部分歐洲分離物的核酸序列相似性為94.1%~100%,該方法可以實時、高靈敏度地檢測樣品中病毒RNA的拷貝數, 并對我國的樣品具有較為廣泛的適用性。
目前世界上只獲得了17個PVX分離物的基因組全序列,分別來自中國、日本、韓國、伊朗、俄羅斯、英國、荷蘭、南美和阿根廷。其中包含3個中國分離物,PVX-1985 (EU571480)、FX21 (EF423572)和Taiwan(AF272736)。世界上的PVX包含4個株系,集合17個PVX分離物的系統進化分析發現PVX群體分為兩大類群,一個來自歐亞群體,另一個來自美洲群體。它們的寄主分別有馬鈴薯、煙草、千日紅等。PVX-1985的基因組全序列與歐洲和亞洲其他分離物的核苷酸相似性在94.2%以上,氨基酸相似性在97.1%以上,而與美洲分離物的核苷酸相似性為80.2%,氨基酸相似性為90.3%[15]。但以侵染性克隆為基礎,該病毒在單個或多個寄主中侵染循環的進化特點仍是研究空白。本試驗建立的PVX實時熒光定量PCR體系可以定量檢測到PVX的RNA拷貝數,為研究該病毒在單一寄主或多個寄主侵染循環中的進化特點奠定基礎,具有實用價值。
[1]AdamsMJ,AntoniwJF,FauquetCM.MolecularcriteriaforgenusandspeciesdiscriminationwithinthefamilyPotyviridae[J].ArchivesofVirology, 2005, 150(3):459479.
[2]HarrisonBD.Advancesingeminivirusresearch[J].AnnualReviewofPhytopathology, 1985, 23(1):5582.
[3]BanceVB.ReplicationofPotato virus XRNAisalteredincoinfectionswithPotato virus Y [J].Virology, 1991, 182(2):486494.
[4]JamalA,NasirIA,TabassumB,etal,MolecularcharacterizationofcapsidproteingeneofPotato virus XfromPakistan[J].AfricanJournalofBiotechnology, 2012,11(74):1385413857.
[5]NyalugweEP,WilsonCR,CouttsBA,etal.BiologicalpropertiesofPotato virus Xinpotato:EffectsofmixedinfectionwithPotato virus Sandresistancephenotypesincultivarsfromthreecontinents[J].PlantDisease,2012, 96:4354.
[6]AwasthiLP,SinghSP,ChaubeyAN,etal.MolecularcharacterizationofpotatovirusesthroughRT-PCRandelectron-microscopy[J].CaribbeanJournalofScienceandTechnology, 2014, 2:405410.
[7]NosheenQ,HameedS,MughalSM,etal.SerologicalidentityofPotato virus X (PVX)andPCRcharacterizationofitscoatprotein(CP)gene[J].InternationalJournalofPlantPathology,2013, 2(2):9296.
[8]朱國春, 朱國慶. 用DTBA方法檢測馬鈴薯病毒[J]. 中國馬鈴薯, 2000, 14(1):5960.
[9]吳興泉, 陳士華, 謝聯輝. 馬鈴薯X病毒的分子鑒定與檢測技術[J]. 河南農業科學, 2006(2):7275.
[10]袁青, 殷幼平, 王中康. 馬鈴薯病毒病分子檢測技術研究進展[J]. 中國馬鈴薯, 2003, 17(1):3336.
[11]AryaM,ShergillIS,WilliamsonM,etal.Basicprinciplesofreal-timequantitativePCR[J].ExpertReviewofMolecularDiagnostics,2005, 5(2):209219.
[12]JiaHongge,PangYongqi,FangRongxiang.Agroinoculationasasimplewaytodeliveratobaccomosaicvirus-basedexpressionvector[J].ActaBotanicaSinica, 2003, 45(7):770773.
[13]GrammatikakiG,AvgelisA,SonninoA.BehaviorofpotatogametoclonalplantsagainstthenecroticstrainofpotatoYpotyvirus[J].RussianJournalofPlantPhysiology, 2007, 54(4):507512.
[14]RobertsIM,HarrisonBD.Detectionofpotatoleafrollandpotatomop-topvirusesbyimmunosorbentelectronmicroscopy[J].AnnalsofAppliedBiology, 1979, 93(3):289297.
[15]YuXiaoqing,JiaJinlei,ZhangChengling,etal.Phylogeneticanalysesofanisolateobtainedfrompotatoin1985revealedPotato virus XwasintroducedtoChinaviamultipleevents[J].VirusGenes, 2010, 40(3):447451.
(責任編輯:楊明麗)
Developmentandapplicationofareal-timequantitativePCRsystemfordetectionofPotato virus X
ShangXiaonan,WuBeilei
(StateKeyLaboratoryforBiologyofPlantDiseasesandInsectPests,InstituteofPlantProtection,ChineseAcademyofAgriculturalSciences,Beijing100193,China)
Potato virus X (PVX)isanimportantvirusinfectingmanysolanaceouscrops.Inordertodevelopareal-timequantitativePCRsystemforspecificdetectionofPVX,thestandardcurvewasestablishedsuccessfullybasedontherecombinantplasmidandhighefficientandspecificprimersderivedfromcoatproteingenesequenceofPVX-1985isolate.WesuccessfullydetectedthecopyquantityofRNAfromPVXintheNicotiana benthamianawhichinjectedbytheAgrobacteriumC58C1containingpCaPVX440plasmid.
Potato virus X(PVX);real-timequantitativePCR;Nicotiana benthamiana
20150319
20150514
中國農業科學院科技創新工程(2014)
E-mail:blwu@ippcaas.cn
S432.41;S532
ADOI:10.3969/j.issn.05291542.2016.03.029

