TNIP1基因多態性與漢族人系統性紅斑狼瘡相關性研究
王勇 韓建文 李鴻斌 阿拉騰楚魯 孫志強 呂新翔 烏日娜
010050呼和浩特,內蒙古醫科大學附屬醫院風濕科(王勇、李鴻斌),皮膚科(韓建文、阿拉騰楚魯、孫志強、呂新翔、烏日娜)
·論著·
TNIP1基因多態性與漢族人系統性紅斑狼瘡相關性研究
王勇 韓建文 李鴻斌 阿拉騰楚魯 孫志強 呂新翔 烏日娜
010050呼和浩特,內蒙古醫科大學附屬醫院風濕科(王勇、李鴻斌),皮膚科(韓建文、阿拉騰楚魯、孫志強、呂新翔、烏日娜)
目的 探討人腫瘤壞死因子α誘導蛋白3(TNFAIP3)相互作用蛋白1(TNIP1)基因多態性與漢族人系統性紅斑狼瘡(SLE)的遺傳關聯性。方法 收集284例漢族人SLE和630例漢族人對照,選擇TNIP1基因區域120個單核苷酸多態性(SNP),利用連接酶檢測反應(LDR)對其進行基因分型,對分型數據利用PLINK 1.07和Haploview軟件進行統計分析。結果 經過數據質控,最終105個SNP的分型數據進入最終統計分析。rs3805433C、rs12516176C、rs6869605C和rs4958882G的等位基因頻率在SLE組(參考等位基因頻率0.301~0.306)高于對照組(參考等位基因頻率0.221~0.225),差異有統計學意義(OR:1.50~1.53,均P<4.72×10-4)。這4個SNP間存在強連鎖不平衡(r2≥0.871,D′≥0.938),且與既往報道的SLE相關SNP rs10036748間存在中等程度的連鎖不平衡(r2≥ 0.073,D′≥ 0.868)。單倍型分析發現,單倍型(H2:CCCGT)在病例組中的頻率(0.290)顯著高于對照組(0.210),差異有統計學意義(OR=1.54,P<4.72×10-4)。結論 TNIP1基因多態性與漢族人SLE具有相關性。
紅斑狼瘡,系統性;多態性,單核苷酸;遺傳關聯研究;基因,TNIP1
系統性紅斑狼瘡(SLE)是一種典型的自身免疫性疾病,表現為自身抗體產生、免疫功能紊亂、多個組織器官損傷。近年來遺傳學研究發現了一大批SLE易感基因[1]。研究提示,人腫瘤壞死因子α誘導蛋白 3(TNFAIP3)相互作用蛋白 1(TNIP1)基因多態性與漢族人及歐洲人群的SLE均有相關性[2]。本研究根據HapMap 3期數據庫及我們前期完成的漢族人SLE全基因組關聯分析研究結果[2],選擇TNIP1基因區域的120個單核苷酸多態性(single nucleotide polymorphisms,SNP),利用連接酶檢測反應(ligase detection reaction,LDR)對其進行基因分型,探討TNIP1基因多態性與漢族人SLE的相關性。
資料和方法
一、資料
1.研究對象:從內蒙古醫科大學附屬醫院皮膚科及風濕免疫科收集漢族SLE患者284例,年齡11~72(35.22±11.39)歲,發病年齡 9~72(30.00±10.59)歲,女男比例為11.2∶1。從我院體檢中心收集漢族健康對照630例,年齡17~80(35.98±15.96)歲,女男比例為11.8∶1。所有患者符合美國風濕病學會(ACR)SLE 診斷標準[3]。284 例 SLE 臨床表型分布情況見表1。所有對照排除SLE及其他自身免疫性疾病,排除SLE及自身免疫病家族史。采集外周靜脈血5 ml,乙二胺四乙酸(EDTA)抗凝,置于-80℃冰箱保存備用。本研究已通過內蒙古醫科大學附屬醫院倫理委員會批準,并在實施過程中遵守赫爾辛基宣言,所有病例及對照均簽署知情同意書。
2.SNP選擇:根據HapMap 3期數據庫,選擇位于 TNIP1基因區域的 SNP(Chr5:150369701~150461190)。根據在中國人群中最小等位基因頻率(MAF)>0.01,篩選出149個SNP。然后根據我們既往的全基因組關聯分析研究結果,去除29個SNP(既往研究中P>0.1)[2]。最后對120個SNP進行基因分型。
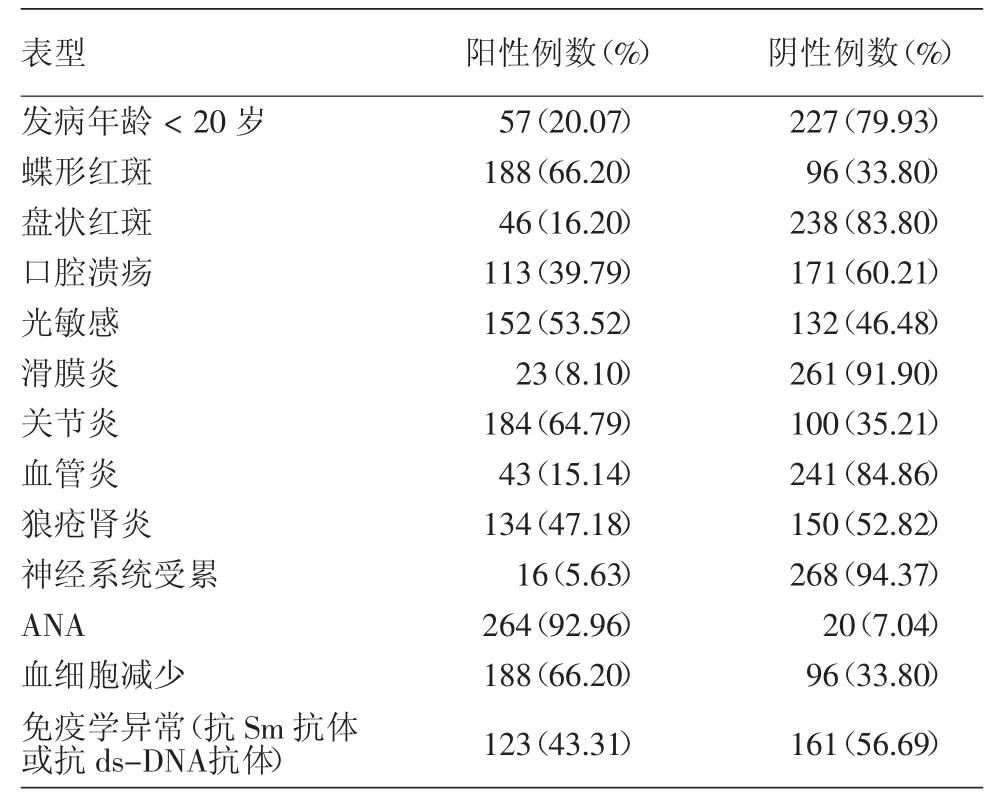
表1 284例系統性紅斑狼瘡患者臨床表型情況
二、方法
1.基因組DNA提取:利用基因組DNA提取試劑盒[康寧生命科學(吳江)有限公司]提取患者及對照基因組DNA,用分光光度儀分別于260 nm和280 nm波長處檢測吸光度,計算DNA濃度,并用凝膠電泳檢測DNA質量,所有DNA樣本均合格。
2.引物及探針的合成:用Primer 3 online(Version 0.4.0)軟件(http://frodo.wi.mit.edu/)和 Oligo(Version 6.31)軟件設計特異性聚合酶鏈反應(PCR)引物和LDR探針,針對每個SNP位點設計2條引物,要盡量保證所有SNP位點引物的T值相同。根據LDR探針設計原則設計LDR的上下游探針[4-5],上游探針5′端用磷酸化修飾。PCR引物由上海瀚宇生物工程有限公司合成,LDR探針由上海英駿生物技術有限公司合成。
3.多重 PCR:PCR 反應體系 20 μl,1 μl模板,2 μl緩沖液,0.6 μl Mg2+,2 μl dNTP,0.2 μl TaqDNA聚合酶,2μl引物混合液,加雙蒸水補足。PCR反應條件:95℃預變性 2 min;94℃ 30 s,56℃退火 1.5 min,65℃30 s,35個循環;最后65℃延伸10 min。
4.多重 LDR:LDR 反應體系 10 μl,1 μl Buffer緩沖液,1 μl探針混合液,0.05 μl Taq DNA 連接酶,4 μl PCR產物,加雙蒸水補足10 μl。探針混合液濃度為2 μmol/L。LDR反應條件:95℃2 min預變性,94℃ 15 s,50℃ 25 s,進行 40個循環。
5.測序與基因分型:采用ABI3730測序儀測序分析反應產物,用Genemapper軟件進行數據分析,判讀各位點上各樣本的基因分型。測序工作由上海翼和應用生物技術有限公司完成。
6.統計分析:所有SNP分型數據質控利用PLINK 1.07軟件完成(http://pngu.mgh.harvard.edu/purcell/plink/)[6]。符合以下任意一條的 SNP 被剔除:分型得率<90%;MAF<1%;不符合Hardy-Weinberg平衡定律(P≤0.01)。經過質控后,計算每個SNP的等位基因頻率,χ2檢驗比較病例組和對照組等位基因頻率及基因型頻率,并計算等位基因的相對危險度估計值比值比OR及其95%可信區間(CI)。對差異顯著的SNP進行兩兩間連鎖不平衡檢驗,計算兩兩間的 r2和 D′值,如果 r2> 0.5和 D′> 0.8,則認為SNP間存在強連鎖不平衡。利用Haploview軟件進行單倍型分析。所有統計檢驗均為雙側概率檢驗,根據Bonferroni多重檢驗校正原理,P<4.72×10-4(0.05/105)為差異有統計學意義。
結 果
一、樣本及SNP質控
經過質控,15個SNP被剔除,其中1個SNP的MAF<0.01,14個 SNP經過 Hardy-Weinberg平衡檢驗,P≤0.01。最終284例SLE患者及630例對照的105個SNP的分型數據進入最終統計分析。SLE組與對照組間年齡、性別差異無統計學意義(P>0.05),具有可比性。
二、等位基因頻率比較及連鎖不平衡檢驗
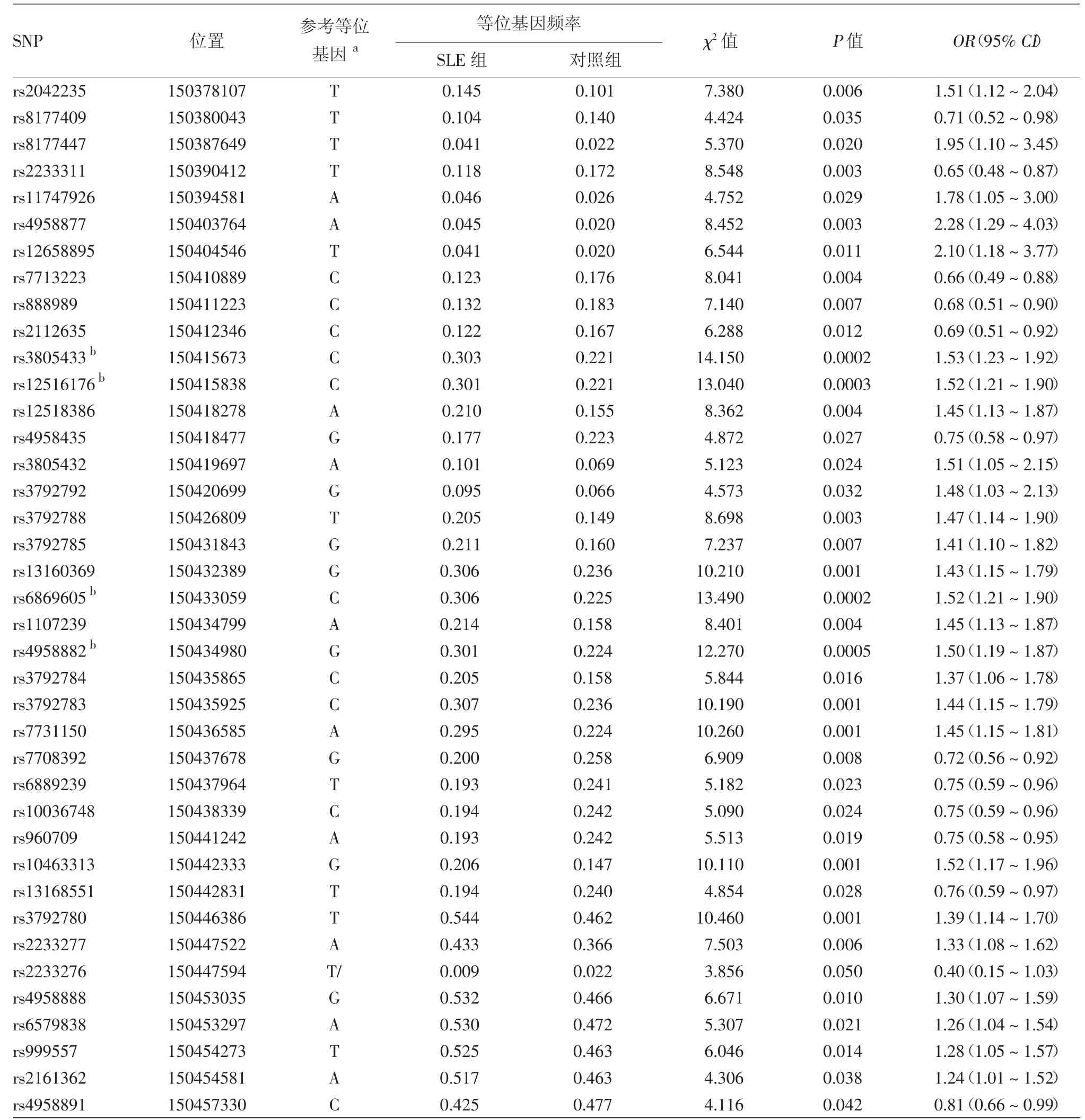
表2 TNIP1基因區域39個SNP在系統性紅斑狼瘡組和對照組間的等位基因頻率比較
經過 χ2檢驗,4 個 SNP(rs3805433、rs12516176、rs6869605、rs4958882)的等位基因頻率在病例組和對照組間的差異有統計學意義(P<4.72×10-4)。另外35個SNP的等位基因頻率在病例組和對照組間差異有統計學意義(P<0.05),但未達到Bonferroni校正檢驗水平(P>4.72×10-4),包括既往漢族人群中的全基因組關聯分析研究報道的相關SNP(rs10036748,P=0.024),見表 2。另外 66個 SNP等位基因頻率在病例組和對照組間差異無統計學意義(P>0.05,數據未顯示),對rs3805433、rs12516176、rs6869605、rs4958882 及 rs10036748 這5個SNP進行兩兩間連鎖不平衡檢驗,前4個SNP兩兩間均存在強連鎖不平衡,而這4個SNP與rs10036748存在中等程度的連鎖不平衡,見表3。Logisitic回歸分析顯示,當控制rs10036748作用后,其余4個SNP的等位基因頻率差異均無統計學意義(P>0.21)。
三、單倍型分析
對 rs3805433、rs12516176、rs6869605、rs4958882及rs10036748這5個SNP進行單倍型分析,結果顯示,單倍型(H2:CCCGT)的頻率在SLE組中的頻率(0.290)顯著高于對照組(0.210),差異有統計學意義(P < 4.72× 10-4),見表 4。
四、臨床表型分析
對病例組進行臨床表型分層分析,分別按照發病年齡<20歲、蝶形紅斑、盤狀紅斑、口腔潰瘍、光敏感、滑膜炎、關節炎、血管炎、狼瘡腎炎、神經系統受累、抗核抗體、血細胞減少、抗Sm抗體以及抗ds-DNA抗體共13項臨床/實驗室指標分為陽性組和陰性組。對兩組間上述5個SNP等位基因頻率及單倍型頻率進行比較,均未發現差異有統計學意義(P>0.05)。
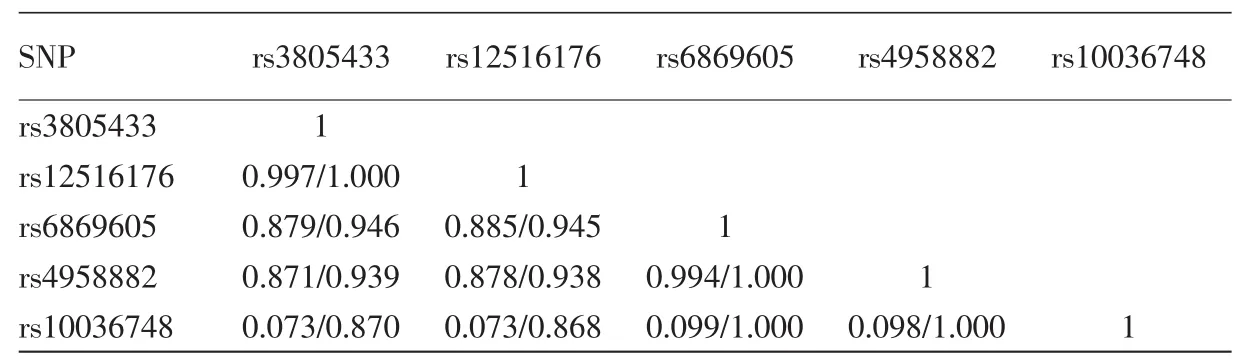
表3 漢族人系統性紅斑狼瘡組和對照組間差異有統計學意義的5個SNPa兩兩間的連鎖不平衡分析(r2/D′)
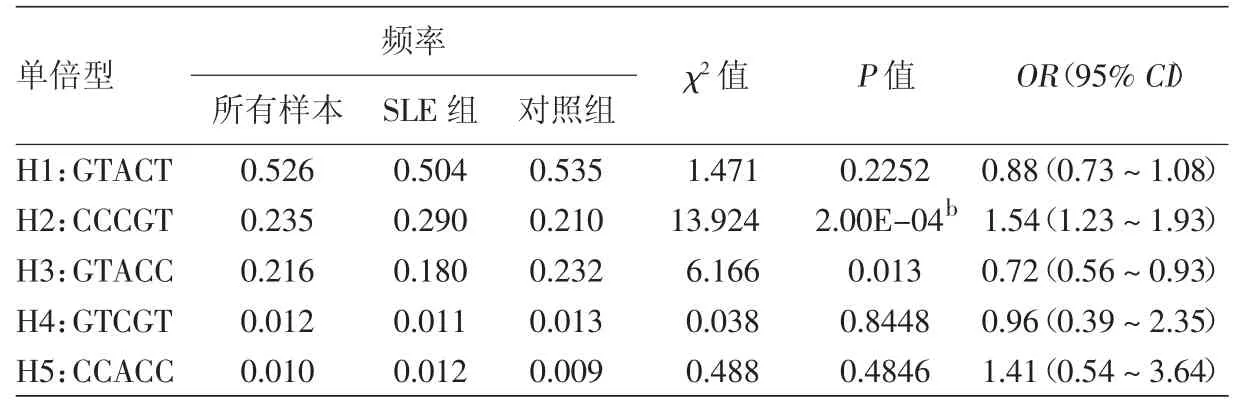
表4 漢族人系統性紅斑狼瘡(SLE)組和對照組間差異有統計學意義的5個SNPa單倍型分型
討 論
TNIP1基因位于5號染色體,編碼蛋白A20結合核因子κB抑制蛋白1(ABIN1)。TNIP1與其配體TNFAIP3相互作用可以下調核因子κB(NF-κB)病理通路的活性[7]。TNIP1 mRNA在外周血淋巴細胞、脾臟、骨骼肌、腎臟中均有表達[8]。NF-κB 的激活可以誘導TNIP1的表達,而TNIP1表達過度可通過腫瘤壞死因子(TNF)抑制 NF-κB 的活性[7]。
TNIP1和TNFAIP3兩個相互作用的基因曾被證實是多種自身免疫性疾病的易感基因,如類風濕性關節炎、乳糜瀉、潰瘍性結腸炎、銀屑病和 SLE[9-10]。TNIP1基因多態性與中國人SLE的相關性已在多項研究中有報道[2,11-13],提示 NF-κB 病理通路在多種自身免疫病的發生、發展中有著重要的作用。基因-基因交互作用分析也發現,NFKB1基因多態性rs28362491和TNIP1基因多態性rs3792783與SLE的相關性有疊加作用,二者均參與NF-κB病理通路[14]。TNIP1基因多態性可能通過影響基因表達或功能引起NF-κB病理通路異常,從而參與SLE的發病。
本研究連鎖不平衡分析結果提示,新發現的4個SLE 相關 SNP(rs3805433、rs12516176、rs6869605、rs4958882)與既往報道的rs10036748存在中等程度的連鎖不平衡,logisitic回歸分析提示,這5個SNP與SLE的相關性可能來自于同一個疾病關聯信號。
Adrianto 等[12]研究發現,在 TNIP1基因的啟動子區域存在2個SLE相關的危險單倍型。本研究發現的危險單倍型(H2:CCCGT)位于 TNIP1基因的5′UTR到內含子5的范圍內(長度約為22.7 kb),位置上與既往報道的危險單倍型有重疊[12]。在另外一項漢族人群的SLE關聯分析研究中,Zhang等[15]發現的SLE危險單倍型也位于TNIP1基因的5′UTR區域。幾項研究發現的SLE危險單倍型部分有重疊,均指向TNIP1基因的5′UTR區域,但是這些SLE危險單倍型間的具體關系仍未能明確,它們是否代表著同一個關聯信號仍需要進一步的研究來闡明。但可以明確的一點是,無論是歐洲人群還是漢族人群中,TNIP1基因均是SLE的一個易感基因,且在SLE的發病機制中發揮著重要的作用。
在根據臨床表型分層分析的結果中,我們未發現在各臨床表型中上述5個SNP及單倍型頻率的差異。2010年He等[16]在一項大樣本量的基因型-表型分析研究中發現,rs10036748與光敏感具有相關性,但未發現與其他臨床表型間的相關性。本研究臨床表型分析陰性結果可能與樣本量偏小有關。
Caster等[17]的小鼠動物模型研究發現TNIP1基因突變[D485N],當表達未激活的ABIN1時,小鼠可以發生類似自身免疫病的癥狀,并且出現狼瘡腎炎的表現。另外,對兩個人種的遺傳關聯研究發現,歐洲人群中與狼瘡腎炎相關性最強的SNP是rs7708392,非洲裔人群中是rs4958881。本研究中SNP rs7708392在SLE和對照中的頻率差異未達到Bonferroni多重檢驗校正水平(P<4.72×10-4),而rs4958881在既往我們進行全基因組關聯分析研究中沒有發現相關性(P=0.7626)[2],因此本次實驗未對其進行分型實驗。
本研究通過對TNIP1基因區域的105個SNP進行分型、統計分析,發現4個SLE相關的SNP和1個SLE危險單倍型,證實了TNIP1是漢族SLE的易感基因。但存在以下幾點不足:①由于樣本量有限,本研究的研究效力不足,一些與SLE關聯性較弱的SNP可能被遺漏;②本研究沒有覆蓋低頻SNP,僅僅對TNIP1基因區域MAF大于0.01的SNP進行了分析,低頻SNP與疾病的相關性需要更大樣本量的遺傳關聯研究來完成;③本研究發現的4個SLE相關SNP可能不是影響SLE易感性的原因SNP,需要更加精細的測序分析來鑒定SLE相關的原因SNP;④由于僅僅對單個位點進行關聯分析研究,基因-基因交互作用研究仍有待進一步開展。
總之,本研究通過遺傳關聯分析研究,在TNIP1基因區域發現4個SLE相關SNP及一個危險單倍型,證實了TNIP1基因多態性與漢族人SLE的相關性,但仍需進一步的精細定位分析及功能學研究來闡明TNIP1基因在SLE發病機制中的作用。
[1]Deng Y,Tsao BP.Genetic susceptibility to systemic lupus erythematosus in the genomic era[J].Nat Rev Rheumatol,2010,6(12):683-692.DOI:10.1038/nrrheum.2010.176.
[2]Han JW,Zheng HF,Cui Y,et al.Genome-wide association study in a Chinese Han population identifies nine new susceptibility loci forsystemic lupus erythematosus [J].Nat Genet,2009,41(11):1234-1237.DOI:10.1038/ng.472.
[3]Hochberg MC.Updating the American College of Rheumatology revised criteria for the classification ofsystemic lupus erythematosus[J].Arthritis Rheum,1997,40(9):1725.
[4]Thomas G,Sinville R,Sutton S,et al.Capillary and microelectrophoretic separations of ligase detection reaction products produced from low-abundantpointmutationsin genomic DNA [J].Electrophoresis,2004,25(10-11):1668-1677.DOI:10.1002/elps.200405886.
[5]Yi P,Chen Z,Yu L,et al.Development of a PCR/LDR/capillary electrophoresis assay with potential for the detection of a betathalassemia fetal mutation in maternal plasma[J].J Matern Fetal Neonatal Med,2010,23 (8):920-927.DOI:10.3109/14767050903387060.
[6]Purcell S,Neale B,Todd-Brown K,et al.PLINK:a tool set for whole-genome association and population-based linkage analyses[J].Am J Hum Genet,2007,81 (3):559-575.DOI:10.1086/519795.
[7]Verstrepen L,Carpentier I,Verhelst K,et al.ABINs:A20 binding inhibitors of NF-kappa B and apoptosis signaling [J].Biochem Pharmacol,2009,78(2):105-114.DOI:10.1016/j.bcp.2009.02.009.
[8]Fukushi M,Dixon J,Kimura T,et al.Identification and cloning of a novel cellular protein Naf1,Nef-associated factor 1,that increases cellsurface CD4 expression[J].FEBS Lett,1999,442(1):83-88.DOI:10.1016/S0014-5793(98)01631-7.
[9]Maxwell JR,Gowers IR,Wilson AG.Complex genetic association of 6q23 with autoimmune rheumatic conditions [J].Arthritis Res Ther,2009,11(2):107.DOI:10.1186/ar2663.
[10]Ramos PS,Criswell LA,Moser KL,et al.A comprehensive analysis of shared loci between systemic lupus erythematosus(SLE)and sixteenautoimmune diseases reveals limited genetic overlap[J].PLoS Genet,2011,7(12):e1002406.DOI:10.1371/journal.pgen.1002406.
[11]Zhong H,Li XL,Li M,et al.Replicated associations of TNFAIP3,TNIP1 and ETS1 with systemic lupus erythematosus in asouthwestern Chinese population [J].Arthritis Res Ther,2011,13(6):R186.DOI:10.1186/ar3514.
[12]Adrianto I,Wang S,Wiley GB,et al.Association of two independent functional risk haplotypes in TNIP1 with systemic lupus erythematosus[J].Arthritis Rheum,2012,64 (11):3695-3705.DOI:10.1002/art.34642.
[13]Zuo XB,Sheng YJ,Hu SJ,et al.Variants in TNFSF4,TNFAIP3,TNIP1,BLK,SLC15A4 and UBE2L3 interact to confer risk of systemic lupuserythematosus in Chinese population[J].Rheumatol Int,2014,34(4):459-464.DOI:10.1007/s00296-013-2864-3.
[14]Cen H,Zhou M,Leng RX,et al.Genetic interaction between genes involved in NF-κB signaling pathway in systemic lupus erythematosus[J].Mol Immunol,2013,56 (4):643-648.DOI:10.1016/j.molimm.2013.07.006.
[15]Zhang DM,Cheng LQ,Zhai ZF,et al.Single-nucleotide polymorphism and haplotypes of TNIP1 associated with systemic lupuserythematosus in a Chinese Han population[J].J Rheumatol,2013,40(9):1535-1544.DOI:10.3899/jrheum.121391.
[16]He CF,Liu YS,Cheng YL,et al.TNIP1,SLC15A4,ETS1,RasGRP3 and IKZF1 are associated with clinical features of systemic lupuserythematosus in a Chinese Han population[J].Lupus,2010,19(10):1181-1186.DOI:10.1177/0961203310367918.
[17]Caster DJ,Korte EA,Nanda SK,et al.ABIN1 dysfunction as a genetic basis for lupus nephritis[J].J Am Soc Nephrol,2013,24(11):1743-1754.DOI:10.1681/ASN.2013020148.
Association analysis between TNIP1 gene polymorphisms and systemic lupus erythematosus in a Chinese Han population
Wang Yong,Han Jianwen,Li Hongbin,Alateng Chulu,Sun Zhiqiang,Lyu Xinxiang,Wu Rina
Department of Rheumatism,Affiliated Hospital of Inner Mongolia Medical University,Hohhot 010050,China(Wang Y,Li HB);Department of Dermatology,Affiliated Hospital of Inner Mongolia Medical University,Hohhot 010050,China(Han JW,Alateng CL,Sun ZQ,Lyu XX,Wu RN)
Objective To investigate association between polymorphisms of the tumor necrosis factor α-induced protein 3 (TNFAIP3)interacting protein 1 (TNIP1)gene and systemic lupus erythematosus(SLE)in a Chinese Han population.Methods Blood samples were collected from 284 patients with SLE of Han nationality(SLE group)and 630 healthy controls of Han nationality (control group).Ligase detection reaction (LDR)was performed to determine the genotypes of 120 single nucleotide polymorphisms (SNPs)in the TNIP1 gene.Data were analyzed with the PLINK 1.07 package and Haploview software.Results After quality control,data on 105 SNPs underwent statistical analysis finally.Four SNPs including rs3805433,rs12516176,rs6869605 and rs4958882 in the TNIP1 gene were significantly associated with SLE (OR:1.50-1.53,allP<4.72×104),and there was a significant increase in the frequency of rs3805433 C,rs12516176 C,rs6869605 C and rs4958882 G alleles in the SLE group (0.301-0.306)compared with the control group(0.221-0.225).There was strong linkage disequilibrium between these 4 SNPs(r2≥ 0.871,D′≥0.938).In addition,moderate linkage disequilibrium was observed between these 4 SNPs and a previously reported SLE-related SNP rs10036748 (r2≥ 0.073,D′≥ 0.868).The frequency of the haplotype H2:CCCGT was significantly higher in the SLE group than in the control group(0.290 vs.0.210,OR=1.54,P < 4.72 × 10-4).Conclusion TNIP1 gene polymorphisms are associated with SLE in Chinese Han population.
Lupus erythematosus,systemic;Polymorphism,single nucleotide;Genetic association studies;Genes,TNIP1
Han Jianwen,Email:hanjianwen1981@hotmail.com
韓建文,Email:hanjianwen1981@hotmail.com
10.3760/cma.j.issn.0412-4030.2016.03.003
國家自然科學基金(31160192、81160189);內蒙古自治區衛生和計劃生育委員會醫療衛生科研計劃項目(201303057);內蒙古自治區高等學校“青年科技英才支持計劃”(NJYT-14-B17);內蒙古醫科大學青年創新基金(NY2011QN005)
Fund programs:National Natural Science Foundation of China (31160192,81160189);Medical and Health ResearchProjectof Health and Family Planning Commission of Inner Mongolia Autonomous Region of China(201303057);Program for Young Talents in Science and Technology in Higher Education Institutions of Inner Mongolia Autonomous Region of China(NJYT-14-B17);Youth Innovation Fund of Inner Mongolia MedicalUniversity(NY2011QN005)
2015-07-02)
(本文編輯:周良佳 顏艷)

