miR-210在馬氏珠母貝外套膜礦化機制中的結構和功能預測
潘曉艷,鄭 哲,田榮榮,焦 鈺,杜曉東
(廣東海洋大學水產學院,廣東 湛江 524025)
miR-210在馬氏珠母貝外套膜礦化機制中的結構和功能預測
潘曉艷,鄭 哲,田榮榮,焦 鈺,杜曉東
(廣東海洋大學水產學院,廣東 湛江 524025)
為進一步探究Pm-miR-210在馬氏珠母貝貝殼形成的作用,利用miR-RACE技術驗證Pm-miR-210的成熟體序列并進行序列分析,通過生物信息學分析來獲得Pm-miR-210的前體序列,并進行組織定量和靶位點預測。結果表明,Pm-miR-210的miR-RACE結果與高通量測序結果一致,利用轉錄組數據庫與Pm-miR-210成熟體序列比對,獲得的前體序列長度為80 bp并具有典型的頸環結構。多序列比對結果顯示,Pm-miR-210成熟體序列的種子區域在不同物種間具有極高的保守性。熒光定量結果得出,Pm-miR-210在外套膜邊緣膜和中央膜中均有表達,但表達量差異不具統計學意義(P > 0.05)。靶向預測分析結果顯示,鈣調蛋白(calmodulin)、鈣調磷酸酶-A亞基(calcineurin A subunit)、N19以及Shematrin-3為Pm-miR-210的候選靶基因。
馬氏珠母貝;Pm-miR-210;靶向預測;貝殼形成
microRNA(miRNA)是一類內生性、在序列結構上具有很大保守性、長度為21~25 nt的非編碼單鏈小分子RNA[1-2]。miRNA 主要是通過其種子序列(5' 端第2—8 nt)與靶mRNA 的3'UTR進行完全或不完全互補配對,從而抑制mRNA的翻譯或直接使其降解或沉默,行使轉錄后調控功能[3-6]。如果miRNA與其靶mRNA完全互補,作用方式和功能與siRNA非常類似,則最后切割靶mRNA[7]。目前,發現的數以千計的 miRNA通過對靶基因的調控[8-10],參與了包括生長發育、細胞分化、細胞調亡等幾乎所有己知細胞生理過程[11-14],并與生物體的感染、腫瘤、代謝性疾病等多種病理過程有關[15]。
miRNA-210是miRNA中的一種類型,作為一種靶基因的沉默子,主要與癌癥及癌細胞轉移的研究有關[16-17]。有研究報道,miRNA-210位于人體染色體的11p15.5位點上[18],通過參與機體的上皮間質轉化過程(epithelial-mesenchymal transition,EMT)[19],使得癌細胞和干細胞一樣具有了浸染和轉移的能力。但最新研究發現,miRNA-210還可能與骨髓間充質干細胞分化有關[20]。
馬氏珠母貝(Pinctada martensii),又稱合浦珠母貝(Pinctada fucata),屬軟體動物門珠母貝屬,是我國南方沿海地區人工培育海水珍珠的最常用貝類[21]。在前期的實驗中本課題組發現馬氏珠母貝中也存在miR-210[22],鑒于miR-210在調節成骨細胞礦化以及調控人骨髓間充質干細胞分化中發揮作用,推測miR-210在貝殼形成過程中行使了相應的功能。為了進一步探究miR-210在馬氏珠母貝礦化中的作用機制,本研究利用miR-RACE、生物信息學及實時熒光定量PCR技術對Pm-miR-210進行序列驗證和分析以及組織定量檢測,并進行Pm-miR-210靶位點基因的篩選,為貝類礦化機制研究提供理論依據。
1 材料與方法
1.1 材料與試劑
實驗用馬氏珠母貝取自湛江市徐聞縣廣東岸華集團珍珠養殖基地,選取活力旺盛、生長狀況良好的貝體進行實驗。馬氏珠母貝外套膜邊緣膜和中央膜材料離體后立即放入液氮速凍,之后于-80℃冰箱保存備用。RNAiso for Small RNA購自Takara,Trizol購自Invitrogen公司,SYBR? Select Master Mix 購自Applied Biosystems公司,GeneJET PCR Purification Kit 購自Thermo公司、DNA Marker、Reverse Transcriptase M-MLV反轉錄酶、Taq 聚合酶、pMD19-T vector購自大連寶生物工程有限公司,大腸桿菌DH5α為本實驗室自備。
1.2 方法
1.2.1 Small RNA提取和模板 cDNA制備 采用RNAiso for Small RNA試劑盒及說明書提取馬氏珠母貝外套膜的small RNA,得到的small RNA需經過0.01 g/mL 瓊脂糖凝膠電泳檢測其完整性,并使用紫外分光光度計來檢測其濃度。選取質量較好的small RNA,參照TaKaRa Poly(A) Polymerase說明書進行后續的3' 端加Poly A尾和5' 端加接頭實驗,然后通過M-MLV反轉錄酶以及通用引物制備cDNA模板。整個實驗過程所用耗材均要求RNase-free。
1.2.2 miR-RACE的擴增 分析已獲得的馬氏珠母貝轉錄組數據庫中 Pm-miR-210,采用上述步驟合成的cDNA模板,通過Primer Premier 5.0軟件分別對Pm-miR-210設計3' miR-RACE、5' miR-RACE特異性引物GSP1和GSP2,結合3' miR-RACE和5' miR-RACE所對應的通用引物進行PCR擴增,引物序列如表1。采用莖環法[23],設計莖環引物。PCR擴增體系及反應程序均參照TaKaRa公司提供的說明書進行。依據不同引物的退火溫度Tm值調整。PCR擴增產物通過0.01 g/mL瓊脂糖凝膠電泳檢測后,將目的基因片段連接到pMD19-T載體上,16℃放置16 h。之后轉化到感受態細胞中,菌落PCR后挑取陽性單克隆送往上海生工測序[24]。

表1 引物序列及用途Table 1 Primer sequences and application
1.2.3 Pm-miR-210序列分析 經測序驗證 PmmiR-210的成熟體序列后,通過Clustal W2在線軟件進行序列同源性分析,然后從馬氏珠母貝基因組數據庫找到包含Pm-miR-210在內的scaffold序列,利用 M-fold 軟件在線預測其二級結構,得到Pm-miR-210前體序列及頸環結構。
1.2.4 熒光定量PCR檢測Pm-miR-210的表達
利用 Trizol法提取馬氏珠母貝外套膜邊緣膜和中央膜的總RNA,經0.01g/mL瓊脂糖凝膠電泳檢測其完整性,并使用紫外分光光度計測定A260/A280值,之后對總RNA進行篩選,使用質量較好且條帶完整無損的總 RNA進行熒光定量PCR模板的制備。在此過程中使用莖環引物對Pm-miR-210進行特異性反轉錄。把反轉錄生成的cDNA模板稀釋100倍,以U6作為內參[22],按照如下反應體系進行熒光定量 PCR:SYBR Mix 5 μL,cDNA 0.5 μL,上下游引物各0.4 μL,ddH2O 3.7 μL。反應條件為:95℃ 7 min ,95℃15 s,Tm 10 s,72℃35 s,40個循環。采用 2-△△Ct法計算Pm-miR-210在馬氏珠母貝外套膜邊緣膜和中央膜中的相對表達量,并且根據SPSS 20.0進行兩組間的差異性統計分析[25]。
1.2.5 生物信息學進行Pm-miR-210的靶位點基因預測 從NCBI數據庫中下載具有完整3' UTR序列的馬氏珠母貝礦化相關基因,利用靶位點預測軟件RNA-hybrid預測Pm-miR-210的靶位點與礦化相關基因的3'UTR作用位點,以自由能小于-20 kcal/mol 為篩選標準,獲得Pm-miR-210的靶基因。
2 結 果
2.1 Pm-miR-210的序列克隆及分析
利用miR-RACE技術擴增得到Pm-miR-210的3'端序列和5'端序列,測序結果顯示,測序結果與預測序列完全一致,說明高通量測序的準確度很高。通過Clustal W2在線軟件,將人類(Homo sapiens)、家鼠( Mus musculus)、果蠅( Drosophila pseudoobscura)和海蠕蟲(Capitella teleta)的miR-210成熟體序列同馬氏珠母貝Pm-miR-210序列進行同源性比較,結果顯示物種間miR-210在種子區域(5'端第2-8nt)完全一致,Pm-miR-210與果蠅的成熟體同源性達到82%,只有4個堿基的差異。與人類和家鼠的成熟體同源性也高達73%,雖與海蠕蟲相比較,同源性較低,但也達到了64%。說明Pm-miR-210與其他物種的miR-210序列具有較高的保守性(圖1)。并且,采用M-fold在線軟件獲得了Pm-miR-210前體序列的二級結構,發現其與典型的miRNA一樣,具有發夾結構(圖2)。
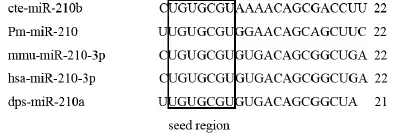
圖1 序列比對結果Fig.1 Sequence alignment results
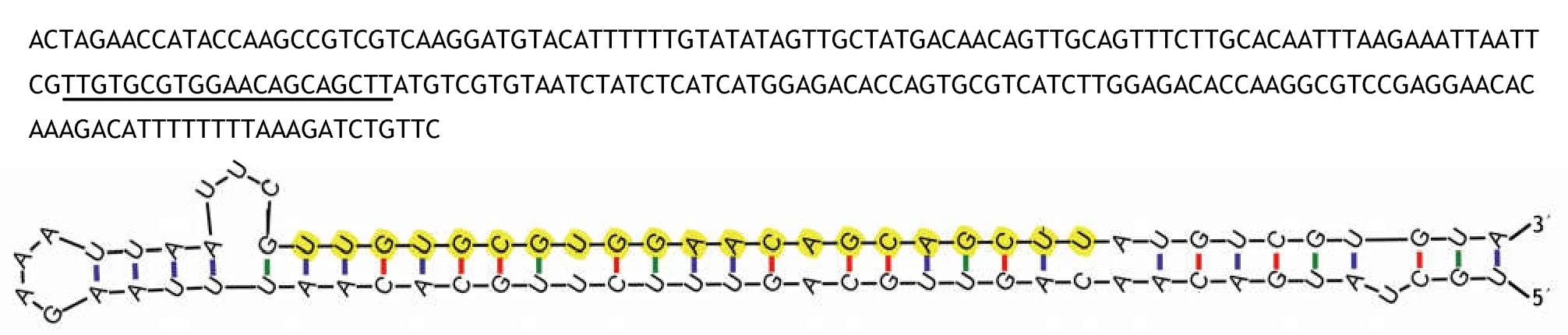
圖2 前體序列及二級結構Fig.2 Precursor sequence and two-stage structure
2.2 Pm-miR-210的外套膜表達量分析
提取馬氏珠母貝外套膜邊緣膜和中央膜的RNA后,使用莖環引物特異性反轉錄成cDNA模板進行實時熒光定量 PCR。熒光定量結果顯示,Pm-miR-210在外套膜邊緣膜和中央膜都有較高表達,CT值分別為11.9和11.1,而且在邊緣膜的含量比在中央膜的含量高出0.442631,但差異不具有統計學意義(P>0.05)。
2.3 馬氏珠母貝Pm-miR-210靶基因的篩選
將馬氏珠母貝轉錄組數據庫中礦化相關的unigene序列靶點篩選后得出Pm-miR-210對多個靶位點都具有不同程度的調控作用,結合miR-210的文獻報道及在線預測軟件 RNAhybrid顯示的最小自由能,最終確定鈣調蛋白(calmodulin)、鈣調磷酸酶-A亞基(calcineurin A subunit)、N19以及shematrin-3為Pm-miR-210的候選靶基因(圖3)。

圖3 候選靶基因Fig.3 Candidate target genes
3 討 論
本實驗以課題組前期研究中構建的馬氏珠母貝miRNA轉錄組數據庫中miR-210的序列為基礎,通過高通量測序后獲得Pm-miR-210序列,通過與其他物種的成熟體序列比對得出,Pm-miR-210成熟體的序列的保守性較高,在種子區域(miRNA 5'端第2-8個核苷酸)完全一致,說明miR-210在進化過程中比較保守。研究表明,“種子”序列[26]和靶基因的堿基互補配對對于miRNA發揮調控功能至關重要。從熒光定量結果來看,Pm-miR-210在外套膜中央膜和邊緣膜中均有表達,證實了Pm-miR-210在外套膜中的存在,初步推測它參與了棱柱層和珍珠層的生物礦化。
馬氏珠母貝的貝殼及珍珠的形成是一個十分復雜的生物礦化過程,外套膜是礦化作用中最關鍵的組織。外套膜細胞分泌的有機基質(基質蛋白)和鈣離子等前驅物經過沉淀最終形成貝類的貝殼和珍珠[27,28]。一般來說,外套膜邊緣區外表皮細胞主要分泌形成棱柱層,外套膜套膜區及中央區外表皮細胞主要分泌形成珍珠層[29]。從熒光定量結果得出,Pm-miR-210在外套膜的邊緣膜和中央膜有較高的表達量,CT值分別為11.9和11.1。因此推測miR-210可能參與了馬氏珠母貝的礦化過程,即參與了珍珠層和棱柱層形成的表達調控。
與貝類貝殼形成的礦化機制相似,生物體骨骼的形成也是一個礦化作用的過程。骨髓間充質干細胞(Bone Mesenehymal Stem Cells,BMSCs)具有強大的增殖能力和多向分化潛能,在適宜的體內或體外環境下可分化為成骨細胞[30],成骨細胞分泌的骨基質與鈣離子經過一系列的礦化作用,最終形成新骨。在這一過程中,一些miRNA的調控會影響成骨細胞的分化,進而干擾或抑制礦化生理過程。Mizuno等[31]利用實時定量PCR法得出,在間充質干細胞ST2自然增殖過程中,miR-125b表達上調,且隨著表達量的增加,細胞的增殖作用逐漸減弱,由此推斷,miR-125b可能起到抑制ST2增殖的作用。Wang等[32]發現在hFOB1.19(人SV40轉染成骨細胞)成骨分化的過程中,miR-27表達水平上升,隨后將miRNA-27或其模擬物轉染hFOB1.19,實時定量PCR結果顯示ALP和骨鈣素mRNA表達水平顯著升高,提示 miR-27對成骨分化有正調控作用。同樣,在貝類珍珠層形成中,miRNA也參與了表達調控。鄭哲[33]等研究表明,miR-2305通過靶向調控馬氏珠母貝外套膜pif177和pearlin的表達而對珍珠質的形成產生影響。
本研究中的Pm-miR-210是否參與貝類的礦化作用?許嘯等[20]研究表明,miR-210基因在成骨細胞分化過程中表達顯著性上調,且具有誘導骨髓間充質干細胞分化為成骨的作用,分化形成的成骨細胞會分泌骨基質,骨基質經過礦化作用最終形成骨骼[34]。Mizuno等[35]用基因芯片分析BMP-4誘導骨髓基質細胞ST2成骨分化過程中miRNA表達的差異性,發現miR-210表達顯著上調。另外,貝殼的形成受有機基質控制比較復雜,由大約95%的文石碳酸鈣和5%的有機基質交替疊加,層層復合而成,有機基質的含量雖少,卻直接決定了整個貝殼的生物礦化過程[36]。鑒于miR-210參與了骨骼形成的相關靶基因的調控和有機基質在生物礦化中的重要調控機制,因此,我們推測miR-210可能也是通過調控馬氏珠母貝外套膜分泌的有機基質的靶基因,來影響其礦化作用的。本研究中預測的Pm-miR-210的候選靶基因為calmodulin、calcineurin A subunit、N19和shematrin-3。其中,calmodulin和calcineurin屬于鈣離子結合蛋白,通過調控鈣的攝取、轉運和吸收等新陳代謝過程,對貝殼的形成起到關鍵作用[37-38];N19和shematrin-3屬于基質蛋白,主要參與珍珠層和棱柱層形成。Shematrin家族在貝殼礦化過程中起到框架蛋白的作用,N19參與馬氏珠母貝的礦化結晶并負調控珍珠層礦化的形成過程[39-40]。此4種候選靶基因均在貝殼的生物礦化中發揮了有機基質的調控作用,因此可以斷定Pm-miR-210參與了馬氏珠母貝貝殼形成的過程。
RNAhybrid是用來篩選靶位點基因快速有效的方法,但這種方法只是對miRNA靶基因的一個預測,要徹底研究miR-210對靶基因的調控機制,還需要進一步做基因的過表達實驗和基因功能驗證。對于篩選出來的miR-210的靶基因是如何參與馬氏珠母貝外套膜的礦化機制的,更需從激活靶蛋白的信號通路方面進行深入的研究。
[1]BUSHATI N,COHEN S M.microRNA functions [J].Annual review of cell and developmental biology,2007,23:175-205.
[2]史孟婧,趙遠鵬,鄒仲敏,等.MiR-210在缺氧中調控機制的研究進展[J].現代生物醫學進展,2013,13(16):3173-3175.
[3]EULALIO A,HUNTZINGER E,IZAURRALDE E.Getting to the root of miRNA-mediated gene silencing [J].Cell,2008,132(1):9-14.
[4]FILIPOWICZ W,BHATTACHARYYA S N,SONENBERG N.Mechanisms of post-transcriptional regulation by microRNAs:are the answers in sight[J].Nature reviews Genetics,2008,9(2):102-114.
[5]TANG G.siRNA and miRNA:an insight into RISCs[J].Trends Biochem Sci,2005,30:106-114.
[6]方福德.microRNA的研究方法與應用[M].北京:中國協和醫科大學出版社,2008.
[7]KHVOROVA A,REYNOLDS A,JAYASENA S D.Functional siRNAs and miRNAs exhibit strand bias[J].Cell,2003,115(2):209-216.
[8]FARH K,GRIMSON A,JAN C,et al.The widespread impact of mammalian MicroRNAs on mRNA repression and evolution[J].Science,2005,310:1817-1821.
[9]BARTEL D.Micro RNAs:genomics,biogenesis,mechanismand function[J].Cell,2004,116:281-297.
[10]KLOOSTERMAN W,PLASTERK R.The diverse functions of microRNAs in animal development and disease[J].Dev Cell,2006,11:441-450.
[11]SEILA A,SHARP P.Small RNAs tell big stories in Whistler[J].Nat Cell Biol,2008,10:630-633.
[12]KARP X,AMBROS V.Developmental biology.Encountering microRNAs in cell fate signaling[J].Science,2005,310:1288-1289.
[13]LEUNG A,SHARP P.microRNAs:a safe guard against turmoil[J].Cell,2007,130:581-585.
[14]GUARNIERI D,DILEONE R,MicroRNAs:a new class of gene regulators[J].Ann Med,2008,40:197-208.
[15]CROCE C,CALIN G.miRNAs,cancer,and stem cell division[J].Cell,2005,122:6-7.
[16]YU P F,FAN S F,HUANG L,et al.MIR210 as a potential molecular target to block invasion and metastasis of gastric cancer[J].Medical Hypotheses,2015,84:209-212.
[17]IVAN M,HUANG X.miR-210:fine-tuning the hypoxic response[J].Adv Exp Med Biol,2014,772:205-227.
[18]XIONG L,WANG F,HUANG X,et al.DNA demethylation regulates the expression of miR-210 in neural progenitor cells subjected to hypoxia[J].FEBS Journal,2012,279:4318-4326.
[19]TAKIKAWA T,MASAMUNE A,HAMADA S,et al.miR210 regulates the interaction between pancreatic cancer cells and stellate cells[J].Biochem Biophys Res Commun,2013,437:433-439.
[20]許嘯.miRNA-210在人骨髓間充質干細胞誘導成骨分化中的作用及意義[D].西安:第四軍醫大學,2010.
[21]田榮榮,田群莉,杜曉東,等.馬氏珠母貝SRF基因的分子特征及組織特異性表達[J].基因組學與應用生物學,2015,34(2):272-278.
[22]焦鈺,田群莉,杜曉東,等.馬氏珠母貝 LST8基因cDNA的分子特征及表達分析[J].基因組學與應用生物學,2014,33(1):10-15.
[23]石琰.miR-210及其靶基因轉錄因子E2F3與喉鱗狀細胞癌的相關研究[D].太原:山西醫科大學第一臨床醫學院,2012.
[24]潘曉艷,鄭哲,田榮榮,等.馬氏珠母貝組蛋白H3基因的克隆與表達特性分析[J].基因組學與應用生物學,2015,34(7):1421-1428.
[25]JIAO Yu,ZHENG Zhe,DU Xiaodong,et al.Identification and characterization of MicroRNAs in pearl oyster Pinctada martensii by Solexa deep sequencing [J].Marine Biotechnology,2014,16(1):54-62.
[26]顏宇琦,王梅梅,熊亞南,等.miR-210過表達載體的構建及 Northern鑒定[J].河北聯合大學學報(醫學版),2012,14(3):306-308.
[27]張學驁,王建方,吳文健,等.貝殼珍珠層生物礦化及其對仿生材料的啟示[J].無機材料學報,2006,21(2):257-265.
[28] 徐在寬.三角帆蚌不同部位外套膜的比較磯究[J].海洋湖沼通報,1991(1):71-76.
[29]石安靜,張矛,吳中文,等.三角帆蚌珍珠囊形成的研究[J].水產學報,1985,9(3):248-252.
[30]MAJUMDAR M K,THIEDE M A,MOSCA J D,et al.Phenotypic and functional comparison of cultures of marrow- derived mesenchymal stem cells(MSCs)and stromal cells[J].J Cell Physiol,1998,176:57- 66.
[31]MIZUNO Y,YAGI K,TOKUZAWA Y.miR-215b inhibits osteoblastic differentiation by down-regulation of cell proliferation[J].Biochem Biophys Res Commun,2008,368:267-272.
[32]WANG T,XU Z.miR-27 promotes osteoblast differentiation by modulating Wnt signaling[J].Biochem Biophys Res Commun,2010,402:186-189.
[33]鄭哲.馬氏珠母貝珍珠層形成microRNA的篩選和功能的研究[D].湛江:廣東海洋大學,2013.
[34] 劉曉丹,李春風,谷大海,等.成骨細胞的研究進展[J].中國畜牧獸醫,2011,38(7):53-56.
[35]MIZUNO Y,TOKUZAWA Y,NINOMIYA Y,et al.miR-210 promotes osteoblastic differentiation through inhibition of AcvR1b[J].FEBS Lett,2009,13:2263-2268.
[36]DAUPHIN Y.Comparison of the soluble matrices of the calcitic prismatic layer of Pinna nobilis(Mollusca,Bivalvia,Pteriomorpha).Comparative Biochemistry And Physiology a-Molecular And Integrative Physiology.2002,132(3):577-590.
[37]張君誠,孟玉環,等.植物Ca2+-caM信號系統及其調控研究進展[J].重慶師范大學學報,2005,22(4):49-52.
[38]符民桂,唐朝樞.鈣調神經磷酸酶的研究進展[J].生物化學與生物物理進展,2000,27(2):157-159.
[39]YANO M,NAGAI K,MORIMOTO K,et al.Shematrin:A family of glycine-rich structural proteins in the shell of the pearl oyster Pinctada fucata [J].Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology,2006,144:254-262.
[40]YANO M,NAGAI K,MORIMOTO K,et al.A novel nacre protein N19 in the pearl oyster Pinctada fucata[J].Biochemical and Biophysical Research Communications,2007,362(1):158-163.
(責任編輯:陳莊)
The Structure and Function Prediction of miR-210 in the Mineralization Mechanism of Mantle in Pinctada martensii
PAN Xiao-yan,ZHENG Zhe,TIAN Rong-rong,JIAO Yu,DU Xiao-dong
(Fisheries College,Guangdong Ocean University,Zhanjiang 524025,China)
To explore the structure and function of miR-210 and the regulation mechanism in Pinctada martensii,miR-RACE technology was used to verify and analyze the mature sequence of the Pm-miR-210.Bioinformatics analysis was performed to obtain the precursor of the sequence of Pm-miR-210 and predict target genes.The miR-RACE result was consistent with the result of high-throughput sequencing.We got the precursor sequence of Pm-miR-210 with 80 bp neck ring structure by the transcriptome data analysis and the mature sequence of the Pm-miR-210.Multiple sequence alignment revealed that the seeds of miR-210 mature sequence area among species are highly conservative.Pm-miR-210 is highly and similarly expressed in the mantle central and mantle edge(P >0.05).Targeted forecast analysis indicated that calmodulin,calcineurin A subunit,N19 and Shematrin-3 are candidate target genes of Pm-miR-210.
Pinctada martensii;Pm-miR-210;Target prediction;mineralization
S917.4
A
1673-9159(2016)03-0009-06
10.3969/j.issn.1673-9159.2016.03.002
2016-03-21
國家自然科學基金(31272635);國家自然科學基金(41206141);國家自然科學基金(31372526)
潘曉艷(1990—),女,碩士研究生,主要從事珍珠貝分子生物學研究。
焦鈺(1981—),女,博士,主要從海洋生物分子生物學研究。Tel.:0759-2383346,Email:jiaoyu1981@hotmail.com

