蠟狀芽孢桿菌幾丁質脫乙酰基酶克隆和表達
孫玉英,張繼泉,王淑軍,嚴翠
(1.淮海工學院海洋學院,江蘇連云港222005;2.中國科學院海洋研究所實驗海洋生物學重點實驗室,山東青島266071)
蠟狀芽孢桿菌幾丁質脫乙酰基酶克隆和表達
孫玉英1,2,張繼泉2,王淑軍1,嚴翠1
(1.淮海工學院海洋學院,江蘇連云港222005;2.中國科學院海洋研究所實驗海洋生物學重點實驗室,山東青島266071)
從產幾丁質脫乙酰基酶的蠟狀芽孢桿菌(Bacillus cereus M1)中獲得了幾丁質脫乙酰基酶BcCDA基因序列,并在大腸桿菌中實現了其高效分泌表達以及一步親和法純化重組幾丁質脫乙酰基酶BcCDA,經SDS-PAGE檢測該酶為單一條帶,其分子量約為23 kD,所得重組BcCDA的具有脫乙酰基酶活性,其比活力為11 608.31 U/mg。該研究為BcCDA作用機制的進一步研究和BcCDA的利用鑒定基礎。
幾丁質脫乙酰基酶;蠟狀芽孢桿菌;分泌表達;純化
幾丁質(chitin)是由N-乙酰氨基葡萄糖以β-1,4糖苷鍵連接而成的多聚體,是天然多糖中十分重要的一類,其數量僅次于世界上資源最豐富的纖維素,但其利用價值比纖維素更高,是自然界中數量第二多的的含氮類有機化合物,被歐美學術界稱為世界第六生命要素。很多類似于蝦蟹等節肢動物的外殼里都存在幾丁質;像昆蟲、動物體表及消化道里也含有豐富的幾丁質。
據估計,自然界中的幾丁質的生物合成量超過1 000億t,而人類每年所需幾丁質的量也高達3.7× 104t,主要是海洋動物產品加工的廢棄物。另外,海洋中產生的大量廢棄物中,幾丁質將近一億t,而且海洋生物圈中產生的幾丁質占全球幾丁質總量的絕大部分,可以說是一種取之不盡的生物寶庫。
而殼聚糖(脫乙酰甲殼素,chitosan)是幾丁質的一類最重要的衍生物,也是自然存在的唯一一種堿性低聚多糖,可以應用于食品、化妝品、紡織、醫療、印染、保健、廢水處理等許多領域,其利用價值很高,是市場推廣和產品研發的有力組成部分。
目前工業上生產殼聚糖普便采用強堿熱化學法,該方法生產殼聚糖存在較多的缺陷和不足,主要包括以下幾方面:生產過程耗能大、消耗大量強堿,嚴重污染環境,不能獲得質量均一、具有特定脫乙酰度的殼聚糖等。幾丁質脫乙酰酶(chitin deacetylase,CDA)能夠催化幾丁質生成殼聚糖,具有工藝條件溫和、環境友好、產品質量高等優點。采用幾丁質脫乙酰酶生產殼聚糖則可以解決強堿熱化學法制備殼寡糖的諸多缺陷和不足,生產出質量均一、性能穩定的殼聚糖,滿足生物醫學等領域對殼聚糖的高質量要求。目前發現的能夠產生CDA的微生物多數為真菌,從真菌中分離得到的CDA主要為糖蛋白,分子量大多為75 kD~150 kD[1-8]。Srinivasan等[9]從城市污水中分離到1株產堿桿菌屬的細菌Alcaligenes sp.ATCC55938,其可以分泌CDA胞外酶,在大規模發酵體系中比真菌容易培養,生長較快而且不需提取純化CDA,只需與甲殼素共同培養即可生產出殼聚糖,這是關于細菌產生CDA的首次報道。此后產CDA的細菌陸續被報道,有芽孢桿菌[10]、副溶血弧菌[11]、炭疽菌[12]、菜豆炭疽菌[13-16]和紅平紅球菌[17]等。細菌相對于真菌,發酵時間短,基因結構簡單容易被改造。
但是目前酶法生產殼聚糖仍然存在缺少產高活性CDA的菌株的問題。因為產CDA菌株的篩選和誘變工作還進行的較少,較少能夠篩選出高產率的CDA產生菌,而且篩選出的菌株也存在產酶性質不穩定,專一性高,酶活力低等問題。利用基因工程菌異源表達CDA是提高產酶能力的重要手段。作者在前期研究中,從自然界篩選到一株幾丁質脫乙酰基酶高產菌株,蠟狀芽孢桿菌(Bacillus cereus M1),通過生物信息學分析并設計一對蠟狀芽孢桿菌幾丁質脫乙酰基酶高度保守的引物對,獲得了一種幾丁質脫乙酰基酶(BcCDA)基因序列,并在大腸桿菌中實現了其高效分泌表達以及一步法純化重組幾丁質脫乙酰基酶BcCDA。所得重組BcCDA的具幾丁質脫乙酰基酶活性,該研究為BcCDA作用機制的進一步研究和BcCDA的利用鑒定基礎。
1 材料與方法
1.1材料
蠟狀芽孢桿菌(Bacillus cereus M1)由淮海工學院海洋學院海洋微生物與酶工程課題組從自然界中分離并保存。
1.2方法
1.2.1基因克隆
以NCBI中提供的13個B.cereus全基因組序列中注釋為幾丁質脫乙酰基酶(chitin deacetylase,CDA)的序列信息設計引物(BcCDA-dF/BcCDA-dR)用于擴增B.cereus M1的幾丁質脫乙酰基酶基因(BcCDA)。引物具體為:
BcCDA-dF:ATGTTTTTCTTTTTTATTACGAG
BcCDA-dR:TTATTGAACATCTTTACTTTTCG
提取B.cereus M1的DNA,作為模板。
PCR的反應體系為:模板1 μL;5 μL 10×PCR buffer,2 μL dNTP(10 mmol/L),2 μL引物BcCDA-dF(10 μmol/L),2 μL引物BcCDA-dR(10 μmol/L),1 μL Taq DNA聚合酶(5 U/μL),加ddH2O補至體積50 μL。PCR反應條件:95℃預變性5 min;95℃變性30 s,55℃退火30s,72℃延伸2min,30個循環;72℃延伸10min。利用1%的瓊脂糖凝膠電泳對PCR擴增產物進行電泳分析,將PCR產物切膠,利用瓊脂糖凝膠純化試劑盒回收,回收PCR產物連接pMD 19-T載體(按照使用說明書進行),16℃連接30 min。將連接產物(pMD 19-BcCDA)轉化大腸桿菌DH5α感受態細胞,涂布含有100μg/mL氨芐青霉素的LB固體平板,37℃培養過夜,以M13-F/M13-R為引物對,利用PCR方法對LB平板上的單克隆進行檢測,將PCR檢測陽性克隆搖菌后進行DNA測序。
1.2.2重組表達載體pCT7-CHISP6H-BcCDA的構建
結合分泌表達載體pCT7-CHISP6H[18]和In-fusionRHD Clontech kit使用說明書,設計引物擴增其含有Polysacc_deac_1功能域部分的序列(對應氨基酸序列為51-254),引物序列如下:
pCT7-CHISP6H-BcCDAF:CATCACCATCACCA C AAAGGCGACACCTCTAAAAAACA(下劃線部分序列為與線性化載體pCT7-CHISP6H匹配的15 bp序列)。
pCT7-CHISP6H-BcCDAR:AAGCTTCGAATTCAC TTATTGAACATCTTTACTTTTCG(下劃線部分序列為與線性化載體pCT7-CHISP6H匹配的15 bp序列)。
以 pCT7-CHISP6H-BcCDAF/pCT7-CHISP6HBcCDAR為引物對,測序質粒pMD19-BcCDA為模板(100 ng/μL),進行PCR擴增,PCR反應體系和擴增程序如下:
PCR反應體系為50 μL,包括:

PCR擴增程序:94℃預變性4 min;94℃變性30 s,52℃退火30 s,72℃延伸1 min,共30個循環;最后72℃延伸10 min。PCR產物電泳后進行產物回收,利用NanoDrop測定回收片段濃度。
利用PmaC I對質粒pCT7-CHISP6H進行酶切,利用0.8%瓊脂糖凝膠電泳分析酶切產物,回收酶切片段,測定回收片段濃度。
根據In-fusionRHD Clontech kit使用說明,將回收的PCR產物和PmaC I酶切的pCT7-CHISP6H線性化質粒進行反應,具體反應體系與反應條件為:回收的PCR產物1 μL,回收的單酶切載體2 μL,5×In-fusionRHD 2 μL,無菌水5 μL,混勻后,在50℃反應15 min后立即置于冰上,然后將連接產物轉化大腸桿菌DH5α感受態細胞,涂布含有100 μg/mL氨芐青霉素的LB固體平板,37℃培養過夜,以T7-F/T7-ter為引物對,利用PCR方法對LB平板上的單克隆進行檢測,將PCR檢測陽性克隆經搖菌后進行DNA測序,由此篩選到構建的重組載體,命名為pCT7-CHISP6H-BcCDA。
1.2.3幾丁質脫乙酰酶活性測定及蛋白質濃度測定
測定酶活的原理為:以對硝基乙酰苯胺為底物,CDA可脫除對硝基乙酰苯胺的乙酰基生成對硝基苯胺,對硝基苯胺在400 nm處有最大吸收峰。通過測定產物溶液在400 nm處的吸光值,來反映BcCDA的酶活值。具體測定方法為:10 mL具塞試管中加入1 mL 200 mg/L的對硝基乙酰苯胺溶液、3 mL 0.05 mol/L的磷酸鹽緩沖液,50℃水浴3 min,加入1 mL酶液,50℃水浴15 min,沸水浴終止酶促反應,加水定容至10 mL,若出現渾濁現象,在3 000 r/min條件下離心10 min,在400 nm處測定吸光值。空白對照組添加1 mL滅活酶液,其余同上。在該反應條件下每小時產生1 μg對硝基苯胺所需要的酶量定義為1個酶活力單位(U/mL)[19]。
采用Bradford法測定蛋白質的含量[20]。
1.2.4重組BcCDA的誘導表達
抽提pCT7-CHISP6H-BcCDA質粒,轉化BL21(DE3)中,37℃條件下,在含有100 μg/mL氨芐青霉素的5 mL LB液體培養基中搖菌,待菌體濃度達到OD600為0.6左右,添加終濃度為1 mmol/L的IPTG進行誘導表達6 h。4℃,10 000 g離心10 min后,取上清液檢測重組幾BcCDA的活性。
1.2.5重組BcCDA的分離純化及SDS-PAGE分析
重組工程菌經活性檢測具有幾丁質脫乙酰基酶活性后,進行放大試驗,放大至400 mL體積。發酵液離心后,上清液直接利用His標簽蛋白純化預裝柱His-TrapTMFF Crude(5 mL)進行親和層析法純化。具體步驟如下:
HisTrapTMFF Crude(5 mL)柱經10倍柱體積的超純水沖洗后,用5倍柱體積的結合緩沖液(50 mmol/L pH7.2的磷酸氫二鈉-磷酸二氫鈉緩沖液,500 mmol/L NaCl,10 mmol/L咪唑)平衡;發酵液中分別添加5 mol/L咪唑母液、5 mol/L NaCl母液以及0.5 mol/L pH7.2的磷酸氫二鈉-磷酸二氫鈉緩沖液至發酵液中各組分濃度與結合緩沖液中一致,混勻,4℃,10000g離心10min,取上清液上HisTrapTMFFCrude柱,流速控制在3mL/min;上樣結束后,利用結合緩沖液沖洗10倍柱體積;利用洗脫液(50 mmol/L pH7.2的磷酸氫二鈉-磷酸二氫鈉緩沖液,500 mmol/L NaCl,250 mmol/L咪唑)洗脫。經檢測酶活力,親和層析純化的含有重組BcCDA目的蛋白的組分,利用HiPrep 26/10脫鹽柱在AKTA avant 25上進行脫鹽,并利用12%的SDS-PAGE分析純化重組蛋白的純度。
2 結果與分析
2.1蠟狀芽孢桿菌幾丁質脫乙酰基酶基因的克隆
利用引物(BcCDA-dF/BcCDA-dR)成功從B. cereus M1中克隆到一個幾丁質脫乙酰基酶基因(Bc-CDA)全長序列,其序列及編碼氨基酸序列如圖1所示。序列全長765 bp,編碼254個氨基酸,其理論等電點pI為9.46,預測分子量為28 552.69 Da。

圖1 蠟狀芽孢桿菌BcCDA基因及其推導的氨基酸序列Fig.1 BcCDA nucleotide sequence and deduced amino acid
利用Signalp4.1信號肽預測軟件[21]分析,結果顯示該序列沒有信號肽。經在線分析(http://smart.emblheidelberg.de/)蠟狀芽孢桿菌BcCDA基因推導的氨基酸序列特征,發現位置為51-176的氨基酸序列注釋為Polysacc_deac_1功能域(見圖2),其對應的閾值為7e-27。該功能域廣泛存在于各類多糖脫乙酰基酶中,其中幾丁寡糖脫乙酰基酶(chitooligosaccharide deacetylase)中也存在該功能域。

圖2 蠟狀芽孢桿菌BcCDA氨基酸結構特征Fig.2 Structure charaterization of BcCDA deduced amino acid
2.2BcCDA重組表達載體的構建及誘導表達分析
利用本課題組構建的大腸桿菌分泌表達載體pCT7-CHISP6H[18]成功構建了含有BcCDA基因Polysacc_ deac_1功能域的分泌型表達載體pCT7-CHISP6HBcCDA。pCT7-CHISP6H-BcCDA轉化BL21(DE3),經IPTG誘導表達后,經檢測發酵上清液含有幾丁質脫乙酰基酶的活性,其最大比活力為235.43 U/mg。
2.3重組BcCDA的分離純化及SDS-PAGE純度檢測
重組工程菌經活性檢測具有幾丁質脫乙酰基酶活性后,進行放大試驗,放大至400 mL體積。發酵液離心后,上清液直接利用His標簽蛋白純化預裝柱His-TrapTMFF Crude(5 mL)進行親和層析法純化,經線性洗脫后,獲得了洗脫目的蛋白如圖3所示。經檢測酶活力知圖中峰1是BcCDA組分。
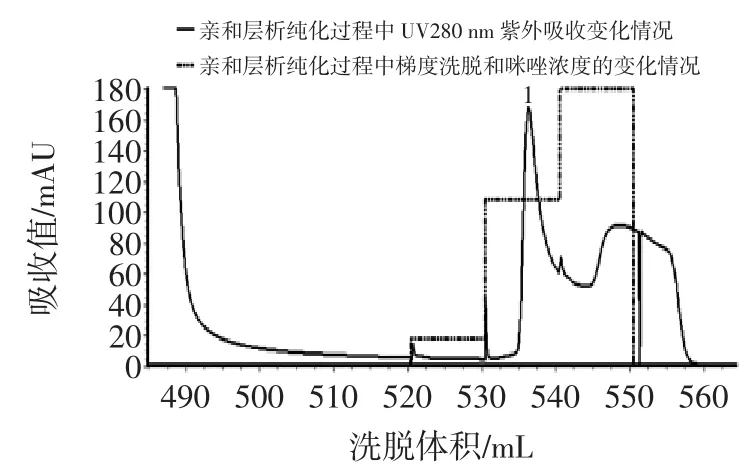
圖3 重組蛋白BcCDA經親和層析純化色譜圖Fig.3 Affinity chromatogram of recombinant BcCDA during purification process
親和層析純化的含有重組BcCDA組分,利用HiPrep 26/10脫鹽柱在AKTA avant 25上進行脫鹽。對分離純化的樣品進行12%的SDS-PAGE分析,結果顯示通過一步親和層析分離可以得到電泳純的重組幾丁質脫乙酰基酶,其分子量大小約為23 kD(見圖4),與預測的重組蛋白理論分子量為23 529.5 Da相一致。純化后溶液中幾丁質脫乙酰基酶的比活力為11608.31U/mg,經親和層析后,其純度提高了49.31倍。

圖4 分離純化的重組幾丁質脫乙酰基酶SDS-PAGE分析結果Fig.4 SDS-PAGE analysis of purified recombinant BcCDA
3 結論與討論
本試驗從產幾丁質脫乙酰基酶的蠟狀芽孢桿菌(Bacillus cereus M1)中獲得了幾丁質脫乙酰基酶Bc-CDA基因序列,并對其序列特征進行了細致分析。利用課題組前期構建的高效分泌表達載體,在大腸桿菌中實現了其高效分泌表達,獲得了具有脫乙酰基活性的重組BcCDA。利用親和層析的方法,獲得了電泳純的重組BcCDA。
目前已經有許多利用基因工程手段進行CDA體外重組表達的報道。Tokuyasu等[22]將Colletotrichum lindemuthianum的CDA基因成功克隆到大腸桿菌,并借助于Streptomyces lividans的甲殼素酶信號肽序列,使CDA分泌到胞外,并且酶活力與野生型真菌的CDA相比沒有明顯差別。Kang等[23]將來自Colletotrichum lindemuthianum的CDA基因,根據畢赤酵母密碼子的偏好性,運用酵母的信號肽構建組成型重組表達載體HMB905A,實現了CDA在畢赤酵母(Pichia pastoris GS115)中的分泌表達,表達水平為77.27 U/mg蛋白,表達的CDA部分被糖基化。Wang等[24]克隆了Aspergillus nidulans的CDA基因,利用pET28a表達載體構建了重組表達質粒,轉化Escherichia coli BL21,經誘導后的酶活力達到4.17 U/mg,但是該CDA以包涵體的形式表達,不利于大規模工業化生產。本研究重組表達的BcCDA在初始發酵液中的最高酶活力為235.43 U/mg,經親和層析后純度提高了49.31倍,比活力達到了11 608.31 U/mg,酶的比活力比較目前微生物源CDA經基因工程菌表達后的酶比活力是相當高的。
本研究所得分泌性重組BcCDA的具幾丁質脫乙酰基酶活性,該研究為BcCDA作用機制的進一步研究和BcCDA的利用鑒定基礎。
[1]Win NN,Stevens WF.Shrimp chitin as substrate for fungal chitin deacetylase[J].Applied Biochemistry and Biotechnology,2001,57(3):334-341
[2]Kim YJ,Zhao Y,Oh KT,et al.Enzymatic deacetylation of chitin by extracellular chitin deacetylase from a newly screened Mortierella sp.DY-52[J].Journal of Microbiology and Biotechnology,2008,18(4):759-766
[3] Gauthier C,Clerisse F,Dommes J,et al.Characterization and cloning of chitin deacetylases from Rhizopus circinans[J].Protein Expression and Purification,2008,59(1):127-137
[4]Jeraj N,Kunic B,Lenasi H,et a1.Purification and molecular characterization of chitin deacetylase from Rhizopus nigrieans[J].Enzyme and Microbial Technology,2006,39(6):1294-1299
[5]Nahar P,Ghormade V,Deshpande MV.The extracellular constitutive production of chitin deacetylase in Metarhizium anisopliae:Possible edge to entomopathogenic fungi in the biological control of insect pests[J].Journal of Invertebrate Pathology,2004,85(2):80-88
[6]Zhang H,Yang S,Fang J,et al.Optimization of the fermentation conditions of Rhizopus japonicus M193 for the production of chitin deacetylase and chitosan[J].Carbohydrate Polymers,2014,101(101):57-67
[7]Cai J,Yang J,Du Y,et a1.Purification and characterization of chitin deacetylase from Scopulariopsis brevicaulis[J].Carbohydrate Polymers,2006,65(2):211-217
[8]Amorim RVS,Ledingham WM,Fukushima K,et a1.Screening of chitin deacetylase from Mucoralean strains(Zygomycetes)and its relationship to cell growth rate[J].Journal of Industrial Microbiology& Biotechnology,2005,32(1):19-23
[9]Srinivasan VR.Biotransformation of chitin to chitosan US,5739015(C12P19/04)[P].1998-04-14
[10]Dai DH,Li W,Hu WL,et al.Effect of medium composition on the synthesis of chitinase and chitin deacetylase from thermophilic Paenibacillus sp.Hul[J].Procedia Environmental Sciences,2011,8(1):620-628
[11]Kadokura K,Rokutani A,Yamamoto M,et al.Purification and characterization of Vibrio parahaemolyticus extracellular chitinase and chitin oligosaccharide deacetylase involved in the production of heterodisaccharide from chitin[J].Applied Biochemistry and Biotechnology,2007,75(2):357-365
[12]Pacheco N,Trombotto S,David L,et al.Activity of chitin deacetylase from Colletotrichum gloeosporioides on chitinous substrates[J]. Carbohydrate Polymers,2013,96(1):227-232
[13]Suresh PV,Sachindra NM,Bhaskar N.Solid state fermentation production of chitin deacetylase by Colletotrichum lindemuthianum ATCC 56676 using different substrates[J].Journal of Food Science and Technology,2011,48(3):349-356
[14]Tokuyasu K,Ohnishi·Kameyama M,Hayashi K.Purification and characterization of extracellular chitin deacetylase from Colletotrichum lindemuthianum[J].Bioscience Biotechnology and Biochemistry,1996,60(10):1598-1603
[15]Tsigos I,Bouriotis V.Purification and characterization of chitin deacetylase from Colletotrichum lindemuthianum[J].Journal of Biological Chemistry,1995,270(44):26286-26291
[16]Shrestha B,Blondeau K,Stevens WF,et al.Expression of chitin deacetylase from Colletotrichum lindemuthianum in Pichia pastoris:Purification and characterization[J].Protein Expression and Purification,2004,38(2):196-204
[17]Sun Y,Zhang J,Wu S,et al.Statistical optimization for production of chitin deacetylase from Rhodococcus erythropolis HG05[J].Carbohydrate Polymers,2014,102(1):649-652
[18]Sun Y,Zhang J,Wang S.Heterologous expression and efficient secretion of chitosanase from Microbacterium sp.in Escherichia coli[J].Indian Journal of Microbiology,2015,55(2):194-199
[19]劉麗萍,趙祥穎,劉建軍,等.一種簡易、高效產幾丁質脫乙酰酶菌種的篩選方法[J].食品與發酵工業,2008,34(1):65-68
[20]Bradford MM.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of proteindye binding[J].Analytical Biochemistry,1976,7(72):248-254
[21]Petersen TN,Brunak S,Von Heijne G,et al.SignalP 4.0:discriminating signal peptides from transmembrane regions[J].Nature Methods,2011,8(10):785-786
[22]Tokuyasu K,Kaneko S,Hayashi K,et al.Prodution of a recombinant chitin deacetylase in the culture medium of Escherichia coli cells[J].FEBS letters,1999,458(1):23-26
[23]Kang L,Chen X,Zhai C,et al.Synthesis and high expression of chitin deacetylase from Colletotrichum lindemuthianum in Pichia pastoris GS115[J].Journal of Microbiology and Biotechnology,2012,22(9):1202-1207
[24]Wang Y,Song JZ,Yang Q,et al.Cloning of a heat-stable chitin deacetylase gene from Aspergillus nidulans and its functional expression in Escherichia coli[J].Applied Biochemistry and Biotechnology,2010,162(3):843-854
Cloning and Recombinant Expression of Chitin Deacetylase from Bacillus cereus
SUN Yu-ying1,2,ZHANG Ji-quan2,WANG Shu-jun1,YAN Cui1
(1.Marine College,Huaihai Institute of Technology,Lianyungang 222005,Jiangsu,China;2.Key Laboratory of Experimental Marine Biology,Institute of Oceanology,Chinese Academy of Sciences,Qingdao 266071,Shandong,China)
A gene encoding chitin deacetylase from Bacillus cereus M1(BcCDA)was obtained.BcCDA was recombinant expressed in Escherichia coli at the secretion form and purified by affinity chromotagraphy.The purity of the purified BcCDA was tested by SDS-PAGE,which molecular weight was approximately 23 kD.The recombinant BcCDA had deacetylase activity,which specific activity was 11 608.31 U/mg.The research would provide a basis for studying the action mechnism and harness of BcCDA.
chitindeacetylase;Bacilluscereus;secretoryexpression;purify
10.3969/j.issn.1005-6521.2016.21.035
碳谷-江蘇海資院海洋先進材料聯合研究中心項目(TG-201402);連云港市第五期“521工程”科研項目
孫玉英(1974—),女(漢),副教授,博士,研究方向:微生物酶技術。
2015-10-04

