奶牛SREBP1蛋白在乳腺上皮細胞的表達定位及對SCD1基因啟動子的轉錄調控
韓立強,王月影,王林楓,朱河水,鐘 凱,褚貝貝,楊國宇
(河南農業大學牧醫工程學院/農業部動物生化與營養重點實驗室,鄭州 450002)
奶牛SREBP1蛋白在乳腺上皮細胞的表達定位及對SCD1基因啟動子的轉錄調控
韓立強,王月影,王林楓,朱河水,鐘 凱,褚貝貝,楊國宇
(河南農業大學牧醫工程學院/農業部動物生化與營養重點實驗室,鄭州 450002)
【目的】固醇調節元件結合蛋白 1( SREBP1)作為核轉錄因子,對于細胞脂肪合成酶基因的表達發揮著重要的調控作用。論文旨在奶牛乳腺上皮細胞中研究SREBP1對于SCD1基因啟動子的轉錄調控作用,為進一步明確SREBP1對于靶基因的轉錄調控機制提供理論基礎。【方法】以荷斯坦奶牛乳腺組織的cDNA為模板,采用分段克隆的方法獲得SREBP1基因的編碼序列,通過重組酶與pcDNA3.1載體進行重組環化構建pcDNA3.1-SREBP1表達載體,將構建的載體測序驗證后提取質粒,轉染奶牛乳腺上皮細胞。以EIF3K基因為內參基因,采用熒光定量PCR檢測SREBP1基因mRNA的表達差異;采用免疫熒光的方法對SREBP1進行標記,以DAPI復染細胞核,激光共聚焦觀察SREBP1蛋白的亞細胞定位;轉染含有不同調控元件的SCD基因啟動子,同時轉染1.0 μg pcDNA3.1-SREBP1作為處理,熒光素酶報告基因系統分析啟動子活性;分別轉染0.25、0.5和1μg的pcDNA3.1-SREBP1載體,分析pGL3-SCD 2 和pGL3-SCD3啟動子活性與SREBP1之間的量效關系。【結果】分段克隆得到的PCR產物分別為1 170、1 116、363和900 bp的片段,經過與pcDNA3.1載體重組后獲得pcDNA3.1-SREBP1表達載體,經酶切和測序驗證,發現除1個無義突變外,與標準序列完全相同,整個序列長度達到3 510bp;將pcDNA3.1-SREBP1載體轉染乳腺上皮細胞后,Real-time PCR檢測發現與轉染空載體的對照組相比,SREBP1基因的mRNA表達倍數增強130.4倍(P<0.001);激光共聚焦觀察發現,DAPI染色的細胞核呈藍色,免疫熒光標記的SREBP1呈綠色,二者融合后呈現青色,共定位在乳腺上皮細胞核中;啟動子活性檢測發現,與pGL3-SCD1、pGL3SCD 2相比,SREBP1處理能夠極顯著增加pGL3-SCD3、pGL3-CD4啟動子的活性(P<0.001),分別比對照組提高了1.0倍和0.7倍,進一步分析發現,在用0.25—1 μg的pcDNA3.1-SREBP1處理后,與pGL3-SCD2的啟動子活性持續下降相比,pGL3-SCD3的啟動子活性從59.81上升到108.43(P<0.001),二者存在劑量效應關系,結合SCD2和SCD 3啟動子上主要的結構差異SRE元件(5′-AGCAGATTGCG-3′),推測此序列可能是SREBP1調控SCD基因啟動子轉錄的結合序列。【結論】克隆構建奶牛SREBP1基因表達載體,亞細胞定位SREBP1蛋白主要在乳腺上皮細胞核中,SREBP1可以與SRE調控元件結合促進SCD1基因啟動子的轉錄。
奶牛;固醇調節元件結合蛋白 1;乳腺上皮細胞;亞細胞定位;轉錄調控
Abstract:【Objective】As a member of nuclear transcription factor, sterol regulatory element binding protein1(SREBP1)plays a significant role in the expression of the lipogenic gene . The objective of this study is to investigate the effect of SREBP1 on the regulation of transcription of SCD1 gene in the mammary epithelial cells, which will provide a fundamental basis for the transcription regulation mechanism of SREBP1 to target gene. 【Method】 The coding sequence (CDS) of SREBP1 gene was cloned using the method of subsection cloning with the cDNA of Holstein mammary tissues as the template. The expression vector pcDNA3.1-SREBP1 was recombined and constructed by recombining vector pcDNA3.1. Plasmids were extracted from the constructed vector after being verified by sequencing, and transfected to mammary epithelial cells. With gene EIF3K as the reference gene, the expressional difference of mRNA in gene SREBP1 was detected with Real-time PCR. SREBP1 was marked with the immunofluorescence method. The cell nucleus was restained with DAPI, and the subcellular localization of protein SREBP1 was observed with laser con-focal microscopy. The SCD1 gene promoters with different regulatory elements and 1.0 μg pcDNA3.1-SREBP1 were restained as treatments, and the promoter activity was analyzed systematically with luciferase reporter gene. Vectors pcDNA3.1-SREBP1 of 0.25 μg, 0.5 μg and 1 μg were transfected respectively. The dose-effect relationship between the promoter activity of pGL3-SCD2 , pGL3-SCD3 and protein SREBP1 was analyzed. 【Result】 The PCR products cloned in subsection were fragments 1170bp, 1116bp, 363bp and 900 bp respectively. The expression vector pcDNA3.1-SREBP1 was obtained by reconstructing the fragments and vector pcDNA3.1. Confirmed by digestion and sequence, the expression vector, except one nonsense mutation, was exactly the same with the standard sequence. The whole sequence length reached 3510 bp. After vector pcDNA3.1-SREBP1 was transfected into mammary epithelial cells, compared with the control group of the empty transfected vector, the expression mRNA of gene SREBP1 increased by 130.4 times (P<0.001)by the real-time PCR detection. As shown in the observation of the laser con-focal microscopy, the cell nucleus dyed with DAPI was blue, and SREBP1 with immunofluorescent labeling was green, both of which were cyan after being merged and were localized in the mammary epithelial cell nucleus. The results of the detection of the promoter activity showed that, compared with those of pGL3-SCD1 and pGL3SCD 2, the treatment with SREBP1 increased the promoter activity of pGL3-SCD3 and pGL3-CD4 significantly (P<0.001), and their promoter activity increased by 1.0 and 0.7 times. The further study showed that, after treatment with 0.25-1μg pcDNA3.1-SREBP1, compared with the continuous decrease of the promoter activity of pGL3-SCD2, the promoter activity of pGL3-SCD3 increased from 59.81 to 108.43(P<0.001). There is a dose-effect relationship between them. Combined with the element SRE(5′-AGCAGATTGCG-3′), the architectural difference between promoter SCD2 and promoter SCD 3, it could be speculated that the sequence was the incorporating sequence in which SREBP1 regulated the transcription of gene promoter SCD1.【Conclusion】 It was verified that the gene expression vector SREBP1 was constructed by cloning, the subcellular localization of protein SREBP1 was in the mammary epithelial cell nucleus, and SREBP1 promoted the regulation of the transcription of SCD1 promoter together with regulatory element SRE.
0 引言
【研究意義】乳腺做為牛、羊等哺乳動物合成分泌乳汁的主要器官,其基因表達調控機制一直是畜牧工作者進行科學研究的重要內容。BIONAZ等采集奶牛乳腺組織分析基因表達譜[1],發現固醇調節元件結合蛋白1(Sterol-regulatory element binding protein1,SREBP1)作為一種轉錄因子,是乳腺中脂肪合成代謝基因表達調控網絡的中心,其他研究發現 一些營養因素可以影響 SREBP1蛋白的活性和脂肪合成酶基因的表達[2-3]。因此研究SREBP1及脂肪合成酶基因在乳腺上皮細胞中的表達調控機制,可以為闡明營養元素影響乳腺脂肪合成的機制提供理論基礎。【前人研究進展】早期在生產中發現對奶牛飼喂油脂會造成奶牛的乳脂率下降,形成生產上低脂乳癥(milk fat depression, MFD)的現象[4],后來發現奶牛在發生MFD時,不僅乳脂肪含量與組成發生變化,而且乳腺組織的脂肪合成關鍵酶的基因表達也受到抑制[4-6],進一步研究發現在降低脂肪合成酶基因表達的同時也能夠影響SREBP1基因mRNA的表達[3,7]。SREBP1作為重要的核轉錄因子,主要調控細胞內的膽固醇濃度和脂類的穩態[8-10]。對于SREBP1調控靶基因的促轉錄功能,通過采用CHIP-chip和CHIP-seq的研究,在人和小鼠全基因組上已發現上百個可能受SREBP1調控的靶基因,其中主要包括硬脂酰輔酶 A去飽和酶(stearoyl-coenzyme A desaturase,SCD)等脂肪合成酶基因[11-13]。SCD是催化飽和脂肪酸脫氫形成不飽和脂肪酸的關鍵酶,在奶牛中主要存在SCD1和SCD5兩種亞型[14],并且SCD1在奶牛乳腺基因表達譜中所占的比例遠遠超過其他脂肪合成酶基因[1]。已有多個研究表明SCD1和SREBP1基因的核酸多態性均與乳脂肪性狀相關[15-16]。【本研究切入點】目前在細胞上研究SREBP1與靶基因的轉錄調控機制多是采用間接研究模式,在培養細胞中加入誘導SREBP1的刺激因素(如胰島素/葡萄糖),通過引起SREBP1及其靶基因mRNA的表達變化來進行推測[17],而這些基因表達變化是SREBP1直接調控還是由于代謝通路的其他因子引起的,目前還不十分清楚[18]。還有研究采用表達SREBP1成熟核蛋白來研究其轉錄調控作用[19],但對于SREBP1全長蛋白在乳腺上皮細胞的轉錄調控機制還缺乏了解,特別是關于奶牛SREBP1在乳腺上皮細胞中調控 SCD1基因轉錄的研究還未見報道。【擬解決的關鍵問題】因此本研究結合前期試驗建立的含不同調控元件奶牛 SCD1基因啟動子[20],通過克隆表達奶牛SREBP1蛋白,在奶牛乳腺上皮細胞中研究SREBP1對于SCD1基因啟動子的轉錄調控作用,有助于闡明SREBP1對于靶基因的轉錄調控機制。
1 材料與方法
相關試驗在2014年1月至2015年4月在河南農業大學農業部動物生化與營養重點實驗室完成。
1.1 材料與儀器
奶牛乳腺組織于2014年1月采自鄭州市屠宰場,乳腺上皮細胞為實驗室保存,4個不同長度 pGL3-SCD1/SCD2/SCD3/SCD4熒光素酶報告基因表達載體為實驗室構建,所用試劑AxyPrep DNA Gel Extraction Kit 試劑盒購自Axygen 公司,ClonExpress Multis kit試劑盒購自Vazyme公司,Lipofectamine2000購自Invitrogen公司,Star XL擴增酶購自Takala公司,雙熒光素酶檢測試劑盒購自Promega公司,DMEM培養基購自Hyclone公司,無血清培養基Opti-mem購自 Gibco公司,標簽蛋白抗體 c-myc Antibody(9E10)購自Santa Cruz公司,熒光二抗Alexa Fluor? 488 Donkey Anti-Mouse IgG (H+L)購自Invitrogen公司,細胞核染料DAPI、抗熒光封片淬滅液購自碧云天,細胞培養板購自 Corning公司,引物合成及測序由上海生工完成。二氧化碳培養箱和 Fluroskan Ascent FL熒光和化學發光檢測儀購自 Thermo公司,激光共聚焦顯微鏡LSM 5 PASCAL購自Carl Zeiss公司。
1.2 方法
1.2.1 奶牛SREBP1基因的克隆 采用TRIZOL提取乳腺組織的RNA,反轉錄成cDNA,根據NCBI公布的奶牛SREBP1基因序列(NM_001113302.1),設計引物時在 SREBP1序列 ATG后加入 c-myc序列(GAACAAAAACTCATCTCAGAAGAGGATCTG)作為標簽蛋白,采用分段克隆 PCR方法對奶牛SREBP1基因進行克隆,具體引物序列見表 1。將SREBP1總共分為四段分別進行擴增,同時PCR擴增pcDNA3.1+序列作為載體序列,分別對目的基因1、2、3、4各片段純化、電泳后定量。
1.2.2 pcDNA3.1-SREBP1載體的構建及鑒定 采用重組酶 Exnase Multis對各片段與載體進行重組環化,重組反應體系:5×CE Multis Buffer 4 μL,vector 108 ng,4個回收片段,Exnase Multis 2 μL,同時做無酶的空白對照,在37℃水浴30 min,置于冰水浴中反應5 min。取反應產物10 μL,加入到感受態細胞中進行轉化,獲得克隆后挑取單菌落擴增,提取質粒進行分別采用HindⅢ、XhoI雙酶切和HindⅢ、XbaI雙酶切鑒定,鑒定正確的質粒送測序公司進行測序。

表 1 引物序列Table 1 The sequences of primer
1.2.3 SREBP1基因mRNA在乳腺上皮細胞中的超表達將培養好的奶牛乳腺上皮細胞經胰酶消化后,按比例分散培養在12孔細胞培養板中,放入37℃、5% CO2培養箱培養,到細胞達到80%的融合度時,棄去培養基,采用轉染試劑(Lipofectamine 2000)轉染pcDNA3.1-SREBP1質粒6 h后,更換新鮮培養基,同時以pcDNA3.1空載體作為對照組,轉染48 h后提取細胞RNA,反轉錄成cDNA,采用熒光定量PCR引物(F-CCAGCTGACAGCTCCATTGA,R-TGCGCGC CACAAGGA)檢測SREBP1基因mRNA的表達,同時檢測奶牛EIF3K基因(F-CCAGGCCCACCAAGA AGAA,R- TTATACCTTCCAGGAGGTCCATGT)作為內參基因[21],計算SREBP1基因的mRNA表達差異倍數。
1.2.4 SREBP1蛋白在乳腺上皮細胞的定位 在24孔板中加入細胞爬片,接種乳腺上皮細胞,細胞培養過夜后,轉染pcDNA3.1-SREBP1質粒,培養24 h后用預冷的PBS洗3遍,4%多聚甲醛固定10 min,PBS洗3遍,透化劑(0.2%Triton100)作用15 min,封閉液(90%PBS+10%胎牛血清)封閉1 h;棄封閉液,用一抗c-MYC Antibody(9E10)(1∶500)4 ℃過夜孵育;PBS洗3遍,加入熒光標記二抗Alexa Fluor? 488 Donkey Anti-Mouse IgG(H+L)(1∶1 000)室溫避光孵育1 h;然后采用DAPI染色液(500μg·mL-1)對于細胞核染色8 min,PBS洗3遍,滴加抗熒光封片淬滅液,封片,晾干放 4℃,在激光共聚焦顯微鏡下觀察SREBP1蛋白亞細胞定位。
1.2.5 SREBP1蛋白對含不同調控元件SCD1啟動子的轉錄調控作用 培養乳腺上皮細胞,以 pGL3-Basic熒光素酶空載體作為對照,轉染實驗室前期構建的4個含有不同調控元件的奶牛 SCD1基因啟動子載體[20],分別是pGL3-SCD1(212bp,序列中有TATA-box元件)、pGL3-SCD2(380bp,有CAAT-box元件)、pGL3-SCD3(416 bp,有SRE元件)和pGL3-SCD4(760 bp,有SRE、Sp1元件)(圖1),同時分別轉染1.0 μg pcDNA3.1-SREBP1質粒作為處理因素,作用24 h后收集細胞加入裂解液,采用熒光和化學發光檢測儀進行熒光素酶活性檢測,采用螢火蟲熒光素酶活性/海腎熒光素酶活性計算啟動子活性,結合調控元件分析SREBP1處理對于啟動子活性的影響。
1.2.6 SREBP1調控SCD1基因啟動子轉錄的劑量關系 培養乳腺上皮細胞,接種12孔細胞培養板后,轉染1.0 μg pGL3-SCD2、pGL3-SCD3啟動子載體,同時分別轉染0.25、0.5、1.0μg的pcDNA3.1-SREBP1質粒作為處理因素,作用24 h后收集細胞,加入裂解液,進行熒光素酶活性檢測并計算啟動子相對活性,分析不同劑量SREBP1的處理對于啟動子活性的影響。
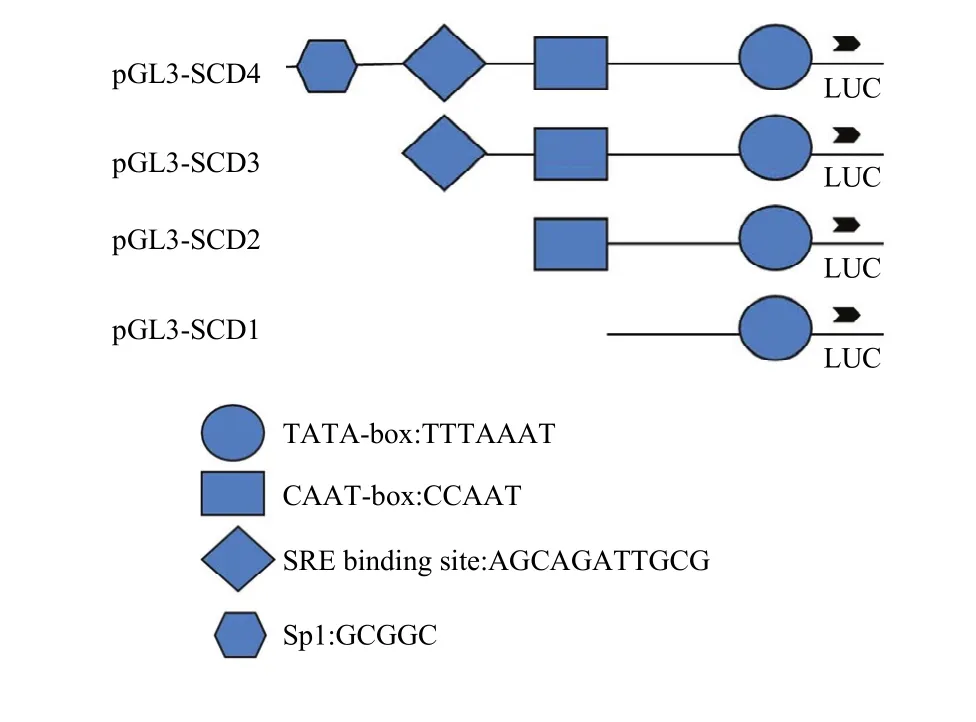
圖1 含不同調控元件的奶牛SCD1啟動子結構Fig. 1 The structure of bovine SCD1 promoter with different regulatory elements
1.2.7 數據統計 試驗中每個處理中重復4次,試驗數據采用SPSS10.0軟件進行統計學分析,**P<0.01,***P<0.001。
2 結果
2.1 SREBP1基因的克隆
對 SREBP1基因采用分段克隆的方法,分別獲得大小約5 400 bp 的pcDNA3.1載體片段和4個不同長度的目的基因片段(圖2),將各個片段切膠回收后進行基因重組獲得連接產物,將連接產物轉化感受態細胞進行陽性克隆篩選,提取陽性細菌質粒后進行酶切鑒定。分別采用HindIII、XhoI雙酶切,獲得3個片段,片段長度分別為5 356、3 057和390 bp,采用HindIII、Xbal雙酶切,獲得2個片段,分別為5 350和3 453 bp(圖3),最后經測序驗證后發現,克隆的序列與 NCBI公布的標準序列相比,除在1 509位有一個無義突變外,其他序列均與標準序列相一致,加上標簽蛋白myc序列,整個表達序列長度達到3 510 bp。
2.2 SREBP1在乳腺上皮細胞中的超表達
將pcDNA3.1-SREBP1質粒轉染乳腺上皮細胞后48h收集細胞,采用熒光定量PCR檢測SREBP1基因的表達豐度,結果發現,與轉染空載體的對照相比,SREBP1基因的mRNA表達顯著升高(圖4),比對照組增加了 130.4倍(P<0.001),表明載體轉染后在奶牛乳腺上皮細胞中能夠高效表達SREBP1基因的mRNA。
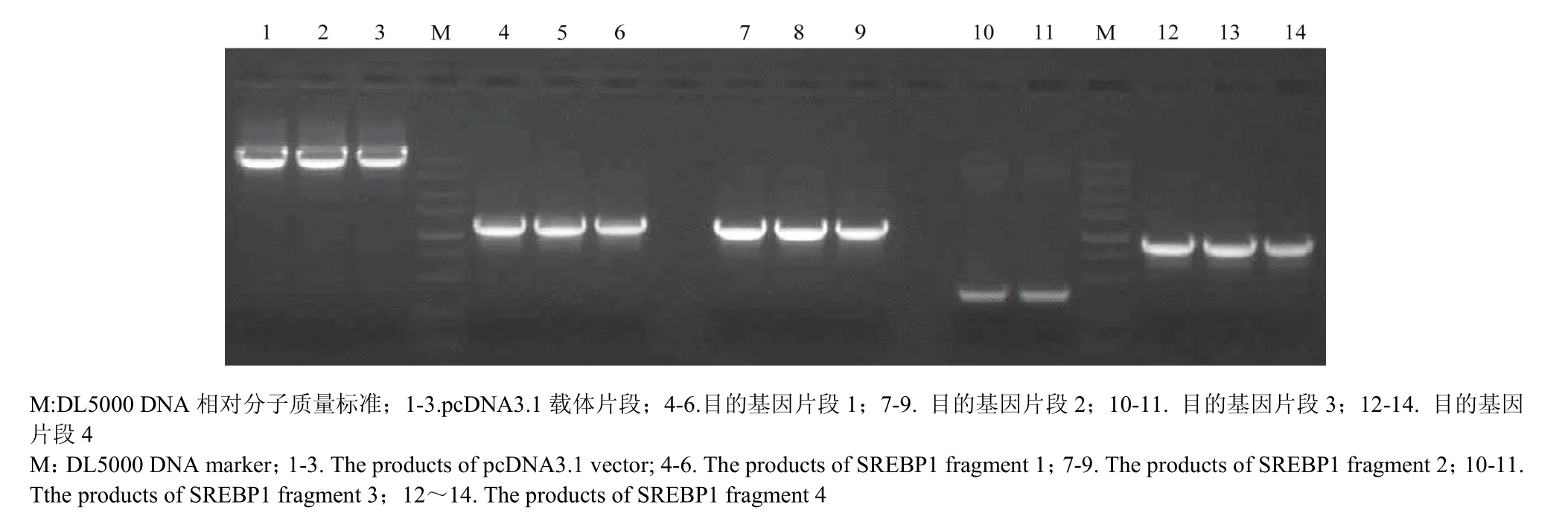
圖2 奶牛SREBP1基因的克隆Fig. 2 The clone of bovine SREBP1
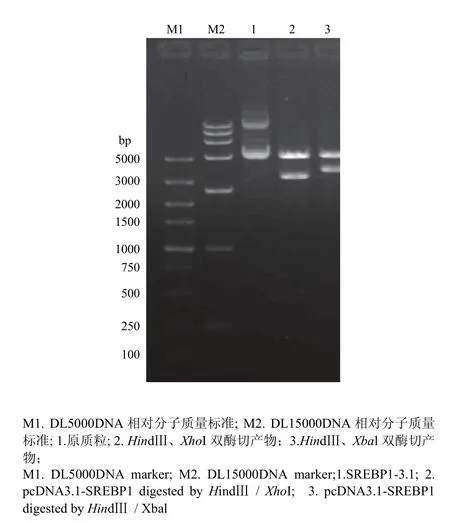
圖3 pcDNA3.1-SREBP1質粒酶切鑒定圖Fig. 3 The restriction enzyme digestion of pcDNA3.1-SREBP1
2.3 SREBP1蛋白在乳腺上皮細胞中的定位
將 pcDNA3.1-SREBP1載體轉染乳腺上皮細胞后,采用激光共聚焦顯微鏡觀察發現(圖 5),進行免疫熒光標記的SREBP1發出綠色熒光,細胞核經 DAPI染色后發出藍色熒光,共定位發現綠色和藍色融合形成一種青色熒光定位在細胞核位置,表明SREBP1作為轉錄因子能夠轉運到乳腺上皮細胞核中進行表達。
2.4 SREBP1蛋白對于不同SCD1啟動子的調控作用
將含不同調控元件的 SCD1基因啟動子 pGL3-SCD1、SCD2、SCD3和SCD4轉染細胞后,同時轉染SREBP1質粒進行處理,檢測啟動子活性發現(圖6),對照組basic啟動子活性為2.32,在SREBP1刺激后啟動子活性降低到1.29(P<0.01),與此類似,SCD1和SCD2啟動子進行SREBP1處理后,熒光值均顯著降低(P<0.01),而對 SCD3、SCD4啟動子進行SREBP1處理后,啟動子活性均有顯著增加。其中SCD3啟動子活性由59.92增加到124.37,增加了1.0倍(P<0.001),SCD4進行SREBP1處理后,啟動子活性由 83.55增加到 145.48,增加了 0.7倍(P< 0.001),表明SREBP1蛋白表達后,顯著增加了SCD3、SCD4啟動子的活性,其中對于SCD3啟動子的活性促進最為明顯。
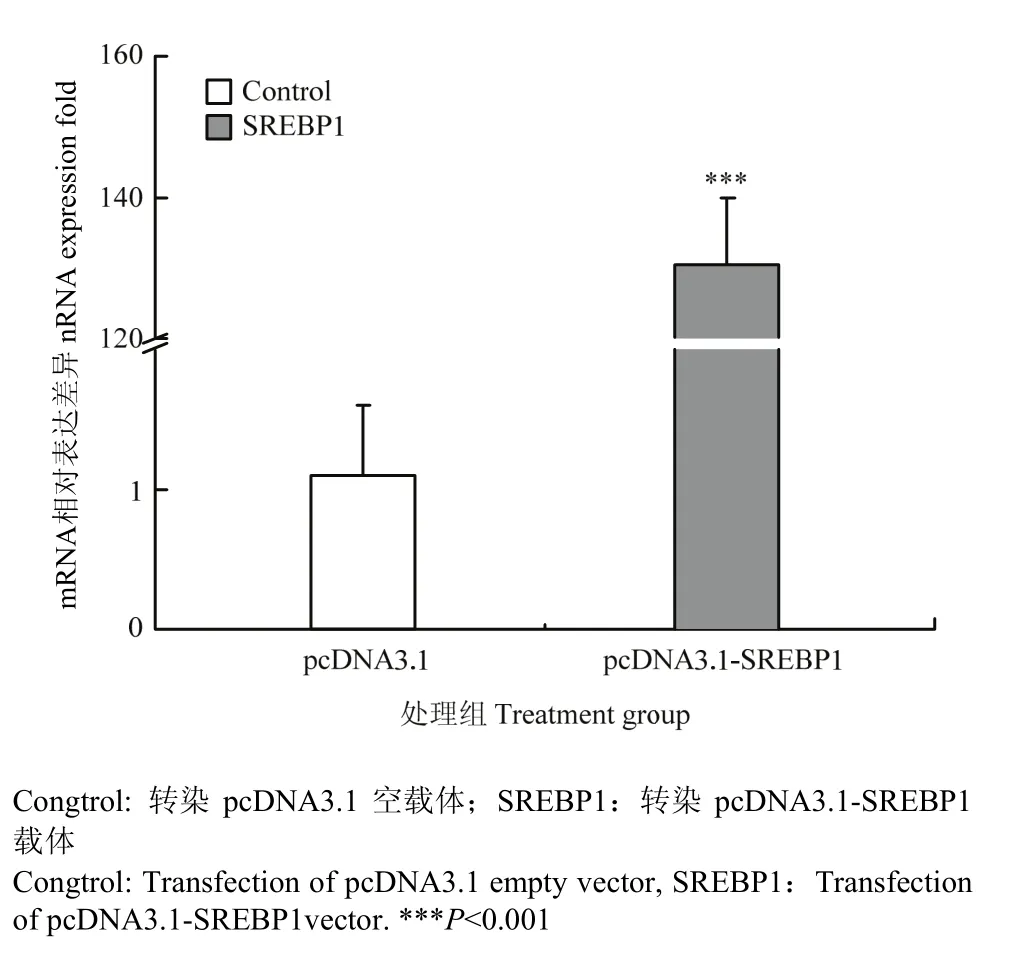
圖4 SREBP1基因mRNA在乳腺上皮細胞中的表達豐度Fig. 4 The mRNA expression of SREBP1 in mammary epithelial cell (n=4)
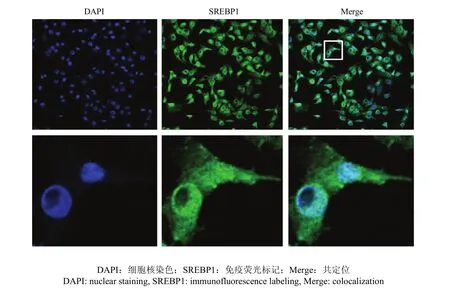
圖5 SREBP1蛋白在乳腺上皮細胞的表達定位Fig. 5 The localization of SREBP1 in mammary epithelial cell

圖6 SREBP1對不同SCD1啟動子活性的調控Fig. 6 The regulation of different SCD1 promoter activities by SREBP1
2.5 SREBP1調控SCD基因啟動子轉錄的量效關系
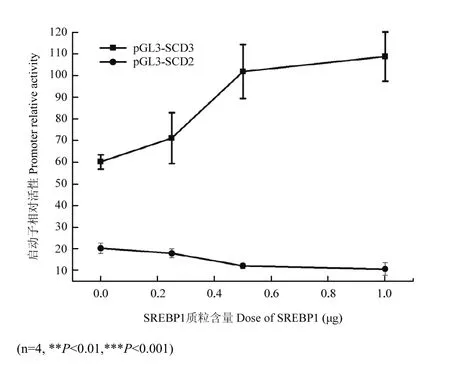
圖7 不同含量SREBP1對pGL3-SCD2、3啟動子活性的調控Fig. 7 The regulation of pGL3-SCD2 and SCD3 promoter activities by different doses of SREBP1
在轉染0.25、0.5、1.0 μg的 SREBP1質粒后,pGL3-SCD2啟動子的活性呈現持續下降的趨勢(圖7),從19.90降低到10.31,而pGL3- SCD3啟動子的活性隨著質粒含量的增加則呈現上升趨勢,其活性值從59.81分別增加到70.76、101.48和108.43(P<0.001),表明SREBP1能夠劑量依賴性的增強pGL3- SCD3啟動子的活性。
3 討論
固醇調節元件結合蛋白(SREBP)屬于“堿性螺旋-環-螺旋亮氨酸拉鏈”轉錄因子家族,其中SREBP1蛋白結構域一般分為3部分,包括480個氨基酸的N-端,80個氨基酸的跨膜結構域和590個氨基酸的C-端[22-23]。本研究中荷斯坦奶牛SREBP1基因的編碼區全長3 440 bp,加上標簽蛋白序列后整個基因長度在3 510 bp左右。由于SREBP1基因序列中含有復雜的二級結構,為提高克隆的準確性和效率,本研究采用分段克隆、基因重組的方法,得到了完整的SREBP1基因編碼區序列,表明此分段克隆重組的方法對于長片段的基因克隆具有較好的效果。經過細胞轉染和熒光定量PCR檢測發現,SREBP1基因的mRNA有顯著表達(圖4),表明載體構建成功。
SREBP1作為在內質網合成的蛋白質,首先合成無活性的前體(大約1 150個AA),然后通過與SCAP的相互作用后進入到高爾基體進行水解活化,暴露出NH2-末端活性結構域形成入核蛋白(大約500個AA),進入到細胞核內與靶基因啟動子結合調控轉錄[24]。因此本研究采用激光共聚焦對表達的SREBP1蛋白進行亞細胞定位后,發現SREBP1能夠在細胞核中表達(圖5),這說明構建的 SREBP1質粒在細胞內表達全長蛋白后能夠水解活化形成入核蛋白,轉運到細胞核內發揮功能。
對于在乳腺上皮細胞中 SREBP1與靶基因的研究發現,SREBP1通過激活PⅢ啟動子來調節乙酰輔酶A羧化酶基因的轉錄[25]。MA等在乳腺上皮細胞中通過構建SREBP1的siRNA抑制載體,能夠降低SCD、ACC和FAS等多種脂肪酸合成酶基因mRNA的表達[26]。本研究采用不同調控元件的 SCD1啟動子,通過在乳腺上皮細胞表達SREBP1后發現pGL3-SCD1-2啟動子的活性顯著降低,而 pGL3-SCD3-4啟動子的活性顯著增加(圖6)。研究表明SREBPs 自身是非常低效的轉錄因子, 只有接近結合位點時才能刺激靶基因的表達[27],而 pGL3-SCD3、4比pGL3-SCD1、2在結構上主要多出了一個SRE調控元件(圖1),表明SRE調控元件可能對于SREBP1調控 SCD1基因轉錄具有重要作用。ZULKIFLI在HEK細胞中采用表達人 SREBP1-c成熟核蛋白來調控羊SCD基因啟動子的轉錄,發現去除羊SCD基因啟動子的SRE元件顯著降低了啟動子活性,其結果與本研究一致[19]。為了進一步驗證SRE調控元件的作用,試驗又采用不同濃度的 SREBP1刺激 pGL3-SCD2、pGL3-SCD3啟動子,結果發現 pGL3-SCD3啟動子的活性增加與 SREBP1的濃度呈現劑量依賴性,結合pGL3-SCD3與pGL3-SCD2啟動子結構上相比只有36bp的差異(其中含有SRE調控元件),說明SREBP1可以通過與SRE元件結合促進SCD1基因轉錄。
啟動子上與轉錄因子結合的位點具有特定模式,稱為模體,一般長度在5—20 bp范圍內。轉錄因子在細胞內可以同時調控多個基因,而在不同基因上的結合位點并不完全相同。研究認為SREBP1結合的經典模體為(5′-TCACNCCAC-3′)/E-和(5′-CANNTG-3′)兩種[13,18]。本研究中 SRE調控元件含有的模體為5′-AGCAGATTGCG-3′(圖1),與經典的SREBP1模體序列并不相符。TABOR 等在HepG2細胞上研究小鼠SREBP1a與鼠SCD基因啟動子的關系,發現了與本研究一致的SRE新模體(5′-AGCAGATTG CG-3′)對于啟動子活性至關重要,而經典模體反而對啟動子活性沒有顯著影響[28],ZULKIFLI比對人、豬、鼠、羊的SCD1基因啟動子序列后發現此模體在不同物種間是高度保守的[19]。這些結果說明SREBP1可能通過與SCD啟動子上(5′-AGCAGAT TGCG-3′)模體的結合,發揮了轉錄因子的促轉錄作用。SEO等在小鼠組織中曾經發現了 1 個新的SREBP1模體,并認為是經典模體的一個功能變體[12],因此本研究中發現的 SREBP1模體序列可能是SCD1基因所特有的一個功能變體,這還需要進一步的研究證實。
4 結論
本研究通過克隆構建奶牛固醇調節元件結合蛋白1真核表達載體,發現固醇調節元件結合蛋白1能夠定位在乳腺上皮細胞核中,并且固醇調節元件結合蛋白1可以與SRE調控元件結合促進SCD1基因啟動子的轉錄。
[1] BIONAZ M, LOOR J J. Gene Networks driving bovine milk fatsynthesis during the lactation cycle. BMC Genomics, 2008, 9: 366.
[2] BERNARD L, LEROUX C, CHILLIARD Y. Expression and nutritional regulation of lipogenic genes in the ruminant lactating mammary gland. Advances in Experimental Medicine and Biology, 2008, 606(1): 67-108.
[3] HARVATINE K J, BAUMAN D E. SREBP1 and thyroid hormone responsive spot 14 (S14) are involved in the regulation of bovine mammary lipid synthesis during diet-induced milk fat depression and treatment with CLA. Journal of Nutrition, 2006, 136(10): 2468-2474.
[4] BAUMAN D E, GRINARI J M. Regulation and nutritional manipulation of milk fat:low-fat milk syndrome. Advanced Experimental Medicine and Biology, 2000, 480:209-216.
[5] PETERSON D G, MATITASHVILI E A, BAUMAN D E. Diet-induced milk fat depression in dairy cows results in increased trans-10, cis-12 CLA in milk fat and coordinate suppression of mRNA abundance for mammary enzymes involved in milk fat synthesis. Journal of Nutrition, 2003, 133(10): 3098-3102.
[6] AHNADI C E, BESWICK N, DELBECCHI L, KENNELLY J J, LACASSE P. Addition of fish oil to diets for dairy cows. II. Effects on milk fat and gene expression of mammary lipogenic enzymes. Journal of Dairy Research, 2002, 69(4): 521-531.
[7] HAN LQ, PANG K, LI H J, ZHU S B, WANG L F. Conjugated linoleic acid-induced milk fat reduction associated with depressed expression of lipogenic genes in lactating Holstein mammary glands. Genetics and Molecular Research, 2012, 11(4):4754-4764.
[8] DAEMEN S, KUTMON M, EVELO C T. A pathway approach to investigate the function and regulationof SREBPs. Genes and Nutrition, 2013, 8(3):289-300.
[9] WEBER L W, BOLL M, STAMPFL A. Maintaining cholesterol homeostasis: sterol regulatory element-binding proteins. World journal of gastroenterology, 2004, 10(21): 3081-3087.
[10] JEON T I, OSBORNE T F. SREBPs: metabolic integrators in physiology and metabolism. Trends in Endocrinology and Metabolism, 2012, 23(2):65-72.
[11] ROME S, LECOMTE V, MEUGNIER E, RIEUSSET J, DEBARD C,EUTHINE V, VIDAL H, LEFAI E. Microarray analyses of SREBP-1a and SREBP-1c target genes identify new regulatory pathways in muscle. Physiological Genomics, 2008, 34(3): 327-337.
[12] SEO Y K, CHONG H K, INFANTE A M, IM S S, XIE X H, OSBORNE T F. Genome-wide analysis of SREBP-1 binding in mouse liver chromatin reveals a preference for promoter proximal binding to a new motif. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(33): 13765-13769.
[13] REED B D, CHAROS A E, SZEKELY A M, WEISSMAN S M, SNYDER M. Genome-wide occupancy of SREBP1 and its partners NFY and SP1 reveals novel functional roles and combinatorial regulation of distinct classes of genes. PLoS Genetics, 2008, 4(7): e1000133.
[14] 張蕊, 張宜輝, 邵丹, 王來娣, 龔道清. 硬脂酰輔酶 A去飽和酶基因的功能與調控. 生命科學, 2013, 25(4):378-382.
ZHANG R, ZHANG Y H, SHAO D, WANG L D, GONG D Q. The function and regulation of stearoyl-CoA desaturase gene. Chinese Bulletin of Life Sciences, 2013, 25(4):378-382. (in Chinese)
[15] CECCHINATO A, RIBECA C, MAURMAYR A, PENASA M, DE MARCHI M, MACCIOTTA N P, MELE M, SECCHIARI P, PAGNACCO G, BITTANTE G. Short communication: Effects of β-lactoglobulin, stearoyl-coenzyme A desaturase 1, and sterol regulatory element binding protein gene allelic variants on milk production, composition, acidity, and coagulation properties of Brown Swiss cows. Journal of Dairy Science, 2012, 95(1): 450-454.
[16] RINCON G, ISLASTREJO A, CASTILLO A , BAUMAN D E, GERMAN B J, MEDRANO J F. Polymorphisms in genes in the SREBP1 signalling pathway and SCD are associated with milk fatty acid composition in Holstein cattle. The Journal of Dairy Reasearch, 2012, 79(1):66-75.
[17] ZHANG Y, CHEN W, LI R, LI Y, GE Y B, CHEN G X. Insu-lin-regulated Srebp-1c and Pck1 mRNA expression in primary hepatocytes from zucker fatty but not lean rats is affected by feeding conditions. PLoS One, 2011, 6(6): e21342.
[18] 湯曉麗, 鄧立彬, 林加日, 張偉龍, 劉雙梅, 魏懿, 梅普明, 汪雁,梁尚. 固醇調節元件結合蛋白 1及其靶基因網絡. 遺傳, 2013, 35(5): 607-615.
TANG X L, DENG L B, LIN J R, ZHANG W L, LIU S M, WEI Y, MEI P M, WANG Y, LIANG S D. Sterol regulatory element binding protein 1 and its target gene networks. Yi Chuan, 2013, 35(5):607-615. (in Chinese)
[19] ZULKIFLI R M, PARR T, SALTER A M, BRAMELD J M. Regulation of ovine and porcine stearoyl coenzyme A desaturase gene promoters by fatty acids and sterols. Journal of Animal Science, 2010, 88(8): 2565-2575.
[20] 韓立強, 曹菁菁, 付彤, 魏占勇, 王亞賓, 楊國宇. 奶牛硬脂酰輔酶 A去飽和酶基因(SCD)啟動子的克隆及活性分析. 農業生物技術學報, 2013, 21(4): 435-440.
HAN L Q, CAO J J, FU T, WEI Z Y, WANG Y B, Yang G Y.Cloning and activity analysis of promoter of bovine stearoyl-CoA desaturase gene(SCD). Journal of Agricultural Biotechnology, 2013, 21(4): 435-440. (in Chinese)
[21] Kadegowda A K G, Bionaz M, Thering B, Piperova L S, Erdman R A. Identification of internal control genes for quantitative polymerase chain reaction in mammary tissue of lactating cows receiving lipid supplements. Journal of Dairy Science, 2009, 92 (5): 2007-2019.
[22] BROWN M S, GOLDSTEIN J L. The SREBP pathway: regulation of cholesterol metabolism by proteolysis of a membrane-bound transcription factor. Cell, 1997, 89(3): 331-340.
[23] 付常振,昝林森,王虹,成功,王洪寶,李耀坤,姜碧杰,高建斌,楊寧. 牛SREBP1基因shRNA序列的篩選及其腺病毒載體的構建與鑒定. 中國農業科學, 2013, 46(23): 5026-5036.
FU C Z, ZAN L S, WANG H, CHENG G, WANG H B, LI Y K, JIANG B J, GAO J B, YANG N. Selection of effective SREBP1 shRNA in cattle and the construction of recombinant adenovirus vector. Scientia Agricultura Sinica, 2013, 46(23): 5026-5036. (in Chinese)
[24] YANG T, ESPENSHADE P J, WRIGHT M E, YABE D, GONG Y, AEBERSOLD R, GOLDSTEIN J L, BROWN M S. Crucial step in cholesterol homeostasis: Sterols promote binding of SCAP to INSIG-1, a membrane protein that facilitates retention of SREBPs in ER. Cell, 2002, 110(4): 489-500.
[25] BARBER M C, VALLANCE A J, KENNEDY H T, TRAVERS M T. Induction of transcripts derived from promoter III of the acetyl-CoA carboxylase-alpha gene in mammary gland is associated with recruitment of SREBP-1 to a region of the proximal promoter defined by a DNase I hypersensitive site. The Biochemical Journal, 2003, 375(2): 489-501.
[26] MA L, CORL B A. Transcriptional regulation of lipid synthesis in bovine mammary epithelial cells by sterol regulatory element binding protein-1. Journal of Dairy Science, 2012, 5(7): 3743-3755.
[27] Seo Y K, Jeon T I, Chong H K, Biesinger J, Xie X H, Osborne T F. Genome-wide localization of SREBP-2 in hepatic chromatin predicts a role in autophagy. Cell Metabolism, 2011, 13(4): 367-375.
[28] Tabor D E, Kim J B, Spiegelman B M, Edwards P A. Identification of conserved cis-Elements and transcription factors required for sterol-regulated transcription of Stearoyl-CoA desaturase 1 and 2. The Journal of Biology Chemistry, 1999, 274(29): 20603-20610.
(責任編輯 林鑒非)
Expression and Localization of Bovine SREBP1 Protein and Regulation of the Transcription of SCD1 Promoter in Bovine Mammary Epithelial Cell
HAN Li-qiang, WANG Yue-ying, WANG Lin-feng, ZHU He-shui, ZHONG Kai, CHU Bei-bei, YANG Guo-yu
(College of Animal Science and Veterinary Medicine, Henan Agricultural University,Key Laboratory of Animal Biochemistry and Nutrition of Ministry of Agriculture, Zhengzhou 450002)
bovine; SREBP1; mammary epithelial cell; subcellular localization; gene regulation
ntia Agricultura Sinica
10.3864/j.issn.0578-1752.2016.24.011
2015-07-29;接受日期:2016-10-17
國家“973”項目(2011CB100802)、國家自然科學基金(31072009)、國家轉基因重大專項(2014ZX0801015B)、河南省高等學校重點科研項目(15A230020)、河南省基礎與前沿項目(162300410258)
聯系方式:韓立強,E-mail:qlhan2001@126.com。通信作者楊國宇,E-mail:haubiochem@163.com

