蛋白質中殘基遠程相互作用預測算法研究綜述
張海倉 高玉娟 鄧明華,4,5 鄭偉謀 卜東波
1(中國科學院計算技術研究所 北京 100190)(中國科學院大學 北京 100049)(北京大學定量生物學中心 北京 100871)(北京大學數學科學學院 北京 100871)(北京大學統計科學中心 北京 100871) (中國科學院理論物理研究所 北京 100190)(zhanghaicang@ict.ac.cn)
蛋白質中殘基遠程相互作用預測算法研究綜述
張海倉1,2高玉娟3鄧明華3,4,5鄭偉謀6卜東波1
1(中國科學院計算技術研究所 北京 100190)(中國科學院大學 北京 100049)(北京大學定量生物學中心 北京 100871)(北京大學數學科學學院 北京 100871)(北京大學統計科學中心 北京 100871) (中國科學院理論物理研究所 北京 100190)(zhanghaicang@ict.ac.cn)
蛋白質是由多個氨基酸殘基順序連接而成的長鏈.在天然狀態下,蛋白質并不是無規則的自由狀態,而是自發形成特定的空間結構,以執行其特定的生物學功能.驅動蛋白質形成特定空間結構的主要因素是殘基間的非共價相互作用,包括疏水作用、靜電相互作用、范德華力等.因此,對殘基之間遠程相互作用的準確預測將有助于對蛋白質空間結構的預測,進而有助于對蛋白質生物學功能的了解.在蛋白質進化過程,有相互作用殘基對之間存在一種“共進化”模式,即當一個殘基發生變異時,與其有相互作用的殘基也要發生相應的變異,以維持相互作用,進而維持整體空間結構以及生物學功能.基于上述生物學觀察,研究者開發了多個統計模型和算法以預測殘基對之間的相互作用:1)概述殘基之間遠程相互作用的兩大類基本預測算法,包括無監督學習方法和監督學習方法;2)使用蛋白質結構預測CASP比賽結果來客觀比較上述各類算法的性能,分析各個算法的特點和優勢;3)從生物學觀察和統計模型2個角度分析總結了未來的發展趨勢.
殘基遠程相互作用預測;蛋白質三級結構預測;圖模型;共進化;機器學習
蛋白質是生物體的重要組成成分,行使催化、免疫、細胞信號傳導等重要的生物學功能[1-2].
蛋白質的基本組成單元是氨基酸,常見的氨基酸有20種.蛋白質是以氨基酸為單元,脫水后由肽鍵連接而成的長鏈(氨基酸脫水之后的剩余部分被稱為殘基).因此從計算的觀點看,可以將蛋白質抽象表示成一個字符串序列,其字符集規模為20,其中每一個字符表示一種氨基酸殘基,如圖1所示:
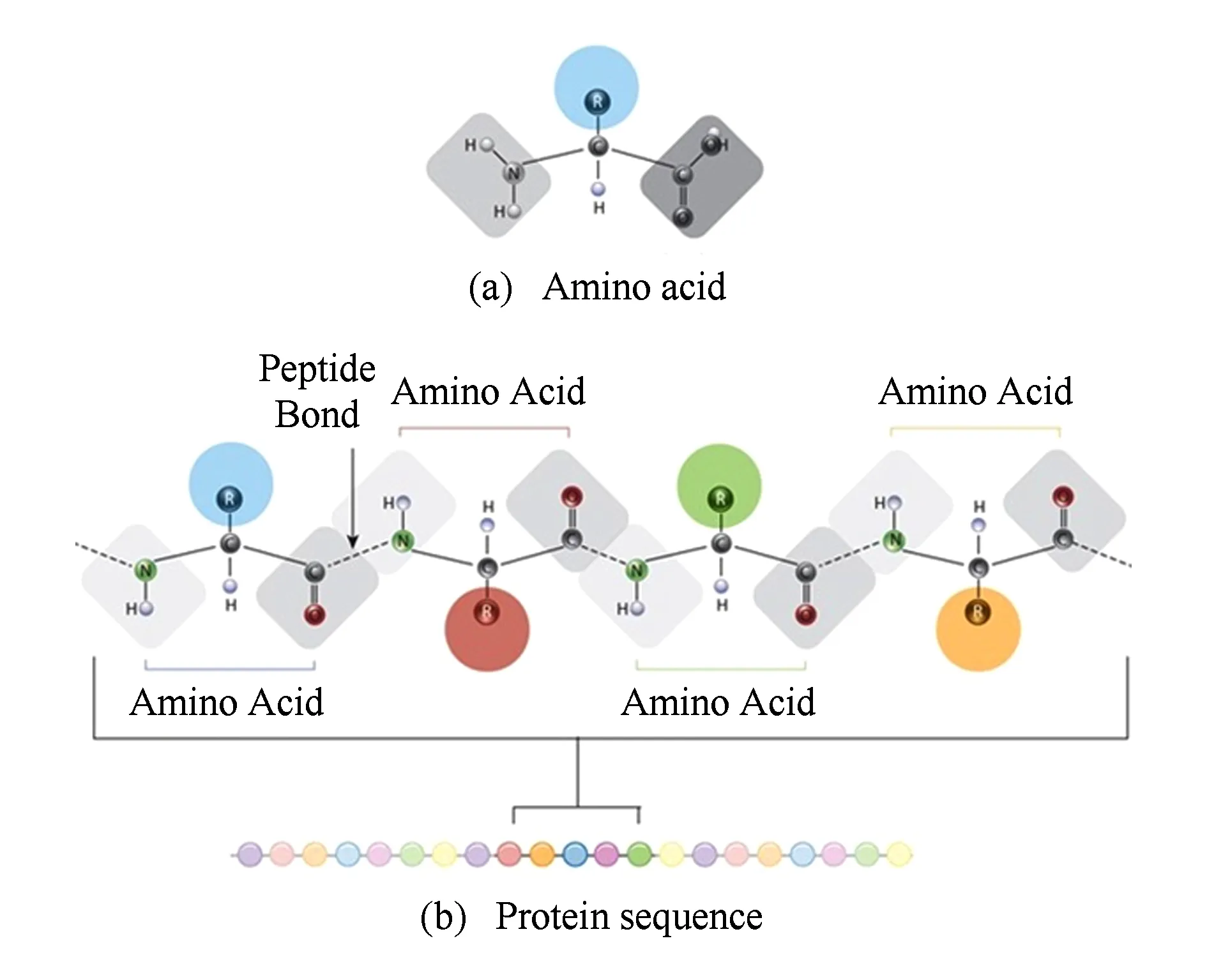
Fig. 1 Illustration of amino acids,peptide bond,and protein sequence圖1 氨基酸、肽鍵以及蛋白質序列示意圖
在天然環境下,蛋白質呈現的并不是松散的、無規則的形態,而是自發折疊成特定的空間結構,其中每個殘基(確切地說是殘基中的每個原子)都有其特定的空間坐標.蛋白質的空間結構決定了其生化功能,因此,認識蛋白質的空間結構對了解其功能至關重要[2].
目前測定蛋白質結構的主要實驗技術包括核磁共振[3]、X-ray晶體衍射[4]和冷凍電鏡[5]等.然而上述實驗測定技術常常受限于蛋白質大小、蛋白質能否結晶以及結構測定的高成本等因素,使得蛋白質結構的測定速度遠遠達不到蛋白質序列的測定速度,因而通過計算的方法預測蛋白質結構具有重要的研究意義.另一方面,從序列出發進行蛋白質空間結構預測是可行的[6]:Anfinsen的經典實驗表明在一般情況下,蛋白質折疊是一個自發過程;或者換句話說,蛋白質的結構信息完全蘊含于其序列之中,從而意味著蛋白質結構預測的可行性[7].
驅動蛋白質序列形成特定空間結構的主要因素是殘基之間的大量非共價相互作用,包括疏水作用、范德華力(van der Waals forces)、離子鍵以及氫鍵等.從具有相互作用的殘基間序列距離來看,上述相互作用可以分作近程相互作用和遠程相互作用2類,其中近程相互作用主導蛋白質形成局部結構,而遠程相互作用則引導局部結構的合理擺放,最終形成穩定的蛋白質空間結構[8].相對于近程相互作用而言,遠程相互作用具有決定整體結構框架的重要作用,從而獲得了更多的關注和研究.本文著重討論殘基間遠程相互作用預測問題.
蛋白質中殘基間是否有相互作用可使用殘基之間的歐氏距離作判據,即有相互作用的殘基之間距離一般較小.通常采用的標準是:當2個殘基的Cβ原子之間歐氏距離小于8?時,則認為這2個殘基具有相互作用,稱之為殘基間接觸(contact),而所有的接觸則形象地表示成接觸圖譜(contact map,見圖2).形式上,一個包含L個殘基的蛋白質接觸圖譜可以表示成一個L×L的矩陣A*:
(1)

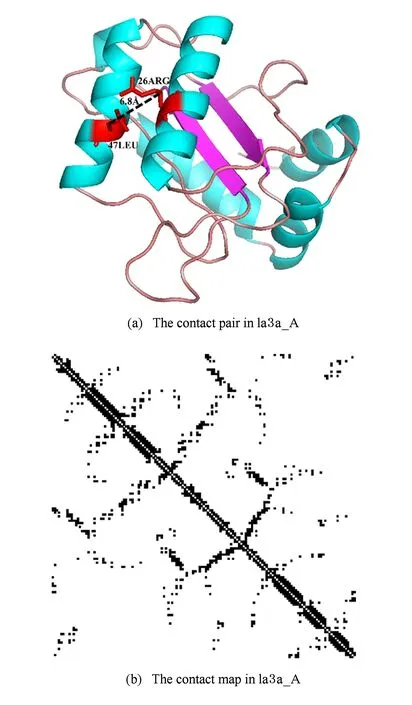
Fig. 2 Illustration of residue-residue contact in a protein 1a3a_A圖2 蛋白質1a3a_A中殘基接觸示意圖
在獲得了殘基間距離信息之后,采用分子動力學模擬(molecular dynamics simulation, MDS)[9]等技術可以有效地反推出各個殘基的空間位置.因此,殘基間相互作用的準確預測成為蛋白質空間結構預測的關鍵環節之一.
在蛋白質進化過程,有相互作用殘基對之間存在一種“共進化”模式,即當一個殘基發生變異時,與其有相互作用的殘基也要發生相應的變異,以維持相互作用,進而維持整體空間結構以及生物學功能.基于上述生物學觀察,研究者提出了多種統計模型和算法以預測殘基間相互作用.從統計學的角度講,由蛋白質序列信息出發預測殘基間遠程相互作用是一個典型的機器學習問題,即預測組成單元間關聯關系的結構學習(structured learning)問題[10].
目前的預測方法主要分為2類:無監督學習方法和監督學習方法.簡要地說,無監督學習方法僅從序列出發抽取出待測蛋白質的進化歷史信息,進而分析2個殘基在進化過程中的共變程度,以共變程度的強弱來推斷殘基間是否存在相互作用.由于這類方法不依賴于已知相互作用的蛋白質集合,因此屬于無監督學習方法的范疇.另一類方法是基于已有的結構信息,依據每個殘基的序列特征和結構特征,采用神經網絡[11]、支持向量機[12-13]等分類模型預測殘基間是否存在相互作用,從而屬于有監督學習方法的范疇.
值得強調指出的是在生物信息學這門交叉學科中,重要的是如何將計算模型和生物學現象相結合.具體到殘基相互作用這個問題而言:1)殘基間相互作用預測大量應用了現有的統計、組合最優化、機器學習等領域的研究成果,是對現有成果的應用和檢驗;2)殘基間相互作用預測問題有其特殊性,主要體現于蛋白質是進化作用的結果.因此,在建模過程中不能簡單照搬現有算法和模型,而是需要考慮進化等生物學觀察,對現有算法和模型作必要的擴展和改進.
換句話說,每一種統計模型和算法的設計都是基于特定的生物學觀察基礎之上的,是對生物學觀察的數學刻畫和描述;另一方面,我們對殘基相互作用的生物學觀察越深刻,則越有助于我們設計更有效的統計模型.
依據上述觀點,本文在介紹每一類算法時,都首先介紹生物學觀察,進而介紹如何基于上述生物學觀察設計統計模型.
本文綜述了目前的蛋白質殘基遠程相互作用預測算法:1)介紹無監督學習方法(又進一步細分為局部模型和全局模型2類);2)介紹監督學習方法,并以國際蛋白質結構預測競賽(critical assessment of protein structure prediction, CASP)的結果來分析比較現有方法;3)總結分析未來的發展趨勢.
1 無監督學習方法的局部模型
1.1 基本思想
1) 生物學觀察.從進化的角度來看,同源蛋白質是指由同一個祖先蛋白質進化而來的后代蛋白質,其結構和功能有一定的保守性,這種保守性由殘基的遠程相互作用來維持.在進化過程中,當有相互作用的殘基對中其中一個殘基發生突變時,另一個常常也發生相應的突變,以維持相互作用,否則不利于蛋白質整體結構的穩定.這種現象被稱之為“有相互作用殘基對的共進化”,如圖3所示.
2) 數學模型設計.通常采用多序列聯配的數學形式來刻畫同源蛋白質之間的同源關系,其中每一列表示由祖先蛋白質中的某個殘基進化生成的殘基(如圖4所示).考慮長度為L的多序列聯配,通常用離散隨機變量Xi(1≤i≤L)表示第i個殘基(亦稱為位點)的氨基酸種類,可取21個離散值(包括20種氨基酸和一個聯配空位),則多序列聯配中的每條序列可看成這些變量的一個觀測樣本.

Fig. 3 The phylogenetic tree and MSA of PF00111圖3 序列家族PF00111的進化樹和多序列聯配
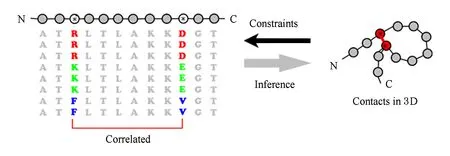
Fig. 4 Illustration of the principal for contact prediction using unsupervised methods圖4 無監督學習方法預測殘基遠程相互作用原理示意圖[14]
基于殘基共進化現象,無監督學習方法的基本思想是首先檢索出與待預測蛋白質序列同源的所有序列,計算出多序列聯配(multiple sequence alignment, MSA),以此來表示待測蛋白質的進化歷史信息;進而分析2個殘基在進化過程中的共進化(或者共變異)程度,以共變程度的強弱來推斷殘基間是否存在相互作用.從統計的角度來講,即是通過多序列聯配中列向量之間的相關性,即2個隨機變量Xi,Xj之間的相關性,從而推斷殘基之間的相互作用.
無監督學習方法可以分為兩大類,即局部模型和全局模型[6].其中,局部模型假設一個殘基對內部的相關性與其他殘基對是獨立的,從而每對殘基單獨計算其相關性;而全局模型則考慮了殘基對之間的關聯關系,對所有的殘基建立統一的全局模型.我們在本節介紹局部模型,在第2節介紹全局模型.
1.2 典型方法
局部模型在計算某對殘基之間的相關性時,不考慮其他殘基對的影響,直接分別計算各殘基對之間的相關性;各種局部模型的差異主要體現在變量相關性的衡量方法不同.下面我們介紹3種典型的局部模型,并分析其優缺點.
1.2.1 典型方法1:共變相關系數

(2)
其中,M是多序列聯配的序列條數,〈·〉為矩陣元素均值,σi為矩陣si所有元素的標準差,如圖5所示.
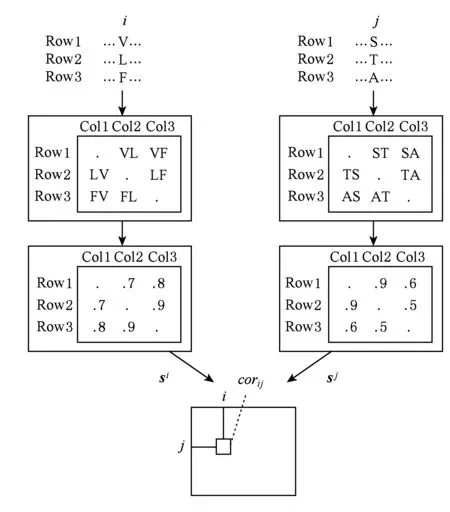
Fig. 5 Illustration of co-mutation extraction for residue pair (i,j)[15]圖5 位點對(i,j)共變信息計算示意圖[15]
實驗表明:將相關系數作為共變度量,在一定閾值下推斷殘基間相互作用,準確率比隨機預測有顯著提高,從而表明由殘基共變性推斷其相互作用的可行性;然而Pearson相關系數只能度量隨機變量間的線性相關關系,從而使得該方法存在一定的局限性.
1.2.2 典型方法2:互信息
Martin等人[16]用互信息(mutual information, MI)識別共進化殘基對.這種方法源于信息論,具體來說,對于某個多序列聯配中的位點i和j,其互信息定義為
(3)
其中,fij(a,b)為位點i出現殘基a且位點j出現殘基b的頻率,fi(a)表示位點i出現殘基a的頻率.與Pearson相關系數相比而言,互信息可以度量變量之間的非線性關系,其值越大表示殘基對間的共進化程度越大,互信息為0則表示2位點獨立進化,或存在保守位點.
Martin等人的實驗結果表明互信息較高的位點對傾向于具有相互作用,但是其效果受限于序列條數和進化偏差造成的背景噪音.因而欲提高預測準確率,需降低背景噪聲的影響,減少對序列條數的依賴,從而為后續研究指明了改進方向.
1.2.3 典型方法3:OMES
Kass等人[17]提出另外一種共變性的度量方法OMES(observed minus expected squared),這種方法基于統計學中的卡方檢驗,通過比較殘基對在2個位點上實際出現次數與期望出現次數之間的差異來定量刻畫殘基對的共進化程度.具體地,其定義為
(4)
其中,Oij(a,b)和Eij(a,b)分別表示殘基對(a,b)出現頻數的觀測值和期望值,M是序列條數.Oij(a,b)可以直接從多序列聯配中統計得到;Eij(a,b)是在假設殘基對間不存在相關性的前提下計算得到的,即Eij(a,b)=Mfi(a)fj(b),其中fi(a),fj(b)分別表示相應位點某氨基酸的出現頻率.OMES的值越大表示2個位點之間的共進化程度越高;對于2個完全獨立進化的位點,OMES的值為0.
我們在GREMLIN數據集[18]上測試OMES方法,并與MI進行了比較;實驗結果表明:MI與OMES的預測性能相當,詳細實驗結果分析參見第4節.
1.3 局部模型的實驗結果及分析
局部模型的優點是簡單、計算速度快;但是也有較大的不足,主要表現為:1)各殘基對之間并不是獨立的,而是存在關聯傳遞的現象,局部模型并沒有考慮這種關聯傳遞現象;2)未考慮序列空間采樣偏差及樣本數不足的影響;3)相關性計算存在大量由進化偏差產生的背景噪聲.
雖然局部模型普遍存在預測準確率偏低的缺陷,然而在一定程度上提取了進化信息,是由序列信息推斷結構約束的早期嘗試,對后續研究具有重要的啟發和借鑒意義.
2 無監督學習方法的全局模型
2.1 基本思想
1) 生物學觀察.局部模型單獨計算各個殘基對的相關性,其暗含的假設是某個殘基對的相互作用是獨立于其他殘基對的.然而一個殘基可能與多個殘基有作用,從而導致關聯傳遞這一普遍存在的現象.如圖6所示,如果殘基A和殘基D共變,殘基D和殘基C共變,那么從序列信息來看,殘基A和殘基D也表現出共變性,然而殘基A和殘基D之間的共變性源于傳遞效應,并非源自殘基A和殘基D的相互作用.這種通過殘基共進化的傳遞效應導致的相關性稱為間接關聯.
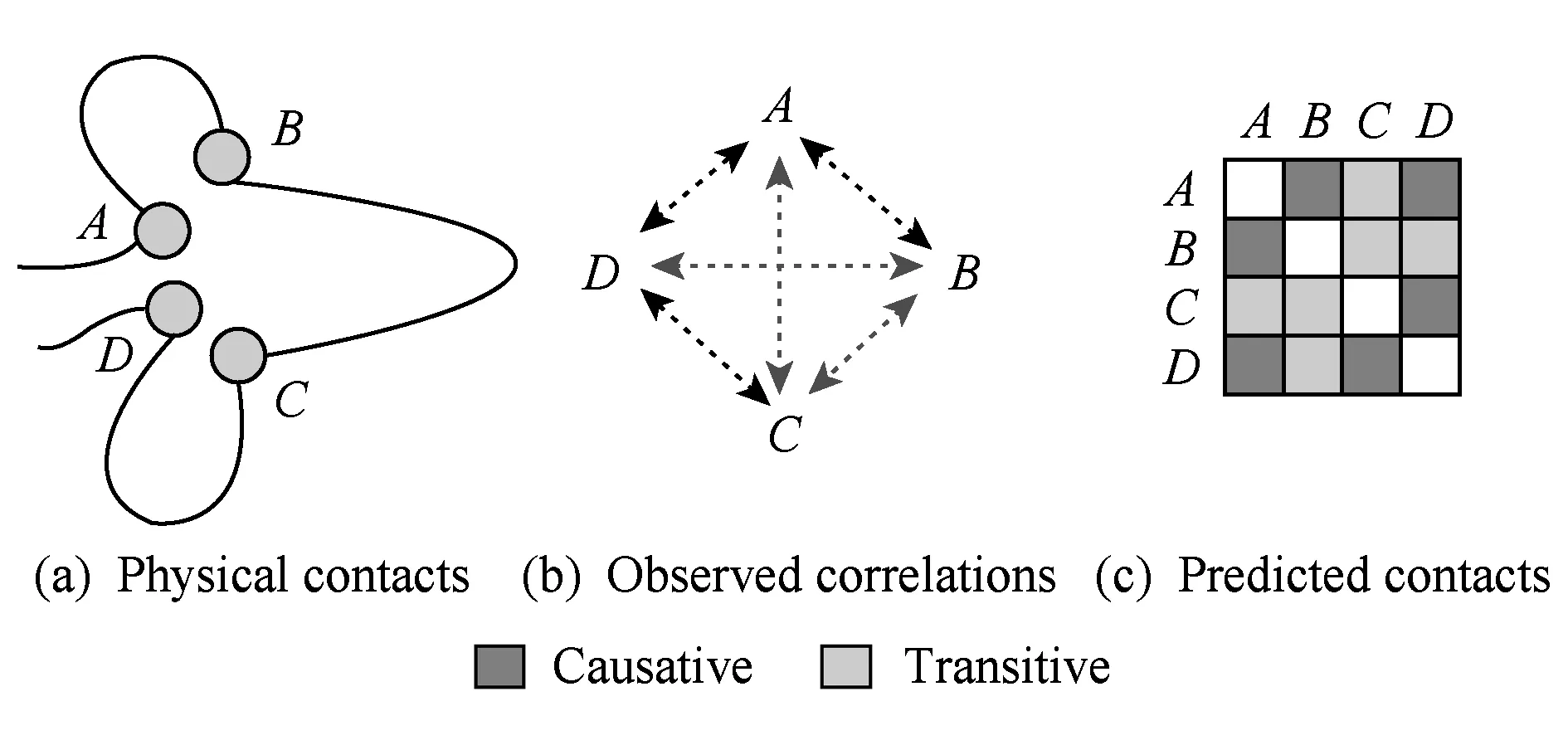
Fig. 6 Illustration of direct and indirect couplings圖6 直接關聯和間接關聯示意圖[25]
2) 數學模型設計.局部模型假設任意2個殘基的共進化和其他殘基是相互獨立的,決定了它只能探測相關性,會受到間接關聯噪聲的顯著影響,從而不能準確提取出真實共進化(接觸)的殘基對.鑒于此,全局模型對所有位點建立全概率模型,同時考慮所有殘基對之間的關聯關系,試圖去除間接關聯的影響,從而避免局部模型的缺陷.
2.2 典型方法
迄今為止已經發展了多種全局模型,比如Markov隨機場模型(Markov random fields, MRF)[19-21]、貝葉斯網絡模型(Bayesian network)[22]、高斯圖模型(Gaussian graphical model)[23-24]和網絡反卷積(network deconvolution)等[25].這些方法的不同之處主要體現在如何對多序列聯配建模,其中貝葉斯網絡模型采用有向圖模型進行建模;Markov隨機場模型和高斯圖模型采用無向圖模型進行建模;高斯圖模型可以看成Markov隨機場模型的特殊形式.
2.2.1 典型方法1:貝葉斯網絡模型
Burger等人提出使用貝葉斯網絡模型預測殘基間相互作用[22].在這種方法中,使用貝葉斯網絡把殘基間的共進化關聯關系表示成依賴關系:當位點i和位點j存在相互作用時,則第i個位置出現殘基Xi的概率依賴于第j個位置出現殘基Xj的概率.這種依賴關系形象地表示成貝葉斯網絡中的一條有向邊,如圖7所示.

Fig. 7 Bayesian network model of a given MSA圖7 貝葉斯網絡模型示意圖
在已知位點之間依賴關系的情況下,可以計算觀察到某個多序列聯配的條件概率;反過來,在給定多序列聯配的情況下,結合依賴關系的先驗分布,可以推斷位點i和位點j之間存在依賴關系(共進化)的后驗概率,最后認為后驗概率高的殘基對具有相互作用.
本節首先描述給定依賴關系的情況下觀察到某個多序列聯配的條件概率計算過程,然后介紹依賴關系的后驗概率的計算過程.
1) 已知殘基間依賴關系計算多序列聯配的條件概率
假設殘基間所有的依賴關系形成有向圖T=(π,V),其中π表示所有有向邊的集合,V={1,2,…,L}表示殘基位點集合.為簡化計算,進一步假設依賴關系圖T是樹狀圖,即在T中存在唯一根節點r,除根節點r外,其余節點i都存在唯一父節點,記其父節點為π(i).
給定殘基間依賴關系T,則觀察到多序列聯配D的條件概率為

(5)
其中,Sij=P(Di,Dj)(P(Di)P(Dj));,表示MSA第i列中各氨基酸出現頻率向量.假設第i列中氨基酸α出現的概率為,且wi服從Dirichlet分布,則:
P(Di)=∫P(Di|w)P(w)dw=
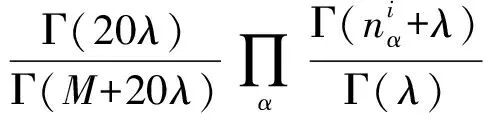
(6)
同理可得:
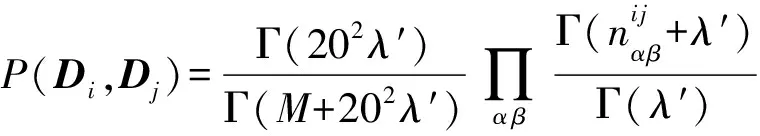
(7)
其中參數λ,λ′是偽計數.
2) 殘基間依賴關系后驗概率的計算

(8)
則式(8)可重寫為
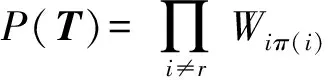
(9)
由式(9)可推出MSA的概率模型為
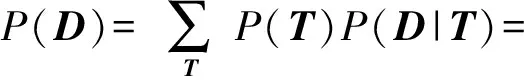
,
(10)
其中,Bjπ(j)=Sjπ(j)Wjπ(j).
使用貝葉斯公式計算T的后驗分布為:P(T|D)=P(D|T)P(T)P(D),進而對于特定的殘基對(k,l)之間有邊Ekl的后驗概率通過對包含Ekl的所有樹的后驗概率求和得到,如圖8所示:
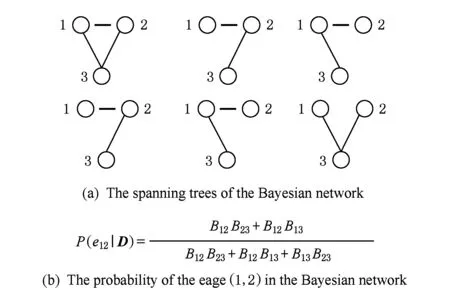
Fig. 8 Illustration of the calculation of posterior probability圖8 后驗概率計算示意圖[22]

(11)

實驗結果表明:以后驗概率P(Eij|D)作為殘基對(i,j)的相關度量能夠去掉間接關聯,和局部模型相比,顯著提高了預測準確率.
2.2.2 典型方法2:Markov隨機場
Markov隨機場是一種無向圖模型,其形式可由最大熵原理推導得到,所以也被稱為最大熵模型.Markov隨機場的優勢在于可以直接刻畫殘基間的遠程相互作用[19-21,27].
設多序列聯配的長度為L,變量Xi表示第i個位置出現的氨基酸,則多序列聯配中的一條序列的生成概率為
P(X1,X2,…,XL)=

(12)
其中:
為配分函數,eij(Xi,Xj)表示位置i處氨基酸Xi和位置j處氨基酸Xj的耦合強度,hi(Xi)為位置i處觀察到殘基Xi的可能性,均為待確定的參數.最終的推斷規則為:耦合強度強的殘基對被預測為具有相互作用.
假設給定包含M條序列的多序列聯配,上述待定參數eij(Xi,Xj)與hi(Xi)可以通過極大似然策略進行估計.然而上述極大似然策略涉及到配分函數Z的計算,其計算是NP難問題,所以發展出多種近似求解方法,包括置信傳播算法、平均場近似算法和偽似然最大化算法,簡要介紹如下:
1) 置信傳播算法bpDCA
Weigt等人[20]用Markov隨機場模型研究蛋白質-蛋白質相互作用,并用置信傳播算法(bpDCA)近似求解模型參數,后來這種方法也被直接借用于殘基間相互作用的推測.
置信傳播算法的基本思想是通過局部信息的多次傳播以逼近全局信息,以此計算配分函數或邊際概率.確切地說,在最大化似然函數過程中,梯度函數的計算涉及邊際概率,而置信傳播算法的核心是解決梯度計算問題.在給定初始參數的情況下,bpDCA迭代執行2個步驟直至滿足收斂條件:
① 用置信傳播算法估計邊緣分布Pi(Xi)和Pij(Xi,Xj)
首先對于每個位置i,迭代求解信息傳遞Pi→j(Xi):

(13)
此處fi(A)為經驗頻率.
然后可獲得邊際分布Pi(Xi)的估計:
Pi(Xi)~exp{hi(Xi)}
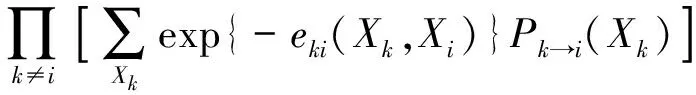
(14)
用類似的方法可得Pij(Xi,Xj)的估計.
② 用梯度下降策略更新參數估計
首先,似然函數的梯度可估計為
Δeij(Xi,Xj)=fij(Xi,Xj)-Pij(Xi,Xj)-

Δhi(Xi)=fi(Xi)-Pi(Xi),
(15)
其中,fi(A)和fij(A,B)為經驗頻率.然后更新參數估計為
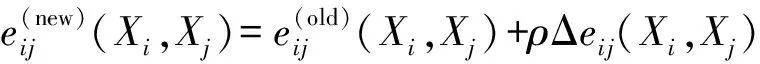
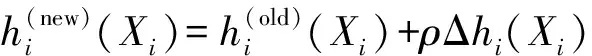
這里ρ為迭代步長.
bpDCA主要有2個缺陷:①速度慢.該算法迭代1次的計算復雜度為O(212L4),即使對長度為60的短蛋白,bpDCA在4核CPU上也需大約運行4 d.②收斂性差.該算法解的漸進性質不能得到有效控制,理論上不能保證其收斂性.
2) 平均場近似算法mfDCA
Morcos等人[19]提出使用平均場近似策略(mfDCA)來近似求解Markov隨機場的參數.平均場近似的基本思想是由簡單可分解分布近似復雜分布,因此其核心在于2個問題:①如何確定簡單分布的形式;②如何衡量簡單分布和復雜分布之間的差異,并找到最接近原始復雜分布的簡單分布.
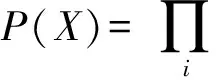
(16)
其中C可由經驗協方差矩陣計算,即Cij(A,B)=fij(A,B)-fi(A)fj(B).此處fi(A)和fij(A,B)為經驗頻率.
mfDCA的優勢是速度快,通過求逆計算耦合參數的時間復雜度是O(L3),比bpDCA速度提高上千倍,從而使大量蛋白質家族的計算成為可能.
3) 極大偽似然算法plmDCA
L?vkvist等人[21]提出偽似然最大化方法估計MRF的參數,其基本思想是用偽似然函數近似似然函數.由于計算偽似然函數梯度的時間復雜度是多項式的,所以可以有效地估計參數.
偽似然函數定義如下:
(17)
上述模型參數數目過多,對長為L的序列來說,模型參數規模為(20×20)×L(L-1)2+20L.當L=100時,模型將有近200萬的參數.為避免過擬合問題,Ekeberg等人在偽似然函數中引入了正則項R(h,e),即通過解決以下優化問題求解參數:
{hPLM,ePLM}=arg min{lpseudo(h,e)+R(h,e)},
(18)

其中,λh和λe分別表示單體項h和雙體項e的正則化參數.
該方法避免了極大似然求解復雜配分函數的問題且當樣本量足夠大時,極大偽似然估計是極大似然估計的一致估計[30],從而能夠保證獲得準確的參數估計,并且和mfDCA相比顯著提高了準確率.
Kamisetty等人[18]在極大偽似然的基礎上,進一步將結構先驗信息引入正則項,開發軟件GREMLIN.實驗結果表明,由于引入結構先驗信息,GREMLIN方法的性能優于plmDCA.
2.2.3 典型方法3:高斯圖模型
高斯圖模型假設多序列聯配中每一條蛋白質序列都服從高斯分布N(μ,Σ),其中高斯分布的協方差矩陣的逆稱為精細矩陣(recision matrix),記作Θ=Σ-1.精細矩陣表征了變量之間的直接關聯信息[31],因而可以通過精細矩陣來預測殘基間的相互作用.在統計學中,通過分析精細矩陣來推斷直接關聯的策略也稱為偏相關分析.
為防止過擬合,通常采用引入正則項的策略來控制模型的復雜度.典型的方法包括PSICOV所使用的圖Lasso以及CoinDCA所采用的成組Lasso,簡要介紹如下:
1) PSICOV使用的圖Lasso策略
Jones等人[24]利用圖Lasso策略推斷精細矩陣,開發了軟件PSICOV.該方法的核心思想是優化含有正則項的似然函數:
(19)
其中,COV為經驗協方差陣.前2項為高斯分布的對數似然,第3項為正則項.正則項的引入有2個作用:①控制模型復雜度,防止過擬合,以避免模型參數過多導致參數推斷的困難;②保證精細矩陣的稀疏性,以此刻畫接觸圖譜的稀疏性.
Jones等人[24]在150個目標蛋白進行測試,結果顯示PSICOV一致優于局部模型和貝葉斯網絡模型.
2) CoinDCA采用的成組Lasso策略
Ma等人[23]對高斯圖模型做了擴展,以融合多個相關家族的進化信息,并開發了軟件CoinDCA.
CoinDCA的基本思想是:假設對于目標蛋白質序列,與其具有相同折疊類型的共有K個家族(可通過同源搜索獲得),由于這K個蛋白家族屬于同一折疊類型,所以可認為它們有類似的蛋白質接觸圖譜;相應地,當用K個高斯分布分別對K個蛋白家族建模時,它們有類似的精細矩陣;成組Lasso的目的是約束這K個高斯分布具有類似的精細矩陣.
具體地,通過優化下式求解這些精細矩陣.
(20)

CoinDCA充分利用了相近家族的進化信息且融合了監督學習(隨機森林)的方法.在PSICOV,CASP10和CASP11數據集上測試的實驗結果表明,這種方法對同源序列少的蛋白預測準確率有顯著提高,減少了對同源序列數目的依賴;而單一地整合多家族信息或者采用隨機森林的方法并不能對預測性能有所改進.
2.2.4 典型方法4:網絡反卷積
在殘基相互作用中消除間接效應,本質上是網絡推斷領域直接作用和間接作用的區分問題[32].Feizi等人[25]提出網絡反卷積(network deconvolution, ND)策略推斷直接相互作用網絡.
網絡反卷積的基本思想是:假設觀測到的相關矩陣Gobs是直接相關矩陣Gdir和間接相關矩陣的疊加,而間接相關可視為直接相關通過多步傳遞得到的(如圖9所示),即:
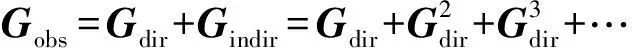
注意到當矩陣Gdir特征值絕對值小于1時,等式右邊收斂,上式有閉合形式:
(21)
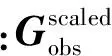
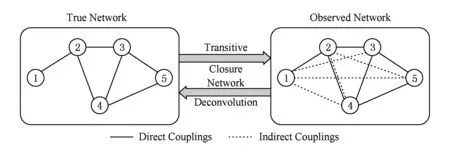
Fig. 9 Illustration of network deconvolution圖9 網絡反卷積意圖[25]
網絡反卷積方法廣泛應用于社交網絡、基因調控網絡等領域的推斷中.Wright等人[32]在殘基相互作用推斷中的結果表明,網絡反卷積策略可有效過濾掉互信息的間接關聯噪聲,而對全局模型mfDCA輸出的直接信息(已去除間接關聯)進行反卷積并沒有顯著改進;而對互信息矩陣反卷積的預測效果不如mfDCA.上述結果說明網絡反卷積的策略具有普適性,但對于特定的殘基相互作用預測問題則仍然需要進行相應的改進.
Sun等人[33]提出了平衡網絡反卷積方法,該方法不需要像原始網絡反卷積方法那樣對Gobs進行線性縮放,其假設
Gobs=Gdir+Gindir=
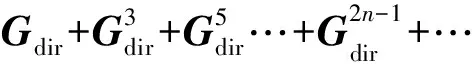
(22)
則:
(23)
可以導出對任意的λobs∈(-∞,+∞),都有|λdir|<1,所以該方法不需要對Gobs進行線性縮放.結果表明,平衡網絡反卷積方法的預測性能優于原始網絡反卷積方法;如何進一步提升預測性能,仍然需要后續有針對性的研究.
2.3 全局模型的實驗結果及分析
我們在GREMLIN數據集上詳細測試了無監督學習方法的性能,如表1、表2所示,結果顯示全局模型優于局部模型,且在全局模型中plmDCA預測性能最優.具體地,我們從實驗結果分析中獲得3個結論:
1) 由于有效地去除了間接效應的影響,全局模型比局部模型革命性地提高了預測準確率.
2) 全局模型之間的預測性能差距相對較小,且不同方法的預測結果有一定程度的不同,將不同方法得到的殘基相互作用信息有效地整合,并用于蛋白質三級結構預測,具有重要意義和廣泛的應用前景.

Table 1 Denoising Performance of LRS for Three LocalMethods on GREMLIN Benchmark
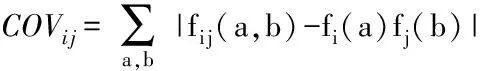

Table 2 Denoising Performance of LRS for Three GlobalMethods on GREMLIN Benchmark
3) 全局模型普遍存在參數多的問題,要得到精確的參數估計需要大量樣本信息,預測效果強烈依賴于同源序列的數目;且復雜的參數估計導致全局模型運行速度較慢,需要開發更加有效的參數估計方法.
3 無監督學習方法中的預處理和后處理
除了統計模型之外,影響遠程相互作用預測性能的因素還包括樣本的不獨立性以及背景噪聲的影響等,因此需要采取預處理和后處理步驟以消除這些因素的影響.通常采用的預處理和后處理步驟簡要陳述如下.
3.1 克服樣本不獨立性的預處理過程
多序列聯配中的樣本是與待測蛋白同源的序列,這些序列來源于同一祖先蛋白質,從而造成了觀測樣本之間的不獨立性,影響模型預測的準確性.為提高準確性,一般對多序列聯配進行2方面預處理:
1) 去冗余.比如去掉與目標蛋白質高度相似(通常采用序列等同度大于90%)的序列.
2) 加權重.對于每條序列,都依據在多序列聯配中與其相似的序列條數賦予權重,其基本思想是:如果一條蛋白質序列具有較多的相似序列,則權重較低;反之則設置較高權重.具體地,對第k條序列來說,首先統計與其序列等同度高于80%的序列數目:
mk=|{n∈{1,2,…,M}|seqid(Xk,Xn)>80%}|,
(24)
進而在統計殘基和殘基對頻率向量時,將每條序列的權重設置為1mk.
3.2 去背景噪聲的后處理過程
同源序列中通常包含由進化造成的較強的背景噪聲.具體來說,如果一個位點突變發生在進化早期,其后代都將延續這個突變,從而導致過高地估計了此位點和其他位點之間的共變性.
通常采用后處理的方法消除這些背景噪聲對相互作用預測的影響,常用的策略簡要介紹如下:
1) 均值乘積校正方法(average product correction, APC)
Dunn等人[34]基于信息論提出對互信息矩陣進行歸一化去噪的方法APC.其基本思想是假設背景噪聲具有如下的均值乘積的形式:

(25)
其中MIi.表示位點i與其他位點互信息的平均值;MI..表示所有位點對互信息的平均值.
經APC去噪后的互信息MIp為
MIpij=MIij-APCij.
(26)
實驗結果表明采用APC技術去除背景噪聲后,能夠有效提高基于互信息對殘基相互作用預測的精度.目前,該策略被推廣應用到其他相關矩陣,已成為全局統計模型標準的后處理步驟.
2) 譜去除方法(spectrum cleaning, SC)
3) 低秩稀疏矩陣分解方法(low rank and sparse matrix decomposition, LRS)
譜去除方法假設背景噪聲來源于相關性矩陣的第一主成分,其秩為1;然而當多序列聯配中的序列是來源于多個家族,則相關性矩陣的其他主成分也會含有背景噪聲[35].基于上述認識和觀察,我們團隊假設背景噪聲是低秩的,同時真實的相互作用是稀疏的.大量統計數據支持上述假設的合理性,即真實相互作用僅占所有可能相互作用的3%~5%[11,24].基于上述生物學觀察,我們設計了統計模型將背景噪聲和真實的相互作用信號區分開來[36].
具體地,對于給定的殘基相關性度量矩陣M,我們認為它是低秩噪聲矩陣和稀疏信號矩陣的疊加,進而用低秩稀疏矩陣分解技術還原真實的相互作用信號矩陣,即求解如下的優化問題:
(27)
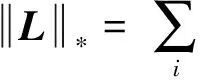
4) 去背景噪音方法的實驗結果及分析
我們在GREMLIN測試集上測試去背景噪音方法的性能,詳細實驗結果表1、表2所示.從表1和表2中可以看出無論序列間隔取值,無論是局部模型或全局模型的具體方法,LRS的去噪效果都一致地優于APC技術,且對局部模型的改善顯著高于全局模型.LRS技術的價值集中體現在對局部模型的顯著改進,使其達到和全局模型mfDCA相近的性能.這是自全局模型提出以來首次發現基于局部度量的方法能夠達到和全局模型可比的效果,也說明只有當有效地去除背景噪音之后,相關性度量才能提取出更加準確的共進化信息.下面我們將從理論上進一步深入分析LRS優于其他去背景噪音方法的原因,主要基于2個事實:
① APC和SC的等價性
SC認為第一主成分表示相關性的整體一致性,能夠刻畫由進化偏差引起的背景噪聲.第一特征值和對應特征向量元素分別近似為
則背景噪音矩陣元素近似為
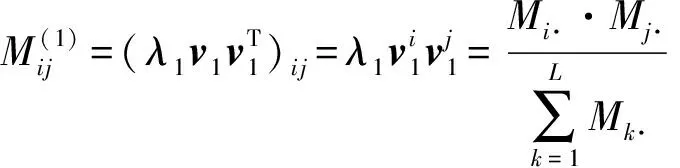
(28)
從上述分析可以看出SC關于背景噪音的近似與APC的平均乘積校正是等價的,都是秩為1的矩陣.
② LRS是SC和APC的擴展和加強
LRS用低秩矩陣近似背景噪聲,是上述2種技術的擴展;另外,用稀疏矩陣表征信號矩陣符合真實殘基接觸的稀疏性事實.這從理論上保證了LRS方法的優越性[36].
我們預期LRS將取代APC成為有效的去除背景噪聲的手段.
4 監督學習方法
4.1 基本思想
1) 生物學觀察.殘基相互作用本身有一定的規律,蛋白質殘基的性質,如二級結構、溶液可及性、疏水性等,對殘基間形成接觸有重要的作用.舉例來說,不同的二級結構對于接觸的分布有重大影響,如Beta正平行和反平行片段之間的殘基接觸呈現出完全不同的模式.
2) 數學模型設計.監督學習方法將殘基間相互作用預測視為機器學習中的分類問題,首先對每個殘基對都提取多種特征(比如保守性、預測的二級結構、溶液可及表面積等),然后在已知殘基相互作用情況的集合上訓練上述特征的權重.
4.2 典型方法
各類監督學習方法的不同主要體現在2方面:1)選取的特征不同;2)采用的機器學習的模型不同.典型的方法包括整數規劃PhyCMAP[37]、隨機森林PconsC系列[38-39]和神經網絡方法MetaPSICOV[11],簡要介紹如下.
4.2.1 典型方法1:整數線性規劃
Xu等人[37]考慮殘基性質對殘基遠程相互作用的重大影響,利用整數線性規劃將殘基相互作用需滿足的物理約束和共進化信息整合起來,開發了軟件PhyCMAP.其基本思想是:1)采用隨機森林技術預測殘基間存在相互作用的概率;2)利用整數線性規劃選擇出概率較大的殘基間相互作用,同時要求這些殘基對滿足一些物理約束.
整數線性規劃的目標函數為
(29)
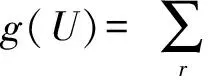
整數線性規劃考慮以下物理約束:殘基i最多參與形成多少殘基相互作用;2個二級結構之間最多會形成多少殘基相互作用;2個strand(形成sheet的片段單元)之間的相互作用具有連續性等.比如當2個strand之間形成正平行sheet時,接觸的相鄰殘基對需要滿足以下約束:Yi,j≥Yi-1,j+Yi+1,j+1-1,其中,i,i±1表示其中一個strand上的殘基,j,j±1表示另一個strand上的殘基.該約束保證2個strand之間形成的相互作用具有連續性.
PhyCMAP由于同時考慮了真實殘基相互作用的限制和共進化信息,其在CASP10和Set600數據集[37]上測試的結果表明PhyCMAP超過了當時比較流行的其他監督學習方法的軟件,例如NNcon[12],SVMcon[40]等.
同時PhyCMAP也有其局限性.其只考慮了局部模型MI輸出的共進化信息,而并沒有考慮更加有效的全局模型輸出的共進化信息.下面介紹的Pconsc和MetaPSICOV方法克服了PhyCMAP的局限性.
4.2.2 典型方法2:隨機森林方法PconsC系列
Skwark等人[39]發現不同的全局模型預測得到的殘基接觸集合有一定差異;而且不同的構建多序列聯配的軟件輸出的多序列聯配也不同,這些不同的多序列聯配也會導致遠程相互作用的預測結果不同.
基于上述觀察,2013年Skwark等人[39]將2種全局模型PSICOV和plmDCA對8種不同多序列聯配的預測結果與其他特征整合,提出了預測殘基間相互作用的隨機森林方法,并開發了軟件PconsC.多序列聯配由HHblits和jackhamme兩種比對軟件取定4種不同的閾值得到;其考慮的殘基對特征包括二級結構預測值、殘基溶液可及表面積、殘基替代向量.實驗結果表明:PconsC具有較高的預測精度,超過PSICOV和plmDCA的預測結果.
基于“相互作用殘基對的成簇性”這一認識,Skwark等人[38]進一步改進PconsC,用多層隨機森林逐步過濾掉孤立的相互作用對,開發了軟件PconsC2.值得指出的是,PconsC2的另一個優勢在于顯著減少了對樣本數的要求,從而首次實現當同源序列少于1 000條時的準確預測.實驗結果表明:PconsC2比已有的phyCMAP具有更優的預測準確率.
4.2.3 典型方法3:神經網絡方法MetaPSICOV
如4.2.1節所述,PhyCMAP結合了結構特征和共進化信息,但是只是引入了局部的共進化信息,并沒有引入更加有效的全局共進化信息.Jones等綜合考慮結構特征和全局的共進化信息,提出了預測殘基間相互作用的神經網絡模型,并開發了軟件metaPSICOV[11].
具體地,metaPSICOV是一個2層前向神經網絡模型:第1層基于二級結構、溶液可及性、殘基替代向量等特征以及PSICOV,mfDCA,plmDCA的預測結果,利用含55個隱單元的神經網絡預測出殘基相互作用概率的粗略估計;第2層以第1層的粗略估計為輸入特征,再加上部分結構特征,使用相同的神經網絡對相互作用概率的估計進行校正.
metaPSICOV根據MSA質量比較準確地權衡共進化特征和傳統特征(如二級結構等)的權重,從而更加有效地整合多種信息,提高預測準確率.結果顯示,metaPSICOV超過PSICOV,mfDCA,plmDCA的預測效果,并在第11屆CASP競賽中獲第1名(詳細分析見第6節).
4.3 監督學習方法的實驗結果及分析
針對監督學習方法的實驗結果分析表明2點結論:
1) 早期的監督學習方法,例如采用支持向量機模型的SVMcon[12]和采用神經網絡模型的NNcon[40]等,由于沒有加入有效的共進化特征,其效果并不比無監督學習方法的全局模型好.
2) 近年來提出的監督學習方法metaPSICOV和Pconsc2等,綜合了多種結構特征和無監督方法輸出的共進化特征,其效果超過了無監督學習方法和早期的監督學習方法,從而表明將無監督方法得到的結果整合到監督學習方法中是當前預測殘基相互作用最有效的策略.
5 現有軟件匯總
第2.4節所述的方法都有相應的服務器為用戶提供殘基相互作用預測服務,我們將這些服務器匯總如表3所示:

Table 3 Overview of Existing Softwares for Protein Contact Prediction
6 CASP比賽中殘基相互作用預測性能分析
CASP競賽是全球范圍內的蛋白質結構預測比賽,現已作為客觀評估蛋白質結構預測質量的標準.從1994年開始,每兩年1屆,迄今已舉辦11屆.目前,CASP競賽包括結構預測、殘基遠程相互作用預測、接觸位點輔助結構預測、結構優化、結構質量評估5個部分.
在蛋白質結構預測領域,大部分軟件采用開源軟件或者免費預測服務的方式,商業軟件較少(比如DNAStar公司開發的NovaFold和BSI公司開發的RAPTOR等).國內研究團隊也多次參加CASP比賽,包括中科院生物物理研究所的Jiang-Server團隊、上海交通大學的Shen-group團隊以及中科院計算所的FALCON團隊.其中本課題團隊開發的FALCON系列軟件在CASP-8中結構預測FR-Hard類上獲得第3名,在CASP-11中結構預測TBM類上獲得第9名.Shen-group在CASP-11殘基接觸預測的FM類蛋白上取得了第2名(以precision評價).
殘基遠程相互作用預測作為CASP競賽的重要部分.在2014年的第11屆CASP比賽中,共有29個軟件參加了殘基相互作用預測[41-45].本文提到的一些經典算法參加了這次比賽,例如采用PhyCMAP方法的RaptorX-Contact軟件、采用MetaPSICOV的CONSIP2軟件以及采用PconsC2方法的MetaPSICOV軟件等.
在CASP11中,參賽軟件大多是基于監督學習的方法.在監督學習方法中,無監督學習全局模型輸出的信息是其重要特征.根據是否含有全局共進化模型信息,可以將這些監督學習方法分為2類:1)不包含全局共進化模型信息的方法:例如PhyCMAP,采用SVMcon方法的MULTICOM-construct、采用DNcon方法的MULTICOM-cluster軟件和采用NNcon方法的MULTICOM-novel軟件等;2)包含全局共進化模型信息的方法:例如采用神經網絡模型的CONSIP2方法、采用隨機森林模型的Pcons-net(PconsC2)、采用SVM模型的Shen-group和RBO_ALEPH[46]等.我們的實驗結果分析表明包含全局共進化模型信息的方法要好于不包含全局共進化模型信息的方法.
CASP競賽對于方法的衡量主要有4個分項:
1) 預測準確率(precision)
根據各方法輸出的分數進行排序,選取TopN作為預測的接觸集合,其他的作為非接觸集合.其中N一般取L10,L5,L2或L等(L為目標蛋白的序列長度),計算正陽性(true positive,TP)和假陽性(false positive,FP),precision的定義為
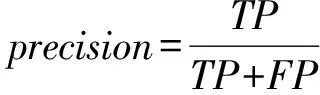
2)Xd值
將氨基酸對之間的距離分成15個區間:(0,4]?,…,(56,60]?.Xd值的定義為
其中,Ppi表示預測的殘基接觸的距離在第i個區間的比例,Pai表示結構所有的殘基接觸對的距離在i個區間的比例.Xd用來衡量真實結構和預測接觸中殘基對距離分布的差異.
3) Matthews相關系數(MCC)
CASP比賽要求各參賽軟件給出每個殘基對接觸的概率.選取0.5作為閾值計算TP,TN,FP(false positive)和FN(false negative).
MCC=(TP×TN-FP×FN)

4) precision-recall曲線下的面積(AUC_PR)
由于上述共進化分析方法給出相關性度量,而非殘基對的接觸概率,Matthews相關系數并不適用于這些方法,所以我們采用precision,Xd值和AUC_PR這3種分項對這些方法進行評估.
我們在CASP11比賽數據集合測試了本文綜述的典型方法以及參加CASP比賽的主要方法,對實驗結果采用自行開發的程序進行了詳細分析(程序和數據下載地址http:bioinfo.ict.ac.cnCOLORS),詳細分析結果按照無模板建模(free modelling, FM)類和有模板建模(template based modelling, TBM)類分別描述如下:
6.1 在FM類目標蛋白上的測試結果
在CASP11比賽中,共有17參賽隊伍提交了超過20個FM類蛋白質域的預測結果.圖10列出了各方法的預測性能,包括precision,Xd和AUC_PR.從圖10我們可以得到如下結論.
1) 綜合全局共進化模型信息的監督方法領先于其他方法.比如CONSIP(metaPSICOV)在3種分項都取得第1名;Shen-group也處于領先地位.以precision和AUC_PR評價,RBO_ALEPH的排名也較靠前.
2) 監督學習方法整體上優于無監督學習方法的效果.例如以precision作評價,無論是包含了全局共進化信息的監督方法(如CONSIP,Shen-group)還是不包含全局共進化信息的監督方法(如MULTICOM-novel,RaptorX,MULTICOM-cluster),都超過了無監督學習的方法(如plmDCA,PSICOV).
3) 在無監督學習方法中,全局模型普遍優于無監督學習方法.
4) 以precision作為評價,LRS技術優于APC技術.
值得指出的是:不同預測方法在不同的評價指標下表現不同,例如RaptorX以precision為評價排名為16,而以Xd為評價排名為2;對于無監督學習方法的去噪音方法,以precision為評價,LRS好于APC;但以AUC_PR為評價,APC好于LRS.其原因在于:LRS只提取顯著的共進化信號,將非顯著的共進化信號的分數設置為0;而APC卻可以對非顯著的共進化信號進行排名,從而造成如果以AUC_PR為評價,APC技術優于LRS技術.

Fig. 10 Prediction performance of different methods on CASP-11 FM targets圖10 典型方法對CASP-11 FM類蛋白的預測性能
6.2 在TBM類目標蛋白上的測試結果
在CASP11比賽中,共有14個參賽組提交了多于60個TBM類別蛋白域的預測結果,如圖11所示:
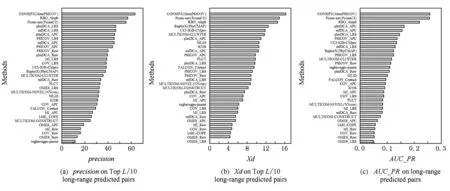
Fig. 11 Prediction performance of different methods on CASP-11 TBM targets圖11 典型方法對CASP-11 TBM類蛋白的預測性能
我們可得到如下結論:
1) 與FM類目標蛋白上的觀察相同,綜合了全局共進化模型信息的監督方法領先于其他方法.
2) 以precision為評價,整體上來講,無監督學習方法中的全局模型優于沒有結合全局模型信息的監督學習方法.例如,plmDCA_LRS和PSICOV_LRS的預測性能優于RaptorX和MULTICOM-cluster.這在很大程度上源于TBM目標蛋白的多序列聯配比FM包含更多的同源序列,從而提供了更準確的共進化信息.
3) 與FM類目標蛋白上的觀察相同,無監督學習方法中的全局模型整體優于局部模型.
6.3 預測準確率與有效同源序列數目的關系
多序列聯配的有效同源序列的數目對預測準確率的影響很大.無監督學習方法中性能較好的全局模型往往具有較多的參數,從而需要大量的同源序列進行參數估計;在監督學習中使用的重要特征也受同源序列數目的影響,例如序列譜、預測的二級結構和預測的可及水表面積等特征.
我們在99個FM類和TBM類目標蛋白上測試同源序列數目對各方法預測準確度的影響,選取經典的無監督學習方法和在CASP11中提交超過90個蛋白域的軟件進行評價.
我們根據Meff將這些目標蛋白分成了3組:1)(0,100],共36個蛋白;2)(100,1 000],共32個蛋白;3)Meff>1 000,共31個蛋白.
如圖12所示,我們可以得到以下結論:
1) 所有方法的預測準確度都隨Meff的提高而提高,但不同方法的提高程度不同.
2) 總體來講,融合全局度量的監督學習方法在3種Meff類別下都領先于其他方法,例如CONSIP2(MetaPSICOV),Pcons-net(Pcons2),RBO_Aleph等.
3) 當Meff較低時,不融合全局度量的監督學習方法優于無監督方法的全局模型.例如MULTICOM-CONSTRUCT,RaptorX-Conact,MULTICOM_CLUSTER優于CCMpred,PSICOV等方法.
4 當Meff較高時,全局模型逐漸超越不含全局模型信息的無監督方法.

Fig. 12 Relationship of mean precision with Meff for different methods on CASP-11 targets(FM and TBM)圖12 典型方法對CASP-11 (FM和TBM)蛋白預測的平均準確率與Meff的關系
7 殘基遠程相互作用預測的應用
殘基共進化分析可在未知蛋白質結構時,僅依據序列推斷殘基間的相互作用,因此在蛋白質結構和功能預測上具有重要的研究意義.
1) 殘基間遠程相互作用的信息能夠有效地促進對蛋白質結構的預測,其典型工作是基于mfDCA的相互作用預測信息開發的蛋白質結構預測軟件DCA-fold[47]和EVfold[14].Hopf等人[48]考慮膜蛋白的結構特性,使用EVfold對DrugBank數據庫中23個已知結構的膜蛋白家族進行結構預測,發現其中20個可以得到高精度預測,進而對Pfam中11個未知結構蛋白質進行結構預測.
2) 由于蛋白質結構中的二硫鍵可以看成一種特殊的殘基相互作用,所以相互作用預測的信息也有助于二硫鍵的預測.Yang等人提取遠程相互作用預測軟件GREMLIN的輸出信息,作為支持向量回歸(support vector regression, SVR)模型的特征之一[49].結果表明其軟件Cyscon優于其他同類軟件.
3) 殘基間相互作用能夠有助于對殘基功能的推斷.一般地,功能位點傾向于是蛋白質中的保守位點;類似地,殘基間的關聯強度也有助于推斷功能位點,比如Marks等人[6,48]計算某特定殘基與其他殘基的累積耦合強度,作為該殘基功能性(功能選擇壓力)的度量,并發現了一些重要的功能元件.
8 相互作用預測方法發展趨勢分析
經過20多年的發展,研究者已經提出了多種殘基相互作用的預測模型,使得預測精度有了顯著提升.然而目前已有的算法依然存在假陽性率較高、對樣本數目顯著依賴等缺陷.在前期工作的基礎上,我們認為殘基間相互作用有3個發展趨勢:
趨勢1. 改進參數估計方法.經典的統計模型如Markov隨機場能夠比較準確地描述蛋白質所有位點的全概率分布,但其參數估計的各種近似策略還有待改善,以進一步縮小與極大似然估計之間的差異,并提高計算效率.
趨勢2. 預測二級結構單元之間的相互作用.傳統遠程相互作用預測方法都是預測殘基間相互作用,這在計算上具有很大便利,但是同時也造成顯著的假陽性.事實上,從預測蛋白質整體結構這個目標來說,判斷二級結構單元之間是否存在相互作用就能夠提供足夠有價值的信息,而不用細化到殘基間是否存在相互作用.
趨勢3. 改進相互作用預測的評價方法.目前的評價方法中,所有殘基對是同等考慮的.然而在蛋白質中,各個殘基的重要性是有顯著差異的,比如二級結構單元之間的相互作用、疏水集團與其他位點的相互作用等具有相對更高的重要性,而突變較多較隨機的不重要的位點則對結構影響不大.一種可能的方案是首先基于多序列聯配給出位點重要性的先驗概率,進而在相互作用預測中有側重地考慮那些重要的殘基,這種有針對性地設置約束將能夠提高結構預測效率和精度.
9 總 結
本文對殘基間遠程相互作用預測進行了綜述,主要介紹了殘基間相互作用預測的機器學習方法,分析了各方法的預測性能,并總結了未來的發展趨勢.
值得指出的是,殘基間相互作用預測是機器學習中結構學習(structured learning)的一個典型問題,因此這方面的研究不僅具有重要的生物學意義,同時能夠推動機器學習領域的研究.
[1]Lodish H F, Berk A, Zipursky S L, et al. Molecular Cell Biology[M]. New York: WH Freeman, 2000
[2]Petsko G A, Ringe D. Protein Structure and Function[M]. London: New Science Press, 2004
[3]Wüthrich K. The way to NMR structures of proteins[J]. Nature Structural & Molecular Biology, 2001, 8(11): 923-925
[4]Kendrew J C, Bodo G, Dintzis H M, et al. A three-dimensional model of the myoglobin molecule obtained by X-ray analysis[J]. Nature, 1958, 181(4610): 662-666
[5]Taylor K A, Glaeser R M. Electron diffraction of frozen, hydrated protein crystals[J]. Science, 1974, 186(4168): 1036-1037
[6]Marks D S, Hopf T A, Sander C. Protein structure prediction from sequence variation[J]. Nature Biotechnology, 2012, 30(11): 1072-1080
[7]Anfinsen C B. Principles that govern the folding of protein chains[J]. Science, 1973, 181(4096): 223-230
[8]Kim De, Dimaio F, Wang R Y, et al. One contact for every twelve residues allows robust and accurate topology-level protein structure modeling[J]. Proteins: Structure, Function, and Bioinformatics, 2014, 82(S2): 208-218
[9]Haile J M. Molecular Dynamics Simulation[M]. New York: Wiley, 1992
[10]Anzai Y. Pattern Recognition and Machine Learning[M]. New York: Academic Press, 2012
[11]Jones D T, Singh T, Kosciolek T, et al. MetaPSICOV: Combining coevolution methods for accurate prediction of contacts and long range hydrogen bonding in proteins[J]. Bioinformatics, 2015, 31(7): 999-1006
[12]Cheng Jianlin, Baldi P. Improved residue contact prediction using support vector machines and a large feature set[J]. BMC Bioinformatics, 2007, 8(1): 11-13
[13]Chen Peng. Analysis and prediction of interactions between residues in proteins[D]. Hefei: University of Science and Technology of China, 2007 (in Chinese)(陳鵬. 蛋白質殘基間的相互作用分析與預測[D]. 合肥: 中國科學技術大學, 2007)
[14]Marks D S, Colwell L J, Sheridan R, et al. Protein 3D structure computed from evolutionary sequence variation[J]. PLoS ONE, 2011, 6(12): 1287-1296
[15]Gobel U, Sander C, Schneider R, et al. Correlated mutations and residue contacts in proteins[J]. Proteins: Structure, Function and Bioinfomatics, 1994, 18(4): 309-317
[16]Martin L C, Gloor G B, Dunn S D, et al. Using information theory to search for co-evolving residues in proteins[J]. Bioinformatics, 2005, 21(22): 4116-4124
[17]Kass I, Horovitz A. Mapping pathways of allosteric communication in GroEL by analysis of correlated mutations[J]. Proteins: Structure, Function, and Bioinformatics, 2002, 48(4): 611-617
[18]Kamisetty H, Ovchinnikov S, Baker D. Assessing the utility of coevolution-based residue-residue contact predictions in a sequence-and structure-rich era[J]. Proceedings of the National Academy of Sciences, 2013, 110(39): 15674-15679
[19]Morcos F, Pagnani A, Lunt B, et al. Direct-coupling analysis of residue coevolution captures native contacts across many protein families[J]. Proceedings of the National Academy of Sciences, 2011, 108(49): 1293-1301
[20]Weigt M, White R A, Szurmant H, et al. Identification of direct residue contacts in protein-protein interaction by message passing[J]. Proceedings of the National Academy of Sciences, 2009, 106(1): 67-72
[21]L?vkvist C, Lan Y, Weigt M, et al. Improved contact prediction in proteins: Using pseudolikelihoods to infer Potts models[J]. Physical Review E, 2013, 87(1): 12707-12929
[22]Burger L, van Nimwegen E. Disentangling direct from indirect co-evolution of residues in protein alignments[J]. PLoS Computational Biology, 2010, 6(1): 10006-10033
[23]Ma Jianzhu, Wang Sheng, Wang Zhiyong, et al. Protein contact prediction by integrating joint evolutionary coupling analysis and supervised learning[J]. Bioinformatics, 2015, 31(21): 3506-3513
[24]Jones D T, Buchan D W, Cozzetto D, et al. PSICOV: Precise structural contact prediction using sparse inverse covariance estimation on large multiple sequence alignments[J]. Bioinformatics, 2012, 28(2): 184-190
[25]Feizi S, Marbach D, Médard M, et al. Network deconvolution as a general method to distinguish direct dependencies in networks[J]. Nature biotechnology, 2013, 31(8): 726-733
[26]Meil? M, Jaakkola T. Tractable Bayesian learning of tree belief networks[C]Proc of the 6th Conf on Uncertainty in Artificial Intelligence. San Francisco, CA: Morgan Kaufmann, 2000: 380-388
[27]Lapedes A S, Bertrand G G, Liu L, et al. Correlated mutations in models of protein sequences: Phylogenetic and structural effects[J]. Lecture Notes-Monograph Series, 1999, 33(1), 236-256
[28]Plefka T. Convergence condition of the TAP equation for the infinite-ranged Ising spin glass model[J]. Journal of Physics A: Mathematical and general, 1982, 15(6): 1971-1985
[29]Georges A, Yedidia J S. How to expand around mean-field thery using high-temperature expansions[J]. Journal of Physics A: Mathematical and General, 1991, 24(9): 2173-2179
[30]Csisz X, R I, Talata Z. Consistent estimation of the basic neighborhood of Markov random fields[J]. The Annals of Statistics, 2006, 34(1): 123-145
[31]Lauritzen S L. Graphical Models[M]. Oxford, UK: Oxford University Press, 1996
[32]Wright S. Correlation and causation[J]. Journal of Agricultural Research, 1921, 20(7): 557-585
[33]Sun Haiping, Huang Yan, Wang Xiaofan, et al. Improving accuracy of protein contact prediction using balanced network deconvolution[J]. Proteins: Structure, Function, and Bioinformatics, 2015, 83(3): 485-496
[34]Dunn S D, Wahl L M, Gloor G B. Mutual information without the influence of phylogeny or entropy dramatically improves residue contact prediction[J]. Bioinformatics, 2008, 24(3): 333-340
[35]Halabi N, Rivoire O, Leibler S, et al. Protein sectors: Evolutionary units of three-dimensional structure[J]. Cell, 2009, 138(4): 774-786
[36]Zhang Haicang, Gao Yujuan, Deng Minghua, et al. Improving residue-residue contact prediction via low rank and sparse decomposition of residue correlation matrix[J]. Biochemical and Biophysical Research Communications, 2016, 472(1): 217-222
[37]Wang Zhiyong, Xu Jinbo. Predicting protein contact map using evolutionary and physical constraints by integer programming[J]. Bioinformatics, 2013, 29(13): 266-273
[38]Skwark M J, Raimondi D, Michel M, et al. Improved contact predictions using the recognition of protein like contact patterns[J]. PLoS Computatioal Biology, 2014, 10(11): 1003-1019
[39]Skwark M J, Abdel-Rehim A, Elofsson A. PconsC: Combination of direct information methods and alignments improves contact prediction[J]. Bioinformatics, 2013, 29(14): 1815-1816
[40]Tegge A N, Wang Z, Eickholt J, et al. NNcon: Improved protein contact map prediction using 2D-recursive neural networks[J]. Nucleic Acids Research, 2009, 37(Suppl 2): 515-518
[41]Kajan L, Hopf T A, Kalas M, et al. FreeContact: Fast and free software for protein contact prediction from residue co-evolution[J]. BMC Bioinformatics, 2014, 15(1): 158-164
[42]Seemayer S, Gruber M, S?ding J. CCMpred-fast and precise prediction of protein residue-residue contacts from correlated mutations[J]. Bioinformatics, 2014, 30(21): 3128-3130
[43]Baldassi C, Zamparo M, Feinauer C, et al. Fast and accurate multivariate Gaussian modeling of protein families: Predicting residue contacts and protein-interaction partners[J]. PLoS ONE, 2014, 9(3): 927-940
[44]Di Lena P, Nagata K, Baldi P. Deep architectures for protein contact map prediction[J]. Bioinformatics, 2012, 28(19): 2449-2457
[45]Monastyrskyy B, D’Andrea D, Fidelis K, et al. New encouraging developments in contact prediction: Assessment of the CASP11 results[J]. Proteins: Structure, Function, and Bioinformatics, 2015, 6(4): 126-140
[46]Schneider M, Brock O. Combining physicochemical and evolutionary information for protein contact prediction[J]. PloS ONE, 2014, 9(10): 1108-1120
[48]Hopf T A, Colwell L J, Sheridan R, et al. Three-dimensional structures of membrane proteins from genomic sequencing[J]. Cell, 2012, 149(7): 1607-1621
[49]Yang Jing, He Baoji, Jang R, et al. Accurate disulfide-bonding network predictions improve ab initio structure prediction of cysteine-rich proteins[J]. Bioinformatics, 2015, 31(23): 3773-3781

Zhang Haicang, born in 1987. PhD candidate from the Institute of Computing Technology, Chinese Academy of Sciences. His main research interests include bioinformatics, algorithm design and machine learning.

Gao Yujuan, born in 1992. PhD candidate from Peking University. Her main research interests include network inference and convex optimization algorithm.

Deng Minghua, born in 1969. Received his BS, MS, and PhD degrees in applied mathematics from Peking University. Professor in the School of Mathematical Sciences, Centre for Quantitative Biology and Center for Statistical Sciences, Peking University. His main research interests include bioinformatics and system biology.

Zheng Weimou, born in 1946. Received his BS degree in the Department of Physics from Peking University and PhD degree from Universite Libre de Bruxelles. Professor of the Institute of Theoretical Physics, Chinese Academy of Sciences. His main research interests include surface physics, stochastic process, nonlinear dynamics, biophysics and bioinformatics.

Bu Dongbo, born in 1973. Received his BS in computer science, MS and PhD degrees from the Institute of Computing Technology, Chinese Academy of Sciences. Professor of the Institute of Computing Technology, Chinese Academy of Sciences. Member of CCF. His main research interests include algorithm design and analysis, SAT problem, and bioinformatics (especially on genome sequencingassembly, protein sequencing via mass spectra, protein structure prediction).
A Survey on Algorithms for Protein Contact Prediction
Zhang Haicang1,2, Gao Yujuan3, Deng Minghua3,4,5, Zheng Weimou6, and Bu Dongbo11
(InstituteofComputingTechnology,ChineseAcademyofSciences,Beijing100190)2(UniversityofChineseAcademyofSciences,Beijing100049)3(CentreforQuantitativeBiology,PekingUniversity,Beijing100871)4(SchoolofMathematicalSciences,PekingUniversity,Beijing100871)5(CenterforStatisticalSciences,PekingUniversity,Beijing100871)6(InstituteofTheoreticalPhysics,ChineseAcademyofSciences,Beijing100190)
Proteins are large molecules consisting of a linear sequence of amino acids. In the natural environment, a protein spontaneously folds into specific tertiary structure to perform its biological functionality. The main factors that drive proteins to fold are interactions between residues, including hydrophobic interaction, Van der Waals’ force and electrostatic interactions. The interactions between residues usually lead to residue-residue contacts, and the prediction of residue-residue contacts should greatly facilitate understanding of protein structures and functionalities. A great variety of techniques have been proposed for residue-residue contacts prediction, including machine learning, statistical models, and linear programing. It should be pointed out that most of these techniques are based on the biological insight of co-evolution, i.e., during the evolutionary history of proteins, a residue’s mutation usually leads its contacting partner to mutate accordingly. In this review, we summarize the state-of-art algorithms in this field with emphasis on the construction of statistical models based on biological insights. We also present the evaluation of these algorithms using CASP (critical assessment of techniques for protein structure prediction) targets as well as popular benchmark datasets, and describe the trends in the field of protein contact prediction.
protein contact prediction; protein tertiary structure prediction; graphical model; co-evolution; machine learning
2015-12-10;
2016-04-14
國家“九七三”重點基礎研究發展計劃基金項目(2012CB316502,2015CB910303);國家自然科學基金項目(11175224,11121403,31270834,61272318,31171262,31428012,31471246);中國科學院理論物理研究所理論物理國家重點實驗室開放工程項目(Y4KF171CJ1) This work was supported by the National Basic Research Program of China (973 Program) (2012CB316502, 2015CB910303), the National Natural Science Foundation of China (11175224, 11121403, 31270834, 61272318, 31171262, 31428012, 31471246), and the Open Project Program of State Key Laboratory of the Institute of Theoretical Physics, Chinese Academy of Sciences (Y4KF171CJ1).
高玉娟(lacus2009@163.com,其對本文的貢獻同第一作者);卜東波(dbu@ict.ac.cn)
TP399

