酮還原酶中立體選擇性還原位點的突變及其產物分析
李凌凌 呂早生 左振宇 楊忠華 劉曜寧 宋采薇
(武漢科技大學化學與化工學院,武漢 430081)
酮還原酶中立體選擇性還原位點的突變及其產物分析
李凌凌 呂早生 左振宇 楊忠華 劉曜寧 宋采薇
(武漢科技大學化學與化工學院,武漢 430081)
為驗證糖多孢紅霉菌聚酮合成酶中酮還原酶(EryKR)的LDD模式序列是否為控制2-甲基環己酮立體選擇性還原的位點,構建了分別異源表達聚酮合成酶模塊1的酮還原酶(EryKR1)、模塊2的酮還原酶(EryKR2)、LDD殘基替換為PQQ(LDD→PQQ)的EryKR1及PQQ替換為LDD(PQQ→LDD)的EryKR2的重組大腸桿菌Escherichia coli BL21(pET28a-eryKR1)、E.coli BL21(pET28a-eryKR2)、E.coli BL21(pET28a-TeryKR1)和E.coli BL21(pET28a-TeryKR2)。SDS-PAGE實驗證明,經IPTG誘導后4個重組菌中都表達出相應的酮還原酶。粗酶液的比酶活分別為1.49 U/mg、0.37 U/mg、0.94 U/mg和0.31 U/mg。利用氣相色譜分別檢測4個重組菌還原2-甲基環己酮體系中產物的立體結構,結果顯示與野生型EryKR1的還原產物以順式-2-甲基環己醇為主不同,LDD→PQQ的突變型EryKR1催化2-甲基環己酮的還原產物主要為反式-2-甲基環己醇,而PQQ→LDD的突變型EryKR2的主要還原產物也由野生型EryKR2的反式-2-甲基環己醇轉變成順式-2-甲基環己醇,證實了LDD模式序列確實為酶中控制2-甲基環己酮立體選擇性還原的位點。
聚酮合成酶;酮還原酶;2-甲基環己酮;突變;立體選擇性
含有環己基的手性醇是很多醫藥、農藥、有機化工產品及材料的重要中間體[1],譬如,合成酸增殖劑的關鍵前體1-苯基-cis-1,2-環己二醇,合成手性藥物的手性砌塊光學純的β-氨基環己醇等。利用基因工程技術構建不對稱還原取代環己酮的生物催化劑,為合成這類手性醇提供了一種安全性、環境相容性、立體選擇性均高的方法,但是,目前關于這方面的報道較少[2]。糖多孢紅霉菌聚酮合成酶的6個模塊中有5個酮還原酶(EryKR),是一個催化羰基不對稱還原的天然酶庫。酮還原酶表現出廣泛的底物譜:酮還原酶不僅能還原天然底物鏈式聚酮鏈及與其相似的底物如(2R,S)-2-甲基-3-羰基戊酰N-乙酰半胱酰胺硫酸酯等[3,4],而且還能以完全不同于鏈式聚酮鏈的含環己基結構的化合物作為底物[5,6],如環己酮、2-甲基環己酮和2-烯丙基環己酮等,顯示了EryKR有望成為立體選擇性還原取代環己酮的手性生物催化劑的巨大潛力。
Ⅰ型聚酮合成酶的酮還原酶根據對鏈式聚酮鏈的立體選擇性還原情況分為A型和B型,糖多孢紅霉菌聚酮合成酶模塊1的酮還原酶(EryKR1)為B型酮還原酶,在第88-103個氨基酸處有一個LDD模式序列,而糖多孢紅霉菌聚酮合成酶模塊2的酮還原酶(EryKR2)為A型酮還原酶,其中沒有這個模式[7]。多種聚酮合成酶的酮還原酶對包括(2R,S)-2-甲基-3-羰基戊酰N-乙酰半胱酰胺硫酸酯在內的若干α-替代的β-羰基雙酮化合物進行的立體選擇性還原研究,發現若這些位點發生了突變,酮還原酶對這些底物的立體選擇性會發生相應的改變,證實了這些酶中存在有控制立體選擇性還原的模式序列[7,8]。
考慮到除了鏈式聚酮鏈,酮還原酶還能以含環己基結構的化合物為底物。為了探討LDD模式序列是否也是控制含環己基結構的化合物立體選擇性還原的位點,本研究利用重疊PCR技術將EryKR1(B型KR)中控制立體選擇性還原的LDD模式序列突變成EryKR2(A型KR)中的相應位點PQQ,并且將EryKR2中的PQQ突變成EryKR1中的LDD,通過比較突變型酮還原酶與野生型酮還原酶以2-甲基環己酮為底物的酶活以及還原產物的立體結構,分析2-甲基環己酮的主要還原產物的立體結構轉變與LDD模式序列的變化之間的關聯,確定LDD模式序列是否為酶中負責控制2-甲基環己酮立體選擇性還原的位點,旨在為指導催化不對稱還原取代環己酮的手性生物催化劑的構建及開發奠定理論基礎。
1 材料與方法
1.1 材料
1.1.1 菌種、質粒和培養基 糖多孢紅霉菌(Saccharopolyspora erythraea)、大腸桿菌表達載體pET28a質粒及重組菌E.coli BL21(pET28a-gdh)為本實驗室保藏;大腸桿菌BL21(E.coli BL21(DE3))為本實驗宿主菌,購買于康為世紀生物科技有限公司。LB培養基[9]:酵母膏5 g/L、蛋白胨10 g/L、NaCl 10 g/L,pH7.0,用于培養大腸桿菌BL21,篩選抗性菌株時,加卡拉霉素終濃度為100 μg/mL;TSB培養基:Tryptic Soy Broth 30 g/L,用于糖多孢紅霉菌的液體培養。
1.1.2 酶和化學試劑 Lysozyme、蛋白酶K、pfu DNA聚合酶、限制性內切酶NdeⅠ和EcoRⅠ、T4DNA連接 酶、DNA ladder Marker、Protein Marker、DNA Loading Buffer、微量瓊脂糖凝膠DNA回收試劑盒及高純度質粒小提試劑盒均為康為世紀生物科技有限公司產品,2-甲基環己酮(98%)和反式-2-甲基環己醇(97%)為Alfa Aesar公司的產品,順式-2-甲基環己醇(98%)購自Aldrich。
1.2 方法
1.2.1 野生型及突變型eryKR1或eryKR2基因的克隆及表達質粒的構建 糖多孢紅霉菌A226基因組DNA的提取參照文獻[6]進行。根據文獻[5]報道的EryKR1和EryKR2的邊界,設計擴增野生型eryKR1基因和eryKR2基因(或突變型eryKR1即TeryKR1基因和突變型eryKR2即TeryKR2基因)的兩端引物,為便于克隆,在正向引物和反向引物前分別加上NdeⅠ和EcoRⅠ酶切位點,其中用于擴增野生型和突變型eryKR1基因的兩端引物為LKR1:5'-gcc atatggacgaggtttccgcgctgcg-3'(gc為保護性堿基,catatg為NdeⅠ酶切位點);RKR1:5'-ggaattctcacgcgcccacc cgcggttcggc-3'(g為保護性堿基,gaattc為EcoRⅠ酶切位點)。
為了采用重疊PCR技術擴增TeryKR1基因,設計中間的將EryKR1酶中的LDD對應的堿基突變成EryKR2酶中的PQQ相應的堿基的正反向引物分 別 TKR1-L:5'-cacgccgccgccacgccgcagcagggcaccg tggacaccctcacc-3',TKR1-R:5'-ggtgtccacggtgccctgctg cggcgtggcggcggcgtggaacac-3';而用于擴增野生型和突變型eryKR2基因的兩端引物為LKR2:5'-gccatatgg acgagctcgacggctggttc-3'(gc為保護性堿基,catatg為NdeⅠ酶切位點),RKR2:5'-ggaattctcaccggtcgcgcag gctctccgtc-3'(gaattc為EcoRⅠ酶切位點,前面g為保護性堿基)。
為了采用重疊PCR技術擴增TeryKR2基因,設計中間的將EryKR2酶中的PQQ突變成EryKR1酶中的LDD的正反向引物分別為TKR2-L:5'-cacgcgg cgggactgctcgacgacgtcgcgatcaac gacatggac-3';TKR2-R:5'-gtcgttgatcgcgacgtcgtcgagcagtcccgccgcgtgcaccac-3';PCR擴增體系和條件參考文獻[6]。
獲得的PCR特異片段產物按照微量瓊脂糖凝膠DNA回收試劑盒的產品說明書回收純化后,按照文獻[9],雙酶切連接到載體pET28a上,再轉化到大腸桿菌BL21中。從篩選到的陽性克隆中利用高純度質粒小提試劑盒提取出重組質粒,經武漢擎科公司測序后,再利用NCBI數據庫提供的核苷酸比對功能進行比對,檢測質粒中克隆的基因片段與目標基因的一致性。
1.2.2 重組酮還原酶的誘導表達和酶活測定 按照1%的接種量將構建的重組菌種子液接種到含100 μg/mL 卡拉霉素的LB液體培養基中,37℃、150 r/min培養3 h左右加入終濃度為1 mmol/L的 IPTG,繼續培養4 h[10]。離心(4℃,10 000 r/min,10 min)收集菌體沉淀,用0.1 mol/L的磷酸鉀緩沖液(pH7.0)洗滌重懸后,超聲波破碎(破胞強度60%,工作3 s間歇5 s,10 min)。離心(4℃,10 000 r/min,10 min)收集上清液(即粗酶液),測定其酶活、蛋白質含量及進行SDS-PAGE電泳分析。測定酶活的反應體系(3 mL):粗酶液20 μL、2 mmol/L NADPH 500 μL、2-甲基環己酮50 μL和0.1 mol/L磷酸鉀緩沖液(pH7.0)。該體系置于37℃反應30 min,期間在340 nm條件下測量NADPH的吸光值變化曲線,根據曲線斜率代入NADPH標準曲線擬合的公式(y=0.004 83x+ 0.004 53,R2=0.999 82,其中y為 340 nm處的吸光值,x為NADPH濃度(μmol/L)),計算NADPH含量變化情況。酶活單位U定義為在本實驗條件下,1 min催化氧化1 μmol NADPH到NADP+所需要的酶量。采用Bradford法測定蛋白質含量[9],根據標準品牛血清白蛋白的濃度與吸光值的標準曲線(y=0.005 79x-0.064,R2=0.997 01,y為595 nm處的吸光值,x為蛋白質含量(μg/mL)),計算上清液中蛋白質濃度。根據文獻[9]進行SDSPAGE分析。
1.2.3 雙重組菌耦合全細胞催化2-甲基環己酮的還原 雙重組菌耦合的發酵體系(10 mL):0.4 g E.coli BL21(pET28a-eryKR1)(或E.coli BL21(pET28a-ery-KR2)、E.coli BL21(pET28a- TeryKR1)、E.coli BL21(pET28a-TeryKR2)),0.1 g E.coli BL21(pET28a-gdh),0.1 mol/L葡 萄 糖,0.2 mmol/L NADPH,10 mmol/L 2-甲基環己酮,0.1 mol/L磷酸鉀緩沖液(pH6.0)。該體系置于30℃、120 r/min振蕩反應6 h。反應液離心(4℃,10 000 r/min,10 min)收集上清并用等體積乙酸乙酯萃取,得到的上層有機相采用Agilent6890型氣相色譜儀檢測分析底物和產物的濃度,并按照文獻[6]計算轉化率、產物收率及對映體過量值(e.e值)。色譜條件為:采用毛細管柱CYCLODEXB(30 m×0.25 mm×0.25 m),載氣為氮氣,進樣量為1 μL,FID檢測器。氣化溫度為250℃,檢測器溫度為300℃;色譜柱初始溫度90℃,維持4 min;升溫速度20℃,終溫為180℃,維持6 min;柱前壓為0.27 MPa,分流比為50∶1。
2 結果
2.1 重組質粒的構建及鑒定
eryKR1基因和eryKR2基因的突變位點兩側的PCR擴增產物TeryKR1-L、TeryKR1-R、TeryKR2-L和TeryKR2-R分別進行1.0%瓊脂糖凝膠電泳檢測,結果如圖1和圖2,可見TeryKR1-L和TeryKR2-L均位于1 000 bp附近,與兩個目標基因突變位點左側的序列長度分別為965 bp和893 bp吻合,而片段TeryKR1-R和TeryKR2-R都略高于500 bp,與兩個目標基因的突變位點右側的序列長度分別為550 bp和553 bp吻合。
突變型eryKR1基因(命名為TeryKR1)和突變型eryKR2基因(命名為TeryKR2)的重疊PCR產物,野生型eryKR1基因和eryKR2基因的PCR擴增產物,分別以1.0 %瓊脂糖凝膠進行電泳檢測,結果如圖3-圖6。產物片段的大小都位于1 000 bp和2 000 bp之間,與目標eryKR1基因和eryKR2基因的序列長度分別為1 461 bp和1 395 bp吻合。
將基因片段eryKR1、eryKR2、TeryKR1和Tery-KR2分別克隆到pET28a載體上,構建了重組質粒pET28a-eryKR1、pET28a-eryKR2、pET28a-TeryKR1和pET28a-TeryKR2。對獲得的重組質粒分別進行雙酶切鑒定(圖7),證實了4個重組質粒中克隆的基因片段均在1 000和2 000 bp之間,與目標片段的大小一致。含有這4個質粒的重組大腸桿菌分別命 名 為E.coli BL21(pET28a-eryKR1)、E.coli BL21(pET28a-eryKR2)、E.coli BL21(pET28a-TeryKR1)和E.coli BL21(pET28a-TeryKR2)。
圖1 eryKR1基因的突變位點兩側的PCR擴增產物的1.0%瓊脂糖凝膠電泳圖
圖2 eryKR2基因的突變位點兩側的PCR擴增產物的1.0%瓊脂糖凝膠電泳圖

圖3 突變型eryKR1基因(TeryKR1)的PCR產物的1.0%瓊脂糖凝膠電泳圖
圖4 突變型eryKR2基因(TeryKR2)的PCR產物的1.0%瓊脂糖凝膠電泳圖

圖5 野生型eryKR1基因的PCR產物的1.0%瓊脂糖凝膠電泳圖
圖6 野生型eryKR2基因的PCR產物的1.0%瓊脂糖凝膠電泳圖
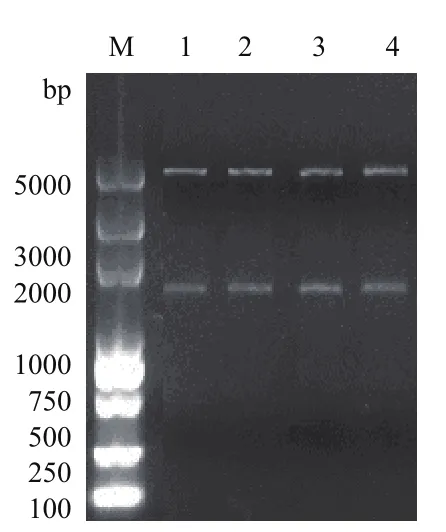
圖7 重組質粒雙酶切鑒定產物1.0%瓊脂糖凝膠電泳圖
利用NCBI數據庫提供的核苷酸比對功能,對重組質粒的測序結果進行序列分析,得出測序結果中克隆的野生型和突變型eryKR1基因、野生型和突變型eryKR2基因與登錄號為AM420293.1報道的糖多孢紅霉菌NRRL2338的基因組DNA中792 285-793 727 bp、796 692-798 068 bp的堿基序列的相似度分別達到100%、99%、100%和99%。
突變型eryKR1和eryKR2基因序列的核苷酸比對結果分別如圖8和圖9,突變型eryKR1基因中對應AM420293.1序列中的第793 209-793 218個堿基ctcgacgac(編碼的氨基酸殘基為LDD)已替換成測序結果中的ccgcagcag(編碼的氨基酸殘基為PQQ);而突變型eryKR2基因中對應AM420293.1序列中的第79 7547-797 556個堿基ccgcagcag(編碼的氨基酸序列為PQQ)已替換成測序結果中的ctcgacgac(編碼的氨基酸序列為LDD),說明質粒pET28a-TeryKR1和pET28a-TeryKR2中克隆的突變型eryKR1和eryKR2基因編碼的氨基酸序列,其中調控底物的立體選擇性還原的氨基酸殘基發生了LDD?PQQ互換。
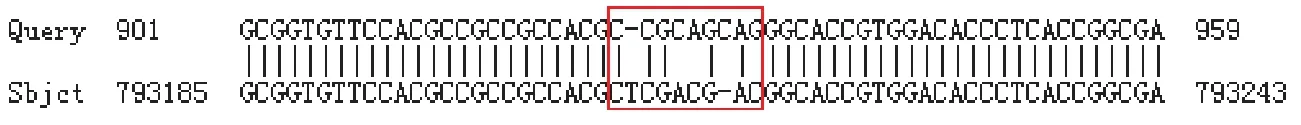
圖8 重組質粒pET28a-TeryKR1的核苷酸比對結果(截圖)

圖9 重組質粒pET28a-TeryKR2的核苷酸比對結果(截圖)
2.2 野生型和突變型酮還原酶在大腸桿菌中的異源表達情況
以E.coli BL21(pET28a)菌為陰性對照,SDSPAGE檢測經IPTG誘導后重組菌E.coli BL21(pET-28a-eryKR1)、E.coli BL21(pET28a-TeryKR1)、E.coli BL21(pET28a-eryKR2)和E.coli BL21(pET28a-TeryKR2)中目標蛋白的表達情況。如圖10和11所示,與陰性對照中的蛋白質條帶不同,經IPTG誘導后的重組菌在45.0 kD和66.2 kD之間都有酮還原酶的表達條帶(目標蛋白的相對分子量為57 kD,包括了pET28a載體表達的His tag片段)。
利用BandScan 5.0軟件分析這些重組菌的總可溶性蛋白的SDS-PAGE圖,可知,重組菌E.coli BL21(pET28a-TeryKR1)中目標蛋白質的表達量占全菌可溶性蛋白質的8.49%(圖10中的孔道4),E.coli BL21(pET28a-eryKR1)中EryKR1的表達量為5.66%(圖10中孔道2),而重組菌E.coli BL21(pET28aeryKR2)(圖11中孔道3)和E.coli BL21(pET28a-TeryKR2)(圖11中孔道5)中表達出來的EryKR2分別占全菌可溶性蛋白質的5.92%和5.04%。取這些孔道對應的重組菌進行后續的酶活和生物催化2-甲基環己酮不對稱還原實驗。
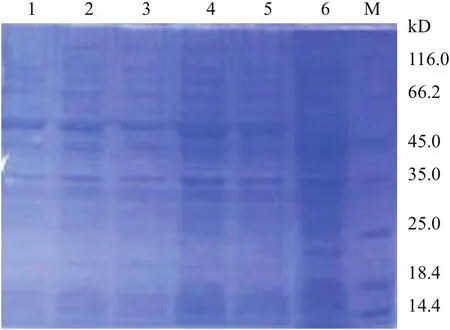
圖10 野生型和突變型酮還原酶EryKR1的SDS-PAGE檢測圖
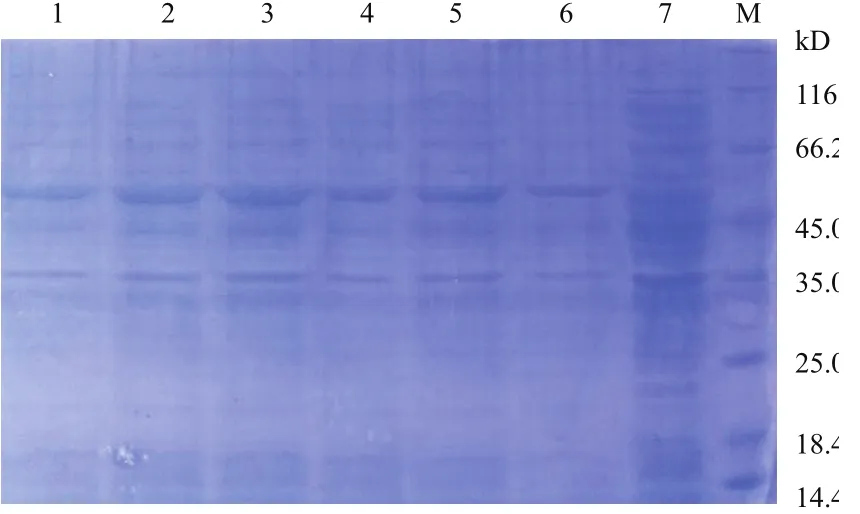
圖11 野生型和突變型酮還原酶EryKR2的SDS-PAGE檢測圖
2.3 野生型和突變型酮還原酶的活性
E.coli BL21(pET28a-eryKR1)、E.coli BL21(pET28a-TeryKR1)、E.coli BL21(pET28a-eryKR2)和E.coli BL21(pET28a-TeryKR2)的重組酮還原酶的粗酶液酶活分別為187.02 U/L、192.94 U/L、51.02 U/L和35.43 U/L,其中的蛋白質濃度分別為125.73 μg/mL、205.52 μg/mL、138.26 μg/mL和 115.68 μg/ mL,由此得出粗酶液的比酶活分別為1.49 U/mg、0.94 U/mg、0.37 U/mg和0.31 U/mg。根據野生型或突變型酮還原酶粗酶液的酶活和比酶活數據,可知控制立體選擇性還原的位點(motif)處的氨基酸序列LDD突變成PQQ(LDD→PQQ)的突變型EryKR1的比酶活,比起野生型EryKR1的略微有所下降,這說明突變對酶的活性有所影響。另外,與EryKR1相比,EryKR2當以2-甲基環己酮作為底物時,酶活較低,而PQQ→LDD的突變型EryKR2與野生型EryKR2的比酶活相較,變化不大。
2.4 雙重組菌耦合催化2-甲基環己酮不對稱還原實驗
氣相色譜檢測構建的四個重組大腸桿菌分別與E.coli BL21(pET28a-gdh)耦合還原2-甲基環己酮的轉化液(圖12),根據底物和產物的峰面積,利用外標法計算底物及產物的濃度,進而得出轉化率、產物產率及對映體過量值(e.e值),數據見表1,可知:與陰性對照的轉化液中沒有還原產物檢出不同(圖12-E),野生型EryKR1催化2-甲基環己酮不對稱還原的產物以順式2-甲基環己醇為主,產率為8.05%,e.e值為58.14%(圖12-A),而突變型EryKR1催化不對稱還原的產物主要為反式,產率為8.54%,e.e值為27.16%(圖12-B);野生型EryKR2催化2-甲基環己酮不對稱還原,產物以反式2-甲基環己醇為主,產率僅為4.00%,e.e值為8.89%(圖12-C),而突變型EryKR2催化不對稱還原的產物主要為順式,產率2.94%,e.e值為13.92%(圖12-D),說明EryKR1和EryKR2中分別發生LDD→PQQ和PQQ→LDD的突變時,催化2-甲基環己酮不對稱還原生成的主要產物的立體結構發生改變,這擴展了LDD 模式序列為酮還原酶中控制鏈式聚酮鏈立體選擇性還原的位點的理論,得出該位點也是控制2-甲基環己酮不對稱還原的模序。
另外,根據酮還原酶催化鏈式聚酮鏈立體選擇性還原的情況,EryKR1為B型酮還原酶,而EryKR2為A型KR,本研究結果還擴展了該理論,即EryKR1和EryKR2催化含環己基結構的化合物還原時形成的主要產物的立體結構也是不同的。
3 討論
對聚酮合成酶的特殊結構、催化機制的研究,有助于了解酶的催化、分子識別及蛋白質與蛋白質相互作用的分子機制[11],其中聚酮合成酶中的酮還原酶如何控制底物的立體選擇性還原的機制一直是研究的熱點。Ⅰ型聚酮合成酶的酮還原酶域(ketoreductase,KR)根據鏈式聚酮鏈立體選擇性還原的情況分為A型和B型,這兩種KR在第88-103和第134-149個氨基酸處的兩個模式序列(motif)有很大區別。B型KR如EryKR1,在第88-10個氨基酸處有一個LDD模式序列,而A型KR如 EryKR2,沒有這個模式。另外,在第134-149個氨基酸處,B型KR有個P144、N148,而A型KR中是W141。若這些位點發生了突變,酶域對(2R,S)-2-甲基-3-羰基戊酰N-乙酰半胱酰胺硫酸酯的立體選擇性會發生相應的改變,證實了這些酶域中存在有控制立體選擇性還原的模式序列[4,7,8]。另外,A型和B型KR的晶體結構分析顯示A型KR中的W殘基與B型KR中的LDD序列分別位于這兩種KR的活性中心裂縫的兩側,說明這兩種類型的KR中底物是從不同的方向被引至酶的活性中心,導致底物中的β-羰基以不同方向結合NADPH,從而生成不同立體結構的產物[12]。而且,與B型KR不同,A型KR的活性中心處于一種井然有序、隨時準備好催化的Lys-2’-O-核糖-Tyr的氫鍵結合的狀態。在底物和輔酶結合時,A型KR會關閉蓋子環(lid loop),限制底物只從一個方向進入活性中心,導致生成的產物具有特定的立體結構[13]。在這個過程中,酶的一些殘基能夠有助于底物的定位[14]。

圖12 雙重組菌耦合還原2-甲基環己酮的轉化液的氣相色譜檢測圖譜
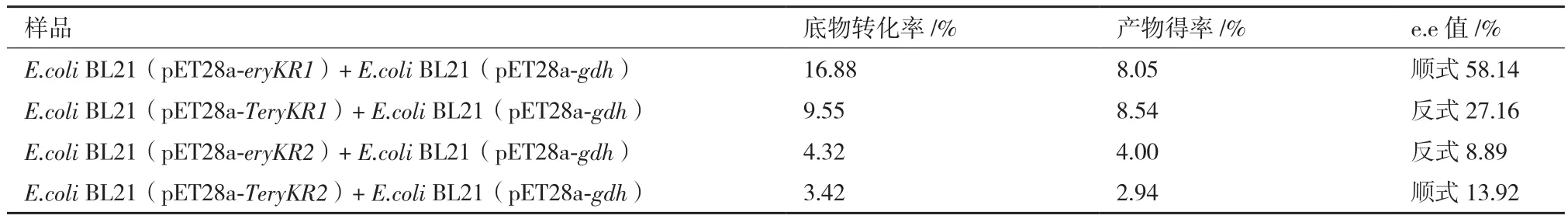
表1 雙重組菌耦合還原2-甲基環己酮的轉化液的氣相色譜檢測結果
聚酮合成酶的酮還原酶域可以還原的底物類型多種多樣,不僅能對類似天然底物——鏈式聚酮鏈的羰基底物有較好的還原能力,如(2R,S)-2-甲基-3-羰基戊酰N-乙酰半胱酰胺硫酯[3],而且還能以那些結構不同于鏈式聚酮鏈的化合物作為底物,如(9RS)-反式-1-萘烷酮[3]、脂環酮[5]等。但是,目前針對聚酮合成酶酮還原酶域的控制立體選擇性還原的機理研究,多以(2R,S)-2-甲基-3-羰基戊酰N-乙酰半胱酰胺硫酸酯、(9RS)-反式-1-萘烷酮為研究底物,對聚酮合成酶的酮還原酶域為何能結合潛手性取代環己酮以及如何控制此反應的立體選擇性機理還未見報道。本研究基于LDD模式序列可能也負責控制取代環己酮類底物的立體選擇性還原的假設,構建了分別異源表達聚酮合成酶模塊1的酮還原酶(EryKR1)、模塊2的酮還原酶(EryKR2)、LDD殘基替換為PQQ(LDD→PQQ)的EryKR1及PQQ替換為LDD(PQQ→LDD)的EryKR2的重組大腸桿菌,考察了野生型模塊1和模塊2的重組酮還原酶對2-甲基環己酮的不對稱還原能力,結果顯示在底物濃度為10 mmol/L的條件下,EryKR1的還原產物主要為順式-2-甲基環己醇,產率為8.05%,e.e值為58.14%,而EryKR2的還原產物主要為反式-2-甲基環己醇,產率為4.00%,e.e值為8.89%,說明即使以2-甲基環己酮為底物,模塊1和模塊2的酮還原酶的立體選擇性還原情況也是不同的。通過比較突變前后的重組EryKR1和EryKR2以2-甲基環己酮為底物的酶活及產物的立體結構,結果發現雖然突變對酶活的影響不大,但是突變后的重組EryKR1催化還原2-甲基環己酮的主要產物由突變前的順式-2-甲基環己醇轉變成反式-2-甲基環己醇,而突變后的重組EryKR2催化的主要還原產物的立體結構則由突變前的反式變成了順式,說明酮還原酶催化2-甲基環己酮還原的主要產物的立體結構變化與酶中LDD模式序列的突變之間存在關聯,得出LDD模序確實為酶中控制取代環己酮類底物立體選擇性還原的位點。
4 結論
本研究構建了分別異源表達聚酮合成酶模塊1的酮還原酶(EryKR1)、模塊2的酮還原酶(EryKR2)、LDD殘基替換為PQQ(LDD→PQQ)的EryKR1和PQQ替換為LDD(PQQ→LDD)的EryKR2的重組大腸桿菌,比較突變型和野生型重組大腸桿菌還原2-甲基環己酮的主要產物的立體結構,得出LDD→PQQ的突變使得EryKR1的主要還原產物從順式-2-甲基環己醇轉變成反式-2-甲基環己醇,而PQQ→LDD的突變可使EryKR2的主要還原產物2-甲基環己醇的立體結構由反式變成順式,確定了聚酮合成酶的酮還原酶的LDD模式序列為控制2-甲基環己酮立體選擇性還原的位點。
[1] Long JX, Shu SY, Wu QY, et al. Selective cyclohexanol production from the renewable lignin derived phenolic chemicals catalyzed by Ni/MgO[J]. Energy Conversion and Management, 2015, 105(15):570-577.
[2] Conti RMD, Porto ALM, Augusto J, et al. Microbial reduction of cyclohexanones[J]. Journal of Molecular Catalysis B:Enzymatic, 2001, 11(4-6):233-236.
[3] Siskos AP, Baerga-Ortiz A, Bali S, et al. Molecular basis of Celmer’s rules:stereochemistry of catalysis by isolated ketoreductase domains from modular polyketide synthases[J]. Chemistry & Biology, 2005, 12(10):1145-1153.
[4] Piasecki SK, Taylor CA, Detelich JF, et al. Employing modular polyketide synthase ketoreductases as biocatalysts in the preparative chemoenzymatic syntheses of diketide chiral building blocks[J].Chemistry & Biology, 2011, 18(10):1331-1340.
[5] Bali S, O’Hare HM, Weissman KJ. Broad substrate specificity of ketoreductases derived from modular polyketide synthases[J]. ChemBiochem, 2006, 7(3):478-484.
[6] 李凌凌, 呂早生, 關海燕, 等. 糖多孢紅霉菌酮還原酶域在羰基還原中的應用[J]. 華中科技大學:自然科學版, 2011, 39(2):72-75.
[7] Baerga-Ortiz A, Popovic B, Siskos AP, et al. Directed mutagenesis alters the stereochemistry of catalysis by isolated ketoreductase domains from the erythromycin polyketide synthase[J]. Chemistry & Biology, 2006, 13(3):277-285.
[8] O’Hare HM, Baerga-Ortiz A, Popovic B, et al. High-throughput mutagenesis to evaluate models of stereochemical control in ketoreductase domains from the erythromycin polyketide synthase[J]. Chemistry & Biology, 2006, 13(3):287-296.
[9] 楊忠華, 左振宇, 黃皓, 等. 生物工程專業實驗[M]. 北京:化學工業出版社, 2014:65-73.
[10] 李凌凌, 呂早生, 左振宇, 等. 嗜酸喜溫硫桿菌硫加氧還原酶基因的克隆、表達及酶活性研究[J]. 生物技術通報, 2015, 31(11):186-194.
[11] Shen B. Polyketide biosynthesis beyond the typeⅠ, Ⅱ and Ш polyketide synthase paradigms[J]. Current Opinion in Chemical Biology, 2003, 7(2):285-295.
[12] Keatinge-Clay AT. A tylosin ketoreductase reveals how chirality is determined in polyketides[J]. Chemistry & Biology, 2007, 14(8):898-908.
[13] Bonnet SA, Whicher JR, Papireddy K, et al. Structural and stereochemical analysis of a modular polyketide synthase ketoreductase domain required for the generation of a cisalkene[J]. Chemistry & Biology, 2013, 20(6):772-783.
[14] Zheng J, Taylor CA, Piasecki SK, et al. Structural and functional analysis of A-type ketoreductases from the amphotericin modular polyketide synthase[J]. Structure, 2010, 18(8):913-922.
(責任編輯 李楠)
Mutation of Amino Acid Motif Involved in Stereoselectivity by Ketoreductases and Analysis of Its Product
LI Ling-ling Lü Zao-sheng ZUO Zhen-yu YANG Zhong-hua LIU Yao-ning SONG Cai-wei
(School of Chemistry and Chemical Engineering,Wuhan University of Science and Technology,Wuhan 430081)
In order to identify whether LDD motif in ketoreductase domain of polyketide synthase from Saccharopolyspora erythraea(EryKR)can account for the stereocontrol of 2-methylcyclohexanone reduction,we constructed the 4 recombinants,Escherichia coli BL21(pET28a-eryKR1)with heterologous expressing ketoreductase in the first module(EryKR1)of polyketide synthase, E.coli BL21(pET28aeryKR2)with heterologous expressing ketoreductase in the second module(EryKR2)of polyketide synthase,E.coli BL21(pET28a-TeryKR1)of the site-mutated EryKR1 in which the nucleotide sequence coding for amino acid residues LDD replaced by PQQ,and E.coli BL21(pET28a-TeryKR2)of the site-mutated EryKR2 while PQQ replaced by LDD. SDS-PAGE demonstrated that ketoreductases were expressed in these 4 recombinants after induction by IPTG. Specific activity of crude enzyme was 1.49 U/mg,0.37 U/mg,0.94 U/mg and 0.31 U/mg,respectively. Gas chromatography analyses of 2-methylcyclohexanone reduction catalyzed by 4 recombinants showed that mutated recombinant E.coli BL21(pET28a-TeryKR1)mainly reduced 2-methylcyclohexanone to trans-2-methylcyclohexanol,unlike the main product of cis-2-methylcyclohexanol catalyzed by wild-type recombinant Escherichia coli BL21(pET28a-eryKR1). Furthermore,main reduction product of mutant E.coli BL21(pET28a-TeryKR2)was cis-2-methylcyclohexanol,rather than the trans-2-methylcyclohexanol by wild-type recombinant E.coli BL21(pET28a-eryKR2),confirming the key role of LDD motif in controlling the stereoselectivity of 2-methylcyclohexanone reduction.
polyketide synthase;ketoreductase;2-methylcyclohexanone;mutation;stereoselectivity
10.13560/j.cnki.biotech.bull.1985.2016-1195
2017-01-05
國家自然科學基金資助項目(21376184),湖北省科技廳基金項目(2009CDA006),武漢科技大學青年科技骨干培育計劃項目(2016XZ014),武漢科技大學大學生科技創新基金研究項目(16ZRA044)
李凌凌,女,博士,副教授,研究方向:生物催化及微生物浸礦技術;E-mail:moonletsmile@163.com

