基于16S rRNA基因的食源性致病菌實(shí)時(shí)熒光定量PCR檢測(cè)研究
劉玲雪++孫紅煒++李凡++徐曉輝++高瑞++楊淑珂++路興波
摘要:本研究以16S rRNA基因作為靶基因,利用TaqMan探針技術(shù)建立了針對(duì)志賀氏菌(Shigella)、沙門氏菌(Salmonella)和金黃色葡萄球菌(Staphylococcus aureus)三種主要食源性致病菌的多重實(shí)時(shí)熒光定量PCR檢測(cè)技術(shù)。結(jié)果表明,建立的熒光定量PCR檢測(cè)體系特異性良好、靈敏度高、簡(jiǎn)便快捷,大大縮短了檢測(cè)時(shí)間,在食源性致病菌快速篩查方面具有良好的應(yīng)用前景。
關(guān)鍵詞:食源性致病菌;16S rRNA基因;多重實(shí)時(shí)熒光定量PCR;TaqMan探針
中圖分類號(hào):TS207.4文獻(xiàn)標(biāo)識(shí)號(hào):A文章編號(hào):1001-4942(2017)06-0126-09
AbstractIn this study, a multiplex real-time quantitative PCR detection technology for the three food-borne pathogens, Shigella, Salmonella and Staphylococcus aureus, had been established utilizing the TaqMan probe and 16S rRNA gene as a target gene. The results indicated that the established multiplex real-time quantitative PCR detection system had good specificity and sensitivity, was simple and efficient, and greatly reduced the detection time, so it had a good prospect in the rapid screening for food-borne pathogens.
KeywordsFood-borne pathogen; 16S rRNA gene; Multiplex real-time quantitative PCR; TaqMan probe
食品種類的日益增多在豐富人們消費(fèi)體驗(yàn)的同時(shí),也帶來了更多的食品安全事件。食品安全事件造成的經(jīng)濟(jì)損失十分驚人。據(jù)相關(guān)部門統(tǒng)計(jì),2015年上半年因各類食品安全事件,經(jīng)濟(jì)損失至少300億元,死傷人數(shù)近萬人[1]。據(jù)統(tǒng)計(jì),在全球各類疾病中,食源性疾病的發(fā)病率位居前列,因食源性疾病而導(dǎo)致死亡的人數(shù)每年約有220萬[2]。據(jù)世界衛(wèi)生組織(WHO)報(bào)道,影響食品安全最主要的因素之一是由病原微生物引起的食源性疾病。通過對(duì)我國(guó)食源性疾病監(jiān)測(cè)系統(tǒng)個(gè)案報(bào)告的資料分析可知,我國(guó)發(fā)生的食品安全事件中有將近一半是由食源性致病菌所導(dǎo)致的[3,4],而有效預(yù)防和控制食源性疾病的重要環(huán)節(jié)是快速檢測(cè)和鑒別食源性致病菌。本研究以食品中常見的三種致病性細(xì)菌——志賀氏菌(Shigella)、沙門氏菌(Salmonella)和金黃色葡萄球菌(Staphylococcus aureus)為對(duì)象,研究建立針對(duì)這三種致病菌的多重實(shí)時(shí)熒光定量PCR快速檢測(cè)技術(shù),這對(duì)及時(shí)切斷病原菌傳播途徑以達(dá)到減少食源性疾病發(fā)生的目的具有十分重要的意義。
1材料與方法
1.1材料
1.1.1供試菌株本研究所用菌株共9株,其中福氏志賀氏菌株購(gòu)自北納創(chuàng)聯(lián)生物技術(shù)有限公司;金黃色葡萄球菌、鼠傷寒沙門氏菌及6種對(duì)照菌株(大腸桿菌、黃單胞菌、枯草桿菌、產(chǎn)氣桿菌、變形桿菌、八疊球菌)均由山東師范大學(xué)生命科學(xué)學(xué)院李世國(guó)老師饋贈(zèng)。將以上菌種分別接種于牛肉膏蛋白胨培養(yǎng)基試管斜面上,存于4℃冰箱中備用。
1.1.2主要儀器設(shè)備臺(tái)式冷凍離心機(jī)(MIKRO 22R),德國(guó)Hettich公司;BioPhotometer Plus儀,德國(guó)Eppendorf公司;PCR八聯(lián)管,美國(guó)應(yīng)用生物系統(tǒng)公司;Step One Plus實(shí)時(shí)熒光定量PCR系統(tǒng),美國(guó)應(yīng)用生物系統(tǒng)公司;微量移液器(0.5~10 μL,10~100 μL),德國(guó)Eppendorf公司。
1.2試驗(yàn)方法
1.2.1細(xì)菌基因組DNA的提取分別采用三種不同的細(xì)菌DNA提取方法,即普通的CTAB法、CWBIO Bacteria Genomic DNA Kit和TIANGEN細(xì)菌基因組DNA提取試劑盒,并對(duì)其提取結(jié)果進(jìn)行比較,選取效果最好的提取方法。最終選用TIANGEN細(xì)菌基因組DNA提取試劑盒法。
1.2.2引物與探針的設(shè)計(jì)根據(jù)GenBank中三種細(xì)菌16S rRNA基因序列,用DNAMAN V6中文版進(jìn)行同源性比對(duì)分析,選擇其高度保守的區(qū)域,根據(jù)引物設(shè)計(jì)的原則,運(yùn)用軟件Primer Premier 5.0設(shè)計(jì)一對(duì)兼并引物[5]。
利用DNAMAN V6軟件對(duì)Shigella、Salmonella和Staphylococcus aureus進(jìn)行同源性序列的分析比對(duì),選取三種菌雙鏈DNA序列中最特異的區(qū)段作為靶序列,用Primer Express V 3.0軟件設(shè)計(jì)各菌的TaqMan探針。交由生工生物工程技術(shù)服務(wù)公司(上海)合成。選擇各條探針的熒光標(biāo)記,5′端報(bào)告基團(tuán)可以為FAM、VIC、NED等,3′端的熒光淬滅基團(tuán)用MGB、BHQ1等。引物和探針見表1。
1.2.3多重?zé)晒舛縋CR反應(yīng)體系的建立采用50 μL反應(yīng)體系,將單重?zé)晒舛縋CR各組分濃度(表2)加倍,將3株菌的探針和模板DNA放入同一個(gè)PCR反應(yīng)管中,成為三重?zé)晒舛縋CR反應(yīng)體系(表3)。擴(kuò)增程序?yàn)?5℃預(yù)變性30 s;95℃變性1 s,60℃退火20 s,40個(gè)循環(huán)。
1.3熒光定量PCR反應(yīng)體系性能評(píng)價(jià)
1.3.1特異性本試驗(yàn)利用建立的多重?zé)晒舛縋CR反應(yīng)體系對(duì)供試3株細(xì)菌及另外6株細(xì)菌(大腸桿菌、黃單胞菌、枯草桿菌、八疊球菌、產(chǎn)氣桿菌、變形桿菌)進(jìn)行熒光定量PCR試驗(yàn),對(duì)該P(yáng)CR體系的特異性進(jìn)行評(píng)價(jià)。
1.3.2靈敏度用BioPhotometer Plus儀測(cè)定提取的細(xì)菌基因組DNA原液濃度,然后稀釋到100 ng/μL,在此基礎(chǔ)上按10倍梯度稀釋,DNA濃度從10 ng/μL到10-5 ng/μL共設(shè)7個(gè)梯度。按照建立的熒光定量PCR體系進(jìn)行試驗(yàn),確定該體系能夠檢測(cè)到的最低DNA濃度,評(píng)價(jià)體系的靈敏度。
1.4模擬樣品試驗(yàn)
1.4.1模擬水樣和牛奶樣本的制備分別挑取3種供試菌單菌落接種到LB瓊脂平板,37℃過夜培養(yǎng),取菌苔至5 mL無菌PBS中制備菌懸液,用無菌PBS 10倍系列稀釋菌懸液至10-8。取10-5、10-6、10-7、10-8四個(gè)梯度各100 μL涂LB瓊脂平板,重復(fù)3次,37℃過夜培養(yǎng),利用平板計(jì)數(shù)法推算原菌懸液的準(zhǔn)確濃度。對(duì)經(jīng)菌落計(jì)數(shù)的菌懸液用無菌PBS 10倍系列稀釋,取濃度為n×107、n×106、n×105、n×104、n×103、n×102、n×101 cfu/mL(1≤n<10,視菌體菌落計(jì)數(shù)結(jié)果而定)的菌液各1 mL分別加入到50 mL離心管中,離心管中加入24 mL飲用水或市售巴斯德消毒的純牛奶(伊利),混勻,得到濃度為n×107~ n×101 cfu/25mL的樣本。
1.4.2模擬水樣本的檢測(cè)針對(duì)各梯度飲用水模擬樣本,2 000×g離心10 min,沉淀小心地重懸于1 mL PBS中,轉(zhuǎn)移到1.5 mL離心管中,13 000×g 離心5 min,棄上清,沉淀用細(xì)菌基因組DNA提取試劑盒提取DNA,最后用50 μL純凈水洗脫,取1 μL為模板,進(jìn)行單重、多重?zé)晒舛縋CR檢測(cè)。
1.4.3模擬牛奶樣本的檢測(cè)針對(duì)各梯度牛奶模擬樣本,每個(gè)離心管中加入2.4 mL triton X-100、1%(W/V)的固體粉末狀胰蛋白酶,漩渦震蕩以徹底混勻,37℃培養(yǎng)30 min,2 000×g離心10 min,沉淀小心地重懸于1 mL PBS中,轉(zhuǎn)移到1.5 mL離心管中,13 000×g離心5 min,棄上清,沉淀用細(xì)菌基因組DNA提取試劑盒提取DNA,最后用50 μL純凈水洗脫。取1 μL作為模板,進(jìn)行單重、多重實(shí)時(shí)熒光定量PCR檢測(cè)。
2結(jié)果與分析
2.1細(xì)菌基因組DNA提取結(jié)果
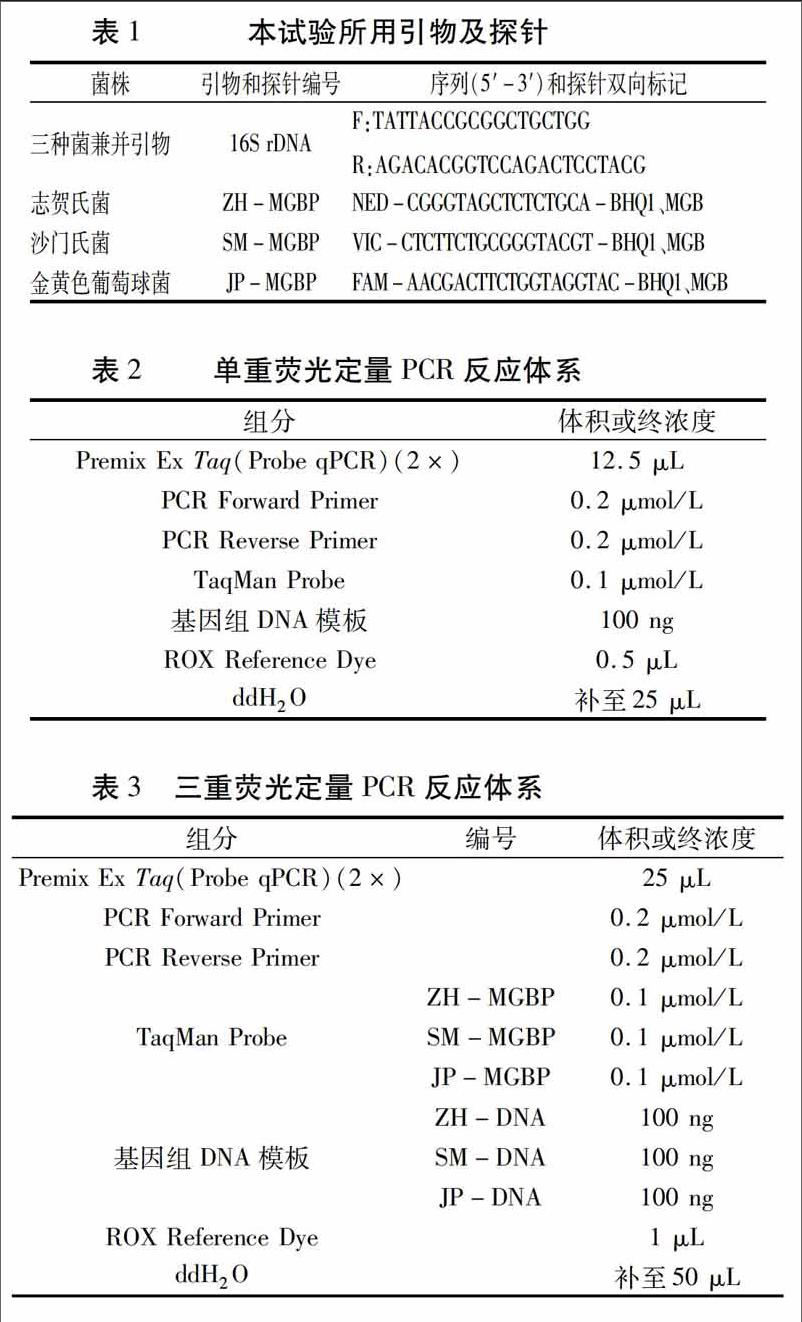
用普通的CTAB法、CWBIO Bacteria Genomic DNA Kit和TIANGEN細(xì)菌基因組DNA提取試劑盒分別提取志賀氏菌、沙門氏菌和金黃色葡萄球菌的基因組DNA,取5 μL進(jìn)行瓊脂糖凝膠電泳分析。由圖1可知,普通的CTAB法提取效果較差,志賀氏菌及金黃色葡萄球菌出現(xiàn)微弱的雜帶,且目的條帶不明亮;CWBIO Bacteria Genomic DNA Kit提取效果也不理想,目的條帶單一但不明亮;TIANGEN細(xì)菌基因組DNA提取試劑盒提取的目的條帶單一且明亮,效果好。故選擇TIANGEN細(xì)菌基因組DNA提取試劑盒來提取病原菌的基因組DNA。
2.2多重實(shí)時(shí)熒光定量PCR反應(yīng)體系建立
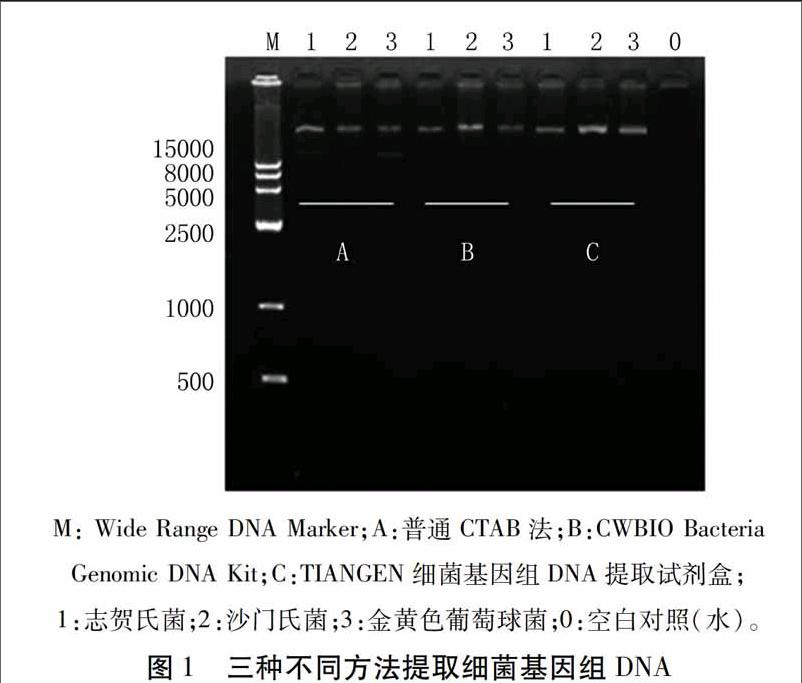
將3株菌的探針和模板DNA放入同一個(gè)PCR反應(yīng)管中,成為三重實(shí)時(shí)熒光定量PCR反應(yīng)體系。在多重?zé)晒舛縋CR反應(yīng)中,個(gè)別菌株的擴(kuò)增曲線可能會(huì)發(fā)生改變,這是由于探針數(shù)目多時(shí)使體系的熒光本底值上升,彼此間產(chǎn)生干擾,但影響不大,只要保證三條擴(kuò)增曲線同時(shí)起峰,曲線間的間距拉大,達(dá)到便于觀察和結(jié)果準(zhǔn)確的目的即可。本研究建立的三重實(shí)時(shí)熒光定量PCR體系達(dá)到了該目的要求,結(jié)果準(zhǔn)確美觀,擴(kuò)增結(jié)果如圖2所示。
2.3實(shí)時(shí)熒光定量PCR性能評(píng)價(jià)
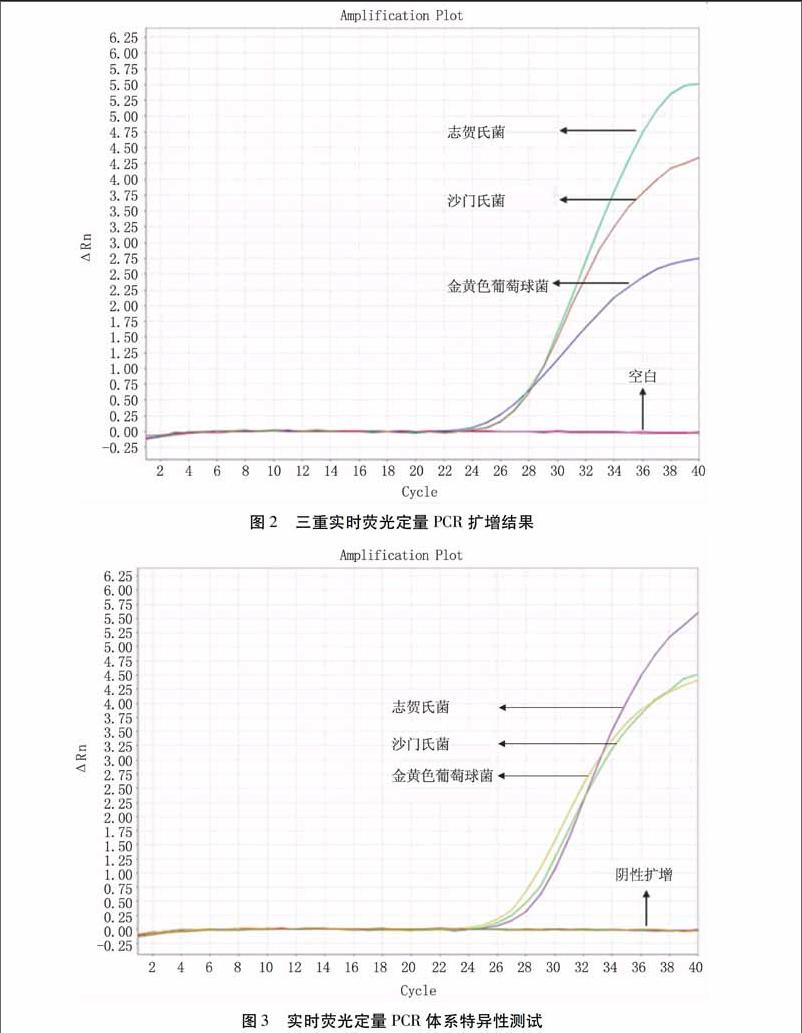
2.3.1特異性實(shí)時(shí)熒光定量PCR試驗(yàn)利用引物和探針的雙重定位作用,使其能專一性地檢測(cè)目標(biāo)菌。利用生物信息學(xué)手段設(shè)計(jì)引物和探針是非常關(guān)鍵的一步,而通過試驗(yàn)對(duì)大量的近緣及遠(yuǎn)緣菌株進(jìn)行特異性驗(yàn)證才能真正保證該體系檢測(cè)的準(zhǔn)確性。由圖3可知,只有目標(biāo)菌株有陽(yáng)性擴(kuò)增信號(hào)產(chǎn)生,其他細(xì)菌和空白對(duì)照均檢測(cè)不到信號(hào)值或信號(hào)值很低,可以認(rèn)定是陰性擴(kuò)增或輕微的污染所致。說明本研究所選引物和探針特異性良好。
2.3.2靈敏度將三種菌的基因組DNA按照10倍梯度稀釋,共設(shè)7個(gè)梯度:10、1、10-1、10-2、10-3、10-4、10-5 ng/μL。結(jié)果(圖4)顯示該熒光定量PCR體系在基因組DNA水平上的檢測(cè)靈敏度可達(dá)到10-4 ng/μL。
2.4模擬樣品檢測(cè)結(jié)果
待測(cè)樣品的處理問題是試驗(yàn)成功與否的關(guān)鍵點(diǎn)和難點(diǎn)。本研究檢測(cè)的是常見的志賀氏菌、沙門氏菌和金黃色葡萄球菌三種食源性致病菌,因此我們選擇飲用水、牛奶兩種樣品作為檢測(cè)對(duì)象。
2.4.1菌液計(jì)數(shù)結(jié)果經(jīng)菌落計(jì)數(shù),結(jié)果見表4,志賀氏菌、沙門氏菌、金黃色葡萄球菌原菌懸液
濃度分別為2.57×109、1.37×1010、5.23×109 cfu/mL。先將沙門氏菌的菌液(1.37×1010 cfu/mL)用PBS稀釋成1.37×109 cfu/mL,再10倍梯度稀釋至1.37×101 cfu/mL;用PBS將志賀氏菌和金黃色葡萄球菌的菌液分別10倍梯度稀釋至2.57×101、5.23×101 cfu/mL。取107~101梯度菌液用作下述試驗(yàn)。
2.4.2模擬水樣的單重實(shí)時(shí)熒光定量PCR靈敏度檢測(cè)結(jié)果以上述梯度稀釋菌液污染飲用水提取的DNA為模板,測(cè)試反應(yīng)體系的靈敏度。結(jié)果如圖5,該反應(yīng)體系在101梯度時(shí)仍有擴(kuò)增信號(hào)出現(xiàn),即最低分別能檢測(cè)到1.37×101 cfu/25mL水樣(沙門氏菌)、2.57×101 cfu/25mL水樣(志賀氏菌)、5.23×101 cfu/25mL水樣(金黃色葡萄球菌)的細(xì)菌。
2.4.3模擬牛奶樣品的單重實(shí)時(shí)熒光定量PCR靈敏度檢測(cè)結(jié)果以上述梯度稀釋菌液污染市售純牛奶(伊利)提取的DNA為模板,測(cè)試實(shí)時(shí)熒光反應(yīng)體系的靈敏度。結(jié)果如圖6,該反應(yīng)體系在102梯度時(shí)仍有擴(kuò)增信號(hào)出現(xiàn),即最低分別能檢測(cè)到2.57×102 cfu/25mL牛奶樣(志賀氏菌)、1.37×102 cfu/25mL牛奶樣(沙門氏菌)、5.23×102 cfu/25mL牛奶樣(金黃色葡萄球菌)的細(xì)菌。
2.4.4模擬水樣多重實(shí)時(shí)熒光定量PCR檢測(cè)結(jié)果在上述模擬水樣單重實(shí)時(shí)熒光定量PCR基礎(chǔ)上,對(duì)三種致病菌進(jìn)行多重實(shí)時(shí)熒光定量PCR檢測(cè),志賀氏菌、沙門氏菌及金黃色葡萄球菌分別選用菌液濃度為2.57×107、1.37×107、5.23×107 cfu/25mL水樣作為模板。結(jié)果如圖7,三種致病菌在CT值25左右均出現(xiàn)擴(kuò)增信號(hào),空白對(duì)照(水)未出現(xiàn)擴(kuò)增信號(hào)。說明該體系特異性良好。
2.4.5模擬牛奶樣品多重實(shí)時(shí)熒光定量PCR檢測(cè)結(jié)果在上述模擬牛奶樣品單重實(shí)時(shí)熒光PCR基礎(chǔ)上,對(duì)三種致病菌建立多重實(shí)時(shí)熒光PCR體系,志賀氏菌、沙門氏菌及金黃色葡萄球菌分別選用菌液濃度為2.57×107、1.37×107、5.23×107 cfu/25mL牛奶樣作為模板。結(jié)果如圖8,三種致病菌在CT值28左右均出現(xiàn)擴(kuò)增信號(hào),空白對(duì)照(未污染牛奶)未出現(xiàn)擴(kuò)增信號(hào)。說明該體系特異性良好。
3討論與結(jié)論
實(shí)時(shí)熒光定量PCR(real-time quantitative polymerase chain reaction)是指在PCR指數(shù)擴(kuò)增期間通過連續(xù)檢測(cè)熒光信號(hào)的強(qiáng)弱來即時(shí)測(cè)定特異性產(chǎn)物的量。其代表性方法TaqMan熒光探針技術(shù)近些年在食品致病菌檢測(cè)方面表現(xiàn)尤為突出,其優(yōu)點(diǎn)在于:采用序列特異性探針,增強(qiáng)了檢測(cè)特異性;可以在單管中檢測(cè)多個(gè)靶標(biāo),適合多重反應(yīng)。例如劉生峰等[6]利用TaqMan探針熒光PCR建立腸出血性Escherichia coli O157∶H7的檢測(cè)方法;國(guó)內(nèi)學(xué)者劉渠等[7]利用相同基因建立的定量PCR體系,可檢出最低 45個(gè)拷貝的細(xì)菌DNA,特異性和重復(fù)性良好。
而本研究采用的新型TaqMan MGB探針比普通TaqMan探針更短,從而降低了成本,另外在3′端標(biāo)記非淬滅熒光分子MGB使熒光本底降低的同時(shí),也使探針雜交穩(wěn)定性極大增加,所以它的結(jié)果更為精確[8]。
本研究針對(duì)食品中常見的三種致病菌Shigella、Salmonella和Staphylococcus aureus建立多重實(shí)時(shí)熒光定量PCR檢測(cè)體系,其中引物的設(shè)計(jì)直接關(guān)系到PCR反應(yīng)的特異性,且對(duì)多重PCR來說,引物對(duì)的復(fù)雜多樣,會(huì)造成引物配對(duì)、擴(kuò)增效率、反應(yīng)條件等諸多方面難以兼顧[9]。本研究在查閱大量關(guān)于特異性保守基因的基礎(chǔ)上,選擇了16S rRNA基因作為其共同的靶基因并設(shè)計(jì)了一對(duì)兼并引物[10],根據(jù)Tm值的差異分析確定可以將其作為熒光定量PCR擴(kuò)增的靶基因和特異性引物,為建立多重?zé)晒舛縋CR檢測(cè)方法奠定了基礎(chǔ)。另外,我們首先利用單重?zé)晒舛縋CR反應(yīng)對(duì)反應(yīng)條件進(jìn)行優(yōu)化,保障反應(yīng)體系具有較高的靈敏度,提高檢測(cè)效率,節(jié)約檢測(cè)成本,從而為食品中多種致病菌的同時(shí)快速檢測(cè)奠定基礎(chǔ)。
為了充分驗(yàn)證該檢測(cè)體系的特異性,本研究采用了陽(yáng)性菌株和干擾菌株的對(duì)照法,從試驗(yàn)結(jié)果來看,建立的檢測(cè)方法具有良好的特異性,沒有出現(xiàn)假陽(yáng)性和假陰性,這也說明了靶基因選擇的科學(xué)性及其引物探針設(shè)計(jì)的正確性。本課題還對(duì)體系的靈敏度進(jìn)行了驗(yàn)證,試驗(yàn)結(jié)果顯示該體系在基因組DNA水平上的檢測(cè)靈敏度可達(dá)到10-4 ng/μL,具有很高的靈敏度。
參考文獻(xiàn):
[1]楊建秀. 食品安全與食源性疾病[J]. 熱帶醫(yī)學(xué)雜志, 2004, 4(2): 201-203.
[2]WHO. Food safety, a world wide challenge[R]. Food Chain, 2001.
[3]龐璐, 張哲, 徐進(jìn). 2006—2010年我國(guó)食源性疾病暴發(fā)簡(jiǎn)介[J]. 中國(guó)食品衛(wèi)生雜志, 2011(6): 560-563.
[4]王茂起, 劉秀梅, 王竹天. 中國(guó)食品污染監(jiān)測(cè)體系的研究[J]. 中國(guó)食品衛(wèi)生雜志, 2006(6): 491-497.
[5]王如景. 三種食源性致病菌多重PCR快速檢測(cè)方法的研究[D]. 河北: 河北農(nóng)業(yè)大學(xué), 2012.
[6]劉生峰, 肖進(jìn)文, 劉利成,等. TaqMan探針熒光PCR檢測(cè)腸出血性大腸埃希菌O157∶ H7方法的建立[J]. 微生物學(xué)雜志, 2012, 32(5): 6-10.
[7]劉渠, 廖靈靈, 徐亞軍,等. 熒光定量PCR檢測(cè)金黃色葡萄球菌腸毒素A方法研究[J]. 中國(guó)衛(wèi)生檢驗(yàn)雜志, 2010(4): 788-789.
[8]伊鋆, 蔡雪鳳. TaqMan熒光探針技術(shù)在食源性致病菌檢測(cè)中的應(yīng)用[J]. 食品工業(yè)科技, 2013(7): 17.
[9]Harmsen D, Karch H. 16S rRNA for diagnosing pathogens: a living tree[J]. ASM News-American Society for Microbiology, 2004, 70(1): 19-24.
[10]李凡, 許恒毅, 李福來. 多重PCR技術(shù)在食源性致病菌檢測(cè)中應(yīng)用的研究進(jìn)展[J]. 食品工業(yè)科技, 2015, 36(21): 372-375.

