不同產地花臉香蘑菌絲體及子實體的RAPD分析
張麗麗+王蘭英+唐萌+楊海茹+胡玲+蔡佳旋+黃艷怡


摘要:采用RAPD分析了5個不同產地的花臉香蘑(Lepista sordida)菌絲體和子實體的DNA多樣性。用12種引物分別對5種樣品進行PCR擴增,從中選出8種引物擴增后的結果進行數據分析,計算樣品間的遺傳相似系數(GS值),進而根據遺傳相似系數矩陣,利用UPGMA法進行聚類分析。結果表明,花臉香蘑菌絲體和子實體分為兩個類群,又將3個不同產地的菌絲體分為2個亞群,為花臉香蘑后期研究提供參考。
關鍵詞:花臉香蘑(Lepista sordida);DNA多樣性;聚類分析
中圖分類號:S646 文獻標識碼:A 文章編號:0439-8114(2017)14-2758-03
DOI:10.14088/j.cnki.issn0439-8114.2017.14.041
Abstract: Using RAPD analysis of the five different regions of mycelia and fruiting body of Lepista sordida DNA diversity. Respectively with 12 primers for PCR amplification, five samples selected by 8 primers' amplification result for data analysis, the genetic similarity coefficient between the sample value(GS),and then according to the genetic similarity coefficient matrix, the cluster analysis using UPGMA method. The results showed that RAPD mycelia and fruiting body Lepista sordida method is divided into two groups, and the mycelium of three different regions can be divided into two subsets, it can be seen that the genetic relationship provide reference for later study of Lepista sordida.
Key words: Lepista sordida; DNA diversity; clustering analysis
花臉香蘑(Lepista sordida)又名紫香花臉蘑,隸屬于傘菌目(Agaricales)白蘑科(Tricholomataceae)香蘑屬(Lepista)[1],花臉香蘑的子實體營養豐富,風味獨特,是一種具有較高開發價值的食用菌[2]。但人工栽培的花臉香蘑存在子實體易碎、不易運輸等缺陷,有待于通過雜交等育種方式進行改良,這就需要對花臉香蘑種質資源進行廣泛采集和科學鑒定,以避免因形態相似或分離失誤而出現偏差[3]。
本研究利用RAPD技術,鑒定花臉香蘑菌株,克服傳統的標記手段和形態分類學的缺點,為花臉香蘑的深入研究提供了強有力的工具和手段,其應用和發展前景廣闊。
1 材料與方法
1.1 材料
花臉香蘑1號菌種購于蘇州北納創聯生物技術有限公司,編號為BNCC117075,采集地為山東;花臉香蘑2號菌種購于蘇州北納創聯生物技術有限公司,編號為BNCC110924,采集地為黑龍江;花臉香蘑3號菌種由吉林大學珠海學院化學與藥學系實驗中心提供;花臉香蘑4號子實體產地為內蒙古大興安嶺阿爾山林區;花臉香蘑5號子實體產地為吉林長白山地區,以上5種菌類原始樣品,樣品編號1~5。
1.2 基因組DNA的提取
采用CTAB法提取樣品基因組DNA,具體步驟參考Zhang等[4]方法進行。
1.3 RAPD分析
1.3.1 RAPD反應體系和擴增條件 PCR擴增體系(20 μL)為DNA模板2 μL、Primer 1 μL、DNA Mix 10 μL、去離子水7 μL。PCR擴增程序為94 ℃預變性 3 min;94 ℃變性30 s,36 ℃退出50 s,72 ℃延伸1.5 min,40個循環;72 ℃延伸10 min,4 ℃保存[5]。
1.3.2 RAPD擴增產物的檢測 利用表1中的12種引物對5個樣品進行PCR擴增,PCR產物在2.0%的瓊脂糖凝膠電泳中檢測。
1.4 數據統計分析
PCR擴增產物電泳位置在凝膠某個相同遷移率位置上有DNA條帶記為1,無DNA條帶記為0,如表2所示,形成RAPD的表型數據矩陣,可以將PCR擴增的結果數字化,便于后續的分析。NTSYS軟件計算相似系數(DICE系數),并且按照遺傳距離進行UPGMA聚類分析[6]。
2 結果與分析
2.1 DNA提取結果
用CTAB方法提取5種樣本的基因組DNA,經2%瓊脂糖凝膠電泳檢測,發現所得的DNA主條帶清晰,沒有出現明顯的拖尾顯現(圖1)。說明提取的DNA保持良好的完整性,花臉香蘑菌絲體及子實體中的多糖等去除的效果良好[7],可以進行下一步RAPD檢測。
2.2 RAPD分析結果
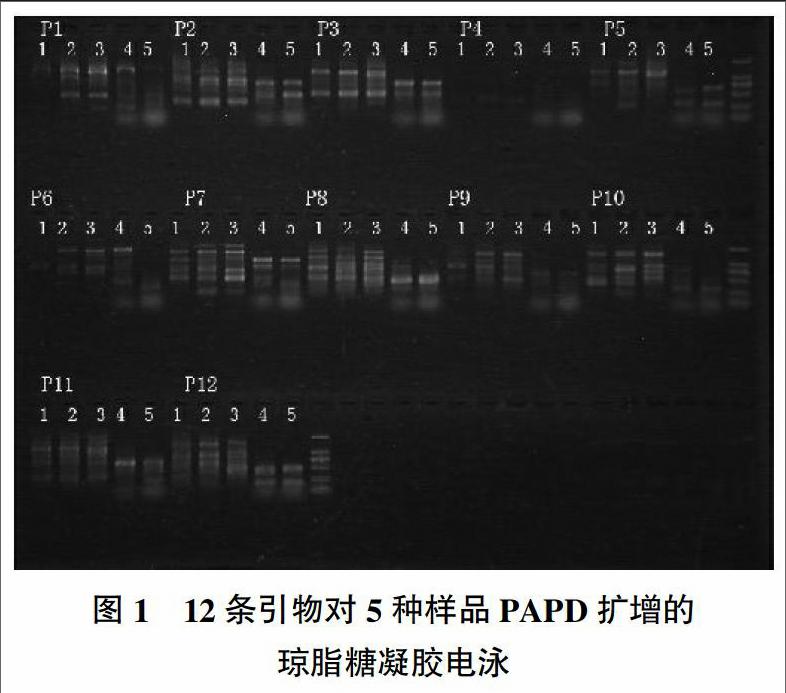
利用RAPD分子標記技術對5個樣品進行遺傳多樣性分析,12條引物擴增如圖2所示。從圖2中可以看出,選出8種條帶清晰的引物進行數據分析,選擇的引物代碼為P2、P3、P5、P7、P8、P10、P11、P12。
2.3 8種引物及其擴增的多態性條帶數
8種引物在5種樣品中共獲得46個擴增條帶,其中多態性條帶有39個,擴增出的多態性條帶百分率為84.78%,說明供試的5種樣品具有豐富的遺傳多態性,對不同的生存環境表現出較強的適應能力,符合花臉香蘑廣泛分布的特點。P7引物擴增出的多態性條帶數最多(7條),而P3引物擴增出的多態性條帶數最少(2條)。不同引物擴增出的清晰條帶數在4~8之間,平均每個引物擴增出的條帶數為5.75條,多態性條帶數為4.88條,多態性條帶百分率為82.90%[8],表3列出了8種引物對5種樣品擴增的多態性條帶數。
2.4 供試材料的親緣關系分析
利用8種引物在5種樣品中獲得46個擴增片段,如表3所示。在NTSYS軟件中計算樣品間的遺傳相似系數(GS值),得到供試材料遺傳相似矩陣。遺傳相似系數越大,表明親緣關系越近,遺傳相似系數越小,表明親緣關系越遠[9]。由表4可知,5種樣品的GS值范圍為0.282 6~0.913 0,變幅為0.630 4。其中4號樣品和5號樣品之間的遺傳相似系數最大為0.913 0,表明這兩個樣品之間的親緣關系較近,遺傳差異性小。3號樣品和4、5號樣品之間的遺傳相似系數最小為0.282 6,表明3號樣品與4、5號樣品之間的親緣關系較遠,遺傳差異性較大,存在較高的遺傳多樣性。
2.5 RAPD分子標記聚類分析
根據遺傳相似系數矩陣,利用UPGMA法進行聚類分析,根據遺傳相似系數對各樣品進行分類分析,聚類圖如2所示。5種樣品分成2類,樣品1、2、3是第一類,樣品4、5是第二類。其中第一類又分為2個亞類,第一亞類為1號樣品,樣品2、3號為第2亞類[10]。
3 結論與討論
4號樣品和5號樣品之間的親緣關系較近,遺傳差異性小。3號樣品與4、5號樣品之間的親緣關系較遠,遺傳差異性較大,存在較高的遺傳多樣性。 5種樣品分成2個類群,樣品1、2、3是第一類群,樣品4、5是第二類群。可見,RAPD將花臉香蘑菌絲體和子實體分為2個類群,2號和3號菌絲體親緣關系比與1號菌絲體親緣關系近。花臉香蘑遺傳多樣性,是由于自身遺傳體系與產地復雜變化的自然生態環境長期共同作用的結果,為花臉香蘑后期開發提供一定的參考。
參考文獻:
[1] 卯曉嵐.中國大型真菌[M].鄭州:河南科技技術出版杜,2000.
[2] 郭 勇,彭衛紅,賈定洪.花臉香蘑的栽培技術[J].食用菌,2007(3):41-42.
[3] 戴彩云,崔曉明.茶園自生食用菌紫丁香蘑的馴化栽培[J].貴州茶葉,1994(1):24.
[4] ZHANG Y F,MOLINA F I. Strain typing of Lentinula edodes by random amplified polymorphic DNA assay[J].Fems Microbiology Letters,1995,131(1):17-20.
[5] WELSH J,HONEYCUTT R J,MCCLELLAND M,et al. Parentagede termination in maize hybirds using the arbitrarily primed polymerase chain reaction(AP-PCR)[J]. Theoretical & Applied Genetics,1991,82(4):473-476.
[6] 孫 勇,林芳燦.中國香菇自然種質遺傳多樣性的RAPD分析[J].菌物系統,2003,22(3):387-393.
[7] 湯 潔.不同種絞股藍的RAPD分析及其抑菌作用的研究[D].合肥:安徽農業大學,2008.
[8] 林 媛.雙孢蘑菇品種間RAPD遺傳多樣性與親緣關系的研究[D].福州:福建農林大學,2008.
[9] 張金霞,黃晨陽,張瑞穎等.中國栽培白靈側耳的RAPD和IGS分析[J].菌物學報,2004,23(4):514-519.
[10] 譚 琦.用RAPD技術對香菇非對稱雜交后代遺傳性狀的分析[J].食用菌學報,2001,8(2):10-14.

