白細胞介素-6受體基因非同義變異rs2228145和中國過敏性鼻炎病人的相關性研究
趙亞麗 張 媛 王成碩 張 羅*
(1.首都醫科大學附屬北京同仁醫院過敏科,北京 100730; 2.北京市耳鼻咽喉科研究所 鼻病研究北京市重點實驗室,北京 100005; 3.首都醫科大學附屬北京同仁醫院耳鼻咽喉頭頸外科,北京 100730)
·鼻部慢性炎癥·
白細胞介素-6受體基因非同義變異rs2228145和中國過敏性鼻炎病人的相關性研究
趙亞麗1,2,3張 媛1,2,3王成碩3張 羅1,2,3*
(1.首都醫科大學附屬北京同仁醫院過敏科,北京 100730; 2.北京市耳鼻咽喉科研究所 鼻病研究北京市重點實驗室,北京 100005; 3.首都醫科大學附屬北京同仁醫院耳鼻咽喉頭頸外科,北京 100730)
目的過敏性鼻炎是鼻部黏膜的慢性炎性疾病,其發病受遺傳和環境因素雙重影響,過敏性鼻炎和哮喘在臨床表現和遺傳基礎具有交叉重疊。本研究擬對在全基因組關聯分析中發現的和哮喘相關的白細胞介素-6受體(interleukin-6 receptor,IL-6R)基因的rs2228145位點進行分析,檢測其和中國過敏性鼻炎病人的相關性。方法收集402例明確診斷為過敏性鼻炎病人,以及416例正常對照,所有個體均為中國漢族。從外周血白細胞提取基因組DNA,應用時間飛行質譜的方法進行基因分型。結果等位基因分析顯示過敏性鼻炎及正常對照在IL-6R基因的rs2228145位點等位基因頻率存在差異(P=0.0039,Pcorrected=0.001 6)。基因型分析進一步驗證了該位點和過敏性鼻炎的相關性,共顯性模型(PA/C vs A/A=0.000,ORA/C vs A/A=0.546,95%CI:0.389~0.766)和顯性模型(PA/C+C/C vs A/A=0.001,ORA/C+C/C vs A/A=0.575,95%CI:0.418~0.790)分析在病例組和對照組差異有統計學意義。結論結果提示和哮喘相關基因IL-6R的rs2228145位點和過敏性鼻炎的發病有關,可降低其發病的風險。
過敏性鼻炎;哮喘;白細胞介素6受體; 關聯分析
過敏性鼻炎(allergic rhinitis,AR)是鼻部黏膜的慢性炎性疾病,發病率在全球有增高的趨勢,可達10%~25%[1],臨床特征為鼻癢、流涕、反復噴嚏及鼻堵[2-3]。有學者[4]認為,過敏性鼻炎的發病受遺傳因素和環境因素的雙重影響,遺傳因素在過敏性鼻炎的發病中具有重要作用,遺傳度可達70%~90%。在過去的十年,連鎖分析和候選基因分析方式確定了少數和過敏性鼻炎相關的基因及位點,然而,這些位點在人群中的可重復性較差。全基因組關聯分析是隨著基因芯片技術的發展而逐漸應用的,對于復雜疾病易感基因的研究可以彌補候選基因局限于特定區域的不足,可以將易感基因定位于較小范圍內,彌補連鎖分析的局限性,可以無偏倚的在全基因組范圍內發現可能和疾病相關的易感基因,具有高效、全面的特點。目前,過敏性鼻炎的全基因組關聯分析較少,發現的比較確定和疾病相關的基因僅有6個:MRPL4、BCAP、C11orf30、LRRC32、FERD3L和ORMDL3[5-7]。
“同一氣道,同一疾病”,過敏性鼻炎和哮喘在表型和發病機制方面均具有相關性。據報道[3],很大一部分過敏性鼻炎病人會有哮喘的臨床表現,并且,過敏性鼻炎可以使得哮喘發病的概率增加[8-9]。過敏性鼻炎和哮喘在遺傳學發病機制上具有部分交叉重疊,部分基因和過敏性鼻炎和哮喘均相關。17q21區域是和哮喘密切相關的區域,為了驗證其和過敏性鼻炎的相關性,Tomita等[7]在兩個獨立的日本過敏性鼻炎人群進行了分析,發現該區域ORMDL3基因和過敏性鼻炎相關。
然而,無論候選基因關聯研究和基于芯片的全基因組關聯研究,發現的位點多數不是和疾病直接相關的位點,而是和功能變異處于連鎖不平衡區域的標記,并且目前發現的過敏性鼻炎的易感的變異僅能解釋部分過敏性鼻炎的發病機制。處于基因編碼區的變異可以改變基因編碼,從而影響其功能,其可能是和疾病直接相關的變異。
白細胞介素-6(interleukin-6,IL-6)是一個復雜的細胞因子,直接參與多種炎性反應。遺傳學研究[10]顯示,其受體基因IL-6R和多種慢性炎性反應性疾病相關。全基因組關聯研究及后續的候選基因分析顯示其非同義變異rs2228145變異可增加哮喘的易感性[11],而對于冠狀動脈粥樣硬化性心臟病(以下簡稱冠心病)[12]、風濕性關節炎[13]以及腹主動脈瘤[14]具有保護性作用。本課題組在前期對于過敏性鼻炎小家系的全基因組測序分析中發現IL-6R基因的rs2228145變異可能和疾病相關(數據尚未發表)。本研究擬在過敏性鼻炎病人中分析哮喘易感基因IL-6R的rs2228145位點疾病的相關性。
1 資料與方法
1.1樣本收集
本研究獲首都醫科大學附屬北京同仁醫院倫理委員會批準,符合世界衛生組織的《赫爾辛基宣言》。所有采集病例及標本的病人及正常對照均簽署了知情同意書,對于未成年人由其監護人代簽。
過敏性鼻炎病人均由有經驗的耳鼻咽喉科醫師診斷。 入選標準,選取中國北京地區長期居住的AR確診病例,所有受試者均出現鼻塞、清水樣涕、鼻癢和噴嚏等癥狀2項或2項以上;查體可見鼻黏膜蒼白、水腫和清水樣分泌物;常見吸入性過敏原的皮膚點刺試驗(skin prick test,SPT)(采用默克公司生產的診斷試劑)和血清特異性IgE(specific IgE,sIgE)(采用Phdia公司的診斷試劑)檢測均顯示至少有一種過敏原陽性(SPT≥++;sIgE≥2級)。吸入性致敏原包括動物毛(倉鼠上皮、狗上皮、兔上皮、貓上皮、豚鼠上皮)、樹1(榛屬、楊屬、榆科、柳屬、榿木)、樹2(樺木、水青岡、櫟屬、懸鈴木屬)、禾本科(天鵝草、鴨茅、黑麥草、梯牧草、龍須草、牛尾草)、禾本科/谷類(禾木科、大麥、燕麥、黑麥、小麥)、艾蒿、蒲公英、大豚草、藜、葎草、刺槐、德國小蠊、松、長葉車前草、新月彎孢屬、白色念珠菌、特異青霉、交鏈孢霉屬、煙曲霉菌、粉塵螨和屋塵螨。排除標準:排除鼻腔良、惡性腫瘤。
收集416例地域匹配的中國漢族正常對照,無過敏性疾病病史或無鼻及鼻腔疾病者作為正常對照,包括210名女性,206名男性。對照的年齡分布為3歲至80歲,平均年齡(35.8±15.0)歲。
1.2單核苷酸多態(singlenucleotidepolymorphism,SNP)選擇
選擇在哮喘的全基因組關聯分析中顯示和疾病相關的IL-6R基因的非同義變異rs2228145位點作為本研究的候選位點,分析其和中國過敏性鼻炎病人的相關性。
1.3基因分型
本研究收集的所有病例及對照均采集外周血,從外周血白細胞提取基因組DNA。基因組DNA提取試劑盒提取DNA(天根生化科技公司,北京)。提取的基因組儲存在4 ℃冰箱備用。應用時間飛行質譜的方法對選取的rs2228145位點進行基因分型,引物及探針應用MassARRAY Assay Design 4.0軟件設計。應用384孔板進行聚合酶鏈反應,采用MassARRAY Typer4.0軟件進行數據分析。每個384孔板應用5個空白及5個重復樣本進行質控。
1.4統計學方法
應用Genetic Power Caculator軟件計算本研究的統計學效力[15]。應用4.1版Haploview 軟件(Broad研究所)進行位點篩選,應用χ2檢驗的方法分析位點基因分型結果在正常人群中是否符合Hardy-Weinberg平衡,對于P值小于0.05的位點將不進行進一步的關聯分析。
應用11.0版STATA統計學軟件(Stata公司)進行病例組和對照組間的關聯分析。過敏性鼻炎病人組和正常對照組等位基因及基因型頻率差異分析采用χ2檢驗。應用10 000 置換的方法對P值進行校正。應用Akaike’s 信息標準(Akaike’s information criteria, AIC)選擇最佳基因型模型。應用非條件的回歸分析計算OR值和95%的可信區間。
2 結果
過敏性鼻炎組及正常對照組樣本的基本信息詳見表1,在本研究納入的過敏性鼻炎病人組中,22例病人具有慢性鼻竇炎病史,281例未合并慢性鼻竇炎;21例合并哮喘病史,282例未合并哮喘,其他99例病例未獲得詳細的病史。在進行DNA提取基因分型中,19例過敏性鼻炎病人和19例正常對照實驗失敗,因此,最終對383例病人及397例正常對照進行了數據的統計分析。詳見表2。
等位基因分析結果如表3。該分析顯示IL-6R基因的rs2228145位點的等位基因頻率在病例組和對照組差異有統計學意義(P=0.0039),P值經10 000次permutation校正后仍具有統計學意義(Pcorrected=0.001 6),提示該位點可能和疾病相關。
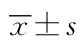
表1 收集AR及正常對照個體的基本信息Tab.1 Demographic characteristics of AR andcontrol subjects [,n(%)]
AR:allergic rhinitis;CRS:chronic rhinosinusitis;*Details about complications with CRS or asthma were not recorded for 99 cases.
基因型分析結果詳見表4。基因型分析采用AIC準則選擇最佳的基因型模型,分別對rs2228145位點基因型按照共顯性模型、顯性模型和隱性模型進行了分析。結果顯示該位點在共顯性模型(A/CvsA/A)病例組和對照組基因型頻率存在差異,PA/C vs A/A=0.000,ORA/C vs A/A=0.546,95%CI:0.389~0.766;同時,顯性模型(A/C+C/CvsA/A)分析病例組和對照組基因型頻率存在差異,PA/C+C/C vs A/A=0.001,ORA/C+C/C vs A/A=0.575,95%CI:0.418~0.790;共顯性模型(PC/C vs A/A=0.072,ORC/C vs A/A=0.662,95%CI:0.423~1.038)盡管沒有達到明顯差異,仍提示基因型頻率在該模型具有不同的趨勢。隱性模型(C/CvsA/A +A/C)分析時未顯示出明顯差異,PC/C vs A/A+A/C=0.718,ORC/C vs A/A+A/C=0.928,95%CI:0.618~1.394。結果提示IL-6R基因的rs2228145(Asp358Ala)位點變異和過敏性鼻炎相關,變異等位基因C編碼氨基酸358Ala對于過敏性鼻炎的發病具有重要的保護性作用(OR<1)。

表2 基因分型及Hardy-Weinberg平衡檢驗結果Tab.2 SNPs genotyped and their Hardy-Weinberg (HWE) thresholds
SNP:single-nucleotide polymorphism;IL-6R: interleukin-6 receptor;AR:allergic rhinitis.

表3 rs2228145位點在AR病例組和對照組等位基因頻率差異分析結果Tab.3 Allele frequencies of rs2228145 investigated among AR and control subjects

表4 rs2228145位點在AR病例組和對照組基因型頻率差異分析結果Tab.4 Genotype of rs2228145 investigated among AR and control subjects
SNP:single-nucleotide polymorphism;AR:allergic rhinitis.
3 討論
過敏性鼻炎和哮喘在臨床表現及遺傳基礎上存在部分交叉重疊,過敏性鼻炎可增加哮喘的發病概率[3,8-9]。因此,本研究選取了在哮喘的全基因組關聯研究[11]中發現的,并在多個研究中[16-17]得到了驗證的和哮喘相關的基因IL-6R的非同義變異rs2228145(Asp358Ala)位點進行分析,探討其和中國過敏性鼻炎的相關性。本研究中筆者發現IL-6R基因的變異位點rs2228145(Asp358Ala)和過敏性鼻炎相關,有意思的是,與哮喘相關研究不同,筆者發現較低頻率等位基因C在過敏性鼻炎的發病中可能發揮重要的保護性作用,而不是增加過敏性鼻炎的患病風險。
IL-6R基因編碼蛋白的配體:IL-6是一種復雜的細胞因子,目前被認為是人類最為多效的細胞因子,可以激活系列細胞,在調節炎性反應中發揮著重要的作用。IL-6R基因變異和多種疾病的發病相關:IL-6R和哮喘相關性是在一項對歐裔人群的大規模的全基因組關聯分析[11]中發現的,在其研究中發現位于內含子區域的rs4129267變異和哮喘發病相關,可增加哮喘的易感性(OR=1.09,Pcombined=2.4×10-8)。變異rs4129267和非同義變異rs2228145位點位于同一個連鎖不平衡的區域,因此,Hawkins等[16]在候選研究中選擇具有氨基酸編碼改變的rs2228145位點在歐裔人群進行了進一步研究,發現rs2228145位點變異等位基因和哮喘肺功能表型相關,這一結果在對于中國哮喘病人的研究[17]中得到結果相似。對于風濕性關節炎的研究[13]顯示IL-6R基因的非同義變異rs8192284和疾病相關(OR=0.9,P=1.3×10-8),該位點與和哮喘相關的rs4129267處于連鎖不平衡區域(r2=0.97,D′=1),該變異的較低頻率等位基因對于風濕性關節炎具有保護性作用。對于腹主動脈瘤的研究[16]顯示IL-6R基因的rs752922位點(可作為rs2228145的tag SNP,r2=0.97)和疾病相關,可降低疾病發病的風險(OR=0.84,P=2.7×10-11)。此外,還發現IL-6R基因的rs2228145和冠心病有關,C等位基因358Ala具有降低該病風險的作用[12]。
IL-6是一種多效的細胞因子,IL-6可控制T細胞的激活,分化反應,促進炎性反應環境的形成,因此其可參與多種自身免疫性和炎性疾病的發生[10]。IL-6與膜結合的IL-6受體相結合可以誘導其與gp130的二聚體的形成,從而促進轉錄因子STAT3和STAT1的磷酸化[18]。循環性IL-6受體由變異的膜結合IL-6受體剪接形成,或者由異構體RNA編碼形成[18]。循環性受體與IL-6結合可直接刺激gp130的表達增高[19]。rs2228145位點位于膜性IL-6R的剪接位置,基因變異后編碼形成循環性IL-6受體,因此,當等位基因發生Asp358Ala變異時,膜結合IL-6R表達降低,sIL-6R增多,并與IL-6R結合,降低STAT3 和 STAT1轉錄因子的活化,從而抑制IL-6誘導的級聯反應,從而導致系列疾病的發生。
IL-6R的rs2228145位點變異在保持膜性IL-6R和sIL-6R的平衡方面發揮重要作用。相同的變異和不同的炎性疾病相關,其作用機制尚很難闡明,可能和其他的變異或者基因的相互作用有關。Revez等[20]研究發現,在IL-6R基因及附近區域,除了rs2228145,rs12083537位點對于sIL-6R水平具有影響作用。其團隊[21]還發現STOML2基因的表達可和IL-6R基因表達相關,兩基因可能在哮喘發病中具有相互調節的作用。IL-6R基因的rs2228145位點變異和過敏性鼻炎的發生相關,可降低過敏性鼻炎的發病風險,其機制尚難闡明,可能也存在其他基因及變異的相互作用,有待于進一步深入研究。
[1] Bousquet J, Khaltaev N, Cruz A A, et al. Allergic rhinitis and its impact on asthma (ARIA) 2008 update (in collaboration with the World Health Organization, G A(2)LEN and AllerGen) [J]. Allergy, 2008, 63 (Suppl 86): 8-160.
[2] Greiner A N, Hellings P W, Rotiroti G, et al. Allergic rhinitis [J]. Lancet, 2011, 378(9809): 2112-2122.
[3] Bousquet J, Van Cauwenberge P, Khaltaev N, et al. Allergic rhinitis and its impact on asthma [J]. J Allergy Clin Immunol, 2001, 108(5 Suppl): S147-334.
[4] van Beijsterveldt C E, Boomsma D I. Genetics of parentally reported asthma, eczema and rhinitis in 5-yr-old twins [J]. Eur Respir J, 2007, 29(3): 516-521.
[5] Moffatt M F, Gut I G, Demenais F, et al. A large-scale, consortium-based genomewide association study of asthma [J]. N Engl J Med, 2010, 363(13): 1211-1221.
[6] Tamari M, Tomita K, and Hirota T. Genome-wide association studies of asthma [J]. Allergol Int, 2011, 60(3): 247-252.
[7] Tomita K, Sakashita M, Hirota T, et al. Variants in the 17q21 asthma susceptibility locus are associated with allergic rhinitis in the Japanese population [J]. Allergy, 2013, 68(1): 92-100.
[8] Leynaert B, Neukirch C, Kony S, et al. Association between asthma and rhinitis according to atopic sensitization in a population-based study [J]. J Allergy Clin Immunol, 2004, 113(1): 86-93.
[9] Linneberg A, Henrik Nielsen N, Frolund L, et al. The link between allergic rhinitis and allergic asthma: a prospective population-based study. The Copenhagen Allergy Study [J]. Allergy, 2002, 57(11): 1048-1052.
[10] Ferreira R C, Freitag D F, Cutler A J, et al. Functional IL6R 358Ala allele impairs classical IL-6 receptor signaling and influences risk of diverse inflammatory diseases [J]. PLoS Genet, 2013, 9(4): e1003444.
[11] Ferreira M A, Matheson M C, Duffy D L, et al. Identification of IL6R and chromosome 11q13.5 as risk loci for asthma [J]. Lancet, 2011, 378(9795): 1006-1014.
[12] Deloukas P, Willenborg C, Kanoni S, et al. Large-scale association analysis identifies new risk loci for coronary artery disease [J]. Nat Genet, 2013, 45(1): 25-33.
[13] Schnabel R B, Kerr K F, Lubitz S A, et al. Large-scale candidate gene analysis in whites and African Americans identifies IL6R polymorphism in relation to atrial fibrillation: the national heart, lung, and blood institute’s candidate gene association resource (CARe) project [J]. Circ Cardiovasc Genet, 2011, 4(5): 557-564.
[14] Harrison S C, Smith A J, Jones G T, et al. Interleukin-6 receptor pathways in abdominal aortic aneurysm [J]. Eur Heart J, 2013, 34(48): 3707-3716.
[15] Purcell S, Cherny S S, and Sham P C. Genetic power calculator: design of linkage and association genetic mapping studies of complex traits [J]. Bioinformatics, 2003, 19(1): 149-150.
[16] Hawkins G A, Robinson M B, Hastie A T, et al. The IL6R variation Asp(358)Ala is a potential modifier of lung function in subjects with asthma [J]. J Allergy Clin Immunol, 2012, 130(2): 510-515.
[17] Wang Y, Hu H, Wu J, et al. The IL6R gene polymorphisms are associated with sIL-6R, IgE and lung function in Chinese patients with asthma [J]. Gene, 2016, 585(1): 51-57.
[18] Kallen K J. The role of transsignalling via the agonistic soluble IL-6 receptor in human diseases [J]. Biochim Biophys Acta, 2002, 1592(3): 323-343.
[19] Chalaris A, Garbers C, Rabe B, et al. The soluble Interleukin 6 receptor: generation and role in inflammation and cancer [J]. Eur J Cell Biol, 2011, 90(6-7): 484-494.
[20] Revez J A, Bain L, Chapman B, et al. A new regulatory variant in the interleukin-6 receptor gene associates with asthma risk [J]. Genes Immun, 2013, 14(7): 441-446.
[21] Revez J A, Matheson M C, Hui J, et al. Identification of STOML2 as a putative novel asthma risk gene associated with IL6R [J]. Allergy, 2016, 71(7): 1020-1030.
Non-synonymousvariantrs2228145ininterleukin-6receptor(IL-6R)geneisassociatedwithallergicrhinitisinChinesesubjects
Zhao Yali1,2,3, Zhang Yuan1,2,3, Wang Chengshuo3, Zhang Luo1,2,3*
(1.DepartmentofAllergy,BeijingTongrenHospital,CapitalMedicalUniversity,Beijing100730,China; 2.BeijingKeyLaboratoryofNasalDiseases,BeijingInstituteofOtorhinolaryngology,Beijing100005,China; 3.DepartmentofOtorhinolaryngologyHeadandNeckSurgery,BeijingTongrenHospital,CapitalMedicalUniversity,Beijing100730,China)
ObjectiveAllergic rhinitis (AR) is a complex chronic inflammatory disease of the nasal mucosa, caused by an interaction between genetic and environmental factors. As evidence suggests that a proportion of individuals with AR also present symptoms of asthma and some genetic variants may have increased susceptibility to both AR and asthma. The objective of this study was to identify whether asthma susceptibility variant rs2228145 in interleukin-6 receptor (IL-6R) is associated with AR in the Chinese population.MethodsA cohort of 402 individuals with physician diagnosed AR and 416 healthy controls were recruited from the Han Chinese population in Beijing. DNA was extracted from the peripheral blood. rs2228145 was genotyped using the sequenom mass array technology platform.ResultsAnalysis of frequency differences of allele between the AR patients and control subjects showed that rs2228145 inIL-6Rwas significantly associated with AR (P=0.0039,Pcorrected=0.001 6). Genotype analysis further confirmed the difference in distribution of this variant between AR patients and controls in both the co-dominant (PA/C vs A/A=0.000,ORA/C vs A/A=0.546,95%CI:0.389-0.766)and dominant (PA/C+C/C vs A/A=0.001,ORA/C+C/C vs A/A=0.575,95%CI:0.418-0.790) models.ConclusionThese results suggest that the rs2228145 inIL-6Rgene is associated with decreased risk for AR.
allergic rhinitis; asthma; interleukin-6 receptor; association studies
國家自然科學基金(81400470,81570895),北京市衛生系統高層次衛生技術人才科研骨干項目(2014-3-015)。This study was supported by National Natural Science Foundation of China (81400470,81570895), Beijing Health Bureau Program for High Level Talents (2014-3-015).
*Corresponding author, E-mail:dr.luozhang@139.com
時間:2017-10-14 16∶06
http://kns.cnki.net/kcms/detail/11.3662.R.20171014.1606.018.html
10.3969/j.issn.1006-7795.2017.05.013]
R765
2017-07-07)
編輯 孫超淵

