高通量測序技術解析五糧液窖泥原核微生物群落結構
趙東,鄭佳,彭志云,呂學蘭,楊康卓,張建敏
(宜賓五糧液股份有限公司 技術研究中心,四川 宜賓,644000)
高通量測序技術解析五糧液窖泥原核微生物群落結構
趙東,鄭佳*,彭志云,呂學蘭,楊康卓,張建敏
(宜賓五糧液股份有限公司 技術研究中心,四川 宜賓,644000)
為清晰認識五糧液特殊窖泥微生態(tài)群落結構及功能,利用Illumina NextSeq 500高通量深度測序平臺首次分析了五糧液不同窖泥原核微生物群落16S rDNA V4高變區(qū)序列;優(yōu)化了QIIME程序并整理和統(tǒng)計樣品序列數目和操作分類單元(OTUs)的數量,分析了不同窖泥中原核微生物群落的豐度、多樣性;利用系統(tǒng)發(fā)育分析確定了優(yōu)勢菌群的系統(tǒng)發(fā)育地位。該研究結果表明,從五糧液窖泥中共獲得7 555 164條高質量目標片段。在相同的測序深度條件下(300 000條),五糧液窖泥原核微生物主要由細菌和古菌構成,共檢出22個門,52個綱,89個目,168個科,330個屬原核微生物,細菌占92.3%,古菌占7.6%。厚壁菌門(Fimicutes)是所有樣品的絕對優(yōu)勢菌群(85.6%),共檢出74個屬,Clostridium、Lactobacillus,Coprococcus,Caldicoprobacter,Soehngenia是豐度最高的細菌屬。結合系統(tǒng)發(fā)育分析確定窖泥中優(yōu)勢菌群為Lactobacillus,Caldicoprobacter,Caloramater,Clostridium,Caloribacterium,Garciella,Eubacterium,Syntrophomonas,Sedimentbacter,Sporanaerobacter,Tissierella,Methanosarcina,Methanobacterium,Methanobrevibacter,Methanoculleus。五糧液窖泥中蘊藏了顯著區(qū)別于其他濃香型白酒窖泥的原核微生物群落,尤其是群系復雜的厚壁菌門細菌,較高豐度的產己酸菌群和促進己酸生成的甲烷菌群。
五糧液;窖泥;原核微生物群落;高通量測序
五糧液,古稱“重碧春、姚子雪曲、雜糧酒等”,是我國傳統(tǒng)固態(tài)發(fā)酵白酒的典型代表。其獨特的“五糧配方、包包曲制曲工藝、雙輪底發(fā)酵、分層起糟、分層蒸酒、量質摘酒”等優(yōu)秀的傳統(tǒng)釀造技藝極大程度促進了我國白酒釀酒工業(yè)發(fā)展。五糧配方的續(xù)糟發(fā)酵是五糧液糟醅發(fā)酵的根本特征,長期不間斷連續(xù)生產,尤其是糟醅的雙輪底發(fā)酵,極大程度上提高了窖池內窖泥與糟醅的厭氧接觸時間,糟醅與窖泥間營養(yǎng)物質、微生物的交換,形成了“以窖養(yǎng)糟、以糟促窖、糟窖互養(yǎng)”的良好互動局面。因此,確定窖泥微生物尤其是原核微生物的結構特征將有助于科學認識五糧液的釀酒微生物菌群代謝特征,為發(fā)酵過程代謝調控提供必要的依據。
窖池微生物群落的研究技術主要包括了傳統(tǒng)分離培養(yǎng)技術、生物標記法和現(xiàn)代分子生物學免培養(yǎng)技術。20世紀90年代以來,吳衍庸等[1]基于傳統(tǒng)分離培養(yǎng)技術剖析了五糧液老窖泥中厭氧微生物的群落結構,結果表明己酸菌和厭氧異養(yǎng)菌等隨窖齡而增加,己酸菌、甲烷菌等隨窖池上、中、下位置順序遞增。趙東等[2]分析了五糧液糟醅發(fā)酵過程中細菌(好氧、兼性厭氧)、酵母和霉菌的變化趨勢。然而,傳統(tǒng)培養(yǎng)法中由于培養(yǎng)基的高度選擇性、微生物的獨特生長特性等,迄今為止僅有微生物總數的0.1%~10%被檢出[3],且此法操作過程費時費力,容易因培養(yǎng)基選擇性、操作者的誤差以及菌株生長條件需求差異等因素掩蓋真實信息,難以定量表征群落多樣性特征及脅迫條件的影響。進入21世紀,聚合酶鏈式反應(PCR)技術開始應用于濃香型白酒微生物群落結構的研究[4-6],一定程度上克服了傳統(tǒng)分離培養(yǎng)技術的缺陷。但諸如PCR-DGGE、PCR-SSCP等手段的低分辨率(僅僅檢出少數優(yōu)勢種群)、條帶共遷移、條帶DNA/RNA回收困難、無法定量確定條帶比例等缺點,難以客觀真實地定量反映窖泥微生物群落的演替規(guī)律。生物標記法,如磷脂脂肪酸(PLFA)[7-8]和麥角甾醇[9]等已應用于研究濃香型白酒微生物群落生物量的分布規(guī)律,但由于菌株間具有相似的PLFA組成等因素,菌株間PLFA相互干擾致使該法無法獲得釀酒體系中微生物群落的組成信息。高通量測序技術是近年來快速發(fā)展并滲透至多學科的有效的分子生態(tài)學手段,無需分離純培養(yǎng)單一菌株,提取體系基因組DNA后,直接在基因水平研究挖掘環(huán)境中微生物群落資源,同時,該方法體系兼具有通量高,覆蓋率高等優(yōu)點[10]。基于測序結果中reads數(序列條數)能進行不同操作分類單元(OTU)的數量的統(tǒng)計,經過OTU與微生物數據庫的比對信息后即可獲得相關微生物的分類信息,若在保證測序深度和測序質量的條件下,即可獲得較為全面的進行微生物群落多樣性評估信息。陶勇等[11]基于焦磷酸測序分析了四川某知名酒廠不同窖齡濃香型白酒窖泥中細菌和古菌的群落結構及多樣性。鄧杰等[12]利用焦磷酸測序技術分析了豐谷窖泥中原核微生物的結構組成。李克亞等[13]基于Illumina MiSeq測序分析了蜀之源和豐谷窖泥中原核微生物的群落結構。各個測序平臺間由于測序原理的不同,獲得樣品測序深度完全不同(序列均在20 000條以內)。同時,樣品間由于測序文庫制備時的偏差致使測序深度具有差異性,直接在不同測序深度進行群落多樣性評估勢必存在一定弊端。因此,為了避免評估微生物群落豐度時出現(xiàn)偏差,需要在文庫制備、測序平臺選擇、測序數據質量控制、生物信息學分析等方面進行層層技術把關。
為了獲得全面的釀酒微生物群落結構特征以及其代謝特征,作為中國白酒領軍企業(yè)的五糧液集團公司于2015年在國內酒類企業(yè)中率先購置了美國Illumina公司NextSeq 500高通量測序儀,該儀器具有靈活的測序類型(宏基因組、宏轉錄組、外顯子、擴增子等)、高的測序通量(中通量20-40Gb,高通量30-120Gb數據量)以及較快的測序速度(12~30 h)等優(yōu)勢[14]。因此,本研究通過高通量深度測序策略進行五糧液窖泥原核微生物16S rDNA V4區(qū)序列進行測序;隨后進行序列重采樣,全面評估窖泥原核微生物群落組成及多樣性。
1 材料和方法
1.1取樣方法
所有樣品均采自五糧液股份有限公司釀酒車間,窖池窖齡范圍為15~50年。6口窖池分別位于不同的車間,所有窖池的生產工藝相同,均為續(xù)糟跑窖發(fā)酵工藝,且各窖池的原輔料品質類型在長期生產中均保持一致為選擇窖池的標準。將窖池窖壁垂直中線處分為2個點,以黃水線為參考基準(距池底高度范圍1.2~1.7 m),即中層和下層窖泥。采樣方法見參考文獻[5],每點采集3個平行樣品,分別取100 g,混合均勻。6口窖池的樣品編號分別從1#至12#(單數為各窖池的中層窖泥,雙數為下層窖泥),其中1#-2#和3#-4#采集自車間一(窖齡約50年)、5#-6#和7#-8#采集自車間二(窖齡約20年)、9#-10#和11#-12#采集自車間三(窖齡約15年),樣品置于真空袋中-20 ℃冷凍保存。
1.2儀器與試劑
NextSeq 500高通量測序儀,Illumina,美國;Qubit 2.0熒光定量儀,Thermo Fisher,美國;T100 梯度PCR儀,Bio-Rad,美國。
土壤DNA提取試劑盒(MP biomedicals,美國),KAPA HIFI Hotstart Readymix高保真熱啟動酶(KAPA Biosystems,美國),Qubit dsDNA HS Assay kit(Thermo Fisher,美國),引物由上海生工生物工程有限公司合成,Agencourt AMPure XP磁珠核酸純化試劑盒(Beckman Coulter,美國),Nextera XT Index Kit v2 Set A 標簽試劑盒(96 Indices,384 Samples,Illumina,新加坡),Nextera XT DNA Sample Preparation Kit 文庫制備試劑盒(24 Samples,Illumina,新加坡),NextSeq 500/550 Mid Output Kit v2 中通量測序試劑盒(300 cycles,Illumina,新加坡)。
1.3基因組DNA提取及PCR擴增
采用土壤DNA提取試劑盒提取樣品總DNA,具體操作步驟見試劑盒使用手冊,然后用Qubit 2.0熒光定量儀測定其濃度,保證每個樣品的DNA質量濃度大于10 μg/μL。
高通量測序區(qū)域采用細菌16S rDNA基因V4區(qū)域(Escherichiacoli位點515-806)的帶有測序接頭的特異性引物對Hyb515F(5’-T ̄C ̄G ̄T ̄C ̄G ̄G ̄C ̄A ̄G ̄C ̄G ̄T ̄C ̄A ̄G ̄A ̄T ̄G ̄T ̄G ̄T ̄A ̄T ̄A ̄A ̄G ̄A ̄G ̄A ̄C ̄A ̄G ̄G ̄T ̄G ̄C ̄C ̄A ̄G ̄C ̄M ̄G ̄C ̄C ̄G ̄C ̄G ̄G ̄T ̄A ̄A-3’)和Hyb806R(5’-G ̄T ̄C ̄T ̄C ̄G ̄T ̄G ̄G ̄G ̄C ̄T ̄C ̄G ̄G ̄A ̄G ̄A ̄T ̄G ̄T ̄G ̄T ̄A ̄T ̄A ̄A ̄G ̄A ̄G ̄A ̄C ̄A ̄G ̄G ̄G ̄A ̄C ̄T ̄A ̄C ̄H ̄V ̄G ̄G ̄G ̄T ̄W ̄T ̄C ̄T ̄A ̄A ̄T-3’),在每對引物的5’末端添加了能與Illumina測序flowcell表面結合以利于產生cluster的接頭序列。PCR擴增具體反應程序如下:94 ℃預變性3min;94 ℃變性20 s,53 ℃退火25 s,72 ℃延伸45 s,共25個循環(huán);72 ℃延伸5 min。利用磁珠法純化PCR擴增產物(純化方法見純化試劑盒),再進行測序文庫構建。
1.4測序文庫構建
利用Nextera XT Index Kit v2 Set A 標簽試劑盒和Nextera XT DNA Sample Preparation Kit文庫制備試劑盒進行樣品上機測序文庫的構建,具體操作步驟見試劑盒操作手冊。使用NextSeq 500高通量測序儀進行雙端測序(Paired-end sequencing),產生讀長為290 bp的序列。
1.5生物信息學分析
1.5.1 序列處理與分析
對測序原始數據通過bcl2fastq軟件獲得相應樣品的原始序列,利用QIIME(Quantitative Insights into Microbial Ecology)對原始DNA序列進行過濾處理,除去嵌合體,并按照最大允許4個堿基錯誤為標準進行序列拼接,最終得到優(yōu)化序列。按照97%相似性將優(yōu)化序列劃分操作分類單元OTUs(Operational Taxonomic Units)。對OTUs表進行重采樣[15],使各樣品獲得一致的序列數,繪制稀釋曲線(Rarefaction curve),計算Chao1豐度指數和Shannon多樣性指數,得到微生物群落結構組成,其中重采樣次數為2次。
Coverage指數的計算公式為:

(1)
式中:n1代表只含有1條序列的OUT數目;N代表抽樣中出現(xiàn)的總序列數目。
Shannon指數計算公式為:
H=-ΣPi×lnPi
(2)
式中:Pi為各種群物種數與樣本總物種數之比。
Chao1指數計算公式為:
Chao1=OTUr+n1(n1-1)/2(n2+1)
(3)
式中:OTUr代表實際OUT數量;n1代表只有1條序列的OUT數量;n2代表只有2條序列的OUT數量。
利用QIIME軟件計算樣品Beta多樣性距離矩陣,并根據Beta多樣性距離矩陣進行主坐標分析(principal coordinate analysis,PCoA)。同時基于各樣品中優(yōu)勢原核微生物的豐度,利用Canoco 4.5軟件進行樣品間關系的主成分分析(principal component analysis,PCA)。
1.5.2 優(yōu)勢序列系統(tǒng)發(fā)育分析
以QIIME中獲得的豐度大于1%優(yōu)勢屬OTUs序列為研究對象,通過RDP classifier(http://rdp.cme.msu.edu/classifier/classifier.jsp)與NCBI BLASTn中查出的同源性最高菌株序列為參考序列,利用MEGA 4.0軟件以ClstralW進行序列比對,以最大相似距離估算系統(tǒng)進化矩陣,采用鄰接法(Neighbor-Joining)構建優(yōu)勢OTU系統(tǒng)發(fā)育樹,其中bootsrap次數為1 000[6]。
2 結果與分析
2.1測序結果
選擇了五糧液6口窖池為研究對象,以黃水線為分界線,比較了干糟對應的中層窖泥和濕糟對應的下層窖泥中原核微生物群落結構。基于Illumina NextSeq深度測序平臺測序,blc2fastq軟件、QIIME程序拼接縫合序列后,12個窖泥獲得了7 659 476條原始序列,質控后得到7 555 164條高質量目標片段(長度范圍283~291 bp),其中序列數最少和最多的樣品分別為7#窖泥(465 263條)和8#窖泥(801 675條)。
由于每個樣品的測序深度不同,若要進行后續(xù)群落多樣性比較,需要進行序列的重采樣。對樣品進行不同序列數采樣(10 000條,20 000條,40 000條,100 000條和300 000條),使每個樣品獲得一致的序列數,計算樣品的稀釋曲線、覆蓋度、多樣性指數等參數。由表1可知,隨著重采樣序列數的增加,樣品覆蓋度增加,當達到30 000條時,覆蓋度達99.5%,且各樣品OTU稀釋曲線區(qū)域平緩(圖1),多樣性指數包括Chao1、Shannon均隨采樣序列的增加而增加,表明300 000條的測序深度能夠比較全面的反映本研究中窖泥原核微生物群落的多樣性。
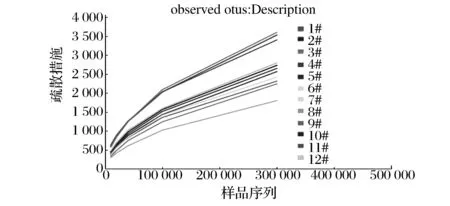
圖1 豐度稀釋曲線Fig.1 Rarefaction curves of pit muds

序列數覆蓋度Chao1指數香農指數OTUs數量100000.966±01313±1155.51±0.04622±0.7200000.977±01812±1845.53±0882±5.7400000.984±02533±1845.55±01257±291000000.990±03972±1165.61±02032±373000000.995±05123±1715.62±03402±13
基于以上重采樣序列,進行目標序列與數據庫的比對,選擇了3種常見的序列比對方法(uclust、blast和RDP),結果如表2所示,可知前2種比對方法在門、綱、科的數量差別不大,在屬上有一定差異性,而RDP比對方法則具有明顯優(yōu)勢。因此,將RDP作為序列比對的主要方法,共檢出22個門,52個綱,89個目,168個科,330個屬原核微生物。

表2 不同序列比對方法結果比較
2.2原核微生物群落多樣性與群落組成
2.2.1 門水平的原核微生物群落多樣性
本研究中,12個窖泥中原核微生物主要是由細菌和古菌構成,其中細菌占92.3%,古菌占7.6%。可見,細菌的豐度和多樣性顯著高于古菌,細菌為優(yōu)勢菌群。由表2可知,共檢出19個門,包括:廣古菌門(Euryarchaeota)、泉古菌門(Crenarchaeota)、酸桿菌門(Acidobacteria)、放線菌門(Acinobacteria)、擬桿菌門(Bacteroidetes)、衣原體門(Chlamydiae)、綠彎菌門(Chloroflexi)、藍細菌門(Cyanobacteria)、芽單胞菌門(Gemmatimonadetes)、黏膠球形菌門(Lentisphaerae)、厚壁菌門(Fimicutes)、硝化螺旋菌門(Nitrospirae)、浮霉菌門(Planctomycetes)、變形菌門(Proteobacteria)、螺旋體門(Spirochaetes)、互養(yǎng)菌門(Synergistetes)、軟壁菌門(Tenericutes)、疣微菌門(Verrucomicrobia)以及其他7種未命名門類(AC1、FBP、OP9、TM7、WS1、WWE)。優(yōu)勢門的豐度見圖2,厚壁菌門(Fimicutes)在所有樣品中均是絕對優(yōu)勢菌群(85.6%),其次為廣古菌門(Euryarchaeota,7.6%)、擬桿菌門(Bacteroidetes,4.7%)、放線菌門(Acinobacteria,1.3%)和變形菌門(Proteobacteria)。1#~6#三對(1#和2#、3#和4#、5#和6#)窖泥中,中層窖泥中厚壁菌門細菌豐度低于下層窖泥,擬桿菌門和泉古菌門則反之。9#~12#兩對(9#和10#、11#和12#)窖泥中厚壁菌門細菌的豐度則是中層高于下層,而泉古菌門的則是下層高于中層。
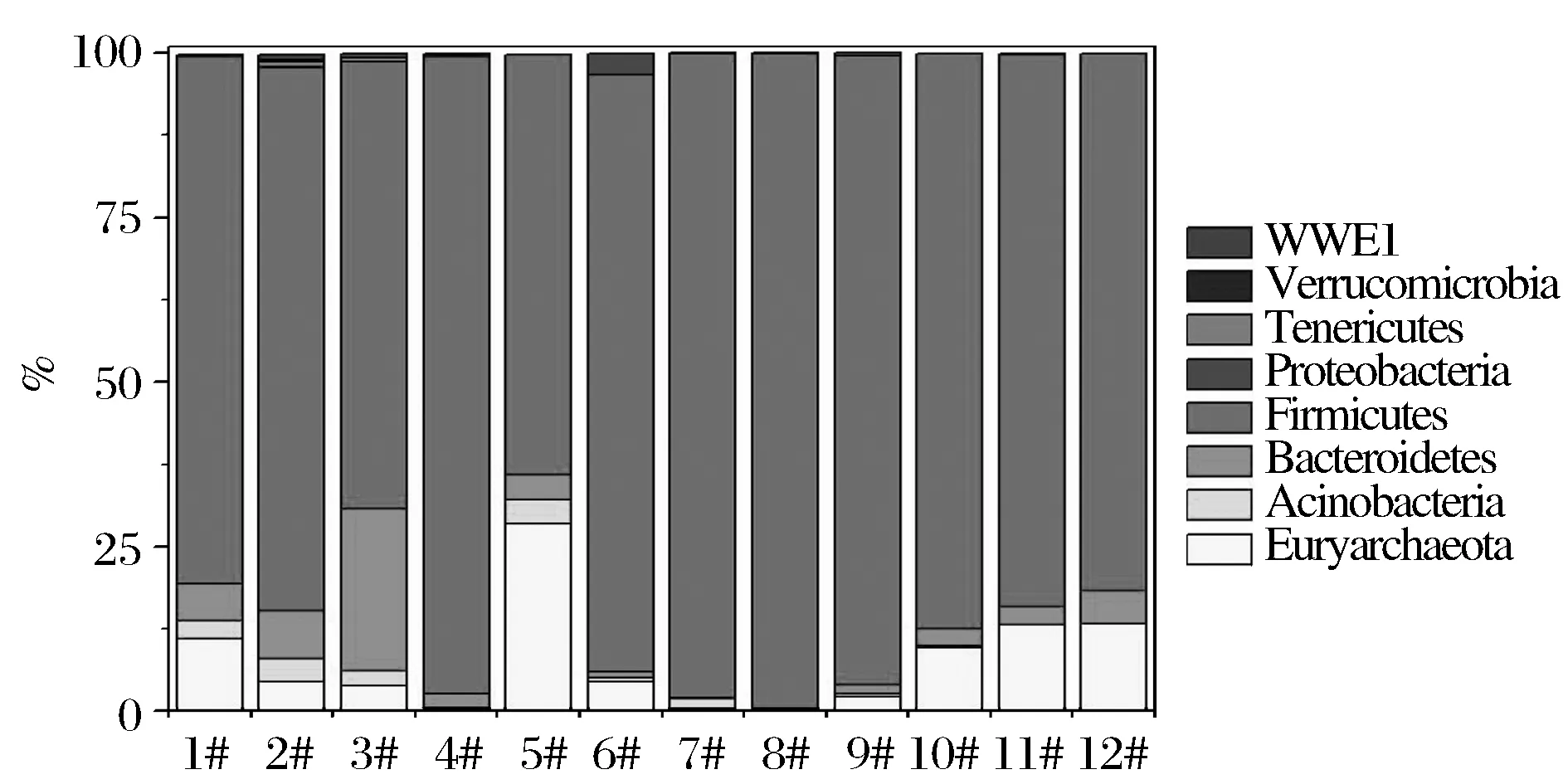
圖2 門水平優(yōu)勢原核微生物菌群Fig.2 Dominantprokaryotic microbial communities at phylum levels in pit muds
2.2.2 屬水平的原核微生物多樣性分析
為進一步清晰的認識五糧液窖泥中重要的功能原核微生物菌群,本研究從屬水平進行系統(tǒng)發(fā)育和統(tǒng)計分析。RDP分析顯示絕大部分序列可歸類到330屬(細菌322屬,古菌8屬),其中豐度大于1%的屬中細菌13個屬(Lactobacillus-OTU975918,Caldicoprobacter-OTU4319602,Caloramater-OTU584933,Clostridium-OTU4342297,Thermoanaerobacterium-OTU4411374,Garciella-OTU742140,Coprococcus-OTU585881,Dorea-OTU349343,Rummincoccus-OTU771083,Syntrophomonas-OTU4390692,Sedimentbacter-OTU805522,Sporanaerobacter-OTU4312474,Tissierella-OTU4320991),古菌4屬(Methanosarcina-OTU264516,Methanobacterium-OTU105600,Methanobrevibacter-OTU22694,Methanoculleus-OTU283331),各優(yōu)勢菌屬的豐度如圖3所示。可見,部分窖池的中層窖泥中原核微生物群落與下層微生物群落結構組成具有一定相似性(如1#和2#、11#和12#),而部分窖池的差異卻很大(5#和6#、7#和8#、9#和10#)。部分窖池的中層窖泥中的甲烷菌群豐度顯著高于下層(1#>2#,3#>4#,5#>6#、11#>12#),而7#和8#中幾乎沒有檢出甲烷菌群,9#中甲烷菌的豐度則小于10#。1#與2#、11#與12#間差異最明顯的是Methanobacterium,3#與4#間主要是Garciella、Caloramator、Ruminococcus和Sporanaerobacter的豐度差別,5#與6#、7#與8#、9#與10#兩兩間的差異十分顯著,主要差異的微生物種屬包括Lactobacillus、Methanobacterium、Ruminococcus。
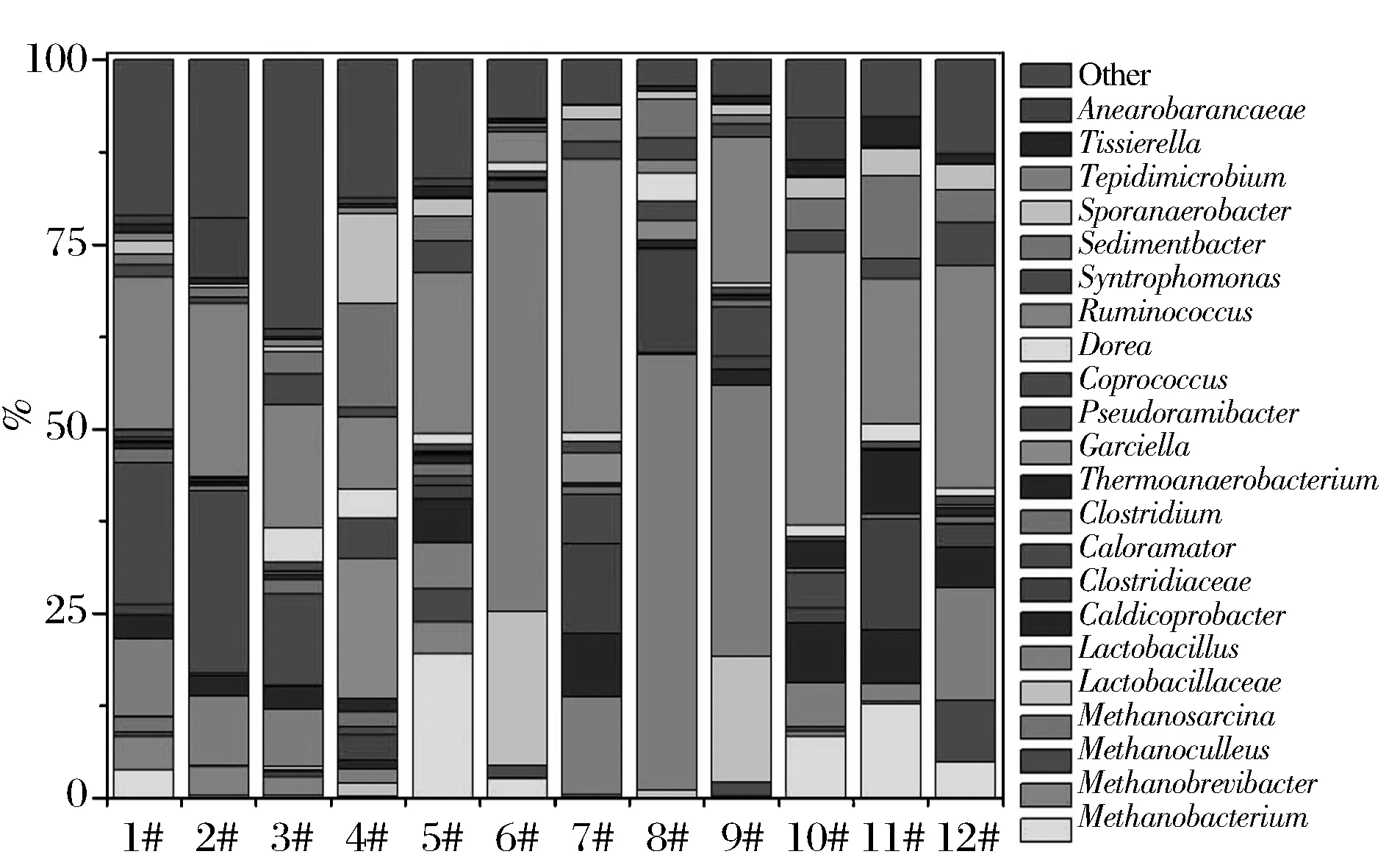
圖3 科屬水平優(yōu)勢原核微生物群落Fig.3 Dominant prokaryotic microbial community at genus levels in pit muds

圖4 五糧液窖泥優(yōu)勢OTU序列系統(tǒng)進化樹Fig.4 Phylogenetic tree of the dominant OTUs sequences in pit muds采用鄰接法對豐度大于1%的OTUs構建系統(tǒng)發(fā)育樹。分支點的數字為分子的Bootstrap值(%)。標尺表示0.05核酸距離,分支長度為相似度。樹枝末端信息包含分類的種名、OTUs編號和NCBI登錄號。
2.2.3 優(yōu)勢序列系統(tǒng)進化分析
上述結果得到了五糧液不同窖泥中總共有17個OTU。RDP Classifier、NCBI Blastn和系統(tǒng)進化分析進一步確認了這17個OTU在屬水平上的分類,優(yōu)勢OTU的16S rDNA序列系統(tǒng)發(fā)育樹如圖4所示。它們與其最近的參考序列相似性分析可知,OTU975918與Lactobacillusacetotolerans相似性100%、OTU742140與Garciellasp.相似性90%、OTU4312474與Soehngeniasp.相似性100%、OTU4320991與unclturedTissierellasp.相似度100%、OTU4390692與Sytrophomonas屬相似度100%、OTU4411374與unclturedCaloribacteriumsp.相似度100%、OTU771083與unculturedClostridumsp.相似性100%、OTU349343與Eubacteriumcontortum相似度99%、OTU264516與Methanosarcina屬相似度100%、OTU283331與Methanoculles屬相似度100%、OTU105600與Mthanobacterium屬相似度100%,根據相似性分析結果可知,以上OTU的歸屬基本可以確定,即OTU4411374從QIIME檢索結果進一步確認為Caloribacterium、OTU585881由Coprococcus→Clostridium、OTU349343由Dorea→Eubacterium、OTU771083由Rummincoccus→Clostridium、OTU4312474由Sporanaerobacter→Soehngenia。
2.2.4 不同窖泥原核微生物群落相似性分析
將不同樣品中屬于相同物種的reads數據,利用weighted unifrace算法計算樣本間距離矩陣,繪制PCoA主坐標分析圖(圖5a)。可知,樣品6#、8#、9#與其他樣品的主要區(qū)別在PC1軸上顯著區(qū)分。基于優(yōu)勢科屬原核微生物的豐度(圖3)構建樣品間關系的主成分分析結果如圖5b所示,與PCoA分析結果類似的,樣品6#、8#、9#位于PC1軸的正半軸,與6#、8#、9#顯著相關的為Lactobacillus和Lactobacillacea。而其余窖泥樣品均位于PC1的負半軸,4#、5#和11#位于PC2軸的正半軸,與之顯著正相關的包括Sedimentbacter、Sporanaerobacterium和Garciella;1#、2#、3#、7#、10#、12#等樣品位于PC2軸的負半軸,與之顯著正相關的包括Caloramator、Ruminococcus、Pseduoramibacter等。
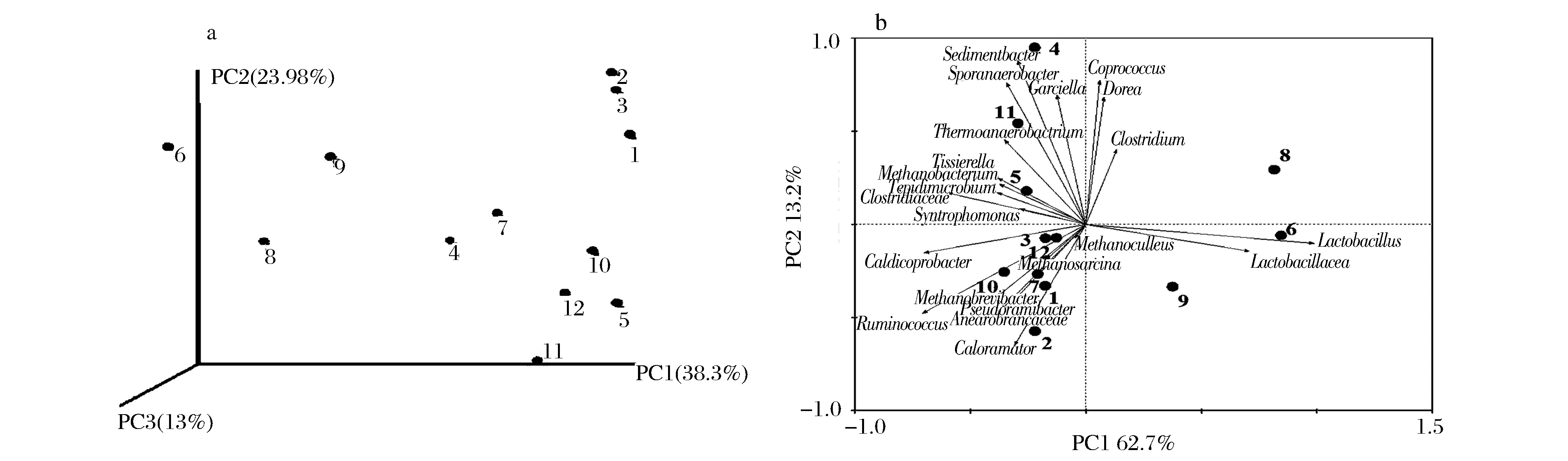
圖5 各窖泥樣品的Weighted unifrace PCoA圖(a)和PCA圖(b)Fig.5 Weighted unifrace PCoA analysis (a) and PCA analysis of pit muds (b)
3 討論
本研究中單個樣品檢出了超過40萬條有效DNA序列。過去文獻報道中,利用Roche 454測序儀獲得窖泥微生物16S rDNA總有效序列范圍為6 568-10 992條[11]。基于Illumina Miseq平臺分析濃香型窖泥的總有效序列范圍為12 556-65 500條[13,16]。可見,采用本研究所述方法體系獲得的測序深度遠遠大于以上2個平臺,故該測序覆蓋度具有明顯的優(yōu)勢(表1)。
厚壁菌門細菌是五糧液窖泥中的絕對優(yōu)勢菌群(圖2),其他同香型窖泥中也檢出了類似的結果。利用MiSeq分析四川蜀之源和豐谷不同窖齡窖泥細菌群落中檢出了8個細菌門和2個古菌門,其中變形菌門和厚壁菌門是絕對優(yōu)勢菌群[13]。利用Roche焦磷酸測序從豐谷不同窖齡窖泥中檢出19個細菌門,其中厚壁菌門占79%[12]。利用焦磷酸測序從四川某知名濃香型酒廠不同窖齡窖泥中檢出19個細菌門和2個古菌門,其中厚壁菌門占66.8%[17]。由此可見,同香型(即濃香型)窖泥原核微生物群落組成大類上具有一定的相似性。
但是,五糧液窖泥中優(yōu)勢細菌科屬的種類、豐度與其他窖泥的差異顯著。本研究檢出了大量細菌屬,尤其是厚壁菌門(Firmicutes),共檢出74個屬,如Clostridium,Bacillus,Lactobacillus,Coprococcus、Caldicoprobacter,Soehngenia,Eubacterium,Planococcus,Solibacillus,Macrococcus,Ruminococcus,Sedimentibacter,Thermoanaerobacterium,Caldicoprobacter等,而在其他酒廠窖泥中厚壁菌門細菌則少于本研究,其檢出了32個屬[11]。同時,兩者間優(yōu)勢菌群的構成以及在總菌群中的占比差異顯著,本研究中優(yōu)勢厚壁菌門細菌屬較多,包括Lactobacillus,Caldicoprobacter,Caloramater,Clostridium,Caloribacterium,Garciella,Eubacterium,Syntrophomonas,Sedimentbacter,Sporanaerobacter,Tissierella(圖3),而后者中的優(yōu)勢厚壁菌門細菌屬則為Lactobacillus(4.23%~62.28%),Bacillus(0.03%~3.63%),Clostridium(0.03%~3.63%),Sedimentibacter(1.57%~4.78%)和Sytrophomonas(0.45%~4.74%)。
以往的研究中發(fā)現(xiàn)酸類、酯類及醇醛類是名優(yōu)濃香型白酒獨特風味的骨架香味成分,酯類主要以乙酯為主(己酸乙酯、乳酸乙酯和乙酸乙酯);同時,優(yōu)質酒中總酸、總酯的含量較普通白酒高數倍,名優(yōu)白酒間風格的區(qū)別在于三大酯類的含量與相互間的比例不同;酸類是酯類產生的基礎,酸多酯高[23]。研究結果發(fā)現(xiàn),Lactobacillus是產生乳酸的主要細菌菌群,Clostridium中Cl.kluyveri是產生己酸、丁酸和H2的主要種屬[18]。本研究中,五糧液窖泥中Clostridium(22%)是優(yōu)勢厚壁菌門細菌。Caldicoprobacter(4.1%)屬厭氧菌,C.oshimai,C.guelmensis均能利用葡萄糖、乳糖等產生乙酸、醋酸、CO2和H2[19]。毛羅菌科Coprococcus(1.5%)細菌屬嚴格厭氧細菌,C.catus能進行丙酸發(fā)酵[20]。Soehngenia(2.7%)細菌中S.saccharolyticagennov.,sp. nov.能代謝酵母膏、半胱氨酸等產生苯乙醛[21]。
甲烷菌常與己酸菌共棲互營生長,并通過種間“氫轉移”作用能有效提高濃香型白酒己酸乙酯等的含量[22]。本研究中檢出了4個優(yōu)勢的古菌屬,其中Methanobacterium、Methanobrevibacter和Methanoculleus屬氫營養(yǎng)型甲烷菌,Methanosarcina可同時利用乙酸和H2。四川某知名濃香型白酒窖泥中優(yōu)勢古菌為Methanosarcina,Methanobacterium,Methanobrevibacter和Methanoculleus[17]。本研究中這些甲烷菌的豐度均高于其他窖泥中的豐度,表明甲烷菌群促進己酸乙酯合成的代謝能力特征較為特殊。由于五糧液獨特的釀酒工藝,如五種糧食配方和雙輪底發(fā)酵工藝,該工藝使得下層糟醅與窖泥間相互接觸的時間較普通糟醅發(fā)酵工藝的時間長1倍甚至更長時間,微生物以及營養(yǎng)物質在兩輪甚至三輪發(fā)酵的過程中在糟醅與窖泥相接處的界面相互交換,窖泥吸收糟醅中的營養(yǎng)物質,再在微生物尤其是產酸細菌的作用下代謝生成較高含量的酸類等,而這些香氣物質又在窖池內發(fā)酵水蒸氣的作用下轉移至糟醅中,糟醅中酵母菌、霉菌等產生的酯化酶類促進酸醇酯化,此外,窖泥本身含有的窖香物質在發(fā)酵過程中轉移至糟醅,此工藝大大提高了白酒中香氣物質的含量以及增加酒體的窖香味。
本研究中,不同空間位置窖泥中原核微生物群落的組成差異明顯(圖3)且與之關聯(lián)的原核微生物具有顯著差別(圖5b),這是由于窖池內糟醅中水分(黃水)從上層糟醅往底層糟醅緩慢浸落,處于底層的窖泥長期處于含水量極高的下層糟醅接觸,糟醅中的部分微生物隨黃水的滴落而遷移至窖池底部,進而下層窖泥中原核微生物群落的結構與中上層既有一定相似性,又有顯著差異。同時本研究發(fā)現(xiàn)有部分窖池中層窖泥的泉古菌門豐度高于下層窖泥,而部分窖池則呈相反情況(圖3),可能的原因有多種,原因之一可能是由于窖齡的差異,窖池內長期厭氧、高酸等環(huán)境造成了窖齡較大的樣品中甲烷菌豐度高于中層窖泥(圖2);原因之二可能是長期生產過程中班組根據每輪次糟醅狀況進行“看糟配料”工藝的差異,即根據每輪次糟醅的實際狀況控制五糧粉、曲粉和水的用量。鑒于本研究中樣品采集量較少,大量樣品采集并歸納總結其中的空間位置規(guī)律將是未來的研究重點。
綜上所述,本研究通過高通量測序手段調查了五糧液不同類型窖泥的原核微生物群落結構與多樣性,發(fā)現(xiàn)由于釀酒環(huán)境及工藝的獨特性,五糧液窖泥中蘊藏了顯著區(qū)別于其他濃香型白酒窖泥的原核微生物群落,尤其是群系復雜的厚壁菌門細菌、較高豐度的產己酸菌群以及促進己酸生成的甲烷菌群。因此,弄清五糧液釀酒生態(tài)各環(huán)節(jié)中微生物群落多樣性以及在傳統(tǒng)工藝操作條件下的微生物群落生理代謝特征、演替規(guī)律以及與環(huán)境的相關性等將是我們下一步重點研究的內容。
[1] 吳衍庸,薛堂榮,陳昭榮,等.五糧液老窖厭氧菌群的分布及其作用的研究[J].微生物學報,1991,31(4):299-307.
[2] 趙東,喬宗偉,彭志云.濃香型白酒發(fā)酵過程中酒醅微生物區(qū)系及其生態(tài)因子演變研究[J].釀酒科技,2007(7):37-39.
[3] AMANN R I,LUDWIG W,SCHLEIFER K H.Phylogenetic identification and in-situ detection of individual microbial-cells without cultivation[J].Microbiology Reviews,1995,59(1):143-169.
[4] 陶勇,徐占成,李東迅,等.窖泥細菌群落結構演替及其與環(huán)境因子的相關性[J].釀酒科技,2011(9):42-46.
[5] ZHENG J,LIANG R,ZHANG L Q,et al.Characterization of microbial communities in strong aromatic liquor fermentation pit muds of different ages assessed by combined DGGE and PLFA analyses[J].Food Research International,2013,54(1):660-666.
[6] ZHENG J,WU C,HUANG J,et al.Spatial distribution of bacterial communities and related biochemical properties in Luzhou-flavor liquor fermented grains[J].Journal of Food Science,2014,79(12):M2 491-M2 498.
[7] 鄭佳,張良,沈才洪,等.濃香型白酒窖池微生物群落特征[J].應用生態(tài)學報,2011,22(4):1 020-1 026.
[8] 劉琨毅,盧中明,鄭佳,等.濃香型白酒窖泥微生物群落PLFA指紋圖譜方法[J].應用與環(huán)境生物學報,2012,18(5):831-837.
[9] 秦臻,鄭佳,彭昱雯,等.生物標記法剖析傳統(tǒng)釀造用大曲微生物群落結構[J].食品科學,2011,32(11):165-170.
[10] 秦楠,栗東芳,楊瑞馥.高通量測序技術及其在微生物學研究中的應用[J].微生物學報,2011,54(4):445-457.
[11] 陶勇,芮俊鵬,李家寶,等.濃香型白酒窖泥中細菌和古菌的組成與多樣性[J].化工學報,2014,65(5):1 800-1 807.
[12] 鄧杰,黃治國,衛(wèi)春會,等.基于高通量測序的濃香型白酒窖池細菌群落結構分析[J].現(xiàn)代食品科技,2015,31(7):50-55.
[13] 李克亞,文章,鄧斌,等.不同窖齡窖泥原核生物多樣性的高通量測序研究[J].食品工業(yè),2016,36(6):121-125.
[14] Sequencing platform comparison tool[OL].http://www.illumina.com.cn/systems/sequencing-platform-comparison.html.
[15] 溫崇慶,何瑤瑤,薛明,等.高通量測序分析DNA提取引起的對蝦腸道菌群結構偏差[J].微生物學報,2016,56(1):130-142.
[16] 于春濤,項明,王蕾.濃香型白酒窖泥變質前后古菌群落差異分析[J].釀酒科技,2016(8):60-64.
[17] TAO Y,LI J,RUI J,XU Z,et al.Prokaryotic communities in pit mud from different-aged cellars used for the production of Chinese Strong-flavored liquor[J].Applied and Environmental Microbioliogy,2014,80(7):2 254-2 260.
[18] SEEDORF H,FRICKE W F,VEITH B,et al.The genome ofClostridiumkluyveri,a strict anaerobe with unique metabolic features[J].PNAS,2008,105(6):2 128-2 133.
[19] BOUANANE-DARENFED A,HANIA W B,HACENE H,et al.Caldicoprobacterguelmensissp.nov.a new thermophilic anaerobic,xylanolytic bacterium isolated from an Algerian hot spring[J].International Journal of Systematic Evolution and Microbiology,2013,63:2 049-2 053.
[20] REICHARD N,DUNCAN S H,YOUNG P,et al.Phylogenetic distribution of three pathways for propionate production within the human gut microbiota[J].ISME Journal,2014,8:1 323-1 335.
[21] PARSHINA S N,KLEEREBEZEM R,SANZ J L,et al.Soehngeniasaccharolyticagen.nov.,sp.nov.andClostridiumamygdalinumsp.nov.,two novel anaerobic,benzaldehyde-converting bacteria[J].International Journal of Systematic Evolution and Microbioliogy,2003,53(6):1 791-1 799.
[22] 吳衍庸,劉光燁.窖泥甲烷細菌與濃香型曲酒[J].釀酒,1988(2):29-32.
[23] 王忠彥,尹昌樹.白酒色譜骨架成分的含量及比例關系對香型和質量的影響[J].釀酒科技,2000(6):93-96.
ProkaryoticmicrobialcommunityinWuliangyepitmudusingNextSeqhigh-throughputsequencingtechnology
ZHAO Dong,ZHENG Jia*,PENG Zhi-yun,LYU Xue-lan, YANG Kang-zhuo,ZHANG Jian-min
(Technology Research Center of Wuliangye Yibin Co. Ltd.,Yibin 644000,China)
In order to well-understand the specific brewing microbial community of Wuliangye pit mud and its function. The 16S ribosomal DNA V4 sequences of prokaryotic community in Wuliangye pit muds from different positions were investigated by Illumina NextSeq 500 next-generation sequencing (NGS) platform for the first time. Numbers of DNA sequences and operation taxonomy units (OTUs) were summarized by optimized QIIME program, and the abundance and diversity indices of prokaryotic microbial community were calculated. A total of 7 555 164 high quality sequences were obtained and under the same sequencing depth, bacteria (92.3%) and archea (7.6%) were determined as main microbial communities. 22 phyla, 52 classes, 89 orders, 168 family, 330 genera were identified by optimized alignment method. Fimicutes was the predominant bacteria in all pit muds with the abundance of 85.6%,Clostridium,Lactobacillus,Coprococcus,Caldicoprobacter, andSoehngeniawere dominant genus. Furthermore, according to the phylogenetic analysis of dominant OTUs (>1%), the dominant genus in pit mud includedLactobacillus,Caldicoprobacter,Caloramater,Clostridium,Caloribacterium,Garciella,Eubacterium,Syntrophomonas,Sedimentbacter,Sporanaerobacter,Tissierella,Methanosarcina,Methanobacterium,Methanobrevibacter, andMethanoculleus. Wuliangye pit mud contained the specific prokaryotic community in comparison with otherStrong-aromaliquors, and it consisted of complex Firmicutes, high percentage of hexanoic acid-production community and methane community. The specific prokaryotic community can promote the formation of hexanoic acid.
Wuliangye; pit mud; prokaryotic community; NGS
教授級高工,碩士生導師,中國釀酒大師(鄭佳研究員為通訊作者,E-mail:zhengwanqi86@163.com)。
固態(tài)發(fā)酵資源利用四川省重點實驗室開放基金(2015GTY005);宜賓市重點科技項目(2016GY009)
2017-02-26,改回日期:2017-05-12
10.13995/j.cnki.11-1802/ts.014156

