熒光定量PCR法計數農家干酪中動物雙歧桿菌乳酸亞種
陳雪,劉韓,裴芳藝,劉振艷,侯惠玲,郭皓,丁琳,
楊文欽1,臧傳剛1,劉宇超1,關宏1*
1(齊齊哈爾醫學院 醫藥科學研究院,黑龍江 齊齊哈爾,161006) 2(齊齊哈爾醫學院,學院辦公室,黑龍江 齊齊哈爾,161006)
熒光定量PCR法計數農家干酪中動物雙歧桿菌乳酸亞種
陳雪1,劉韓2*,裴芳藝1,劉振艷1,侯惠玲1,郭皓1,丁琳1,
楊文欽1,臧傳剛1,劉宇超1,關宏1*
1(齊齊哈爾醫學院 醫藥科學研究院,黑龍江 齊齊哈爾,161006) 2(齊齊哈爾醫學院,學院辦公室,黑龍江 齊齊哈爾,161006)
分別利用熒光定量PCR及平板菌落計數法計數農家干酪中雙歧桿菌的菌體數,通過對結果的比較分析,建立一種適用于快速、敏感、特異的檢測干酪中雙歧桿菌活菌數的方法。利用傳統工藝制備雙歧桿菌農家干酪,在其貯存期間分別采用熒光定量PCR及平板菌落計數法計數干酪中雙歧桿菌的數量。其中,在利用熒光定量PCR法檢測時,對影響PCR定量準確的因素進行系統研究,包括設計雙歧桿菌引物,并對引物特異性進行評價、考察,從干酪基質中提取DNA的數量和質量,建立標準曲線。引物特異性驗證結果表明,引物專一性強。采用試劑盒法從干酪樣品中提取DNA的純度較好,OD260/OD280均在1.75~1.82之間。除貯存第1天外,熒光定量PCR法計數結果比平板計數法高0.39%~2.25%,未見顯著差異。熒光定量PCR具有靈敏、特異、簡便和快速的特點,可用于干酪中雙歧桿菌的定量檢測。
熒光定量PCR;干酪;雙歧桿菌
雙歧桿菌(Bifidobacterium)是一種厭氧的革蘭氏陽性桿菌,它作為維持人體腸道菌群平衡的有益菌[1],被廣泛應用于乳制品的制作中,如發酵乳、冰激凌、酸奶及干酪等[2]。不僅如此,它還具有提高機體免疫力[3]、降低結腸癌發病率、抗腫瘤、延緩衰老等一系列保健功能[4-6],近年來備受人們青睞。與酸奶和發酵乳相比,干酪作為雙歧桿菌等益生菌載體更具有優勢:(1)干酪pH值相對較高,一般在pH 4.8~5.6之間,高于發酵乳的pH 3.7~4.3。(2)厭氧環境。干酪結構致密,內部氧化還原電位極低,其內部環境可以認為是厭氧環境,這有利于雙歧桿菌類嚴格厭氧的益生菌生長。(3)緩沖力強。干酪結構致密且蛋白、脂肪含量高,這為益生菌通過胃腸道提供了保護作用[7-8]。
在益生菌食品中只有當益生菌保持一定活力且具有一定數量時才能發揮其益生作用。然而,市場上銷售的益生菌產品通常會出現益生菌活菌數較低甚至不含益生菌等問題。事實表明,大多數菌株均具有非常相似的表型特性,利用傳統平板培養法計數時不僅費時且選擇性差,只能計數能夠生長的菌株而對處于活的非可培養狀態的細菌無法檢測。此外,在混合發酵體系中,當其他菌株數量占優勢且也能在相同的培養基生長時也無法用平板法檢測其益生菌數[9]。因此,需要建立一種有效和快速的方法來鑒別益生菌同時計數活菌數[10]。實時熒光定量PCR是在PCR定性技術基礎上發展起來的核酸定量技術,在體系中加入熒光基團(通常為熒光染料或探針),實時通過熒光信號的積累監測PCR的進程[11]。該方法以菌體DNA為模板,通過標準曲線來確定菌株在樣品中的數量,具有敏感性高、特異性強、省時等優點,近年來該方法廣泛應用于發酵乳制品中益生菌和致病菌的檢測。
本研究以實驗室保藏的動物雙歧桿菌乳酸亞種QYW-BB06為研究對象,將其作為附屬發酵劑添加到農家干酪中。在干酪貯存的20天內,每隔5天利用熒光定量PCR方法檢測干酪中雙歧桿菌的活菌數,并與傳統平板計數方法作對比。同時,對影響PCR定量準確的因素:引物特異性、在干酪基質中提取DNA的數量和質量、標準曲線的建立進行系統研究,旨為建立一種準確、快速的測定農家干酪中雙歧桿菌數量的方法提供理論依據。
1 材料與方法
1.1材料
1.1.1 菌種
長雙歧桿菌長亞種(Bifidobacteriumlongumsubsp.longum)QYW-LB01,動物雙歧桿菌乳酸亞種(Bifidobacteriumanimalissubsp.lactis)QYW-BB06。以上菌種均保藏于齊齊哈爾醫學院微生態工程技術研究中心。
1.1.2 培養基
(1)脫脂乳培養基:100 g/L 脫脂乳,10 g/L 酵母精;(2)改良MRS培養基:在MRS培養基中添加0.5 g/L的半胱氨酸[12];(3)含莫匹羅星鋰鹽的MRS培養基:在改良MRS培養基中加入莫匹羅星鋰鹽50 mg/L;(4)M17培養基:購買于青島海博生物有限公司。
1.2儀器與設備
Wizard Genomic DNA Purification Kit,Promega公司;SYBR Premix Ex TaqTM,TaKaRa公司;ABI7300實時熒光 PCR 儀,ABI公司;梯度PCR儀、全自動熒光和化學發光成像分析系統,Bio-RAD公司;厭氧操作培養箱,SHEL LAB 公司;二氧化碳培養箱、Nano Drop紫外分光光度計,賽默飛公司。
1.3菌株復蘇及DNA提取
將保藏的菌種分別以3區劃線方式接種到固體改良MRS培養基中,37 ℃、厭氧培養48 h后用滅菌牙簽挑取單菌落,分別接種到1 mL新鮮液體改良MRS培養基中,37 ℃、厭氧培養24 h。按5%的接種量繼續擴培菌體,待菌體活力恢復后備用。
菌體DNA提取方法按照Wizard Genomic DNA Purification Kit的說明書步驟進行提取,將溶菌酶的濃度提高至50 mg/mL。取適量干酪發酵劑(含乳酸乳球菌)用M17培養基擴培菌體后直接提取DNA。
1.4繪制熒光定量PCR標準曲線
取經連續10倍稀釋的已知量B.animalissubsp.lactisQYW-BB06DNA作為熒光定量PCR模板,以其已知量的不同菌數對數值為橫坐標(lg CFU/g),以反應過程中出現熒光信號的初始循環數(Ct)為縱坐標制作標準曲線。標準曲線方程:Y=-3.113x+39.121,斜率為-3.113,線性回歸系數R2為0.995 95,PCR擴增效率E=101/3.113-1=1.095,即109.5%。通過測得的Ct值代入標準曲線方程中求出對應的菌數對數值,再反求其對數,計數樣品中的雙歧桿菌。
1.5引物設計及引物特異性驗證
1.5.1 引物設計
動物雙歧桿菌特異性引物的設計基于延伸因子tal基因的1個片段。引物序列及擴增產物長度如表1所示,由生工生物工程(上海)股份有限公司合成。引物均用TE稀釋制成100 μmol/L的貯存液。

表1 試驗所用引物
1.5.2 PCR法對引物特異性的驗證
采用PCR法,用雙歧桿菌特異性引物擴增目的菌株DNA模板(B.animalissubsp.lactisQYW-BB06)及非目的菌株DNA模板(乳酸乳球菌)。PCR反應體系:模板DNA 1 μL,10×buffer 2 μL,dNTPS(2.5 mmol/L)1.6 μL,雙歧桿菌上下游引物(10 μmol /L)各0.4 μL,r-Taq(5v/μL)0.4 μL,滅菌雙蒸水補足至20 μL,設置3個平行樣品。反應條件:94 ℃ 10 min,94 ℃ 30 s,60 ℃ 30 s,72 ℃ 40 s,72 ℃ 10 min,4 ℃ ∞,橫線處共20個循環。PCR產物經2%瓊脂糖凝膠電泳進行檢測。
1.5.3 熒光定量PCR法對引物特異性的驗證[9]
采用熒光定量PCR法,用雙歧桿菌特異性引物分別擴增目的菌株DNA模板(B.animalissubsp.lactisQYW-BB06)及非目的菌株DNA模板(乳酸乳球菌、B.longumsubsp.longumQYW-LB01)。在反應體系中,各菌株DNA模板量為50 ng。
將目的菌株DNA與非目的菌株DNA混合,且在非目的菌株DNA濃度較高的體系中驗證引物的特異性。目的菌株DNA與非目的菌株DNA濃度比設置為1∶1、1∶50、1∶150及1∶1 000。
熒光定量PCR體系及反應條件:熒光定量PCR反應體系:SYBR premix ExTaq10 μL、正反向引物(10 μmol/L)各0.4 μL、ROX reference dye 0.4 μL、DNA模板2 μL及滅菌雙蒸水6.8 μL補足至20 μL,每個DNA模板設置3個復孔。反應條件:95 ℃ 30 s,95 ℃ 5 s,60 ℃ 31 s,橫線處共40個循環。
1.6農家干酪樣品
樣品制作工藝流程:鮮牛乳→過濾、殺菌(63 ℃,30 min)→冷卻(32 ℃)→接種動物雙歧桿菌乳酸亞種(108CFU/mL接種)→接種商業直投式發酵劑(包含乳酸乳球菌乳酸亞種及乳酸乳球菌乳脂亞種)→添加氯化鈣(0.1 g/L)→添加凝乳酶→凝乳→切割凝塊→靜置→熱燙→排乳清→水洗→堆積→鹽漬→包裝,4 ℃貯存。
1.7計數干酪樣品中的雙歧桿菌
在干酪貯存的第1、5、10、15、20天時分別使用平板菌落計數法及熒光定量PCR法計數干酪中雙歧桿菌菌體數。
1.7.1 從干酪樣品中取樣
無菌條件下稱取10 g干酪樣品與90 mL滅菌的2%檸檬酸三鈉-4%聚乙二醇8 000混合,室溫下放置10 min,勻漿5 min。取1 mL混合溶液,使用含莫匹羅星鋰鹽的MRS培養基,傾注法進行平板菌落計數;另取1 mL混合溶液提取DNA。使用Wizard Genomic DNA Purification Kit提取干酪樣品中DNA,具體操作參照ABDULAMIR等人的方法[14]。利用紫外分光光度法分析DNA的濃度及純度。DNA純度的表達方式為OD260與OD280的比值。
1.8統計學分析
應用SPSS17.0軟件中的方差分析對試驗數據進行差異顯著性分析,當p<0.05為差異顯著;當p< 0.01為差異極顯著。
2 結果與分析
2.1PCR法對引物特異性的驗證結果
選取干酪發酵劑中乳酸乳球菌為非目的模板進行引物特異性驗證。分別以B.animalissubsp.lactisQYW-BB06DNA、乳酸乳球菌DNA、二者以不同比例混合的DNA為模板,以動物雙歧桿菌引物進行PCR反應。PCR產物經2%瓊脂糖凝膠電泳進行檢測。如圖1所示:泳道1、3、4均在100 bp偏上處有1條清晰條帶,與擴增片段大小相符,為116 bp。泳道2為非特異性擴增,沒有擴增條帶,說明動物雙歧桿菌引物不能擴增乳酸乳球菌DNA。泳道3、4為目的DNA與非目的DNA以不同比例混合為模板,均只有一條清晰條帶且大小與特異性擴增(泳道1)相同。說明引物特異性強,在混合模板條件下未發生非特異性擴增。
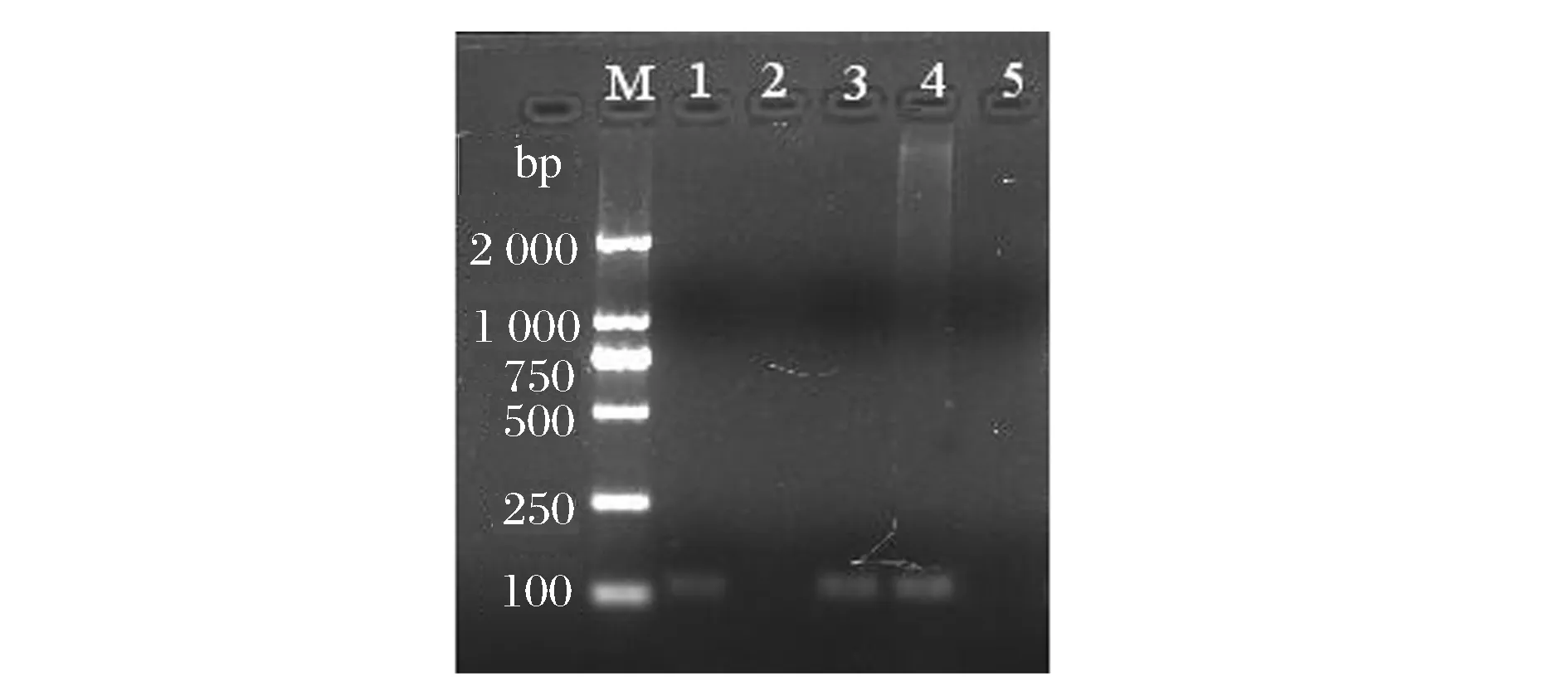
M-DNA marker; 1-動物雙歧桿菌DNA(目的菌株DNA); 2-乳酸乳球菌DNA(非目的菌株DNA);3-1 ng目的菌株DNA與 1 ng非目的菌株DNA混合;4-1 ng目的菌株DNA 與 50 ng非目的菌株DNA混合;5-水圖1 PCR法對引物特異性的驗證結果Fig.1 Verification of primer specificity by PCR
2.2熒光定量PCR方法對引物特異性的驗證結果
采用熒光定量PCR方法對引物特異性進行驗證,選取乳酸乳球菌、B.longumsubsp.longumQYW-LB01 DNA為陰性對照。結果見表2,動物雙歧桿菌引物擴增非目的模板得到的Ct值為29.99和無計算結果(noCt)。動物雙歧桿菌引物擴增目的模板得到的Ct值為14.73。

表2 熒光定量PCR檢測引物特異性
注:1. 在反應體系中,各菌株DNA模板量為50 ng;2. 加粗的Ct值為目的菌株DNA。
在復雜的微生物體系中,熒光定量PCR擴增的目的模板不一定是數量占優勢的菌屬,因此有必要在目的DNA和非目的DNA混合體系中驗證引物的特異性。在含有大量非目的DNA存在的條件下,動物雙歧桿菌引物擴增目的DNA時所得的Ct值差異不明顯(表3)。在目的DNA與乳酸乳球菌DNA混合體系中,所有按不同比例混合的模板所得的Ct值差異不顯著,Ct值的變化(ΔCt)僅從0.01到0.19。在目的DNA與B.longumsubsp.longumQYW-LB01 DNA混合體系中,僅在比例為1∶1 000的體系中ΔCt值較大為1.17,其余按不同比例混合的模板ΔCt值僅從0.02到0.10。說明在含有這2種非目的模板存在的條件下,動物雙歧桿菌引物能夠特異性擴增目的模板。
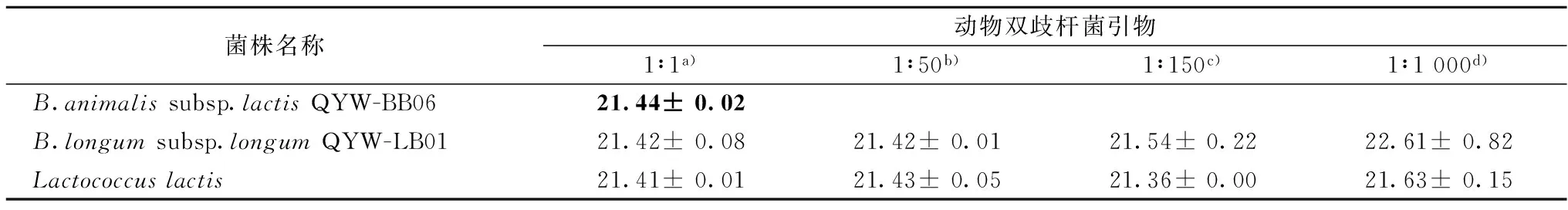
表3 熒光定量PCR檢測引物特異性
注:無非目的DNA模板混合的陽性對照用加粗的Ct值表示;1.a)1 ng 目的 DNA 與 1 ng 非目的DNA 混合;2.b)1 ng目的DNA 與 50 ng 非目的DNA混合;3.c)1 ng 目的 DNA與150 ng非目的DNA混合;4.d)1 ng目的DNA與 1 000 ng非目的DNA混合。
2.3從干酪樣品中提取DNA
從干酪樣品中提取DNA的數量和質量直接影響定量的準確性,所以選擇一種合適的DNA提取方法顯得尤為重要。從干酪樣品中提取DNA的濃度及純度結果見表4。結果顯示,從干酪樣品中提取DNA的純度較高,OD260/OD280值均在1.75~1.82之間。DNA的質量濃度變化較大從16.4~24.6 ng/μL不等。干酪貯存第1天時所提取的DNA量最少,隨貯存時間的延長,提取DNA的量逐漸增多。
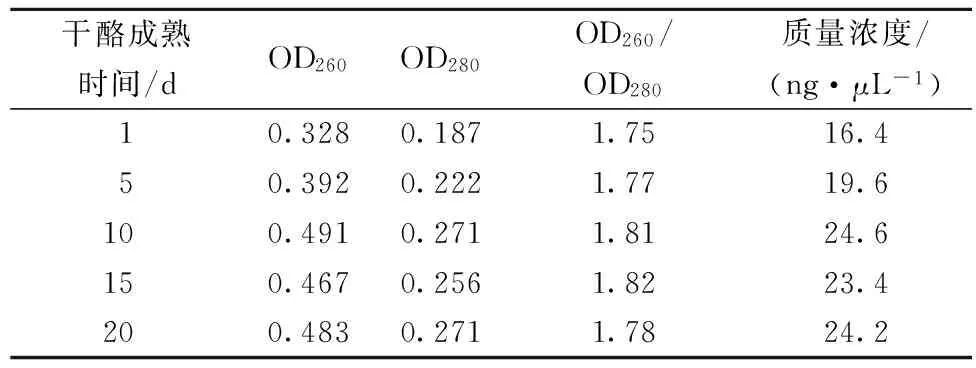
表4 干酪貯存期間提取DNA的純度、濃度檢測結果
2.4熒光定量PCR法與平板計數法計數干酪中雙歧桿菌
農家干酪是一種典型的非成熟、新鮮軟質干酪。由于其貨架期短,所以選擇1、5、10、15、20天進行雙歧桿菌的檢測。分別提取1、5、10、15、20天的干酪樣品DNA(表4),用于熒光定量PCR的檢測。圖3所示,熒光定量檢測與傳統平板菌落計數法檢測結果差異不顯著(p>0.05)。除干酪貯存的第1天外,熒光定量方法檢測雙歧桿菌活菌數均高于平板計數方法,這可能與在貯存第1天從干酪中提取DNA數量較少有關。在貯存期內,干酪樣品中B.animalissubsp.lactisQYW-BB06的活菌數均大于108CFU/mL。

圖3 熒光定量PCR法與平板計數法計數干酪中雙歧桿菌Fig.3 Comparison of Bifidobacterium animalis subsp. lactis count (lg CFU/mL) obtained by real-time PCR and plate count
3 討論
采用平板計數法和熒光定量PCR法分別計數干酪中B.animalissubsp.lactisQYW-BB06數量。在干酪的貯存期內,采用平板計數方法計數雙歧桿菌的活菌數時,活菌數在8.0~8.3 lg CFU/mL內變化。采用熒光定量PCR法計數的雙歧桿菌數時,在干酪貯存的第1天,菌數(8.2 lg CFU/mL)量略低于平板計數法(8.3 log CFU/mL),這可能與提取DNA數量較少有關;在貯存的第5、10、15、20天,熒光定量PCR計數的雙歧桿菌數量(8.1~8.3 lg CFU/mL)均高于平板計數法(8.0~8.2 lg CFU/mL)。這是由于熒光定量PCR法是基于干酪樣品中細菌DNA濃度和純度進行計數的,而死菌、處于活的非可培養狀態的DNA均可被提出,無法區分活菌及死菌,導致所有狀態的菌體DNA一同被擴增,使結果偏高[15]。由此,可以看出所得DNA的濃度和純度對熒光定量PCR計數的雙歧桿菌數量影響較大。大多數乳制品中的DNA是從其制品內所包含的微生物提取得到的,乳制品中DNA的回收率應該是恒定的并且盡可能地高。LARPIN研究指出,干酪是一種“濃縮”的乳制品,營養成分豐富,蛋白質含量一般在20%~35%,脂肪含量通常≥25%,相當于將原料乳中的蛋白質和脂肪濃縮10倍,這種復雜的組成成分會直接導致DNA的提出率降低[16]。所以干酪在為益生菌提供良好保護作用的同時也為提取菌體DNA提高了難度。此外,復雜的乳制品成分中可能還含有PCR反應的抑制因子[17-19]。通常,從乳制品中提取DNA的第一步是將菌體細胞從乳制品中分離出來,這不僅簡化了后續DNA的提取步驟,還消除了大多數與乳制品相關的反應抑制劑。目前,已有許多課題組對此方面進行研究,BARUZZI將干酪樣品與檸檬酸鈉溶液混合,用機械裝置或玻璃珠勻漿使酪蛋白溶解[20]。FLREZ通過多次用緩沖液清洗菌體細胞,并添加Triton X-100或蛋白酶來去除酪蛋白[21]。STEVENS在均漿階段加入聚乙二醇,更好地回收菌體[22]。CHRISTINE采用2%檸檬酸三鈉-4%聚乙二醇8000緩沖液將菌體從干酪中分離出來后提取DNA,得到DNA的濃度較高且純度OD260/OD280值均在1.80~2.11[10]。ABDULAMIR比較了4種從復雜基質中提取DNA的方法,以提取DNA的濃度和純度為評價指標,得出采用Wizard Genomic DNA Purification Kit提取DNA質量最高[14]。通過綜合比較分析,選用2%檸檬酸三鈉-4%聚乙二醇8000緩沖液將菌體從干酪樣品種分離出來,然后利用Wizard試劑盒提取其DNA,獲得DNA純度較高,OD260/OD280值均在1.75~1.82。DNA的質量濃度變化從16.4~23.4 ng/μL。
研究發現,疊氮類染料(疊氮溴化丙錠PMA,疊氮溴化乙錠EMA)可以抑制非活性菌體DNA的PCR擴增,這可進一步降低熒光定量PCR法計數時出現的假陽性結果[23-24]。éMILIE[13]在使用熒光定量PCR方法檢測干酪在制作和成熟期間雙歧桿菌、鼠李糖乳桿菌和瑞士乳桿菌的活菌數時,使用PMA處理從樣品中提取的DNA。結果表明,PMA可以抑制死菌的DNA擴增,這是由于PMA與樣品中死細胞的DNA分子共價交聯,抑制該DNA的PCR擴增。但必須指出的是本法雖然可以抑制死菌DNA的擴增,但無法去除全部已滅活的細胞對該試驗產生的影響,這是由于已經滅活的細菌仍可能具有完整的細胞膜結構,而疊氮類染料是以細胞膜完整性作為細菌活性的檢測標準。因此,疊氮類核酸染料檢測法對不同細菌滅活方式的適用性還有待研究。應當指出,研究使用的兩種計數法得出的結果差異不顯著(p>0.05)。前期研究結果表明,B.animalissubsp.lactisQYW-BB06具有很強的耐酸、耐膽鹽性,這為其在干酪中大量存活提供前提條件[25]。在20天的貯存期內,雙歧桿菌活菌數僅微量降低,活菌數遠大于死菌數。這可能是本文未使用疊氮類核酸染料處理提取的DNA,所用兩種計數方法得出的結果仍差異不顯著的原因。
本研究使用的熒光基團是非特異性的SYBR Green熒光染料,它具有使用方便不必設計復雜探針、反應靈敏價格便宜等優點。但主要的缺點就是容易與非特異性雙鏈DNA結合,產生假陽性的結果。因此,為了減少假陽性結果的出現,就必須盡可能提高引物特異性。干酪是一種典型的多菌種發酵的產物,其發酵劑通常包括2種乳酸乳球菌。所以,在試驗中應用的引物要求只能擴增出目的DNA(B.animalissubsp.lactisQYW-BB06DNA),而不能擴增非目的DNA。SHEU[26]基于tuf基因分別為動物雙歧桿菌動物亞種、動物雙歧桿菌乳亞種、兩歧雙歧、短雙歧、嬰兒雙歧和長雙歧桿菌設計了引物。在熒光定量PCR反應中,每一個目的DNA與其對應的引物都擴增出與預期大小相等的目的片段,且溶解曲線只有單峰。同時,將每一種引物與其非目的菌株DNA混合均得出陰性結果。在CHRISTINE[10]的研究中,給出了更加具體的引物特異性驗證方法。將各菌株引物分別擴增目的菌株DNA(陽性對照)和非目的菌株DNA(陰性對照)或將目的菌株DNA與非目的菌株DNA按不同比例混合,觀察Ct值變化。結果表明,設計的引物特異性強,對目的DNA進行擴增而非目的DNA不擴增。本研究使用的引物參考éMILIE[13]等人的研究,通過普通PCR及熒光定量PCR兩種方法進行特異性驗證。結果表明該引物特異性強,對干酪發酵劑中的乳球菌不擴增(無條帶、無Ct值);當擴增與目的菌株同源性較近的長雙歧桿菌長亞種DNA時,所得Ct>29且Tm與目的菌株不同。當目的菌株DNA與大量非目的菌株DNA混合時,不影響目的菌株DNA的擴增,所得Ct差異不顯著,這與CHRISTINE[10]的研究結果相似。
[1] MASCOL,VANHOUTTE T,TEMMERMAN R,et al.Evaluation of real-time PCR targeting the 16S rRNA andrecAgenes for the enumeration of bifidobacteria in probiotic products[J].International Journal of Food Microbiology,2007,113(3):351-357.
[2] YI C W,ROCH C Y,CHENG C C.Growth and survival of bifidobacteria and lactic acid bacteria during the fermentation and storage of cultured soy milk drinks[J].Food Microbiology,2002,19(5):501-508.
[3] TOJO M,OIKAWA T,MORIKAWA Y,et al.The effects ofBifidobacteriumbreveadministration on campylobacter enterits[J].Acta Paediatr,1987,29(1):160-167.
[4] CANDELA M,PERNA F,CAMEVALI P,et al.Interaction of probioticLactobacillusandBifidobacteriumstrains with human intestinal epithelial cells:Adhesion properties,competition against enteropathogens and modulation of IL-8 production[J].International Journal of Food Microbiology,2008,125(3):286-292.
[5] LINDA JB,DIANIEL DG,FRANK FB.The role of probiotic cultures in prevention of colon cancer[J].The Journal of Nutrition,2000,130(2):410-414.
[6] OUWEHAND AC,SALMINEN S,ISOLAURI E.Probiotics:an overview of beneficial effects[J].Antonie Van Leeuwenhoek,2002,82(3):279-289.
[7] 賈宏信,龔廣予,郭本恒.益生菌干酪的研究進展[J].食品科學,2013,34(15):355-360.
[8] TERRI DB,CELSO GV,HAMID BG,et al.Incorporation of bifidobacteria into cheeses:challenges and rewards[J].International Dairy Journal,2004,14(5):375-387.
[9] CHRISTOPHE M,BOJANA BM.Application of PCR-based methods to dairy products and to non-dairy probiotic products[J].Polymerase Chain Reaction,2012,15:11-50.
[10] CHRISTINE A,FRANCOISE B.Quantitative PCR for the specific quantification ofLactococcuslactisandLactobacillusparacaseiand its interest forLactococcuslactisin cheese samples[J].Food Microbiology,2013,36:286-295.
[11] 朱軍偉,杭鋒,王欽博,等.實時熒光定量PCR技術在乳品微生物學中的應用研究進展[J].安徽農業科學,2014,42(15):4 764-4 766.
[12] MIRANDA RO,NETO GG,FREITAS RD,et al.Enumeration of bifidobacteria using PetrifilmTMAC in pure cultures and in a fermented milk manufactured with a commercial culture ofStreptococcusthermophilus[J].Food Microbiology,2011,28(8):1 509 -1 513.
[13] éMILIE DF,VERONIQUE DL,CLEMENTINE LB,et al.Assessment of probiotic viability during Cheddar cheese manufacture and ripening using propidium monoazide-PCR quantification[J].Food Microbiology,2012,3:1-11.
[14] ABDULAMIR AS,YOKE TS,NORDIN N,et al.Detection and quantification of probiotic bacteria using optimized DNA extraction,traditional and real-time PCR methods in complex microbial communities[J].African Journal of Biotechnology,2010,9(10):1 481-1 492.
[15] EUNICE CI,MIRELLA CS,DIANA T,et al.Comparison of real-time PCR assay and plate count forLactobacillusparacaseienumeration in yoghurt[J].Annals of Microbiology,2015,66(2):1-10.
[16] LARPINS,MONDOLONIC,GOERGESS,et al.Geotrichumcandidumdominates in yeast population dynamics in Livarot,a Frenchred-smear cheese[J].Fems Yeast Research,2006,6(8):1 243-1 253.
[17] NIEDERHAUSERC,CANDRIANU,HOFELEINC,et al.Use of polymerase chain-reaction for detection ofListeriamonocytogenesin food[J].Applied & Environmental Microbiology,1992,58(5):1 564-1 568.
[18] ROSSENL,NORSKOV P,HOLMSTROMK,et al.Inhibition of PCR by components of food samples,microbial diagnostic assays and DNA-extraction solutions[J].International Journal of Food Microbiology,1992,17(1):37-45.
[19] HERMANL,DERIDDERH.Cheese components reduce the sensitivity of detection ofListeriamonocytogenesby the polymerase chain-reaction[J].Netherlands Milk& Dairy Journal,1993,47(1):23-29.
[20] BARUZZIF,MATARANTEA,CAPUTOL,et al.Development of a culture-independent polymerase chain reaction-based assay for the detection of lactobacilli in stretched cheese[J].Journal of RapidMethods Automationin Microbiology,2005,13(3):177-192.
[21] FLOREZAB,MAYOB.Microbial diversity and succession during the manufacture and ripening of traditional,Spanish,blue-veined Cabrales cheese,as determined byPCR-DGGE[J].International Journal of Food Microbiology,2006,110(2):165-171.
[22] STEVENSKA,JAYKUSLA.Direct detection of bacterial pathogens in representative dairy products using a combined bacterial concentration-PCR approach[J].Journal of Applied Microbiology,2004,97(6):1 115-1 120.
[23] BARBAU PE,MAHILLON J,PILLYSER J,et al.Evaluation of viability-qPCR detection system on viable and deadSalmonellaserovarenteritidis[J].Journal of Microbiological Methods,2014,103:131-137.
[24] ELIZAQUIVEL P,AZNAR R,SANCHEZ G.Recent developments in the use of viability dyes and quantitative PCR in the food microbiology field[J].Journal of Applied Microbiology,2013,116(1):1-13.
[25] 陳雪,劉韓,楊文欽,等.雙歧桿菌新鮮干酪的研制及其感官評價[J].中國乳品工業,2015,43(6):24-27.
[26] SHEU SJ,HWANG WZ,CHIANG YC,et al.Use oftufgene-based primers for the PCR detection of probioticBifidobacteriumspecies and enumeration ofBifidobacteriainfermented milk by cultural and quantitative real-time PCR methods[J].Journal of Food Science,2010,75(8):521-527.
AssessmentofBifidobacterialviabilityduringcheeseripeningbyreal-timePCRquantification
CHEN Xue1,LIU Han2*,PEI Fang-yi1,LIU Zhen-yan1,HOU Hui-ling1,GUO Hao1, DING Lin1,YANG Wen-qin1,ZANG Chuan-gang1,LIU Yu-chao1,GUAN Hong1*
1(Research Institute of Medicine and Pharmacy,Qiqihar Medical University,Qiqihar 161006,China) 2(Faculty Office,Qiqihar Medical University,Qiqihar 161006,China)
To establish a simple, sensitive, accurate and rapid detection method for bifidobacteria in cheese, we compared real-time PCR (qPCR) and plate counts.Bifidobacteriumanimalissubsp.lactisQYW-BB06 (cfu) enumerated by qPCR were compared to culturableBifidobacteriumanimalissubsp.lactisenumerated by plate counts at 1, 5, 10, 15 and 20 days of cheese manufacture. The specificity of each primer set was assessed by qPCR and PCR, the yield and purity of DNA extracted from cheese were evaluated, and the standard curve was established. Target DNA was successfully amplified to show a single peak on the amplicon melting curve, non-target DNA was not amplified. High DNA yield and quality were obtained from cheese sample with mean OD260/OD280ratios ranging from 1.75 to 1.82. Besides of the first day of cheese storage, qPCR counts were higher than plate counts, values ranged from 0.39 to 2.25%. Real-Time PCR assay can be used in detection of bifidobacteria quantitatively in cheese.
real-time polymerase chain reaction; cheese;Bifidobacterium
碩士,助理研究員(關宏教授、劉韓工程師為通訊作者,E-mail:1352354944@qq.com,liuhan204@163.com )。
黑龍江省教育廳項目(產B族維生素乳酸菌的篩選及生物特性研究2016-KYYWF-0890);齊齊哈爾市科技局項目(SFGG-201557)
2017-04-18,改回日期:2017-05-23
10.13995/j.cnki.11-1802/ts.014556

