浙江省三個斑鱖野生群體線粒體DNA細胞色素b基因序列的遺傳變異
許曉軍,孟慶輝,陳小明
(浙江省水產技術推廣總站,浙江 杭州 310023)
浙江農業學報ActaAgriculturaeZhejiangensis, 2017,29(10): 1637-1641
許曉軍,孟慶輝,陳小明. 浙江省三個斑鱖野生群體線粒體DNA細胞色素b基因序列的遺傳變異[J].浙江農業學報,2017,29(10): 1637-1641.
10.3969/j.issn.1004-1524.2017.10.06
2017-03-15
2013年度浙江省農業標準化研究項目“浙江省斑鱖種質特性研究”
許曉軍(1979—),男,河南安陽人,博士研究生,高級工程師,從事水生生物遺傳育種方向研究。E-mail: 24587980@qq.com
浙江省三個斑鱖野生群體線粒體DNA細胞色素b基因序列的遺傳變異
許曉軍,孟慶輝,陳小明
(浙江省水產技術推廣總站,浙江 杭州 310023)
對錢塘江上游江山、烏溪江段和甌江上游云和段3個斑鱖(SinipercascherzeriSteindachner)群體線粒體DNA細胞色素b(Cytb)基因片段進行了擴增和測序,比較并分析了其遺傳多樣性和群體遺傳分化。結果顯示,在斑鱖Cytb基因965 bp的部分序列中,A+T含量(52.11%)略高于G+C含量(47.89%),77個個體中發現了16個多態性位點,8種單倍型。江山群體、烏溪江群體和云和群體的單倍型多樣性分別為0.570±0.078、0.668±0.067、0.667±0.032,核苷酸多樣性分別為0.001 8±0.000 3、0.001 9±0.000 2、0.004 3±0.000 6。江山群體與烏溪江群體間的Fst為-0.005 16(P>0.05),表明同為錢塘江上游的兩群體沒有明顯分化;但云和群體與江山群體、烏溪江群體間Fst分別為0.191 23(P<0.01)和0.178 10(P<0.01),表明甌江上游與錢塘江上游斑鱖群體存在顯著的分化。
斑鱖;細胞色素b基因;遺傳變異
斑鱖(SinipercascherzeriSteindachner)隸屬鱸形目(Perciformes)、鱸亞目(Percoidei)、鮨科(Serranidae)、鱖亞科(Sinipercinae)、鱖屬(Siniperca),俗稱肉鱖、褐鱖、巖鱖等,為東亞特有種類,廣泛分布于我國內陸水域,在朝鮮和越南也有分布[1-2]。20世紀末期,國內學者從形態學層次對鱖亞科進行了重新整理,修正了斑鱖命名上的一些同物異名情況,基于同工酶的研究未能發現不同地區斑鱖存在顯著差異[3]。
隨著分子生物學技術的快速發展,21世紀初,線粒體基因測序技術被用于斑鱖的群體遺傳學研究。mtDNA高度變異控制區(control region, CR)和細胞色素b(cytochrome b,Cytb)研究結果顯示,我國各斑鱖野生群體具有較為豐富的遺傳多樣性,各群體間有明顯的遺傳分化,錢塘江群體的分化較為特別[4]。浙江省各主要水系上游區段均有較豐富的斑鱖野生資源分布。天然水域生態環境破壞以及過度捕撈,使斑鱖的野生資源正在下降[5]。加之斑鱖人工繁殖技術的成功和發展[6-7],人工繁育群體逃逸、無序的異地引種等現象日益突顯。使各地天然斑鱖種質資源面臨基因污染的嚴峻考驗。mtDNACytb進化速度適中,是魚類系統發育學、群體遺傳學研究應用較多的重要分子標記[8]。因此,本研究擬通過對我省不同斑鱖野生群體mtDNACytb序列進行分析,初步了解其遺傳多樣性和遺傳分化情況,為對其合理開發和保護管理提供資料和參考。
1 材料與方法
1.1 樣品采集
斑鱖3個地理群體分別采自錢塘江上游江山港段(江山碗窯水庫)、烏溪江段(遂昌烏溪江水庫)和甌江上游大溪云和段(云和緊水灘水庫)。活體運輸至實驗室進行后續實驗,采集地點及樣品數量見表1。
1.2研究方法
1.2.1 基因組DNA提取
表1斑鱖樣品采集情況
Table1Sampling information ofSinipercascherzeriSteindachner
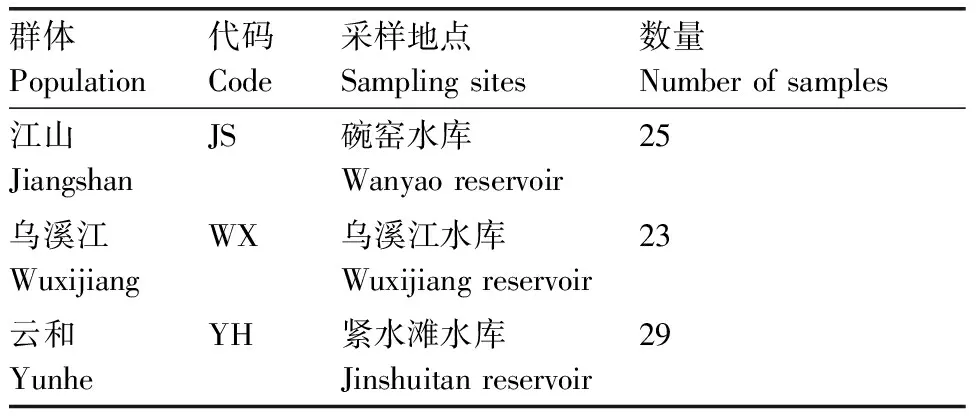
群體Population代碼Code采樣地點Samplingsites數量Numberofsamples江山JiangshanJS碗窯水庫Wanyaoreservoir25烏溪江WuxijiangWX烏溪江水庫Wuxijiangreservoir23云和YunheYH緊水灘水庫Jinshuitanreservoir29
采用肝素鈉溶液浸潤的注射器尾靜脈取血方法抽取200 μL抗凝血。以20 μL抗凝血為起始組織,采用天根血液基因組提取試劑盒進行基因組DNA提取,操作步驟按照試劑盒說明書進行。提取的基因組DNA經電泳檢測后,稀釋至100 mg·L-1于-20 ℃保存備用。
1.2.2 PCR擴增和測序
引物由上海生工生物工程技術服務有限公司合成,序列參考F: 5′-GACTTGAAAAACCACCGTTG-3′,R: 5′-CTCCGATCTCCGGATTACAAGAC-3′。PCR體系為50 μL: TaKaRaExTaq酶(5 U·μL-1) 0.25 μL,10×ExTaqBuffer 5 μL,dNTP Mixture(各2.5 mmol·L-1) 4 μL,模板DNA 1 μL,正反向引物(10 μmol·L-1)各1 μL,加滅菌超純水至50 μL。反應條件為95 ℃預變性5 min,94 ℃變性30 s,56 ℃退火30 s,72 ℃延伸1 min,30個循環;72 ℃延伸10 min;4 ℃保存。
PCR擴增產物用1.5%瓊脂糖電泳檢測,確認樣品擴增成功后,交由上海生工生物工程技術服務有限公司進行產物純化測序。
1.3 數據分析
用BLAST軟件將測序結果與GenBank相關序列進行同源性比較。利用DnaSP 5.0軟件統計單倍型及單倍型多樣性、核苷酸多樣性、中性檢驗值(Tajima’s D)、群體間分化水平(Fst)。利用Arlequin 計算遺傳分化指數(Fst),分子變異分析(AMOVA分析)。利用MEGA 6.0統計堿基組成并構建分子系統樹。
2 結果與分析
2.1 mtDNA Cyt b基因片段堿基組成
對斑鱖3個群體77個體的mtDNACytb基因片段進行比對,除去兩端部分模糊序列后,獲得長度為965 bp的同源序列。經Mega 6分析,發現3斑鱖群體mtDNACytb基因片段堿基組成差異不大,平均堿基組成為A=24.42%,T=27.69%,C=32.90%,G=15.00%。A+T含量為52.11%,略高于G+C含量。詳見表2。
2.2 群體內遺傳多樣性分析
在965 bp長度的核苷酸序列中共檢測到16個多態位點,其中1個變異位點,15個簡約信息位點。77個個體中共檢出8種單倍型(GenBank登錄號:KY608889-KY608896)(表 3)。其中,江山群體3種單倍型,烏溪江群體5種單倍型,云和群體3種單倍型。除1種單倍型為3群體共享,2種單倍型為江山群體與烏溪江群體共享外,其余6種單倍型中有1種為江山群體獨有,3種為烏溪江群體獨有,2種為云和群體獨有(表3)。
表2斑鱖mtDNACytb基因片段堿基組成
Table2Base compositions forCytbsequences of three populations
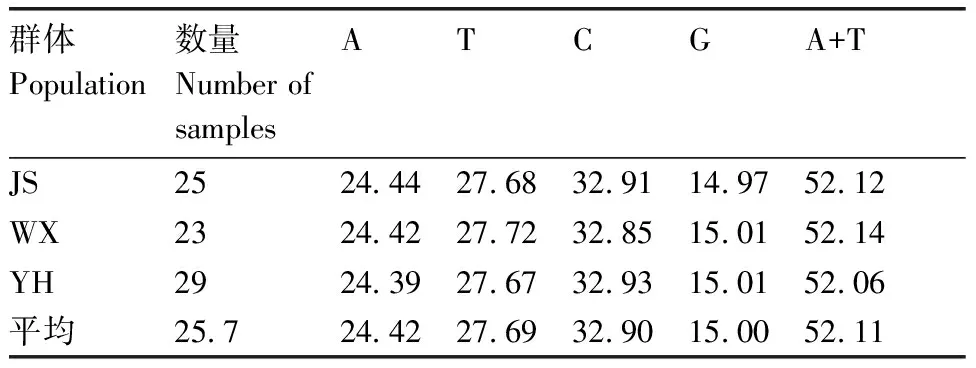
%
3個群體單倍型多樣性為0.570~0.688,烏溪江群體具有最多的單倍型數(5),但云和群體較江山、烏溪江群體具有較高的核苷酸多樣性(0.004 3)和平均核苷酸差異數(4.118)。Tajima’s D檢驗值在0.813 1~1.969 9,均無顯著性差異。詳見表4。
2.3 群體間遺傳分化
不同群體間Fst遺傳分化分析表明,3個斑鱖群體間遺傳分化指數較低,江山群體和烏溪江群體間差異最小,為-0.005 16(P=0.393 43);云和群體與江山群體、烏溪江群體間差異較大,分別為0.191 23和0.178 10,達到了顯著分化水平,見表5。
表3三個群體中的單倍型分布
Table3Haplotypes distribution of three populations

單倍型HaplotypeGenbankaccessionNo.江山Jiangshan烏溪江Wuxijiang云和YunheHap_1KY608889151111Hap_2KY60889078Hap_3KY6088912Hap_4KY6088921Hap_5KY6088931Hap_6KY6088943Hap_7KY60889511Hap_8KY6088967
表4三個群體遺傳多樣性參數統計
Table4Genetic diversity parameters of three populations

群體Population單倍型數NumberofHaplotype單倍型多樣性(Hd)Haplotypediversity核苷酸多樣性(π)Nucleotidediversity平均核苷酸差異數(K)AveragenucleotidedifferenceTajimasD檢驗值TajimasD江山Jiangshan30570±007800018±00003170008131烏溪江Wuxijiang50668±006700019±00002178709352云和Yunhe30677±003200043±00006411819699
根據Fst遺傳分化分析,將江山群體和烏溪江群體歸為一個組群,進行分子變異分析(AMOVA)。分析結果表明,遺傳變異主要來源于群體內(74.05%),組群間遺傳變異占比其次(25.31%),而組群內群體間遺傳變異占比很小(0.64%)(表6)。
表5三個群體間Fst統計值
Table5Fstvalues of three populations
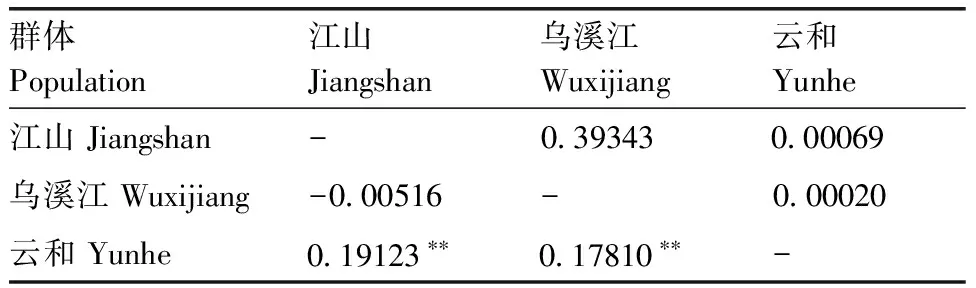
群體Population江山Jiangshan烏溪江Wuxijiang云和Yunhe江山Jiangshan-039343000069烏溪江Wuxijiang-000516-000020云和Yunhe019123??017810??-
對角線上方數值為顯著性P值;對角線下方數值為Fst值。**表示分化水平達到極顯著水平(P<0.01)。
The data above the diagonal showed significantPvalues; the data below the diagonal showedFstvalues. ** indicated that the differentiation level between populations was significant (P<0.01).
表6三個群體分子變異分析結果
Table6Analysis of molecular variance among three populations
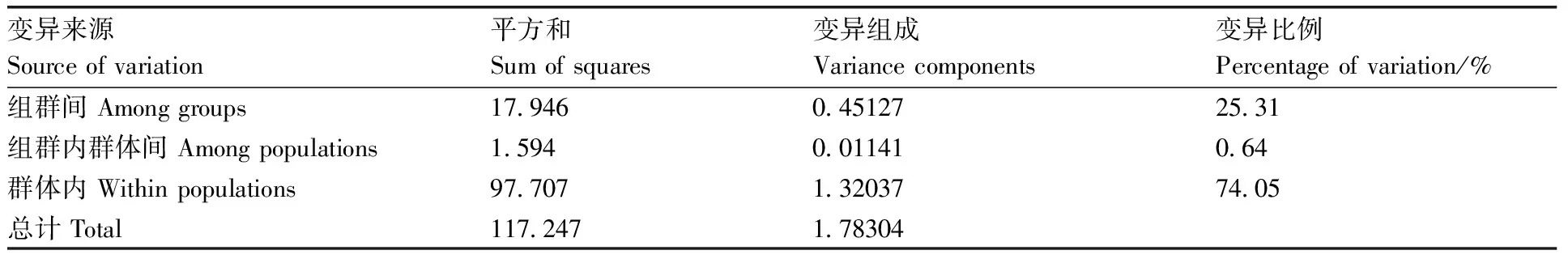
變異來源Sourceofvariation平方和Sumofsquares變異組成Variancecomponents變異比例Percentageofvariation/%組群間Amonggroups179460451272531組群內群體間Amongpopulations1594001141064群體內Withinpopulations977071320377405總計Total117247178304
3 討論
3.1 斑鱖群體的遺傳多樣性
本研究中3個斑鱖群體Cytb序列的堿基組成為A占24.42%、T占27.69%、C占32.90%、G占15.00%,其中G含量顯著低于其他堿基含量,顯示出明顯的Cytb基因共同特征[9]。該結果與在河川沙塘鱧、河鱸、秦嶺細鱗鮭等Cytb中觀察到的結果相似[10-12]。根據線粒體基因序列的單倍型多樣性(Hd)和核苷酸多樣性(π),將海洋魚類的遺傳多樣性分為4 種類型:低Hd(<0.5)和低π(<0.5%),高Hd(>0.5)和低π(<0.5%),低Hd(<0.5)和高π(>0.5%),高Hd(>0.5)和高π(>0.5%)[13]。本研究中3個斑鱖群體遺傳多樣性表現屬于第2種類型,具有較高的單倍型多樣性,但其核苷酸多樣性相對較低,私有單倍型較多。這可能是種群由于環境改變,導致有效種群數量降低,在“瓶頸效應”之后,種群經過短期快速擴張加速積累新突變所致[14]。此結果與斑鱖資源由于捕撈和環境污染,種群資源由各水系中上游廣泛分布轉為僅在上游分布的歷史事實相符。
3.2 斑鱖群體遺傳分化
群體遺傳分化系數Fst是利用遺傳信息反映各群體間遺傳分化程度的指標,數值越大表示兩個群體間遺傳分化水平越高。若Fst<0.05,表示群體幾乎沒有遺傳分化;若0.05
[1] 周才武,楊青,蔡德霖. 鱖亞科SINIPERCIXAE魚類的分類整理和地理分布[J]. 動物學研究,1988,9(2):113-126.
ZHOU C W, YANG Q, CAI D L. On the classification and distribution of the Sinipercinae fishes (Family Serranidae)[J].ZoologicalResearch, 1988, 9(2):113-126. (in Chinese with English abstract)
[2] 李思忠. 鱖亞科魚類地理分布的研究[J]. 動物學雜志,1991,26(4):40-44.
LI S Z. Geographic distribution of the Sinipercinae fishes[J].ChineseJournalofZoology, 1991, 26(4):40-44. (in Chinese with English abstract)
[3] 孔曉瑜,周才武. 鱖亞科Sinipercinae魚類的LDH同工酶的比較研究[J]. 青島海洋大學學報,1992,22(1):103-109.
KONG X Y, ZHOU C W. Comparative studies on LDH isozyme in Sinipercinae fishes of China[J].JournalofOceanUniversityofQingdao, 1992, 22(1):103-109. (in Chinese with English abstract)
[4] 王偉偉,趙金良,李思發. 我國斑鱖六個群體mtDNACytb序列的遺傳變異[J]. 動物學研究,2006,27(6):589-593.
WANG W W, ZHAO J L, LI S F. Genetic variation of the mitochondrial DNACytbamong six populations ofSinipercascherzeriin China[J].ZoologicalResearch, 2006, 27(6):589-593. (in Chinese with English abstract)
[5] 梁旭方.國內外鱖類研究及養殖概況[J]. 水產科技情報,1996,23(1):13-17.
LIANG X F. Study on mandarin fish and its culture home and abroad[J].FisheriesScience&TechnologyInformation, 1996, 23(1):13-17. (in Chinese with English abstract)
[6] 曾可為,王青云,高愛銀,等.斑鱖的生物學及繁殖生物學的研究[J]. 內陸水產,2005,30(2):21-23.
ZENG K W, WANG Q Y, GAO A Y, et al. Biology and reproduction study onSinipercascherzeri[J].InlandFisheries, 2005, 30(2):21-23. (in Chinese with English abstract)
[7] 凌繼忠.斑鱖人工繁育技術總結[J]. 漁業致富指南,2004(16):33-34.
LING J Z. Artificial breeding technology ofSinipercascherzeri[J].FisheryGuidetobeRich, 2004(16):33-34. (in Chinese)
[8] 郭新紅,劉少軍,劉巧,等.魚類線粒體DNA研究新進展[J]. 遺傳學報,2004,31(9):983-1000.
GUO X H, LIU S J, LIU Q, et al. New progresses on mitochondrial DNA in fish[J].ActaGeneticaSinica, 2004, 31(9):983-1000. (in Chinese with English abstract)
[9] ROGERS A R, HARPENDING H. Population growth makes waves in the distribution of pairwise genetic differences[J].MolecularBiologyandEvolution, 1992, 9(3):552-569.
[10] 謝楠,馮曉宇,郭水榮,等.河川沙塘鱧線粒體細胞色素b(Cyt b)基因序列分析[J]. 現代農業科技,2010(19):288-290.
XIE N, FENG X Y, GUO S R, et al. Sequences analysis of mitochondrial cytochrome b gene fragment ofOdontobutispotamophila[J].ModernAgriculturalScienceandTechnology, 2010(19):288-290. (in Chinese with English abstract)
[11] 武菲,王翠華,胡文革,等.基于細胞色素b基因河鱸養殖群體與野生群體遺傳多樣性分析[J]. 淡水漁業,2015,45(6):16-21.
WU F, WANG C H, HU W G, et al. An analysis of the gene diversity based on the mitochondrial cytochromebgene sequence of wild and cultivated populations ofPercaFluviatilis[J].FreshwaterFisheries, 2015, 45(6):16-21. (in Chinese with English abstract)
[12] 張艷萍,王太,杜巖巖,等.秦嶺細鱗鮭人工繁育群體與野生群體遺傳變異分析[J]. 水生生物學報,2014,38(5):828-833.
ZHANG Y P, WANG T, DU Y Y, et al. Analysis of the genetic diversity of cultured and wildBrachymystaxLenokTsinlingensispopulations based on mtDNA D-loop and Cytb[J].ActaHydrobiologicaSinica, 2014, 38(5):828-833. (in Chinese with English abstract)
[13] GRANT W S, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J].Heredity, 1998, 89(5):415-426.
[14] AVISE J C. Phylogeography[M]. Cambrige, Massachusetts: Harvard University Press, 2000.
[15] WRIGHT S. The interpretation of population structure by F-Statistics with special regard to systems of mating[J].Evolution, 1965, 19(3):395-420.
GeneticvariationofmitochondrialDNACytbamongthreeSinipercascherzeripopulationsinZhejiangProvince
XU Xiaojun, MENG Qinghui, CHEN Xiaoming
(ZhejiangFisheriesTechnicalExtensionCenter,Hangzhou310023,China)
The mtDNACytbgene ofSinipercascherzeriSteindachner from the upper reaches of Qiantang River (Jiangshan population and Wuxijiang population) and the upper reaches of Oujiang River (Yunhe population) were amplified and sequenced to assess the genetic diversity and genetic structure. The results showed that the average content of A+T (52.11%) was higher than that of G+C (47.89%) in the aligned 965 bp partial sequences. A total of 16 polymorphic sites were detected and 8 haplotypes were recovered out of 77 individuals. The haplotype diversity of Jiangshan, Wuxijiang and Yunhe populations were 0.570±0.078, 0.668±0.067 and 0.667±0.032,respectively, while the nucleotide diversity of the three populations were 0.001 8±0.000 3、0.001 9±0.000 2、0.004 3±0.000 6,respectively. TheFstvalue between Jiangshan population and Wuxijiang population was low (Fst=-0.005 16,P>0.05), which indicated that the two upper reaches of Qiantang River populations did not have significant genetic differentiation. While statistically significant genetic differentiations were observed between Yunhe population and Jiangshan population (Fst=0.191 23,P<0.01), and between Yunhe population and Wuxijiang population (Fst=0.178 10,P<0.01), which indicated significant genetic differentiations between the upper reaches of Qiantang River and the upper reaches of Oujiang River.
SinipercascherzeriSteindachner; cytochrome b gene; genetic variation
S937;Q75
A
1004-1524(2017)10-1637-05
(責任編輯盧福莊)

