利用MALDI-TOF MS鑒定芽苗菜中解鳥氨酸拉烏爾菌和肺炎克雷伯菌的研究
魏 超,代曉航,郭靈安,劉 煒
(四川省農科院分析測試中心,四川 成都 610066)
基質解析電離飛行時間質譜(MALDI-TOF MS )是一種軟電離有機質譜,通過基質吸收激光的能量,并將部分能量傳遞給樣品,幫助樣品離子化,根據其質量與電荷的比(質核比m/z)進行檢測[1]。MALDI-TOF MS通過基質輔助電離微生物蛋白質,使其片段化成大分子多肽,而獲得肽指紋圖譜進行檢測,因為微生物蛋白質具有特異性和保守性,不受培養基和培養條件影響,所以其多肽指紋圖譜也具有特異性和保守性。根據得到的不同微生物的特征性譜峰,通過檢索特征性質譜峰數據庫或與已知微生物的質譜峰比較,達到對微生物進行快速檢測、鑒定和分型的目的[2]。從分子生物學的角度看,核酸決定蛋白的表達,但蛋白質的豐度受其穩定性、翻譯效率和其他蛋白質的豐度調控以及后修飾等影響,因此,基于蛋白質水平的微生物分析相比核酸水平進行的分析可獲得更多的微生物信息[1,3-4]。解鳥氨酸拉烏爾菌(Raoultellaornithinolytica)因與克雷伯菌屬在基因水平的差異被從克雷伯菌屬中的解鳥氨酸克雷伯菌分離出來[5-6],此菌可引起溶血癥,在患者的痰液、尿液、膽汁中均有檢出[7-9],其毒力基因blaKPC-2與肺炎克雷伯菌相同[10],此外解鳥氨酸拉烏爾菌的一些分離菌次生代謝產物具有抗腫瘤活性,常被用作工程菌株[11]。肺炎克雷伯菌(Klebsiellapneumoniae)在自然界中廣泛分布于土壤、水、果蔬,也見于人和動物的腸道中,該菌常被分離于醫學臨床標本中,在適宜或特定的條件下,能引起人感染發病,致病作用較廣泛[5],此外由于拉烏爾菌屬與克雷伯菌屬特征生化反應比較接近,所以在商業化鑒定系統中常發生誤判的情況[12-13]。
本實驗采用MALDI-TOF MS方法對芽苗菜中解鳥氨酸拉烏爾菌和肺炎克雷伯菌進行鑒定和蛋白質特征性分析,目的在于優化數據庫使其更適合農產品非常見微生物的檢測與危害性評估。
1 實驗部分
1.1 儀器、試劑與材料
MALDI-TOF-MS(島津,AXIMA Confidence),微生物鑒定儀(生物梅里埃公司,ATB),生化培養箱(寧波東南生物儀器有限公司,DHP-9082),PCR擴增儀(賽默飛世爾科技(中國)有限公司,SimpliAmp)。
蛋白質緩沖水(BPW)、麥康凱瓊脂(MAC)、伊紅美藍瓊脂(EMB)(廣東環凱微生物科技有限公司);乙醇、乙腈(色譜純,Fisher Scientific);甲酸(色譜純,Tedia公司);α-氰基-4-羥基肉桂酸(CHCA)(Sigma公司 );微生物鑒定生化試劑條(生物梅里埃公司,ID32E),實驗用水為去離子水。
芽苗菜(金絲柳苗,松柳苗)30份樣品來自成都大型超市,超市分布覆蓋大成都范圍。
1.2 實驗方法
1.2.1微生物的盲篩稱取25 g樣品,225 mL BPW均質后36 ℃培養16 h,增菌液劃線于EMB和MAC培養基36 ℃培養24 h,每份樣品每種培養條件分別挑取6個單菌落用于進一步純化,36 ℃培養36 h后進行質譜鑒定。
1.2.2分離微生物的質譜鑒定樣品前處理:在1.0 mL乙醇水溶液(體積比70∶30)中加入10 μL接種環移取適當菌量,振蕩30 s后離心1 min,棄溶液,加入0.75 mL乙酸-水溶液(體積比5∶25)振蕩20 s,于25 ℃、40 kHz超聲10 min,加入0.75 mL乙腈振蕩均勻,4 000 r/min離心1 min取上清液1 μL點至靶板,揮干加入1 μL CHCA,揮干。
質譜條件:電噴霧離子源(ESI),氮氣激光器,固定聚焦337 nm,檢測器線性模式-電子倍增管(Multiple dynode),激光束能頻75~80 Hz,收集范圍2 000~20 000(m/z),每個樣品200次收集峰疊加,校準品ATCC 8739大腸桿菌,每16個樣品校正2次,分離微生物獲得的質譜圖數據導入MALDI-TOF/SARAMIS微生物鑒定系統與其中的超級譜圖進行多重比較分析,獲得微生物的質譜鑒定結果。
1.2.3微生物標識峰優化及相關性分析收集質譜圖譜導入MALDI-TOF/SARAMIS微生物鑒定系統,采用SARAMIS Premium軟件中的Superpectrum和Taxonomy進行標記峰足跡圖繪制、標識峰整理與微生物同源性分析。
1.2.4微生物的生化鑒定“1.2.1”中EMB和MAC純化單菌落劃線到營養瓊脂培養基上,36 ℃培養24 h,挑取營養瓊脂培養菌落至0.85%生理鹽水比濁管,振蕩分散,將菌液調至0.5麥氏單位,每孔55 μL分裝于ID32E 試劑條試劑杯中,36 ℃培養24 h,ATB系統自動讀取試劑條信息并給出最終結果。
1.2.5微生物16srDNA的相關分析25 μL PCR反應體系,擴增引物27f:5′-AGAGTTTGATCCTGGCTCAG-3′和1492r:5′-TACGGCTACCTTGTTACGACTT-3′,PCR反應條件為預變性95 ℃ 5 min;94 ℃ 30 s、60 ℃ 30 s、72 ℃ 30 s,共進行35個循環;延伸72 ℃ 7 min。PCR產物進行雙向測序并拼接,獲得序列結果并提交NCBI比對,采用MAGA7.0軟件最大簡約法(Maximum parsimony)進行序列分析構建發育樹。
2 結果與討論
2.1 解鳥氨酸拉烏爾菌及肺炎克雷伯菌的分離與鑒定
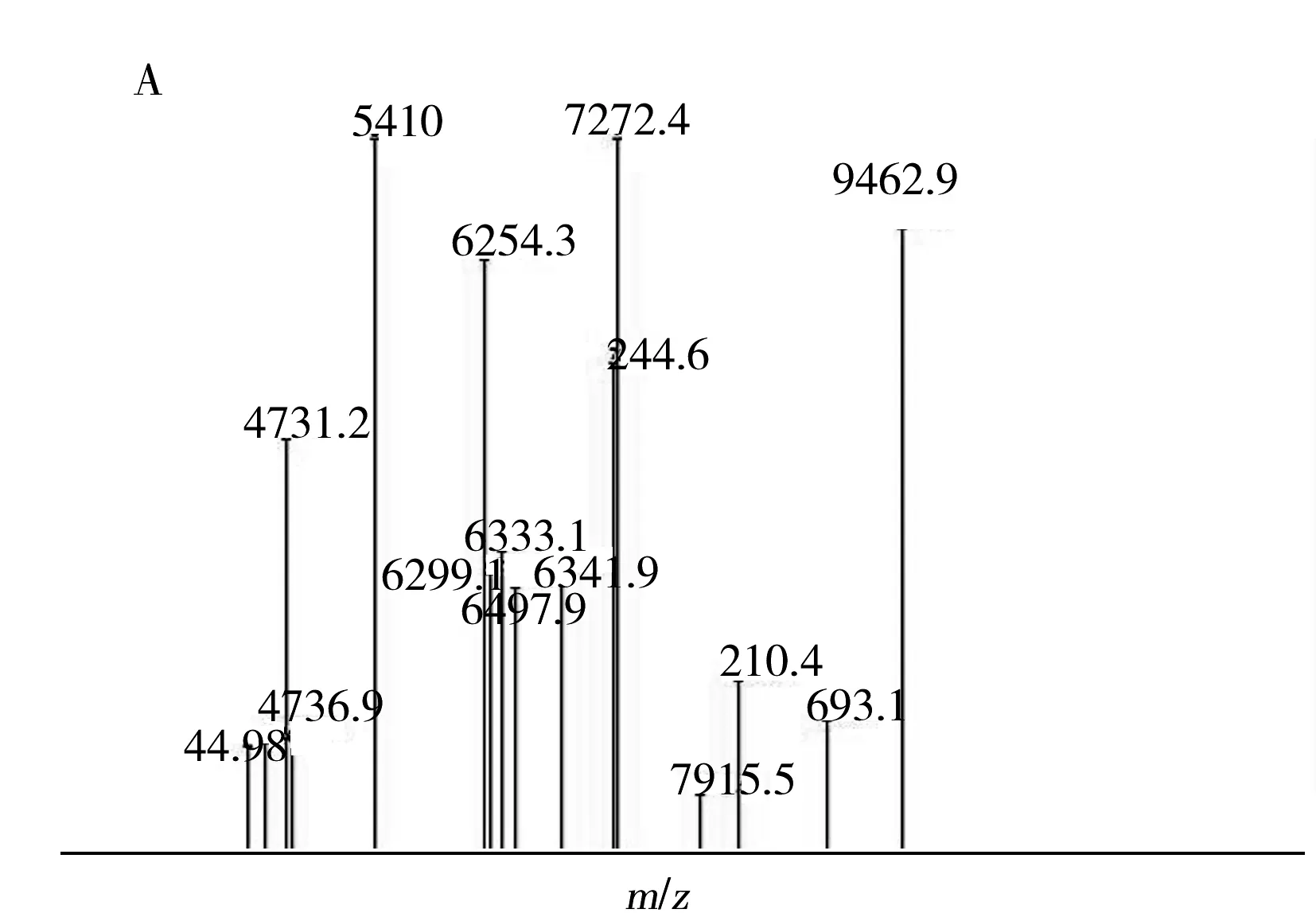
采用兩種分離培養基在30份樣品中分離得到解鳥氨酸拉烏爾菌12株(其中EMB培養基2株,MAC培養基10株,來自10份樣品),解鳥氨酸拉烏爾菌的檢出率為33.3%。肺炎克雷伯菌分離得到15株(其中MAC分離得到10株,EMB分離5株,來自13份樣品),分離率43.3%,兩種微生物的質譜圖見圖1,質譜對兩種微生物獲得很好的鑒定結果。取得有效結果的兩種微生物的生化圖譜見表1,部分質譜鑒定為解鳥氨酸拉烏爾菌的微生物生化結果為克雷伯菌(Klebsiellaspp)。
因生化反應的相似拉烏爾菌屬經常被誤認為克雷伯菌屬細菌[5,13]。已有研究中芽苗菜中解鳥氨酸拉烏爾菌和肺炎克雷伯菌檢出率較高,由生化圖譜可以看出兩種微生物在32個生化反應中大部分表現一致(表1),僅鳥氨酸脫羧酶和天門冬素芳胺酶生化反應不一致,其中鳥氨酸脫羧酶陽性為鑒定拉烏爾菌屬內解鳥氨酸種的特征性反應,天門冬素芳胺酶實驗在拉烏爾菌屬其余種中也可能表現為陰性。雖然本實驗對兩種微生物的生化鑒定取得了有效的結果,但部分質譜鑒定解鳥氨酸拉烏爾菌的微生物生化鑒定結果有一定的差異,獲得結果為克雷伯菌。實驗結果表明兩種微生物較難區分,與已有文獻一致。
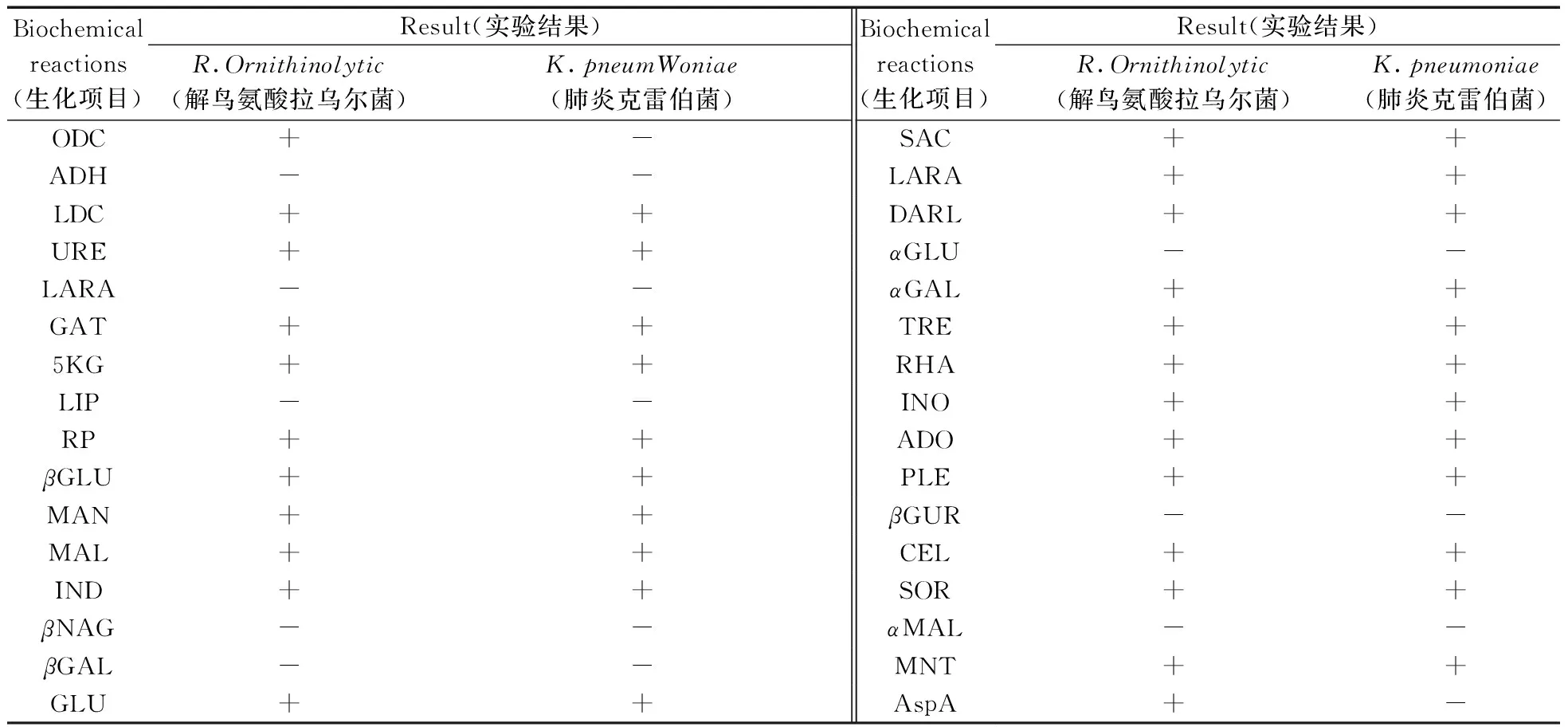
表1 解鳥氨酸拉烏爾菌和肺炎克雷伯菌的生化圖譜Table 1 Biochemistry map of K.pneumoniae and R.ornithinolytica
ODC(鳥氨酸脫羧酶),AHD( 精氨酸雙水解酶),LDC(賴氨酸脫羧酶),URE(尿素酶),LARL(L-阿拉伯醇),GAT(d-半乳糖酸鹽同化),5KG(5酮基·葡萄糖酸鈉),LIP(酯酶),RP(酚紅),βGLU(β-葡萄糖苷酶),MAL(d-麥芽糖),IND(吲哚),βNAG(β-氮乙酰葡萄糖胺),βGAL(半乳糖苷酶),GLU(d-葡萄糖),SAC(蔗糖),LARA(L-阿拉伯糖),DARL(D-阿拉伯醇),αGLU(α-葡萄糖苷酶),αGAL(α-半乳糖苷酶),TRE(d-海藻糖),RHA(L-鼠李糖),INO(肌醇),ADO(側金盞花醇),PLE(古老糖),βGUR(β-葡萄糖醛縮酶),CEL(d-纖維二糖),SOR(d-山梨醇),αMAL(α-麥芽糖苷酶),MAN(d-甘露醇),MNT(丙二酸鹽),AspA(L-天門冬素芳胺酶)
2.2 解鳥氨酸拉烏爾菌和肺炎克雷伯菌質譜特征峰的研究
足跡圖可直觀表現微生物質譜標識峰的位置(包括<0.08的相對偏差),芽苗菜中分離的12株解鳥氨酸拉烏爾菌以及肺炎克雷伯菌的標識峰足跡圖顯示,在小質核比(m/z<5 000)區域內標識峰比較密集,隨著質核比增大,標識峰逐漸減少,但盡管鑒定為同種微生物,其標識峰的數目、質核比軌跡仍相差較多,即便數據庫中的超級譜圖間差距也比較大。



圖1 微生物鑒定質譜圖譜Fig 1 Microorganism mass spectra of MALDI-TOF MS A:R.ornithinolytica(解鳥氨酸拉烏爾菌),B:K.pneumoniae(肺炎克雷伯菌),C:Calibration E.coli ATCC 8739(標準大腸桿菌 ATCC8739)
對芽苗菜中分離的解鳥氨酸拉烏爾菌的全部標識峰進行擬合(包含<0.08的相對偏差),12株微生物共有18組質核比標識峰完全擬合(圖2A),再添加4組超級譜圖數據后,16組解鳥氨酸拉烏爾菌完全擬合的標識峰為16組(圖2B),減少為質核比(m/z)5 599.6和5 618.3(包含<0.08的絕對偏差)的兩組標識峰,這兩組峰響應值較高可認為是芽苗菜分離菌種的特有譜峰,完全擬合的16組標識峰可認定為解鳥氨酸拉烏爾菌的特征峰,將16組峰上傳至SARAMIS Premium數據系統,對其加權以提高系統對解鳥氨酸拉烏爾菌的分辨能力。
對分離的肺炎克雷伯菌的全部標識峰進行擬合(包含<0.08的相對偏差),15株微生物共有16組標識峰完全擬合(圖3A),再添加4組超級譜圖數據后,19組肺炎克雷伯菌完全擬合的標識峰有10組(圖3B),減少的5組標識峰中有3組峰的響應值較低,剩余2組峰的響應值較高可認為是芽苗菜分離菌種特有譜峰,完全擬合的10組標識峰可認定為解鳥氨酸拉烏爾菌的特征峰,將其上傳至SARAMIS Premium數據系統,對其加權以提高系統對解鳥氨酸拉烏爾菌的分辨能力。
質譜鑒定微生物是根據其特征峰擬合進行數據計算獲得鑒定結果,數據庫的容量和特征峰的權重決定鑒定結果的準確性,SARAMIS Premium是一個開放平臺,本實驗對生化相似的兩種微生物進行特征峰整理和加權,還可針對更多樣的農產品特征微生物進行研究。


圖4 解鳥氨酸拉烏爾菌聚類分析樹Fig.4 R.ornithinolytica cluster dendrogram by MALDI-TOF MSA:cluster dendrogram of 12 strains of R.ornithinolytica;B:cluster dendrogram of R.ornithinolytica with weighted feature peak(A:12株解鳥氨酸拉烏爾菌進行聚類分析;B:解鳥氨酸拉烏爾菌征峰加權后聚類分析圖)
2.3 解鳥氨酸拉烏爾菌及肺炎克雷伯菌系統發育學的研究
對芽苗菜中分離的12株解鳥氨酸拉烏爾菌進行聚類分析(圖4A),對其特征峰加權后的聚類分析結果見圖4B,對比兩張分析圖,在特征峰加權后微生物的相關系數有所提高,兩張圖中一級分支基本一致,但更高級的分支和相對距離發生了變化。在圖4B中,13和14號圖譜為超級譜圖庫中同源樣品,聚類分析結果也顯示其相關系數>100%;1和6號為芽苗菜分離解鳥氨酸拉烏爾菌,聚類分析結果顯示其相關系數>100%,經溯源來源于同份芽苗菜樣品的不同培養基的分離。肺炎克雷伯菌的聚類分析樹與特征峰加權后分析樹與圖4的分析結果相近,加權特征峰后相關系數也有所提高,一級分支基本不變,但高級分支和相關位置發生了一定變化,聚類結果顯示一級分支的5組微生物,除2組數據庫中同源微生物,另外3組6株肺炎克雷伯菌為芽苗菜中分離,經溯源來自于3份芽苗菜樣品的不同培養基。可見MALDI-TOF MS方法可以用于微生物的溯源,但其溯源的準確性還需進一步優化SARAMIS Premium系統。
2.4 解鳥氨酸拉烏爾菌與肺炎克雷伯菌相似性的研究
由生化鑒定結果可以看出,兩種微生物的生化反應非常接近,容易誤判,利用SARAMIS Premium將兩種微生物的質譜圖譜進行相關分析(圖5),兩種微生物雖有部分相交但在三四級分支中仍可分辨。圖5顯示兩種微生物的相關性仍較高,最后一級分支系數仍大于50%,與“2.3”聚類分析樹中種內最后一級分支相關系數接近,可見兩種微生物在質譜圖中較接近但可分辨。

對兩種微生物進行16SrDNA測序并建立相關分析系統發育樹(圖6),系統發育樹可將兩種微生物分于兩個分支,雖然遺傳距離很接近但仍有一定差異,兩種微生物在基因水平上區分明顯。
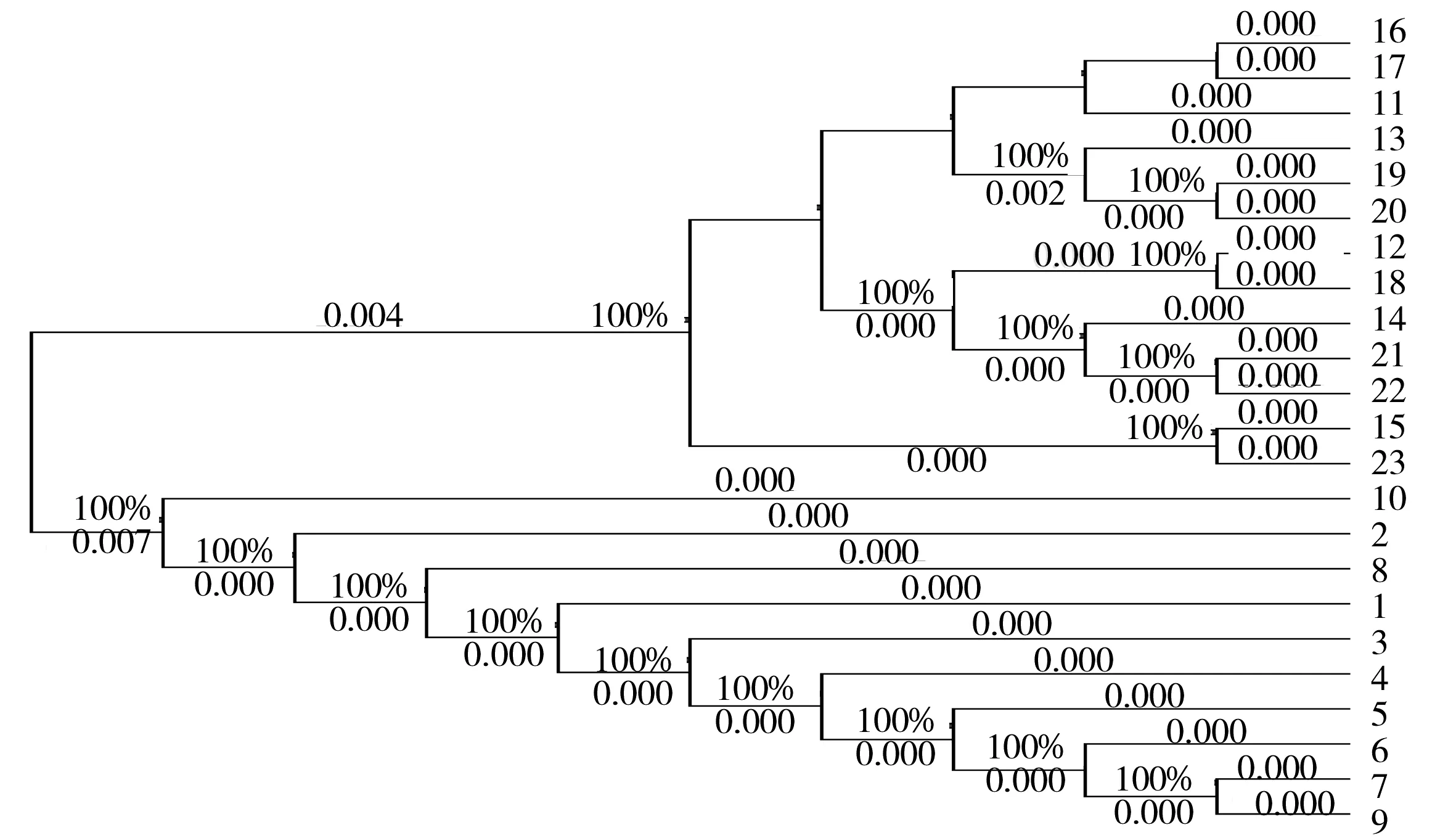
圖6 解鳥氨酸拉烏爾菌和肺炎克雷伯菌的16srDNA相關分析樹Fig.6 K.pneumoniae and R.ornithinolytica related analysis tree by 16srDNA1-10:R.ornithinolytica;11-23:K.pneumoniae
3 結 論
本文針對芽苗菜中分離率最高且相似程度較高的兩種微生物解鳥氨酸拉烏爾菌和肺炎克雷伯菌進行了MALDI-TOF MS鑒定相關分析并優化了標識峰,實驗結果表明,MALDI-TOF MS在農產品相似微生物鑒定上準確度較高,但其溯源和相關分析能力比傳統16SrDNA 方法還有一定差距,對開放數據庫系統ARAMIS Premium進行添加和優化是提高MALDI-TOF MS對農產品微生物檢測和溯源準確性的主要方法。
[1] Richard J S.ProteinsandProteomics:ALaboraryManual.New York:Cold Spring Harbor Laboratory Press,2003:397-412.
[2] Smith H I,Hecker M.MicobialProteomics:FunctionalBiologyofWholeOrganism.John Wiley & Sons,2006:5-7,311-315.
[3] Tekippe M E,Burnham C A.EuropeanJournalofClinicalMicrobiology&InfectiousDiseases,2014,33(12):2163-2171.
[4] Luo Y,Siu G K,Yeung A S,Chen J H,Ho P L,Leung K W.JournalofMedicalMicrobiology,2015,64(1):18-24.
[5] Fang H,Chen C Z,Zhang X J.PathogenceBacteriaofEnterobacteriaceae.Beijing:China Agriculture Press(房海,陳翠珍,張曉軍.腸桿菌科病源細菌.北京:中國農業科學技術出版社),2011.
[6] Eefje de J,Arjan S J,Nathalie S B,Rob J R.DiagnosticMicrobiology&InfectiousDisease,2013,75(4):431-433.
[7] García-Lozano T,Pascual Plá F J,Aznar O E.MedicinaClinica,2013,141(3):138-147.
[9] Seng P,Boushab B M,Romain F,Gouriet F,Bruder N,Martin C.InternationalJournalofInfectiousDiseases,2016,45:65-71.
[11] Fioka M J,Lewtak K,Rzymowska J,Grzywnowicz K,Huasstasiak M,Sofińskachmiel W.Pathogens&Disease,2013,69(1):49-61.
[12] Al-Hulu S M,Al-Charrakh A H,Al-Saadi M A K.Med.J.Babylon.,2009,7(4):42-47.
[13] Kanki M,Yoda T,Tsukamoto T,Shibata T.Applied&EnvironmentalMicrobiology,2002,68(7):3462-3468.

