應用可視芯片技術高通量鑒別8種魚成分
韓建勛,陳 穎*,王 斌,張九凱,馬秀麗
(1.中國檢驗檢疫科學研究院 農產品安全研究中心,北京 100176;2.中國農業大學 食品科學與營養工程學院,北京 100083)
受經濟利益的驅動,全球魚肉市場以次充好、摻假問題層出不窮。有報道稱,德國產鰈科魚的抽檢標識不符比例達25%;在美國波士頓和洛杉磯,魚肉虛假標識的比例分別高達48%和55%[1]。我國媒體也曾曝光國內超市將油魚冒充鱈魚的事件。魚肉摻假問題不僅影響國際間進出口貿易秩序,而且會降低社會誠信度,損害消費者利益。
對于未加工魚類,消費者很容易靠外觀、形態、花紋及顏色進行區分,但經絞碎、研磨、烘烤、油炸等加工處理的無骨魚、魚糜、魚肉罐頭或生鮮調理食品等,則很難利用形態學鑒別。近年來科技人員采用光譜[2-4]、質譜[5-6]、生物傳感[7]、PCR(Polymerase chain reaction)[8-9]、DNA條碼[10-13]等現代分析技術在魚肉真偽鑒別方面開展了廣泛研究,取得了不錯的成果。有研究采用近紅外光譜技術,結合主成分分析和BP人工神經網絡技術建立了青、草、鰱、鳙、鯉、鯽、魴7 種淡水魚的品種鑒別模型,準確率達96.4%[4]。該技術前處理簡單、檢測速度快,但魚類的光譜曲線易受不同養殖環境、不同產地等因素的影響,因此數據庫模型的構建需大量代表性樣本。也有文獻通過構建電化學生物傳感體系,檢測大西洋鮭與虹鱒特異PCR擴增產物產生的差異循環伏安信號,從而鑒別魚肉樣品來源[7],但實驗檢測電極易吸附非特異DNA,進而影響結果的特異性。隨著分子生物學技術的發展,以PCR為基礎的DNA分析技術已成為物種鑒定的主流。有文獻提出以COI(Cytochrome C oxidase I)基因為靶基因,通過建立大西洋鮭的SYBR-Green 熔解曲線方法,可準確鑒別大西洋鮭、紅鮭、銀鮭與大馬哈魚及其制品[14],方法簡便、成本低,但一次擴增只能檢測1個物種,通量較低。也有報道利用大西洋鮭線粒體CR(Control region)區段設計特異性LAMP 引物,建立了大西洋鮭種類鑒定的LAMP 檢測方法[15]。LAMP方法簡便快速,但特異性引物設計難度較大。近幾年,DNA 條形碼技術在物種鑒別領域發展迅速。有研究以魚COI基因為目標基因,應用DNA條形碼技術調查深圳批發市場和超市零售魚肉制品的種類來源,發現77份魚肉制品中有28份樣品的標簽標示不符,錯標率達36.36%[13]。DNA 條形碼技術鑒別物種雖然準確率和通量高,但必須依賴DNA 測序技術,步驟多,成本高。可視芯片技術作為一種利用特異探針雜交產生可見信號的基因芯片技術,具有操作簡單、通量高和結果肉眼可見的特點,已在病原微生物[16]、食源性過敏原[17]以及轉基因作物[18]檢測中得到應用。
本文針對市場中常見的多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚以及東星斑魚8種魚類,采用可視芯片技術建立了一種魚肉及其制品中8種魚源性成分的高通量快速檢測方法,旨在為魚肉制品的溯源和安全監管提供新的技術支撐。
1 實驗部分
1.1 材 料
1.1.1樣品大黃魚(Larimichthyscrocea)由遼寧出入境檢驗檢疫局檢驗檢疫技術中心提供,青石斑魚(Epinephelusawoara)與東星斑魚(Plectropomusleopardus)由廈門出入境檢驗檢疫局檢驗檢疫技術中心提供,暗紋東方鲀(Takifuguobscurus)由福建出入境檢驗檢疫局檢驗檢疫技術中心提供,金槍魚(Thunnusthynnus)由上海出入境檢驗檢疫局檢驗檢疫技術中心提供,多寶魚(Psettamaxima)、帶魚(Benthodesmuselongatus)以及銀鯧魚(Pampusargenteus)購自北京超市。所有魚肉樣品均在-20 ℃保存備用。
1.1.2引物與探針所有引物與探針均根據多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚以及東星斑魚的小清蛋白基因序列比對結果設計。為避免雜交反應出現假陰性,且作為探針陣列的定位點,實驗中需要合成一段由20個腺嘌呤組成的陽性對照(5′-ALD-AAAAAAAAAAAAAAAAAAAA-3′-Biotin)。其中,魚肉通用-反向引物5′端進行生物素(Biotin)標記,在探針和陽性對照的5′端進行醛基(ALD)修飾,以使探針和陽性對照能與芯片表面修飾的氨基結合固定。

表1 引物及探針序列Table 1 The sequence of primers and probes
1.1.3試劑與儀器EX Taq DNA polymerase、dNTPs、10×PCR 緩沖液購自寶生物工程(大連)有限公司;CTAB裂解液(20 g/L CTAB,1.4 mol/L NaCl,0.1 mol/L Tris、20 mmol/L Na2-EDTA,pH 8.0)、CTAB沉淀液(5 g/L CTAB,0.04 mol/L NaCl,pH 8.0)、1.2 mol/L NaCl等為自行配制;蛋白酶K、三氯甲烷、異丙醇、無水乙醇等購自北京宏捷有限公司。
微量移液器(10、20、100、1 000 μL,德國Eppendorf公司),精密烘箱(Venticell,德國 MMM公司),恒溫均勻器(Thermomixer Comfort,德國Eppendorf公司),電子天平(BS,德國Sartorius公司),離心機(5804R,德國Eppendorf公司),PCR擴增儀(Mastercycler gradient,德國Eppendorf公司),生物芯片點樣系統(AD3200,美國BioDot公司)。
1.2 實驗方法
1.2.1樣品DNA提取取研磨過的魚肉原料或市售樣品0.1 g至2.0 mL離心管中,后續DNA提取參照文獻中的CTAB法[17]。
1.2.2常規PCR擴增反應體系為25 μL:DNA模板5 μL;10×PCR反應緩沖液2.5 μL;dNTPs 2.0 μL;正/反向引物(10 μmol/L)各0.5 μL;EX Taq DNA polymerase 0.3 μL,用無菌水補至總體積為25 μL。
擴增參數為:95 ℃預變性5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35個循環;72 ℃延伸5 min。

圖1 魚類探針的可視芯片點樣圖Fig.1 Schematic diagram of capture probes for fish species on the chip1.Turbot(多寶魚);2.Silvery pomfret(銀鯧魚);3.Tuna(金槍魚);4.Obscura puffer(暗紋東方鲀);5.Indigo hamlet(青石斑魚);6.Hairtail(帶魚);7.Large yellow croaker(大黃魚);8.Miniatus grouper(東星斑魚);P.Positive control(陽性對照)(biotin-dA20)
1.2.3可視芯片的制備與雜交芯片的制備與雜交程序參照文獻[16]的方法。利用生物芯片點樣儀將多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚、東星斑魚以及陽性對照等探針以十字形圖案方式進行點樣(圖1)。
1.2.4可視芯片特異性分析單組分魚肉特異性分析:為驗證魚類探針的特異性,采用魚肉通用引物對分別擴增多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚、東星斑魚等DNA,擴增條件同“1.2.2”所述,將PCR產物分別與芯片進行雜交反應,用無菌水作空白對照。
多組分魚肉特異性分析:將多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚、東星斑魚8種魚類DNA進行等濃度(10 ng/μL)混合,利用魚肉通用引物進行擴增,擴增條件同“1.2.2”所述,將PCR產物與芯片進行雜交反應,用無菌水作空白對照。
1.2.5可視芯片靈敏度分析將已測定濃度的多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚、東星斑魚等8種魚肉DNA,用無菌水稀釋至10 ng/μL,取10 μL 10 ng/μL DNA至90 μL無菌水中,得到1 ng/μL的DNA濃度,由此依次類推得到0.1、0.01、0.001 ng/μL的DNA濃度。按照“1.2.2”條件進行PCR擴增,擴增產物分別與芯片進行雜交反應,用無菌水作空白對照。
1.2.6市售樣品檢測為確證可視芯片方法的準確性及適用性,選取7份市售魚肉加工樣品:鮮得味金槍魚罐頭、伊納寶金槍魚罐頭、五香黃花魚罐頭、三文魚松、古龍茄汁沙丁魚、石斑魚肉、東星斑魚肉,DNA提取后按照“1.2.2”條件進行擴增,PCR產物分別與芯片進行雜交反應,將檢測結果與樣品標簽進行比對。
2 結果與討論
2.1 引物及探針設計
目前常用于魚肉種類鑒別的基因有線粒體COI基因、CR區段以及16S rDNA等,但經序列比對發現這些基因并不適用于同時設計8種魚類的通用引物及特異探針。考慮到作為魚類主要過敏原的小清蛋白不僅廣泛存在于魚類骨骼纖維中,而且具有物種特異性,現已成為研究不同物種之間親緣關系的重要工具[19],因此選擇小清蛋白基因作為魚肉種類鑒別的靶標基因。
首先自行克隆測序了暗紋東方鲀(T.obscurus)、東星斑魚(C.miniata)、青石斑魚(H.indigo)、多寶魚(P.lethostigma)、大黃魚(L.xanthurus)、銀鯧魚(P.argenteus)以及帶魚(T.haumela)的小清蛋白基因序列,提交GenBank數據庫后分別獲得序列號MF671767、MF671768、MF671769、MF671770、MF671771、MF671772、MF671773。同時從GenBank數據庫下載金槍魚的小清蛋白基因序列(GenBank:FN544082.1)。采用生物信息學軟件ClustralX比對不同品種魚的小清蛋白基因序列,在基因保守區設計了魚通用引物,在非保守區設計了不同品種魚的特異性探針。序列比對結果如圖2所示。
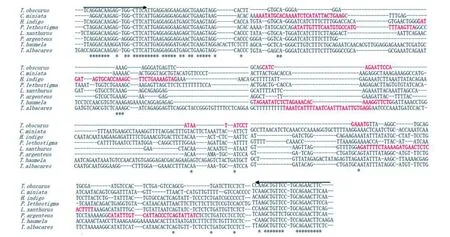
圖2 魚類小清蛋白基因序列比對結果Fig.2 The results of the sequence alignments of parvalbumin gene in fishthe nucleotides followed arrows indicate the position of fish universal forward and reverse primer,the specific probes for eight fish species are shown in red color(箭頭對應序列代表魚的通用正/反向引物序列,紅色加粗標記序列代表特異性探針序列)
2.2 可視芯片特異性結果
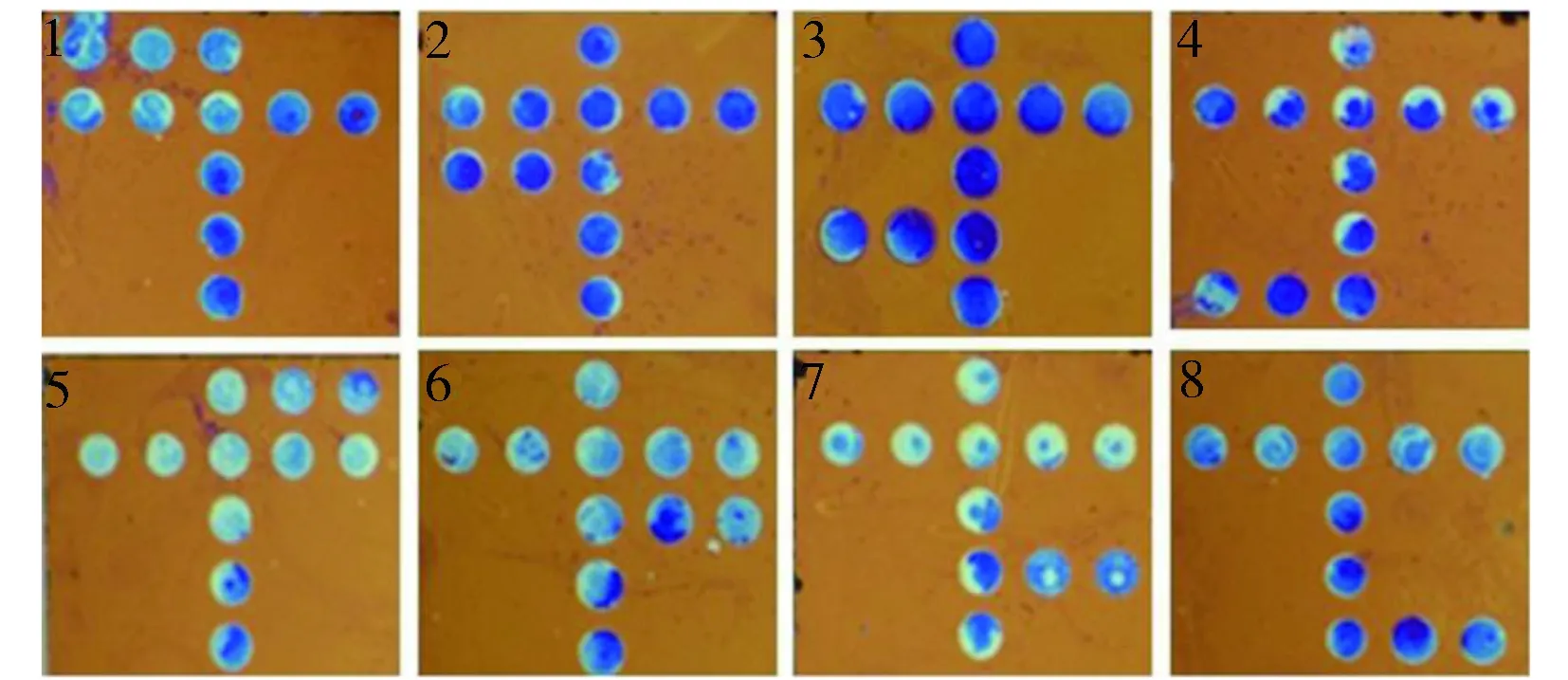
圖3 單組分魚肉可視芯片特異性結果Fig.3 Specificity detection result of single-fish species on thin-film biosensor chips1.Turbot;2.Silvery pomfret;3.Tuna;4.Obscura puffer;5.Indigo hamlet;6.Hairtail;7.Large yellow croaker;8.Miniatus grouper
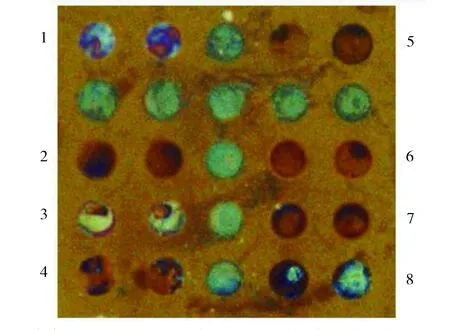
圖4 多組分魚肉可視芯片特異性結果Fig.4 Specificity detection result of multiple-fish species on thin-film biosensor chips1.Turbot;2.Silvery pomfret;3.Tuna;4.Obscura puffer;5.Indigo hamlet;6.Hairtail;7.Large yellow croaker;8.Miniatus grouper
2.2.1單組分魚肉特異性分析依照實驗預先設計的十字型芯片圖案,陽性對照位點全部呈現明顯的信號,多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚、東星斑魚8種魚的靶標探針位點也均呈現明顯信號,與陽性對照的信號相當,且與背景信號明顯區分,而同一張芯片上的其他非靶標探針位點則完全與背景一致,沒有任何信號(圖3),說明本研究設計的探針可特異性檢出多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚以及東星斑魚成分。
2.2.2多組分魚肉特異性分析圖4是可視芯片高通量檢測魚類的特異性結果。芯片中陽性對照的信號全部清晰顯現,所有魚類探針位點的信號明顯,均可與背景清晰地區分,說明該方法可同時檢出8種魚類成分。實驗也發現,不同魚的探針位點雜交信號強度存在差異性,其原因可能是靶標基因擴增產物與特異探針的雜交反應受到其他擴增產物或探針的干擾,但檢測信號與背景均可明顯區分,不影響其高通量檢測8種魚類的準確性,且整個可視芯片檢測過程僅需約35 min。相比一次擴增只能檢測1個物種的實時熒光PCR方法、LAMP方法等,該方法具有明顯的通量優勢;相較通量更高的測序技術,其優勢在于結果肉眼可見,檢測時間短,一般測序技術上機檢測1個樣本至少需2~3 h,且測序數據需經序列比對才能獲得最終檢測結果,耗時較長,數據分析專業性較強。

圖5 多寶魚的可視芯片靈敏度檢測結果Fig.5 The detection limit result of Turbot on thin-film biosensor chips1-5:10,1.0,0.1,0.01,0.001 ng/μL
2.3 可視芯片靈敏度結果
將多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚以及東星斑魚DNA分別稀釋至10、1.0、0.1、0.01、0.001 ng/μL,將其PCR產物與可視芯片分別進行雜交反應。以多寶魚為例,當DNA為0.01 ng/μL時,芯片上特異結合位點產生微弱信號,當DNA為0.001 ng/μL時,芯片上特異結合位點無信號產生,與背景顏色一致,表明方法檢測多寶魚的靈敏度為0.01 ng/μL(圖5)。同樣,得到銀鯧魚、暗紋東方鲀、帶魚、東星斑魚的檢測靈敏度為0.01 ng/μL,青石斑魚、金槍魚以及大黃魚的檢測靈敏度為0.1 ng/μL。
2.4 市售樣品檢測結果
為驗證方法的可行性,利用已建立的可視芯片方法對7份市售樣品開展多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚以及東星斑魚等成分的檢測,結果見表2。兩份金槍魚罐頭均檢出標識的金槍魚成分,東星斑魚肉檢出標識的東星斑魚成分,說明該檢出結果與標簽一致;五香黃花魚罐頭檢出大黃魚成分,石斑魚肉檢出青石斑魚成分,但樣品標簽中未明確標識大黃魚、青石斑魚成分;三文魚松以及古龍茄汁沙丁魚兩份樣品標簽中無上述8種魚成分標識,且樣品中也均未檢出8種魚類成分。以上說明,本研究建立的可視芯片方法可用于市售魚肉樣品中8種魚源性成分的檢測。

表2 市售樣品的檢測結果Table 2 The detection results of commercial products
+:detected;-:no detected
3 結 論
針對魚肉市場常見的魚類品種摻假問題,本研究以多寶魚、銀鯧魚、金槍魚、暗紋東方鲀、青石斑魚、帶魚、大黃魚以及東星斑魚8種魚類為研究對象,根據小清蛋白基因序列設計了8種魚的通用引物以及特異性探針,利用可視芯片技術建立了一種快速、準確、靈敏、便捷的可同時檢測8種魚類的高通量檢測方法,為我國魚肉市場的安全監管以及檢驗檢疫口岸的現場快速通關提供了新的技術支撐。
[1] China Agricultural News Network.Fish Fraud Was Serious in European and American Which Was Reported by the Media,and 39% of Seafood in New York Involved the Adulteration(中國農業新聞網.媒體稱歐美魚肉造假嚴重 紐約海鮮39%涉假).[2013-02-21].http://www.farmer.com.cn/xwpd/gjdt/201302/t20130221_810789.htm.
[2] Veliogˇlu H M,Temiz H T,Boyaci I H.FoodChem.,2015,172:283-290.
[3] Almeida F S,Lima S M,Andrade L H C,Súarez Y R.Appl.Spectrosc.,2012,66(7):782-785.
[4] Xu W J,Liu R,Hong X S,Xiong S B.Trans.Chin.Soc.Agric.Eng.(徐文杰,劉茹,洪響聲,熊善柏.農業工程學報),2014,30(1):253-261.
[5] Maasz G,Takács P,Boda P,Varibiro G,Pirger Z.Sci.TotalEnviron.,2017,601/602:317.
[6] Mazzeo M F,Siciliano R A.J.Proteom.,2016,147:119-124.
[7] Zhao Z X,Xu G C,Li J T,Yang S Y.Biotech.Bull.(趙紫霞,徐桂彩,李炯棠,楊世勇.生物技術通報),2017,33(6):54-61.
[8] Luekasemsuk T,Panvisavas N,Chaturongakul S.Toxicon,2015,102:43-47.
[9] Mueller S,Handy S M,Deeds J R,George G O,Broadhead W J,Pugh S E,Garrett S D.FoodControl,2015,55:39-42.
[10] Paracchini V,Petrillo M,Lievens A,Gallardo A P,Martinsohn J T,Hofherr J,Maquet A,Silva A P B,Kagkli D M,Querci M,Patak A,Angers-Loustau A.FoodControl,2017,79:297-308.
[11] Dhar B,Ghosh S K.Gene,2017,627:248.
[12] Chang C H,Lin H Y,Ren Q,Lin Y S,Shao K T.FoodControl,2016,66:38-43.
[13] Wang M,Liu H,Huang H,Zhao X M,Shi Q,He S P,Sun Y.FoodSci.(王敏,劉葒,黃海,趙曉萌,石瓊,何舜平,孫穎.食品科學),2015,36(20):247-251.
[14] Li X G,Zhao F,Ma L P,Sun Y,Zhou D Q.J.Chin.Inst.FoodSci.Technol.(李新光,趙峰,馬麗萍,孫永,周德慶.中國食品學報),2014,(3):170-176.
[15] Feng J L,Ye J,Meng L,Jiang X N,Dai Z Y.J.Nucl.Agric.Sci.(馮俊麗,葉劍,孟璐,姜曉娜,戴志遠.核農學報),2017,31(5):868-875.
[16] Zhao J Y,Bai S L,Huang W S,Chen Y.FoodFerment.Ind.(趙金毅,白素蘭,黃文勝,陳穎.食品與發酵工業),2008,34(8):141-144.
[17] Wang W,Han J X,Wu Y J,Yuan F,Chen Y,Ge Y Q.J.Agric.FoodChem.,2011,59(13):6889-6894.
[18] Bai S L,Zhang J,Li S C,Chen H D,Terzaghi W,Zhang X,Chi X R,Tian J,Luo H X,Huang W S,Chen Y,Zhang Y C.J.Agric.FoodChem.,2010,58(15):8490-8494.
[19] Nong X X,Bin S Y,Meng T,Chen D X,Liu X L,Zhang J S.Lett.Biotechnol.(農小獻,賓石玉,蒙濤,陳敦學,劉希良,張建社.生物技術通訊),2011,22(6):887-891.

