基于全基因組的卷柏復蘇相關AP2/ERF轉錄因子基因家族分析△
徐志超,浦香東,宋經元
(中國醫學科學院 北京協和醫學院 藥用植物研究所/國家中醫藥管理局 中藥資源保護重點研究室,北京 100193)
復蘇植物能夠在脫水95%以上的生理情況下生存且遇水復蘇,在蕨類植物和苔蘚植物中常見,被子植物中數量極少[1-2]。復蘇植物極度耐旱的特點成為國際研究的熱點之一,被認為是抗旱基因挖掘及利用的模式植物。截至目前多個復蘇植物的基因組已公布,如單子葉植物復活草Oropetiumthomaeum[3]、維柯薩Xerophytaviscosa[4]和雙子葉植物旋蒴苣苔Boeahygrometrica[5],為抗旱基因的挖掘提供資源。
卷柏Selaginellatamariscina,又稱“九死還魂草”,為石松綱卷柏科卷柏屬植物,旱時卷曲,遇水而榮,是典型的復蘇植物[6-7]。卷柏屬包括約700個種,《中國植物志》中記錄有66種卷柏[8],2015版《中華人民共和國藥典》以卷柏和墊狀卷柏S.pulvinata入藥,其干燥全草炒炭后具有活血通經的功能[9]。卷柏屬不同物種間遺傳分化極其顯著,其生理特點也顯著不同,如卷柏為耐旱型[10],而江南卷柏為干旱敏感型[11]。
AP2/ERF轉錄因子在植物生長發育、脅迫響應中發揮重要的調控作用,包括干旱、高鹽、低溫等逆境脅迫響應[12]。AP2/ERF轉錄因子家族至少含有一個高度保守的AP2結構域,根據結構域的數量和識別序列不同,分為AP2、ERF、RAV、DREB、SOLOIST等5個亞家族[13]。已報道多個基于全基因組分析AP2/ERF轉錄因子家族的研究,如擬南芥[14]、水稻[15]、葡萄[16]、丹參[17]等,然而低等植物AP2/ERF轉錄因子的挖掘研究較少。本研究基于課題組前期卷柏基因組的研究數據,挖掘卷柏的AP2/ERF轉錄因子,篩選與卷柏復蘇密切相關的轉錄調控因子。
1 材料和方法
1.1 卷柏基因組、轉錄組數據
卷柏基因組組裝、注釋及轉錄組測序工作由本課題組完成。卷柏基因組測序原始數據(PacBio RSII測序平臺)以及卷柏干旱及復水的轉錄組數據(Illumina HiSeq4000測序平臺)已上傳至NCBI(National Center for Biotechnology Information)的SRA(Sequence Read Archive)數據庫,登錄號:SAMN07840812(BioSample ID)、PRJNA416064(BioProject ID)。
1.2 基于蛋白序列比對的卷柏AP2/ERF轉錄因子鑒定
下載擬南芥AP2/ERF轉錄因子的AP2、ERF、DREB、RAV、SOLOIST亞家族成員,采用BLASTP比對卷柏基因組蛋白序列,Evalue設置為1E-5;采用PFAM數據庫隱馬爾科夫模型HMM:PF00847搜索卷柏基因組數據庫,Evalue設置為1E-5。以gff文件格式從基因組數據庫中提取所有AP2/ERF序列,根據NCBI網站BLASTP比對的結果,利用APOLLO軟件手動校正序列。校正后,保存每個AP2/ERF轉錄因子的CDS、DNA、蛋白序列信息。在蛋白質數據庫PROSITE(http://prosite.expasy.org/)中確認每個成員的保守結構域序列,用DNAMAN軟件比對分析其保守性。
1.3 AP2/ERF系統進化、基因結構和保守基序分析
將卷柏和擬南芥AP2/ERF家族成員的蛋白序列采用MUSCLE做多序列比對,構建NJ(Neighbour-Joining)樹,選擇Jones-Taylor-Thornton(JTT)模型,重復次數1000次。基于CDS序列及對應基因組序列,利用在線服務器Gene Structure Display Server(GSDS 2.0)(http://gsds.cbi.pku.edu.cn/index.php)對基因結構進行分析。利用MEME(Suite version 4.9.1)(http://meme-suite.org/)對AP2/ERF轉錄因子保守基序進行分析,具體原則如下:E值小于2×10-30,基序重復次數不限,氨基酸個數10~200個。
1.4 AP2/ERF轉錄因子家族基因的差異表達分析
本研究分析卷柏干旱及復水處理的9個轉錄組數據,分別為干旱(hydration,HD)、70%相對含水量(Relative water content,RWC)、50% RWC、30% RWC、15% RWC、5% RWC、復水2 h(rehydration,RH)、RH 4 h、RH 6 h。基于轉錄組數據,通過HiSAT2和Cufflinks軟件計算AP2/ERF轉錄因子的FPKM值[18]。按公式(1)計算RWC。
(1)
式中:Fwt為干旱時間點的卷柏質量;Dwt為卷柏在65 ℃烘干2 d后的質量;FTwt為卷柏復水24 h后的質量[11]。
2 結果和分析
2.1 卷柏AP2/ERF轉錄因子家族預測及系統進化分析
在卷柏基因組中共預測到68個AP2/ERF轉錄因子基因,基因數量遠低于高等植物中AP2/ERF的基因數量,如擬南芥(147)、葡萄(132)、水稻(164)、丹參(170)。卷柏AP2/ERF轉錄因子的長度范圍在72 aa(ST44G415)到587 aa(ST311G14)之間,其cDNA長度在219 bp到1764 bp之間。
用卷柏和擬南芥AP2保守結構域建進化樹,AP2/ERF轉錄因子家族被分為5個亞家族,分別為AP2、DREB、ERF、RAV和SOLOIST,各亞家族成員個數分別為23、16、18、8和 3,如圖1所示。擬南芥、葡萄和丹參的SOLOIST基因只有一個,水稻中未發現SOLOIST基因,但卷柏中發現3個,認為SOLOIST基因在進化過程中發生基因數量的收縮。高等植物AP2、ERF、DREB的亞家族成員遠遠多于卷柏中,證明進化過程中該亞家族存在巨大的基因擴張。利用DNAMAN軟件對擬南芥和卷柏的DREB亞家族成員保守結構域序列進行比對分析,發現AP2/ERF保守結構域(YRG和RAYD元件)在卷柏和擬南芥中高度保守,如圖2所示。
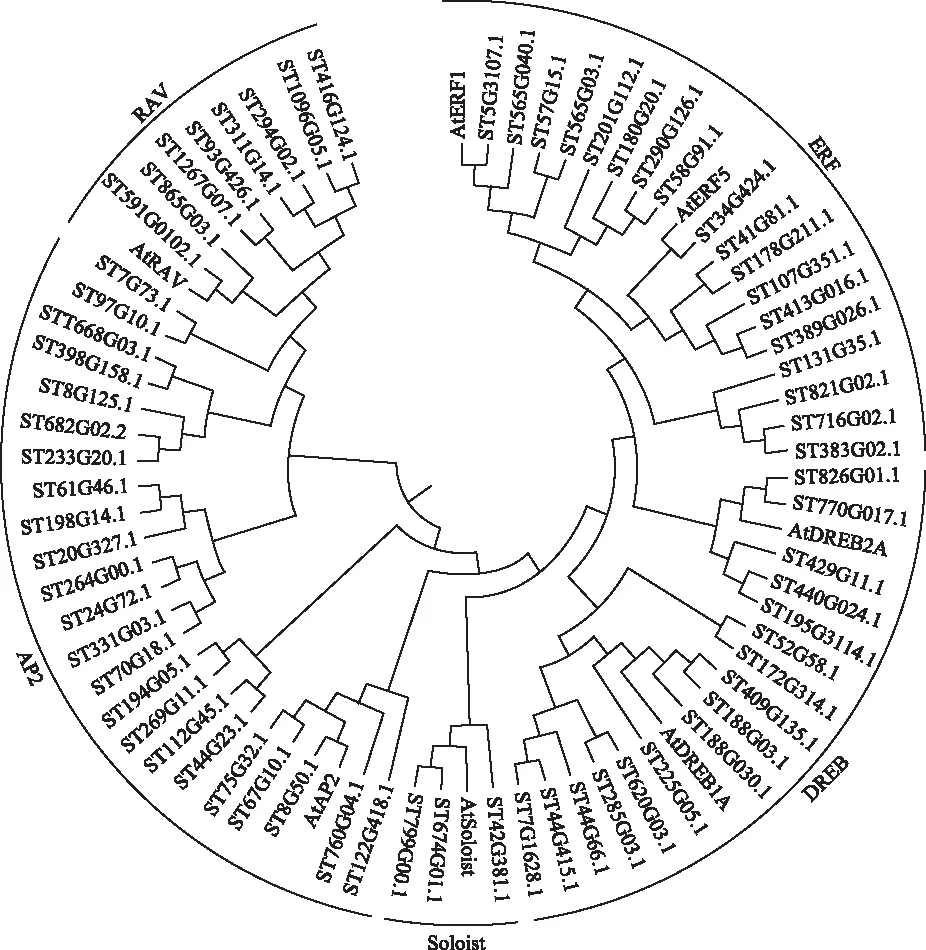
圖1 卷柏AP2/ERF基因家族系統進化分析
2.2 卷柏AP2/ERF基因家族的基因結構分析
AP2/ERF基因家族的基因結構顯示不同亞家族基因結構差異較大,兩個RAV亞家族基因(ST294G02和ST311G14)所含內含子數最多(14個),而大部分DREB、ERF亞家族成員不含內含子,如圖3所示。AP2/ERF家族成員的保守基序顯示AP2/ERF各亞家族之間序列相對保守,亞家族內部有一定差異。位于同一個亞家族內含有相同基序的成員,可能具有相似功能。
2.3 AP2/ERF轉錄因子基因的差異表達分析
AP2/ERF轉錄因子的DREB亞家族是干旱應答元件的結合蛋白,與植物干旱、高鹽等逆境密切相關。本研究對卷柏進行干旱脅迫及復水處理,HD(hydration)、70% RWC、50% RWC、30% RWC、15% RWC、5% RWC、RH 2 h(rehydration)、RH 4 h、

圖2 卷柏AP2/ERF亞家族數量分布(A)及DREB亞家族AP2/ERF保守結構域比對(B)
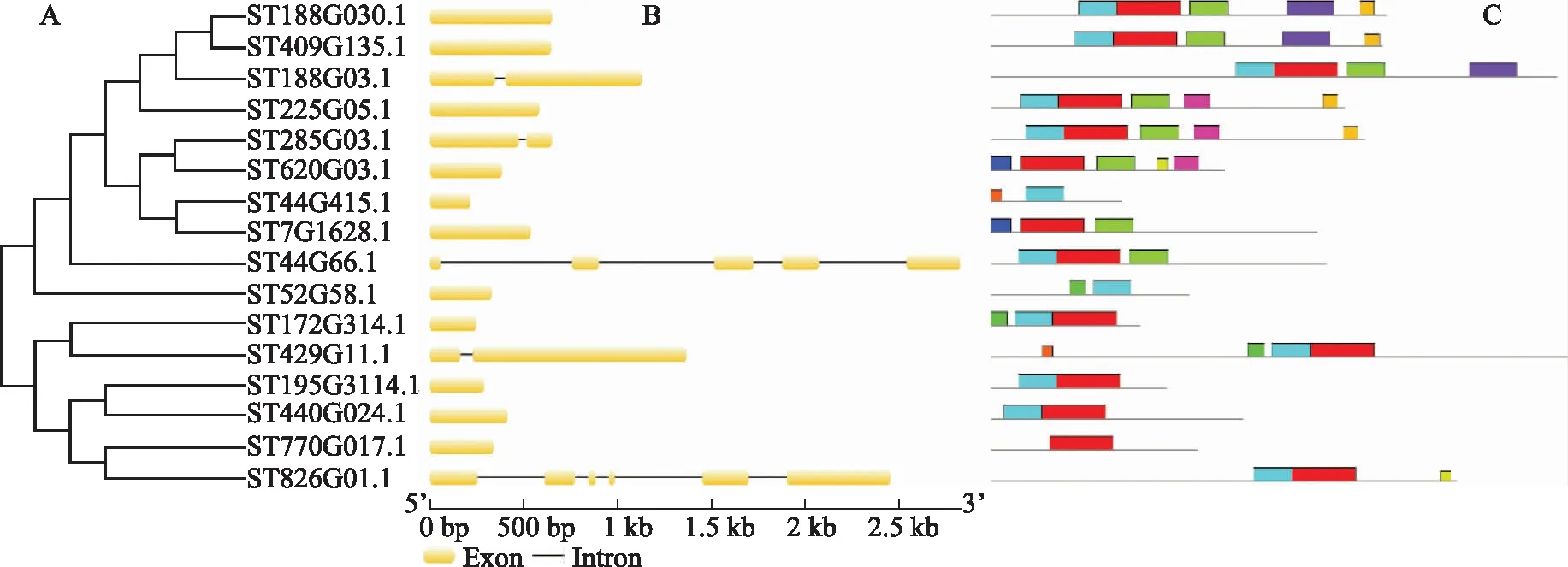
注:A.亞家族成員建樹;B.亞家族成員基因結構;C.亞家族成員蛋白保守基序分布。圖3 卷柏DREB亞家族基因結構特征和保守基序分布
RH 6 h處理的9個轉錄組數據(共100 G)比對到卷柏基因組,計算FPKM值,篩選AP2/ERF基因的表達譜,如圖4所示。FPKM值越大,說明基因表達豐度越高,反之則說明基因表達豐度越低。由于基因表達FPKM值差異顯著,將FPKM值用scale函數做Z-score標準化處理,顯示基因在不同處理下的表達差異并聚類。從基因表達模式來看,HD、70% RWC和RH 6 h的AP2/ERF轉錄因子基因表達聚在一起,而干旱脅迫嚴重的50% RWC、30% RWC、15% RWC、5% RWC、RH 2 h(rehydration)、RH 4 h的基因表達聚為另一枝。ST429G11、ST44G415和ST565G040基因的表達聚在一起,響應干旱脅迫最為敏感的一枝,其中ST429G11和ST44G415屬于DREB亞家族,ST565G040為ERF亞家族。
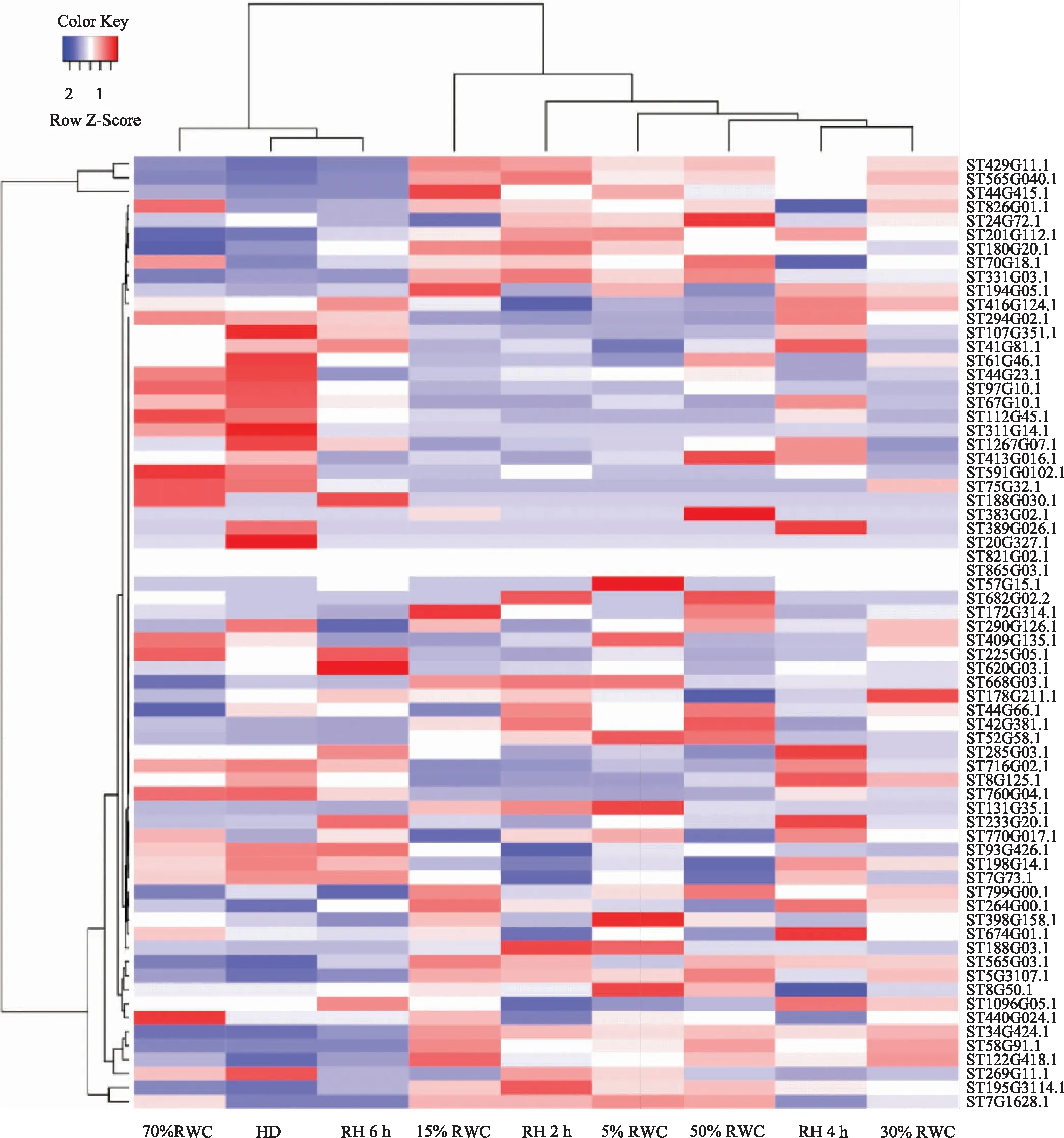
圖4 AP2/ERF轉錄因子在卷柏干旱脅迫及復水處理下的表達圖譜
47%的AP2/ERF基因家族成員在干旱脅迫及復水處理下顯示高表達(FPKM>10),15%的AP2/ERF基因家族成員表達沉默(FPKM<1),推測為沉默基因或假基因。17個基因與卷柏干旱及復水表型顯著正相關,呈共表達,在干旱脅迫后顯著高表達[log2(50%RWC/HD)>1],且復水后基因表達降低,其中包括7個DREB亞家族、6個ERF亞家族和4個AP2家族成員,如圖5所示。推測在卷柏干旱脅迫后顯著高表達的AP2/ERF轉錄因子在卷柏生長發育及脅迫響應進程中起重要的調控作用。
卷柏中表達最高的兩個AP2/ERF轉錄因子成員均屬于DREB亞家族,分別為ST429G11和ST44G415,其FPKM值在5%RWC狀態下高達2833和3977,分別是HD狀態下的12倍和74倍。啟動子結合元件顯示ST429G11的啟動子序列含有AREB轉錄因子結合的ABA響應元件(ABRE,ACGTGG)、GRF7轉錄因子結合的GTE元件(TGTCAGG)和熱激元件(HSE,AAAAAATTAC)的結合位點;ST44G415的啟動子序列含有 GRF7轉錄因子結合的GTE元件(TGTCAGG)的結合位點。研究推測ST429G11為ABA非依賴的調控途徑中響應干旱脅迫的重要調控因子。

注:不同的線條顏色代表17個干旱脅迫相關的AP2/ERF編碼基因。圖5 候選參與卷柏干旱脅迫響應的AP2/ERF基因共表達
3 討論
本研究基于卷柏全基因組測序,對其AP2/ERF家族成員進行系統鑒定,篩選參與卷柏干旱脅迫進程密切相關的AP2/ERF轉錄因子。研究發現卷柏DREB亞家族成員主要參與其非生物脅迫響應進程如干旱脅迫,與已報道的其他植物的DREB功能相一致。
在卷柏基因組中共鑒定到68個AP2/ERF轉錄因子,遠遠低于其他高等植物AP2/ERF家族成員數量,與低等植物齒肋赤蘚Syntrichiacaninervis的數量(80)接近[19],低等植物到高等植物AP2/ERF基因數量的擴張反映了植物在進化過程中響應非生物脅迫和生物脅迫的轉錄因子調控的重要性及復雜性。此外,通過多序列比對及保守基序分析發現,卷柏AP2/ERF轉錄因子的AP2/ERF結構域與高等植物擬南芥AP2/ERF的存在差異,但核心保守結構域一致,表明該轉錄因子在物種進化中基因結構具有較高的保守性。
DREB亞家族和ERF亞家族結構相似,均具有一個AP2/ERF亞家族,且一般不含有內含子,主要區別在于保守AP2結構域的第14和19位氨基酸不同。兩個亞家族功能亦相似,在植物響應生物及非生物脅迫時發揮重要的調控作用。在藥用植物領域,干旱、高鹽等非生物脅迫或生物脅迫顯著影響藥用植物品質,AP2/ERF轉錄因子的調控作用也陸續被發現,同樣主要來自于DREB和ERF亞家族成員的調控,如長春花ERF亞家族成員ORCA3可以調控萜類吲哚生物堿代謝途徑中的多個關鍵酶,影響長春堿的產量[20]。本研究發現:卷柏的DREB和ERF亞家族系統進化關系較近,聚在一大枝;卷柏的干旱脅迫響應極為敏感的AP2/ERF轉錄因子基因絕大部分來自于DREB和ERF亞家族。從復蘇植物卷柏干旱響應調控因子AP2/ERF成員的結構保守性推測其在卷柏干旱脅迫進程中的調控機制與高等植物相似,但仍需要進一步的功能鑒定。卷柏AP2/ERF轉錄因子的挖掘將豐富植物干旱響應的基因資源庫,為卷柏復蘇分子機制的解析奠定基礎,同時有助于研究植物進化過程中AP2/ERF轉錄因子的基因擴張或功能分化。
[1] Giarola V,Hou Q,Bartels D.Angiosperm plant desiccation tolerance:hints from transcriptomics and genome sequencing[J].Trends Plant Sci,2017,22(8):705-717.
[2] Farrant J M,Moore J P.Programming desiccation-tolerance:from plants to seeds to resurrection plants[J].Curr Opin Plant Bio,2011,14(3):340-345.
[3] Vanburen R,Bryant D,Edger P P,et al.Single-molecule sequencing of the desiccation-tolerant grassOropetiumthomaeum[J].Nature,2015,527:508.
[4] Costa M C D,Artur M A S,Maia J,et al.A footprint of desiccation tolerance in the genome ofXerophytaviscosa[J].Nat Plants,2017,3(4):17038.
[5] Xiao L,Yang G,Zhang L,et al.The resurrection genome ofBoeahygrometrica:A blueprint for survival of dehydration[J].Proc Natl Acad Sci USA,2015,112(18):5833-5837.
[6] Banks J A.Selaginellaand 400 Million Years of Separation[J].Annu Rev Plant Biol,2009,60:223.
[7] Korall P,Kenrick P.The phylogenetic history of Selaginellaceae based on DNA sequences from the plastid and nucleus:extreme substitution rates and rate heterogeneity[J].Mol Phylogenet Evol,2004,31(3):852-864.
[8] 中國科學院《中國植物志》編委會.中國植物志:第6卷[M].北京:科學出版社,2004:313
[9] 國家藥典委員會.中華人民共和國藥典:一部[S].北京:中國醫藥科技出版社,2015:226
[10] Wang X,Chen S,Zhang H,et al.Desiccation Tolerance Mechanism in Resurrection Fern-AllySelaginellatamariscinaRevealed by Physiological and Proteomic Analysis[J].J Proteome Res,2010,9(12):6561-6577.
[11] Yobi A,Wong B W,Xu W,et al.Comparative metabolic profiling between desiccation-sensitive and desiccation-tolerant species ofSelaginellareveals insights into the resurrection trait[J].Plant J,2012,72(6):983-999.
[12] Licausi F,Ohme-Takagi M,Perata P.APETALA2/Ethylene Responsive Factor(AP2/ERF)transcription factors:mediators of stress responses and developmental programs[J].New Phytol,2013,199(3):639-649.
[13] 季愛加,羅紅梅,徐志超,等.藥用植物轉錄因子AP2/ERF研究與展望[J].科學通報,2015(14):1272-1284.
[14] Sakuma Y,Liu Q,Dubouzet J G,et al.DNA-binding specificity of the ERF/AP2 domain ofArabidopsisDREBs,transcription factors involved in dehydration-and cold-inducible gene expression[J].Biochem Biophys Res Commun,2002,290(3):998-1009.
[15] Nakano T,Suzuki K,Fujimura T,et al.Genome-wide analysis of the ERF gene family inArabidopsisand rice[J].Plant Physiol,2006,140:411-432.
[16] Licausi F,Giorgi F M ,Zenoni S,et al.Genomic and transcriptomic analysis of the AP2/ERF superfamily inVitisvinifera[J].BMC Genomics,2010,11:719.
[17] Ji A J,Luo H M,Xu Z C,et al.Genome-wide identification of the AP2/ERF gene family involved in active constituent biosynthesis inSalviamiltiorrhiza[J].Plant Genome,2016,9(2):1-11.
[18] Trapnell C,Roberts A,Goff L,et al.Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J].Nat Protoc,2012,7:562-578.
[19] Li X,Zhang D,Gao B,et al.Transcriptome-wide identification,classification,and characterization of AP2/ERF family genes in the desert mossSyntrichiacaninervis[J].Front in Plant Sci,2017,8(47):262.
[20] Wang C T,Liu H,Gao X S,et al.Overexpression of G10H and ORCA3 in the hairy roots ofCatharanthusroseusimproves catharanthine production[J].Plant Cell Rep,2010,29:887-894.

