基于熵值的冠心病基因網絡模塊劃分方法評價與模塊功能相似度分析
顧 浩,陳寅螢,王朋倩,王 忠
(中國中醫科學院1.中醫臨床基礎醫學研究所,3.中藥研究所,北京 100700;2.中國中醫科學院廣安門醫院科研處,北京 100053)
冠心病是冠狀動脈粥樣硬化使血管腔狹窄或阻塞或(和)因冠狀動脈功能性改變(痙攣)導致心肌缺血缺氧或壞死而引起的一類心臟病[1]。目前已知的冠心病致病基因達上百個,且關系網絡復雜,利用單基因單靶點的一對一分析,很難發現核心致病基因群及其主要的生物學功能[2]。利用網絡模塊劃分方法,可以更好地分析疾病網絡的結構和功能,有助于發現疾病關鍵基因群[3]。
隨著組學技術和大數據的發展,生命科學在人體和疾病的生物分子層面積累了大量的數據[4-5],網絡為大量生物信息的整合提供了有效手段[6-7]。基于生物網絡的模塊劃分方法,成為網絡簡化分解和抽提新知識的重要途徑[8-9]。從網絡節點在模塊中的分布重疊情況,模塊劃分方法可以分為重疊模塊和非重疊模塊劃分方法。目前,常用的重疊模塊劃分方法主要包括MCLiQUE,Cluster one,NCMine,PEW?CC和Fuzzifier cluster等算法[10]。非重疊模塊劃分方 法 包 括 Community cluster(glay),Connected component cluster,MCL cluster,MCODE cluster,Spectral clusters of protein sequences和AP等[11]。
面對眾多的模塊劃分方法,如何選擇一個優化的模塊劃分方法對網絡進行結構和功能解析,從而得到一個相對穩定可靠的結果,是目前模塊研究的重要內容。有研究者提出了網絡結構熵的概念,可以有效地刻畫無尺度網絡的無序性[12-15]。本研究團隊前期利用網絡結構熵計算方法,對中藥成分干預腦缺血的藥理網絡進行了模塊劃分結果的熵值計算[15]。用最小網絡結構熵刻畫節點間的關系,熵越小,節點間越相似,那么識別出來的功能模塊就越穩定,這為本研究進一步篩選疾病網絡模塊劃分方法提供了可借鑒的經驗。
本研究以冠心病為例,利用重疊模塊和非重疊模塊等多種常用模塊劃分方法,解析冠心病基因網絡;通過最小網絡結構熵值,判定適用于冠心病基因的模塊識別的最佳方法;利用杰卡德相似系數(Jaccard similarity coefficient)分析冠心病網絡和模塊網絡功能的相關性,為簡化和理解疾病網絡提供一種可行方案。
1 數據來源與方法
1.1 數據來源
2017年6月10日,以“coronary heart disease”為關鍵詞檢索美國NCB(I生物科技信息中心基因數據庫,National Center for Biotechnology Infor?mation,https://www.ncbi.nlm.nih.gov/gene/),收集到302個冠心病相關基因。
利用String數據庫的文本挖掘功能,發現上述基因之間的相關關系,從而構建基因關系網絡。將302個冠心病相關基因,輸入String數據庫,選擇智人(Homo sapiens)屬,能夠識別其中284個基因。在此基礎上,以綜合積分(score≥0.4)為條件篩選基因之間的相關關系,得到1個由284個節點和3039條邊構成的冠心病相關基因關系網絡,相關參數見表1。從表1可以看出,網絡中共有284個節點,3039條邊,平均節點度21.4,平均局部聚集系數0.531,期望邊數881條。
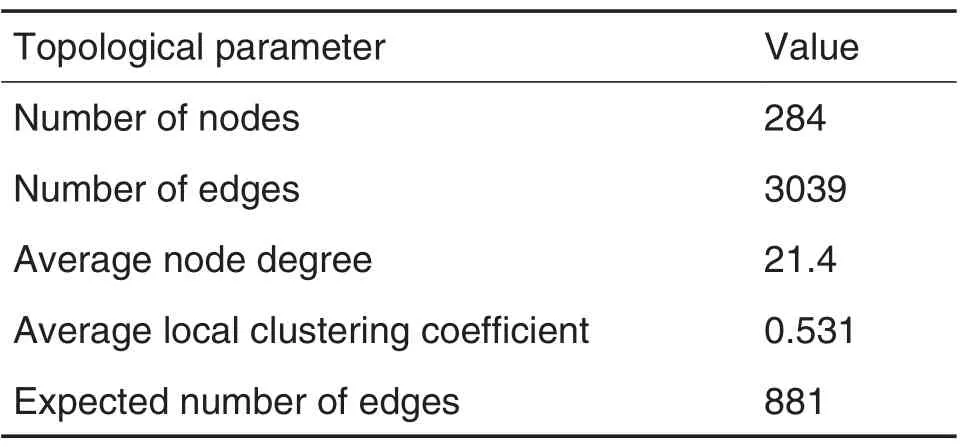
Tab.1 Topological parameters of coronary heart disease related gene network
本研究利用多種模塊劃分方法對冠心病網絡進行了模塊劃分,并利用David數據庫(DAVID Bioin?formatics Resources 6.8,https://david.ncifcrf.gov/summary.jsp)進行了冠心病網絡的相關通路富集分析。
1.2 研究方法
1.2.1模塊劃分方法
本研究對前述的11種常用方法均進行了計算,除AP算法不能成功劃分出模塊,其他方法均可。因此,本研究在能劃分出模塊方法的基礎上進一步開展熵值計算。
1.2.2熵值計算方法
網絡結構熵(network structure entropy)[15],計算公式如下:
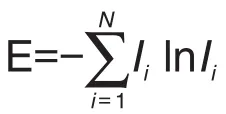
其中,N為網絡中節點數目,Ii為第i個節點的重要度。網絡結構熵由節點的連接度分布確定,因此,節點的重要度定義為每個節點連接度占所有節點連接度總和的比例。
1.2.3杰卡德相似系數
杰卡德相似系數用來度量2個集合之間的相似性,被定義為2個集合A和B的交集元素在A和B的并集中所占的比例,稱為2個集合的杰卡德相似系數,用符號J(A,B)表示。公式如下:
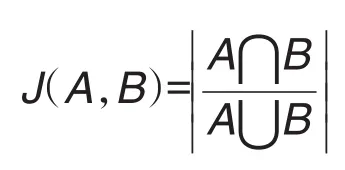
利用David數據庫富集模塊的相關通路,可以得到每個模塊的通路集合。要比較模塊相似性,可以通過比較不同模塊的通路集合的相似系數,評價模塊之間功能的相關性。
1.3 研究技術路線
根據NCBI數據庫收集的冠心病相關基因通過String數據庫進行基因關系網絡的構建。針對冠心病基因關系網絡,利用12種重疊模塊和非重疊模塊的劃分方法,進行網絡模塊劃分,得到12個模塊劃分結果網絡。利用網絡信息熵的計算方法對12個結果進行評價,確定熵值最低的劃分結果以及相應的劃分方法為MCODE cluster。用MCODE cluster方法劃分共得到11個模塊,利用David數據分析平臺進行基因本體(gene ontology,GO)功能富集分析,分析冠心病基因網絡和11個模塊的生物信號通路。利用杰卡德相似系數比較劃分前后網絡的生物信號通路之間的相似度,從而評價網絡模塊劃分結果是否與原網絡功能一致(圖1)。
2 結果
2.1 模塊劃分方法比較
基于11種模塊劃分方法劃分模塊,然后計算模塊網絡熵值(表2)。熵值越小,網絡的混亂度越低,越趨于穩定。上述方法劃分模塊后,計算熵值,比較熵值大小,發現MCODE cluster方法得到熵值最低,為4.33637。說明MCODE cluster方法比較適合冠心病基因關系網絡的模塊劃分。因此,選取MCODE cluster方法劃分的模塊網絡為研究對象,進行下一步計算和分析。
2.2 基于最優算法MCODE cluster的模塊劃分結果
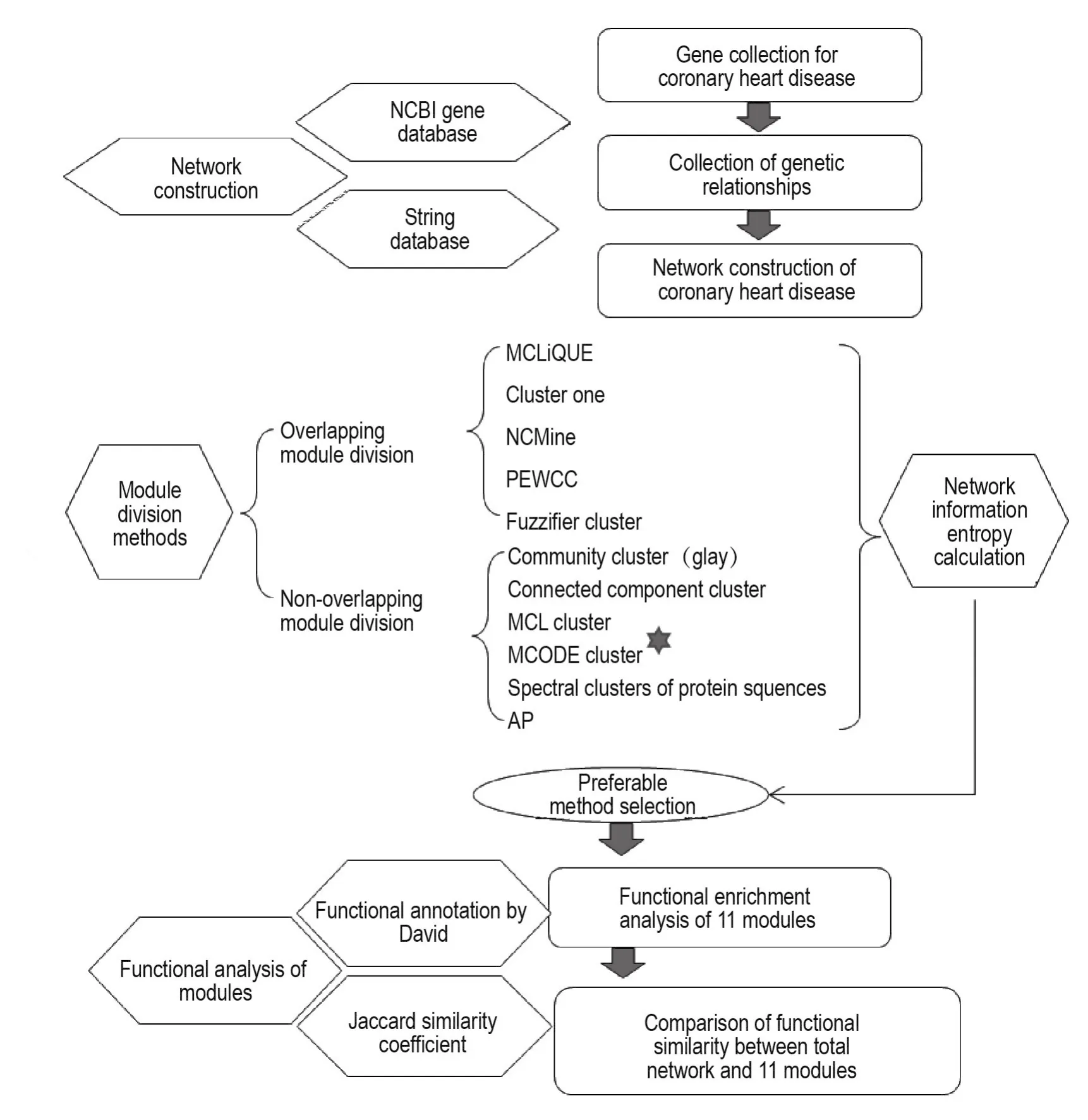
Fig.1 Work flow diagram for gene network construction and analysis of coronary heart disease.The work flow diagram consists of four parts:the construction of the disease gene network,the module division based on 11 methods,the module division method evaluation by entropy value,and the function analysis of the module division result.★:the method with the lowest entropy.

Tab.2 Entropy calculation of results of module division
基于MCODE cluster方法,劃分模塊網絡結果如圖2。從圖2可以看出,11個內部相互連接、外部相對獨立的網絡集團即為模塊。基于MCODE cluster方法,冠心病基因網絡被劃分為11個模塊,詳細信息如表3所示。圖2中第1排從左到右依次為1~5號模塊,從表3中可以看到相關模塊內包含的基因名稱。圖1第2排從左到右,依次為6~11號模塊。其中,模塊1包含51個節點,926條邊,是網絡中最大的模塊。模塊8,9,10和11均包含3個節點,3條邊,是網絡中最小的模塊。
2.3 基于David數據庫冠心病基因功能富集分析
為探討原網絡和劃分后的模塊網絡在生物功能上的相關性,基于David生物信息分析平臺,對冠心病原網絡和劃分后11個模塊網絡進行基因的相關信號通路富集分析。最終富集結果發現,冠心病原網絡中基因涉及52條KEGG信號通路(P<0.05),由于篇幅有限,表4中展示其中15條通路。
11個模塊富集結果總共涵蓋冠心病相關通路中的38條通路,覆蓋率達73.1%。在功能上說明劃分后的模塊能夠表達原疾病網絡的絕大部分功能。圖3可以看出11個模塊分別在冠心病通路中所占的比例分布。模塊1占46.6%,模塊2占1.6%,模塊3占7.8%,模塊4占4.7%,模塊5和6占0.0%,模塊7占10.9%,模塊8,9和10占0.0%,模塊11占1.6%。
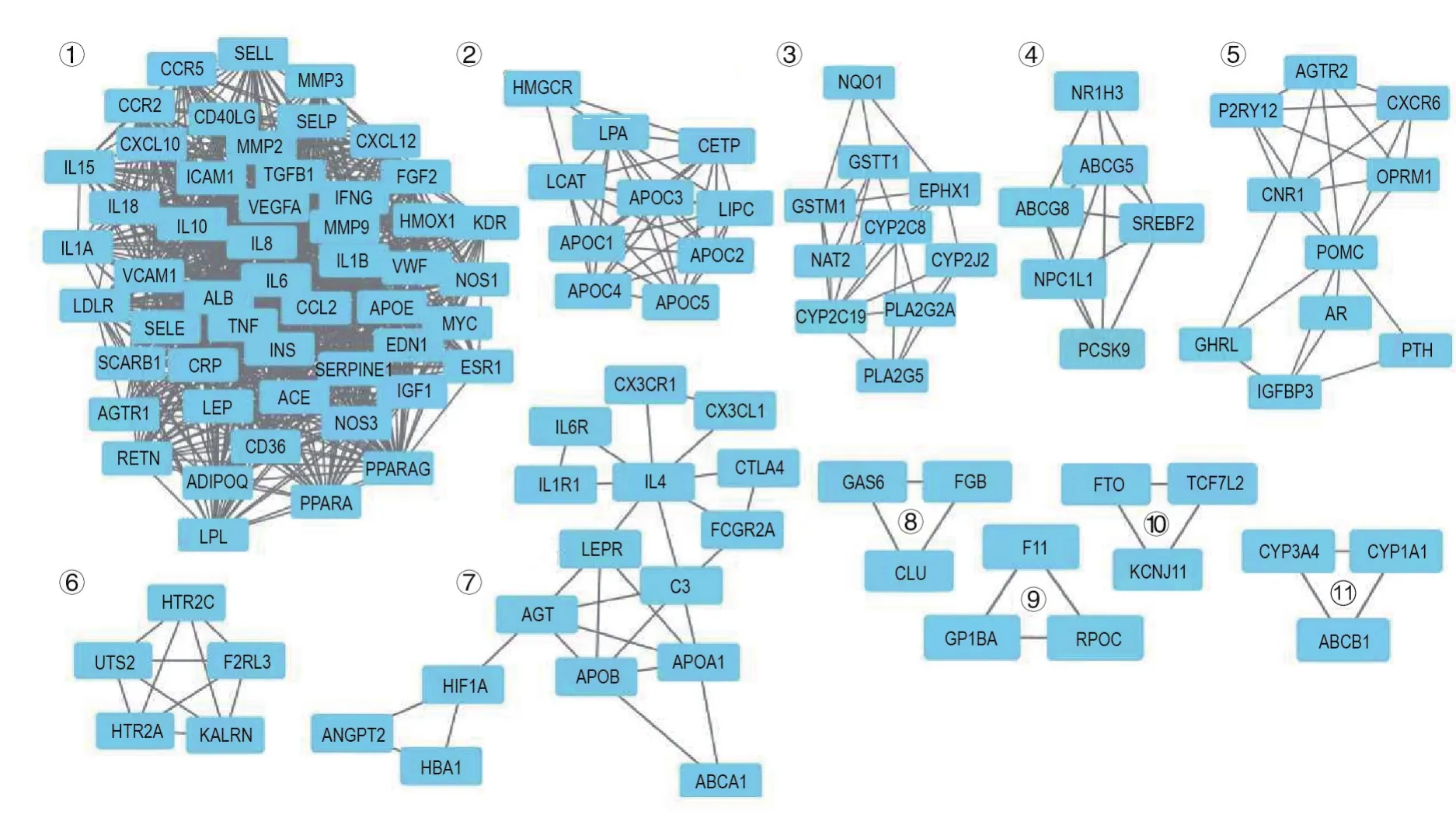
Fig.2 Eleven modules of coronary heart disease network divided by MCODE method.SEL:selectin;CCR:C-C motif chemokine receptor;MMP:matrix metallopeptidase;CXCL:C-X-C motif chemokine ligand;IL:interleukin;ICAM:intercellular adhesion molecule;TGFB:transforming growth factor beta;FGF:fibroblast growth factor;VEGFA:vascular endothelial growth factor A;IFNG:interferon gamma;KDR:kinase insert domain receptor;VCAM:vascular cell adhesion molecule;VWF:von Willebrand factor;LDLR:low density lipoprotein receptor;ALB:albumin;NOS:nitric oxide synthase;CCL:C-C motif chemokine ligand;TNF:tumor necrosis factor;SCARB:scavenger receptor class B;CRP:C-reactive protein;EDN1:endothelin 1;INS:insulin;SERPINE:serpin family E;ESR1:estrogen receptor 1;AGTR:angiotensin Ⅱ receptor;LEP:leptin;ACE:angiotensin I converting enzyme;IGF:insulin like growth factor;RETN:resistin;ADIPOQ:adiponectin;C1Q and collagen domain containing;PPAR:peroxisome proliferator activated receptor;HMGCR:3-hydroxy-3-methylglutaryl-Co A reductase;CETP:cholesteryl ester transfer protein;LCAT:lecithin-cholesterol acyltransferase;APO:apolipoprotein;LIPC:lipase C;hepatic type;NQO:NAD(P)H quinone dehydrogenase;GST:glutathione S-transferase;EPHX1:epoxide hydrolase 1;CYP:cytochrome P450;NAT:N-acetyltransferase;PLA:phospholipase A;NR1H:nuclear receptor subfamily 1 group H;ABC:ATP binding cassette;SREBF:sterol regulatory element binding transcription factor;NPC1L:NPC1 like intracellular cholesterol transporter;PCSK:proprotein convertase subtilisin/kexin;CXCR:C-X-C motif chemokine receptor;P2RY12:purinergic receptor P2Y12;CNR:cannabinoid receptor;POMC:proopiomelanocortin;AR:androgen receptor;GHRL:ghrelin and obestatin prepro?peptide;PTH:parathyroid hormone;IGFBP:insulin like growth factor binding protein;HTR:5-hydroxytryptamine receptor;UTS:urotensin;F2RL:F2R like thrombin or trypsin receptor;HTR:5-hydroxytryptamine receptor;KALRN:kalirin RhoGEF kinase;CX3C:C-X3-C motif chemokine;CTLA:cytotoxic T-lymphocyte associated protein;FCGR:Fc fragment of IgG receptor;HIF1A:hypoxia inducible factor 1 alpha subunit;ANGPT:angiopoietin;HBA1:hemoglobin subunit alpha 1;GAS:growth arrest specific;FGB:fibrinogen beta chain;CLU:clus?terin;FTO:alpha-ketoglutarate dependent dioxygenase;TCF:transcription factor;F11:coagulation factor XI;GP1BA:glycoprotein Ib platelet alpha subunit;PROC:protein C,inactivator of coagulation factors Va and VIIIa;KCNJ:potassium voltage-gated channel subfamily J.
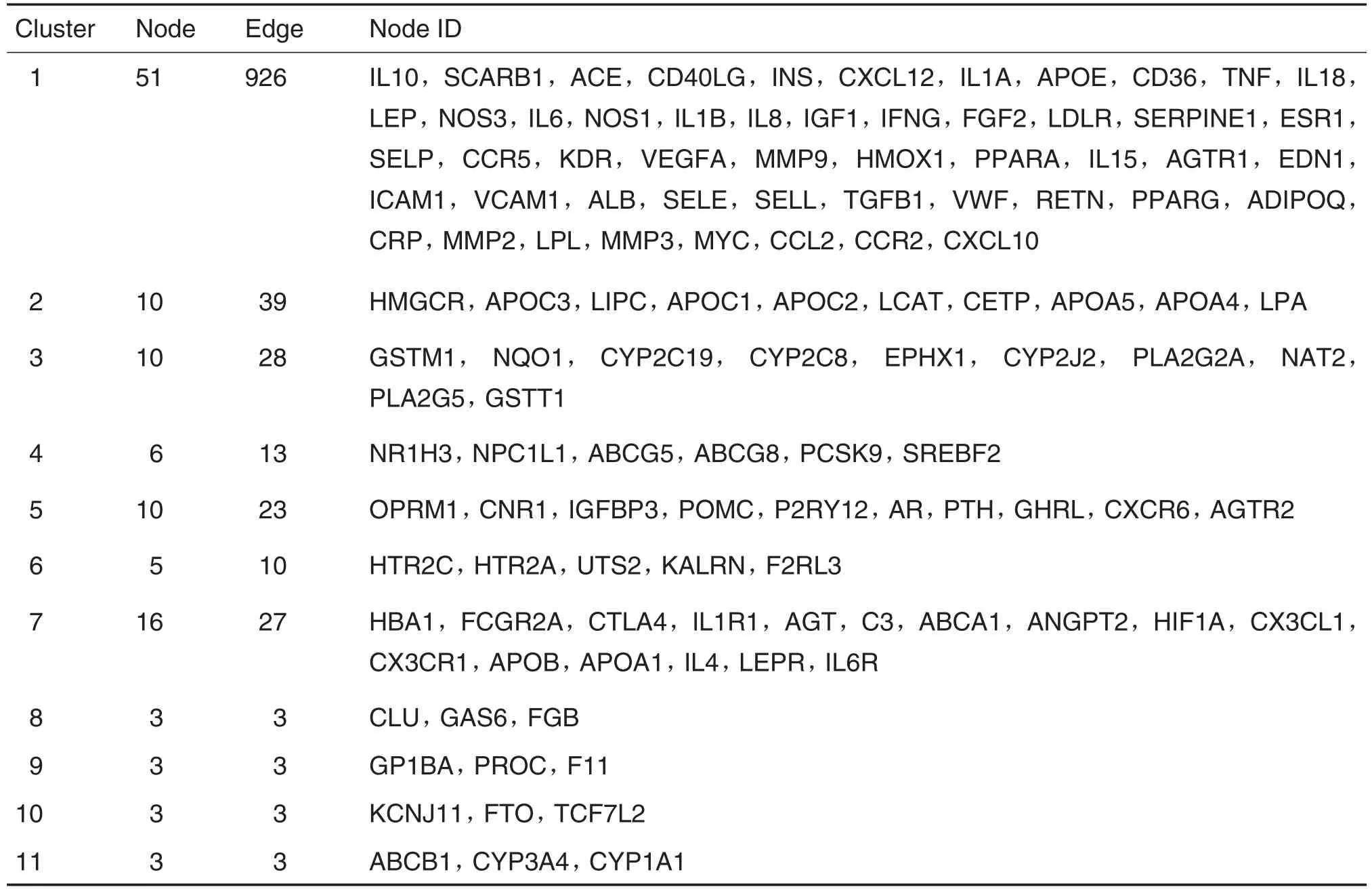
Tab.3 Eleven modules of coronary heart disease network
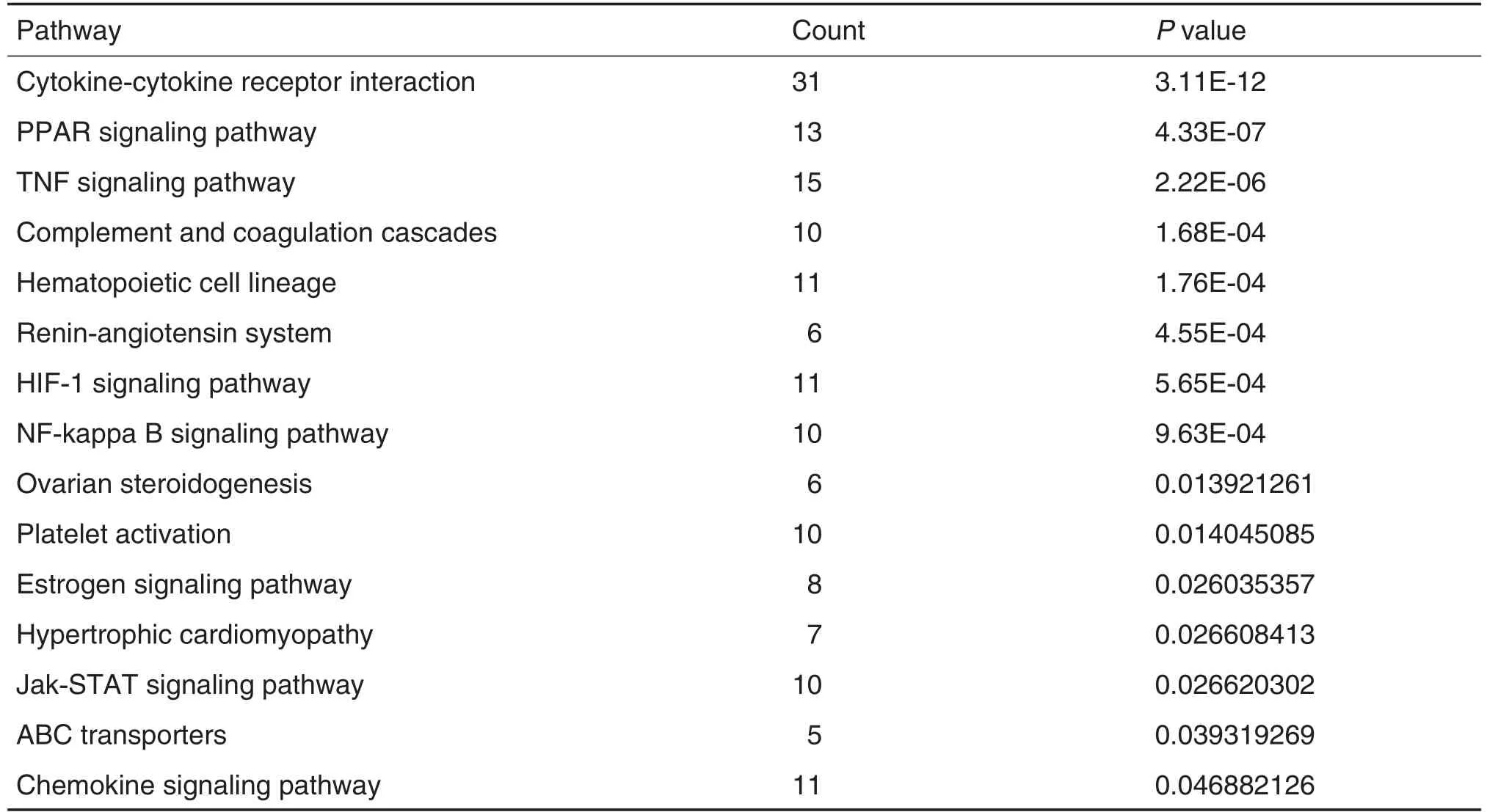
Tab.4 Pathway enrichment of coronary heart disease related genes
MCODE cluster模塊劃分方法對劃分出的網絡模塊進行了評分和排序,密度大模塊評分高,根據評分高低對模塊編號和排序[16]。按照這一評分方案作為判定模塊主次的依據,則該方法劃分的主要模塊為1號模塊,其涉及通路包括過氧化物酶體增生物激活受體信號通路、腫瘤壞死因子信號通路、造血細胞譜系、雌激素信號通路、肥厚性心肌病、Jak-STAT信號通路和趨化因子信號通路等30個信號通路,占冠心病相關通路的46.6%。因此,主要模塊與冠心病功能一致度較高。
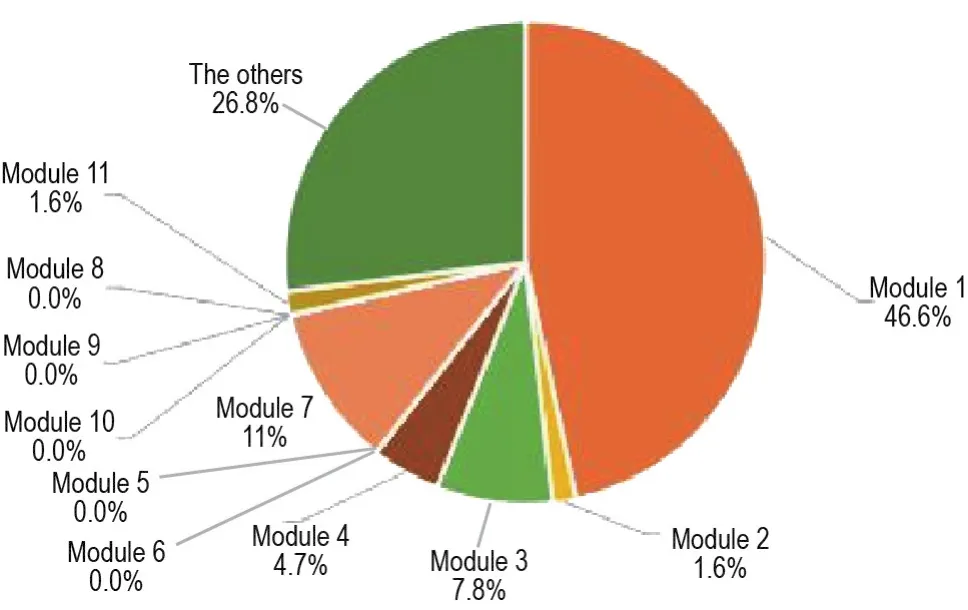
Fig.3 Distribution of module-related pathways in coro?nary heart disease.
3.4 利用杰卡德相似系數模塊功能相似度分析,驗證基于熵值所獲得的冠心病基因網絡模塊劃分結果的合理性
無論模塊如何劃分,最終目的都是在復雜網絡中找到結構和功能相對獨立的基團。熵值的方法從網絡結構和能量角度,評價了模塊劃分方法的生物穩定性。基于杰卡德相似系數的模塊功能相似度分析,可以評價各個模塊與疾病功能的相關性。因此,本部分采用杰卡德相似系數,從生物功能角度,驗證基于熵值所獲得的冠心病基因網絡模塊的合理性。
表5中顯示了11個模塊所富集到的通路數量、與疾病總網絡相比較的交集和并集,以及通過交集與并集計算的杰卡德相似系數。同時列出了11個模塊基因數與疾病總網絡基因數的比例。
利用折線圖4,可以更直觀地顯示基因占比和杰卡德相似系數的關系。橫坐標表示模塊編號,縱坐標代表模塊網絡與疾病總網絡相比較,在通路功能和基因數量上所占的比例,即杰卡德相似系數和模塊基因數占比。
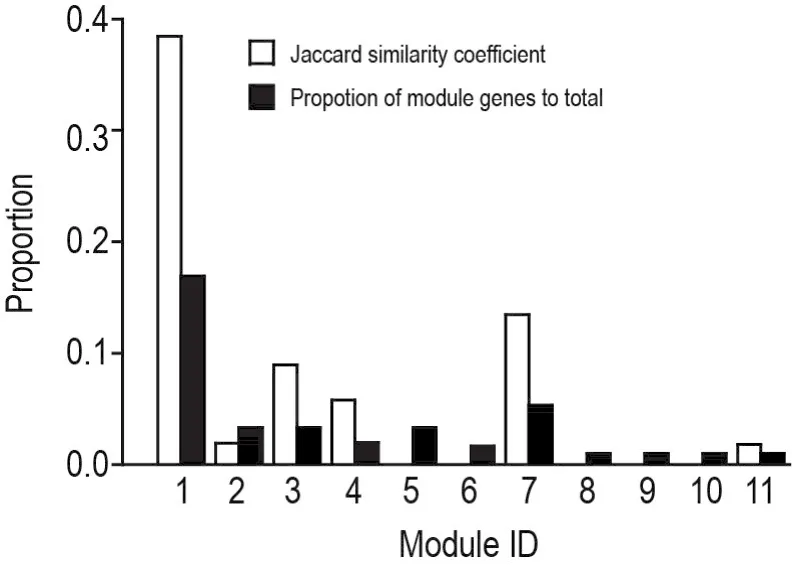
Fig.4 Comparison of number and function of genes between coronary heart disease and its modules.
從結果可以看出,杰卡德相似系數在模塊1,3,4,和7中,相對于基因占比平均高出1倍以上。模塊1基因數占比接近0.17,但代表疾病相關通路占比的杰卡德相似系數卻超過0.38。同理,模塊3,4和7均用較少的基因富集到較多的功能。這一結果表明,模塊在功能富集上具有一定優勢。模塊2,5,6,8,9,10和11基因占比與功能杰卡德相似系數基本一致,差異較小。通過此方法,還可判斷功能富集能力較高的模塊,從而為評價主要模塊和次要模塊提供另一個可參考指標。
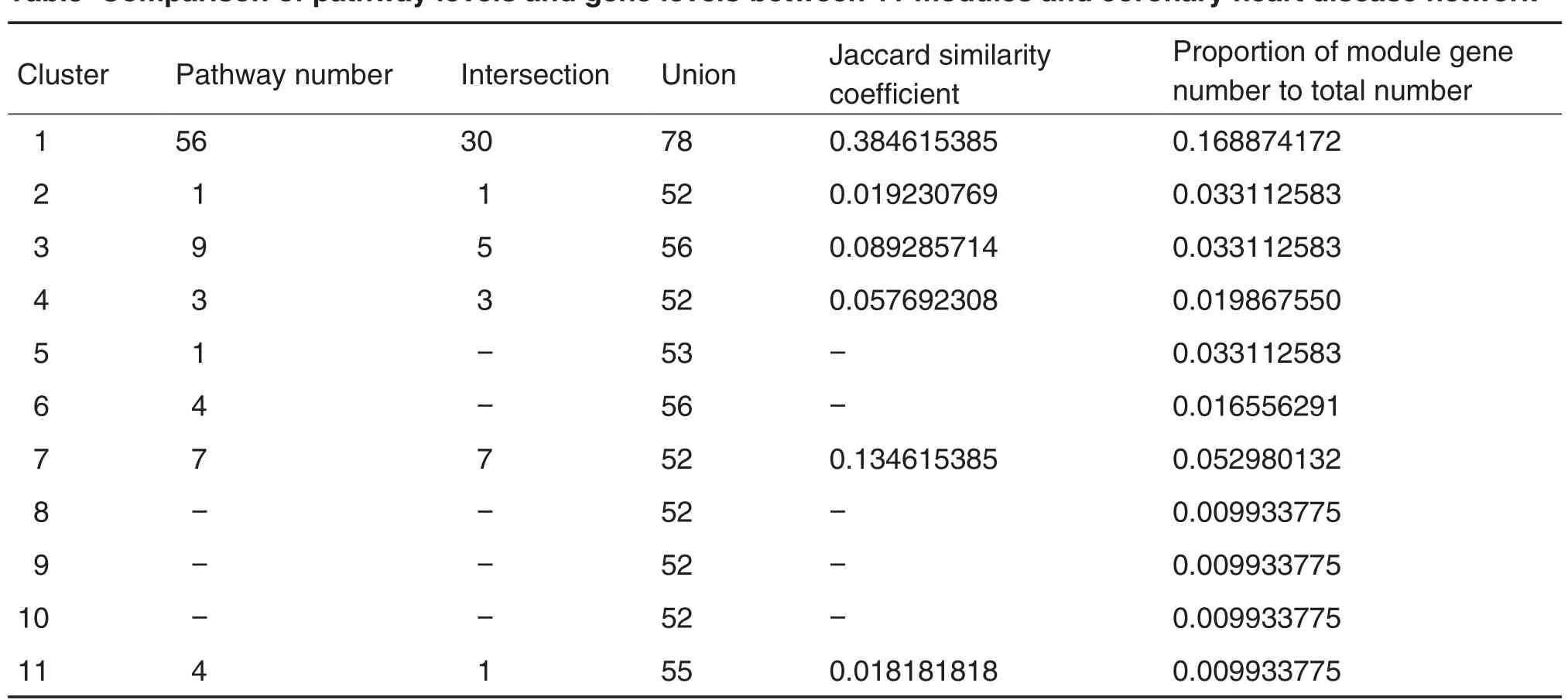
Tab.5 Comparison of pathway levels and gene levels between 11 modules and coronary heart disease network
3 討論
基于生物網絡的模塊劃分方法,旨在實現將大量分子作功能分區的劃歸,化繁為簡,達到從不同尺度理解生命活動的層級和結構的目的,從而針對分區進行有效的干預和調控。但目前模塊劃分方法多樣,如何評價和選擇適合疾病網絡的模塊劃分方法,是值得醫藥學關注的問題。
本研究通過多種常用模塊劃分方法,對冠心病基因網絡進行了模塊劃分,劃分結果通過網絡結構熵值計算進行評價,發現MCODE cluster方法劃分出11個模塊,網絡熵值為4.33637,在幾種方法中熵值最小,較為適用于冠心病基因網絡的模塊識別。基于David數據庫進行基因功能富集分析,發現冠心病相關基因涉及52條KEGG信號通路(P<0.05),11個模塊總共能涵蓋冠心病相關通路中的38條通路,覆蓋率達到73.07%,說明模塊網絡能夠表達原疾病網絡的絕大部分功能。利用杰卡德相似指數分析,發現第3,4和7模塊都用較少的基因,富集到較多的功能,這一結果表明模塊在功能富集上有一定優勢。
本研究使用的網絡結構熵方法,具有明確的物理意義,熵值越小,網絡的混亂度越低,越趨于穩定,適用于對所有模塊劃分結果網絡的穩定性評價。通過對冠心病基因網絡的實踐,表明該方法能夠為基因相關疾病網絡的簡化和解析提供依據,并為進一步基于疾病模塊的組合藥物設計與開發奠定基礎。

