大豆抗大豆花葉病毒病基因研究進展
王大剛,李凱,智海劍
?
大豆抗大豆花葉病毒病基因研究進展
王大剛1,李凱2,智海劍2
(1安徽省農業(yè)科學院作物研究所/安徽省農作物品質改良重點實驗室,合肥 230031;2南京農業(yè)大學大豆研究所/國家大豆改良中心/ 作物遺傳與種質創(chuàng)新國家重點實驗室,南京 210095)
大豆花葉病毒(soybean mosaic virus,SMV)病是嚴重危害世界大豆((L.) Merr.)生產的主要病害之一。近十年來,國內外關于大豆對SMV抗病基因的遺傳標記定位、候選抗病基因的分析及大豆抗SMV的調控網絡等研究取得許多新進展。大豆對SMV的抗性遺傳主要分為數量抗性和質量抗性,其中數量抗性的遺傳主要由1對加性主基因+加性-顯性多基因共同控制;對不同SMV株系的質量抗性遺傳分別由1對不同的顯性基因控制。標記定位研究發(fā)現,大豆對SMV數量抗性位點主要分布在大豆的第6、10和13等染色體上。22個對SMV具有單顯性質量抗性的基因位點已被標記定位在大豆的第2、6、13和14染色體上,且定位的多數抗病基因位點兩側標記間的物理距離都在1 Mb以內。其中第13染色體上的基因位點數最多,有、、R、R和R等10個,定位在第2染色體上的基因位點有8個,如、R、R、R和R等,第6和14染色體上各有2個基因位點,分別為R、R和、R。參考大豆全基因組序列(http://www.phytozome.net/soybean),利用生物信息學方法、表達譜分析及克隆測序技術等進一步縮小了大豆抗SMV候選基因的篩選范圍。目前,在大豆第2染色體上確定的抗SMV候選基因主要有8個:、、、、、、和,在第6染色體上的是,在第13和14染色體上的抗SMV候選基因分別有9個和6個:、、、、、、、和、、、、、。基于病毒誘導的基因沉默VIGS(virus induced gene silencing,VIGS)和轉基因操作等技術,研究發(fā)現抗SMV相關基因、、等參與大豆對SMV的抗性,屬于正調控因子;而和等則增加大豆對SMV的易感性,為負調控因子。在綜合SMV抗病基因的相關研究基礎上,構建了基于和介導對SMV極端抗性的調控網絡模型。介導的大豆對SMV極端抗性調控模型的建立為大豆抗SMV信號網絡的研究提供了新的方向。介導的大豆對SMV極端抗性的主要機制是通過ABA信號的傳導,從而使胞間連絲處的胼胝質沉積以抑制病毒從最初侵染的細胞向健康細胞的轉移。本文系統綜述了SMV抗病基因方面的最新研究成果并對該領域未來的研究方向進行了展望,以期為大豆抗SMV分子設計育種和抗病基因的機理研究提供參考。
大豆;大豆花葉病毒;抗病基因;標記定位;功能研究
大豆((L.) Merr.)是世界上食用植物油和植物蛋白的主要來源,生長過程中受多種病害侵染。其中,大豆花葉病毒(soybean mosaic virus,SMV)病對大豆的產量和品質均可造成嚴重危害。由于SMV分布廣、危害重、化學藥劑難以防治,一直是大豆種植面臨的主要問題。選育和種植抗病品種是控制該病最經濟、有效且對環(huán)境友好的防治方法。
篩選新的抗病種質并發(fā)掘新的抗病基因是拓寬大豆抗病遺傳基礎、成功培育抗SMV品種的關鍵。國家和各省大豆新品種審定實行SMV抗性“一票否決”制[1],對抗SMV育種起到了極大的推進作用。隨著大豆抗SMV研究的深入,王大剛等[2]、李凱等[3]曾對國內外學者在SMV抗源篩選、抗性遺傳方式和抗病基因分子標記定位等方面的研究進行了闡述。
本文主要聚焦SMV抗病基因的標記定位、候選抗病基因的分析及抗SMV相關基因的功能鑒定和參與的調控網絡等研究,通過系統的梳理和總結,以期為大豆分子設計育種、抗SMV功能基因的利用和新基因的發(fā)掘提供參考。
1 大豆對SMV抗病基因的標記定位
大豆對SMV的抗性主要存在數量抗性和質量抗性兩類[4]。數量抗性遺傳主要由1對加性主基因+加性-顯性多基因共同控制[5],利用中豆29×中豆32的重組自交系RIL(recombinant inbred lines,RIL)群體,郭丹丹等[6]研究還發(fā)現大豆對SMV株系SC3的數量抗性遺傳由2對等加性主基因控制。大豆對SMV質量抗性遺傳,國內外普遍認為是由1對顯性抗病基因控制[7-11]。通過抗病品種與感病品種的正反交組合研究發(fā)現,大白麻對SMV株系SC4及齊黃22對SC8均是由單顯性核基因控制,且抗性不受細胞質影響[12]。
迄今為止,已經命名和定位的大豆抗SMV單顯性基因位點主要有“”(resistance of soybean mosaic virus,)系列和“R”(resistance of soybean mosaic virus in China,R)系列兩類。其中“”系列抗病基因位點主要有、、和等,由美國、加拿大和韓國等學者鑒定、命名和定位[13-28](表1);“R”系列抗病基因位點主要有R、R—R、R—R、R、R、R—R、R等,由中國和印度學者鑒定、命名和定位[27-47](表1)。
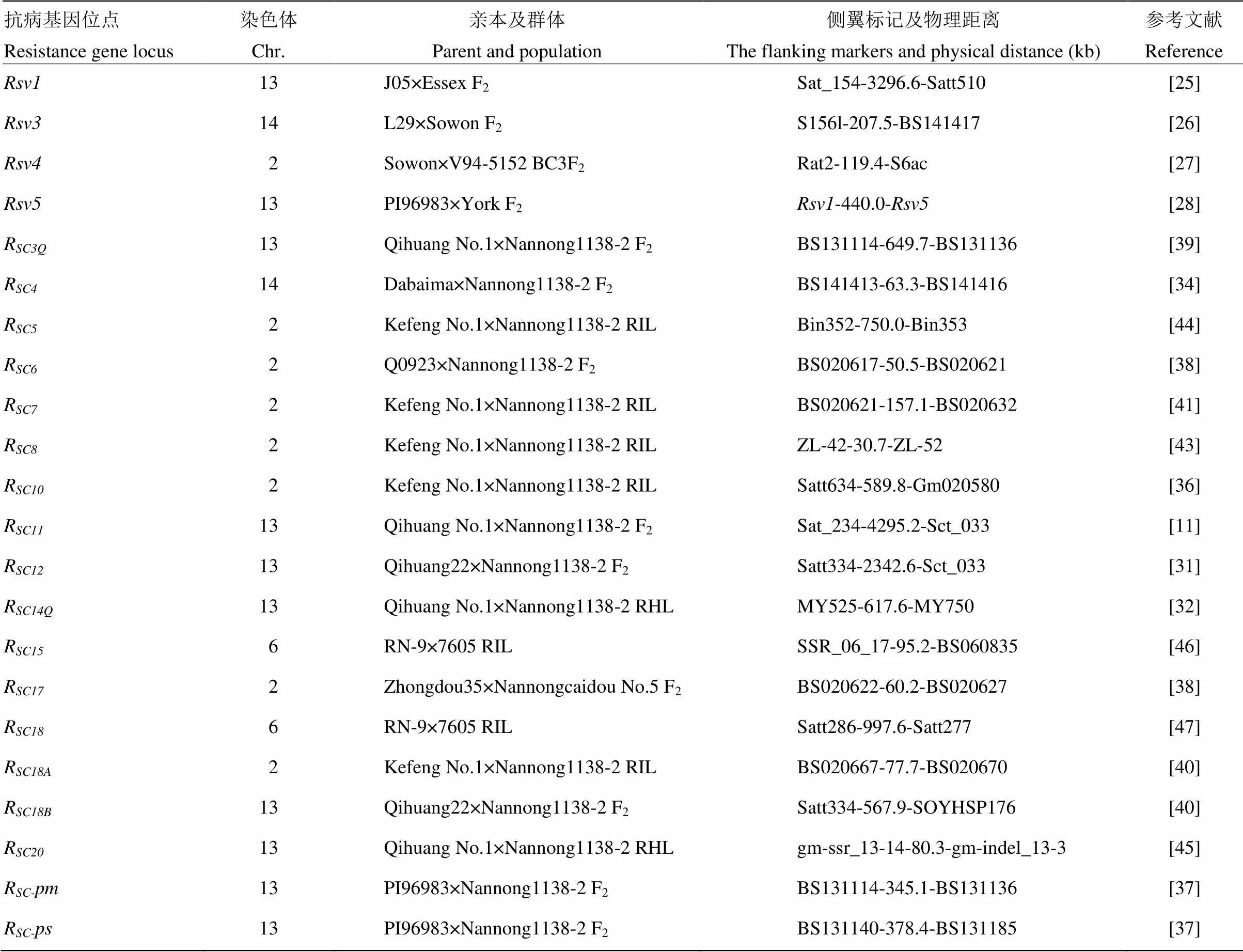
表1 大豆花葉病毒(SMV)抗病基因位點的標記定位
BARCSOYSSR_02_、BARCSOYSSR_06_、BARCSOYSSR_13_和BARCSOYSSR_14_簡寫為BS02、BS06、BS13和BS14。下同
BARCSOYSSR_02_, BARCSOYSSR_06_, BARCSOYSSR_13_ and BARCSOYSSR_14_ abbreviated as BS02, BS06, BS13 and BS14 respectively. the same as below
由表1可知,目前國內外已標記定位的SMV抗病基因位點有22個,主要分布在大豆第2、6、13和14染色體上,其中第13染色體上基因位點最多,有10個,定位在第2染色體上的基因位點有8個,而在第6和14染色體上均有2個SMV抗病基因位點。除少數幾個基因位點兩側分子標記的物理距離較遠外,多數抗病基因位點兩側分子標記的物理距離均在1 Mb以內(表1)。
SMV抗病基因位點最早由Yu等[15]利用SSR(simple sequence repeats,SSR)標記SM176定位在大豆F連鎖群(第13染色體)上,遺傳距離為0.5 cM。利用RFLP(restriction fragment length polymorphism,RFLP)、RAPD(random amplified polymorphic DNA,RAPD)和SSR標記,Gore等[18]構建了包含和花生斑駁病毒抗病基因位點在內的6.8 cM精細遺傳圖譜。Shi等[25]構建J05與Essex雜交的F2群體,進一步將位點定位在SSR標記Sat_154和Satt510之間,遺傳距離分別為0.2和2.3 cM(表1)。參考大豆Willimas82全基因組序列(http://www.phytozome.net/soybean,Wm82.a2.v1),通過分析發(fā)現SMV抗病基因位點所在的物理區(qū)間起止位置為28 506 083—31 802 676 bp,物理距離3 296.6 kb(表1)。盡管是位點的一個等位基因[20],但其在大豆第13染色體上的物理位置并不確定[48]。利用Suweon97和Willimas82的F2群體,Ma等[48]研究發(fā)現Suweon97攜帶的抗病基因定位在標記BARCSOYSSR_13_1114和BARCSOYSSR_13_1115之間,與PI96983攜帶的位點屬于不同的區(qū)域。兩標記間的物理距離為97.5 kb(http://www.phytozome.net/soybean,Wm82.a2.v1)。
位點被定位在大豆連鎖群B2(第14染色體)上標記A519和M3Satt之間,遺傳距離分別為0.9和0.8 cM[19],而基于標記A519開發(fā)的3個SNP(single nucleotide polymorphism,SNP)標記A519-460、A519-956和A519-1165與緊密連鎖[24]。Shi等[25]發(fā)現SSR標記Sat_424與大豆J05攜帶的緊密連鎖,遺傳距離為1.5 cM。利用新開發(fā)的標記,Suh等[26]進一步將定位在分子標記S156l和BARCSOYSSR_14_1417之間,遺傳距離分別為0.3和0.5 cM,候選區(qū)域在第14染色體上的起止位置為46 852 283—47 059 763 bp(http://www.phytozome.net/ soybean,Wm82.a2.v1),物理距離207.5 kb(表1)。
Hayes等[49]利用BSA(bulked segregant analysis,BSA)法和F2群體將抗病基因定位在大豆D1b連鎖群(第2染色體)上,SSR標記Satt558和Satt542與之間的遺傳距離分別為7.8和4.7 cM。利用比較作圖策略,Hwang等[50]進一步將定位在EST(expressed sequence tag,EST)標記AI856415-g和AW471852A之間,遺傳距離分別為2.8和2.4 cM。基于大豆基因組鳥槍測序法,Saghai Maroof等[51]將鎖定在1.3 cM的精細作圖區(qū)域內。2016年,Ilut等[27]對區(qū)域進行了深度重測序,發(fā)現與兩側的標記Rat2和S6ac的遺傳距離均為0.2 cM(表1),起止位置為12 044 285—12 163 994 bp(http://www. phytozome.net/soybean,Wm82.a2.v1),物理區(qū)間119.7 kb(表1)。
美國大豆品種York攜帶的抗病基因早先被命名為[52],被推測是PI96983攜帶抗病基因位點的等位基因,2個品種對SMV株系G1–G7的抗性反應存在差異[52]。Klepadlo等[28]利用PI96983()×York()構建包含3 000個F2:3家系的群體檢測了2個抗病基因之間的等位性關系,結果發(fā)現和是2個緊密連鎖的基因位點,遺傳距離為2.2 cM,物理距離約為440 kb,最后作者將York攜帶的抗病基因重新命名為[28]。
科豐1號是中國科學院遺傳與發(fā)育生物學研究所從河北地方大豆品種系統選擇育成,對中國的18個SMV株系(SC1–SC13、SC16和SC18—SC21)和美國的7個株系(G1—G7)均具有抗性[53-55]。利用科豐1號×南農1138-2組配的F2、RIL和剩余雜合系RHL(residual heterozygous lines,RHL)群體,Adhimoolam等[44]、Fu等[29]、Yan等[41]、Wang等[33]、Zhao等[43]、李春燕等[36]、郭東全等[56]和Li等[40]先后將科豐1號對SMV株系SC5、SC7、SC8、SC10、SC13、SC18攜帶的抗病基因R、R、R、R、R、R定位在大豆的第2染色體上(表1和圖1)。此外,陽小鳳等[38]將大豆品系Q0923對SC6株系具有的抗病基因R和中豆35攜帶對SC17的抗病基因R也均定位在第2染色體上(表1和圖1)。
根據大豆Willimas82的參考基因組序列(http:// www.phytozome.net/soybean,Wm82.a2.v1),結合上述標記定位的結果分析可以發(fā)現,大豆第2染色體上的SMV抗病基因位點主要分布在11 188 722—13 104 514 bp(表1,圖1)。其中有6個SMV抗病基因位點的候選區(qū)段交叉重疊,集中分布在12 044 285—12 313 444 bp約269.2 kb的區(qū)間內,推測這6個基因位點可能緊密連鎖或具有等位關系。進一步對這一區(qū)間分析發(fā)現,該區(qū)間內有20個候選基因(—)(圖1,表2),但均不具有NBS-LRR(nucleotide binding site-leucine rich repeat,NBS-LRR)抗病基因保守結構域。
齊黃1號是山東省農業(yè)科學院從山東壽張地方品種系統選擇育成的另一個對SMV具有廣譜抗性的品種,對中國的18個SMV株系(SC1—SC8、SC11—SC14、SC16—SC18和SC20—SC22)和美國的7個株系(G1—G7)也均具有抗性[53-55]。利用F2、RIL和RHL群體,Zheng等[39]、白麗等[11]、Li等[30]、Ma等[31-32]、Li等[40]和Adhimoolam等[45]將齊黃1號及其衍生系齊黃22攜帶的抗SMV基因R、R、R、R、R和R均定位在大豆的第13染色體上(表1和圖2)。Yang等[37]將美國抗源PI96983攜帶對SMV株系SC3、SC6、SC17的抗病基因pm以及對SC7的抗病基因Rscps也均定位在第13染色體上相鄰的區(qū)域內(表1和圖2)。

表2 抗病基因位點Rsv4、RSC5、RSC6、RSC7、RSC8和RSC17在大豆第2染色體上可能的候選基因a
a數據來源于大豆的全基因組數據庫(http://www.phytozome.net/soybean,Wm82.a2.v1,2018年3月12日),加粗字體表示第2染色體上可能性較大的SMV抗病候選基因。下同
aThe data from the soybean reference genome sequence (http://www.phytozome.net/soybean, Wm82.a2.v1, March 12, 2018),the bold font indicates the putative candidate genes to SMV in soybean on chromosome 2. the same as below
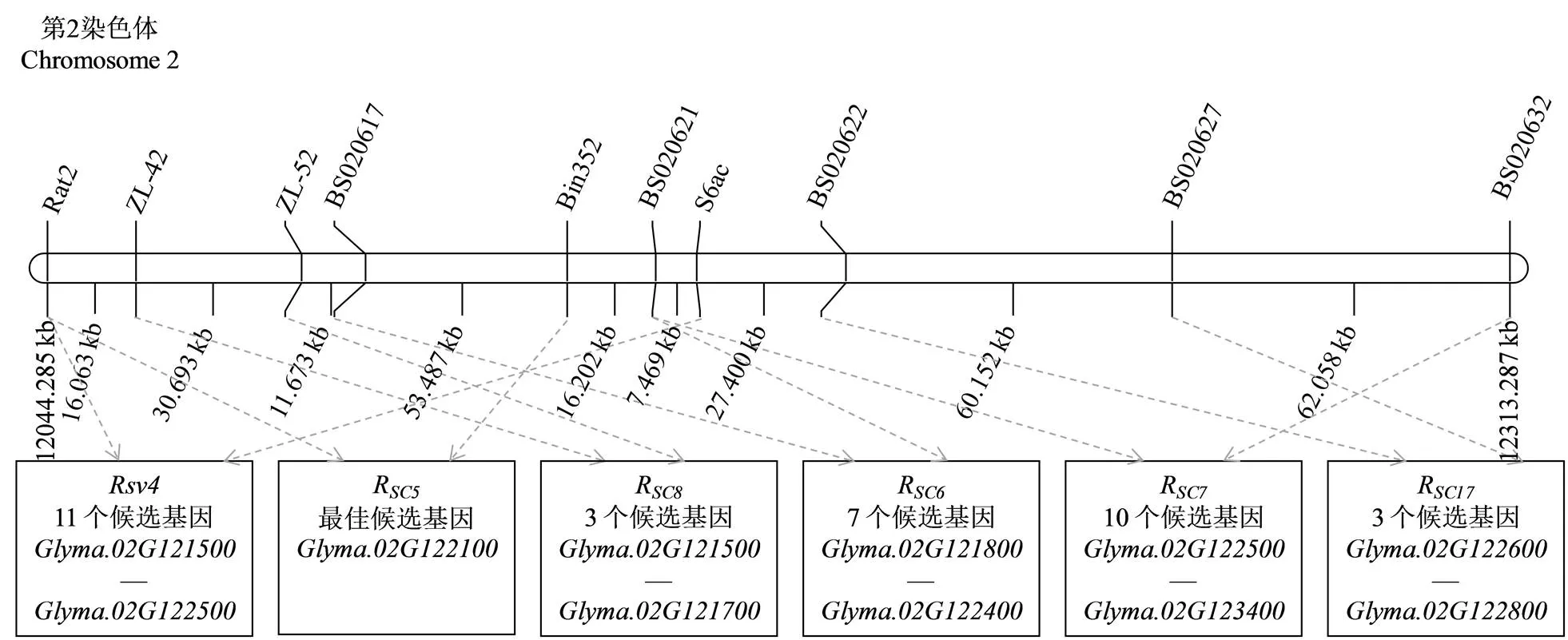
虛線箭頭部分表示單個SMV抗病基因的物理區(qū)間;最下面的框內是SMV抗病候選基因的個數及范圍。下同

圖2 大豆抗SMV基因在第13染色體上的遺傳、物理圖譜及抗病候選基因
基于大豆Willimas82的參考基因組序列(http:// www.phytozome.net/soybean,Wm82.a2.v1),結合標記定位結果分析發(fā)現,國內外定位于第13染色體上的SMV抗病基因位點的候選區(qū)段主要在27 656 895—31 951 960 bp成簇存在(圖2),物理距離為4.295 Mb,且這一區(qū)段內富集著許多具NBS-LRR結構域的候選基因(表3)。從圖2可以發(fā)現,在4.295 Mb的物理區(qū)段內,SMV抗病基因位點的候選區(qū)段均存在相互重疊的現象,相較于第2染色體上候選區(qū)段內的基因數,第13染色體上候選區(qū)段內的基因數量更多,推測該候選區(qū)段內的多數基因連鎖較為緊密,難以發(fā)生交換。
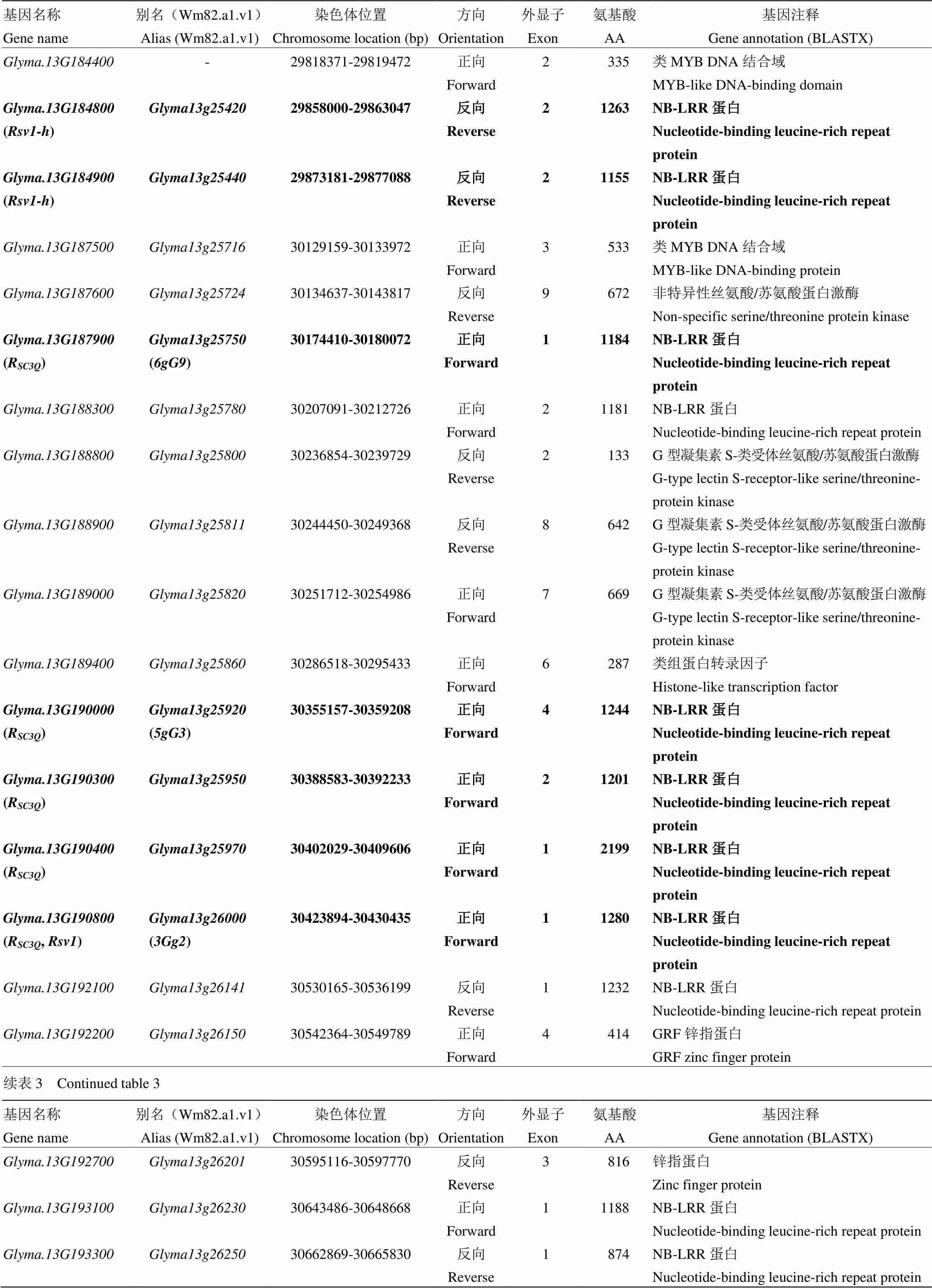
表3 SMV抗病基因位點Rsv1、Rsv5、RSC3Q、RSC11、RSC12、RSC14Q、RSC18B、RSC20、Rsc-pm和Rsc-ps在大豆第13染色體上部分可能的候選基因
RN-9是繼科豐1號和齊黃1號后發(fā)現的又一個廣譜抗源,對15個SMV株系均表現極端抗性,特別是對能感染所有鑒別寄主的SC15株系表現抗病[35,47]。利用RN-9×7605的RIL群體,Yang等[35]將RN-9攜帶的對SMV株系SC15具有的抗病基因R定位在大豆第6染色體上,該基因與SSR標記Sat_213和Satt286之間的遺傳距離分別為8.0和6.6 cM。2017年,Ren等[46]進一步將R鎖定在標記SSR_06_17和BS060835之間,遺傳距離分別為1.44和0.63 cM(表1),起止位置為15 635 537—15 730 967 bp(http://www. phytozome.net/soybean,Wm82.a2.v1),物理區(qū)間為95.2 kb(表1,圖3-a)。
此外,李凱等[47]將RN-9攜帶對SC18的抗性基因R定位在標記Satt286和Satt277之間,遺傳距離分別為6.12和4.69 cM,物理區(qū)間為997.6 kb。參考大豆Willimas82的基因組序列(http://www.phytozome. net/soybean,Wm82.a2.v1),分析發(fā)現抗病基因R和R在第6染色體上的候選區(qū)段沒有重疊的區(qū)域(圖3-a)。其中,在95.4 kb的R候選基因區(qū)段內存在5個候選基因(—),且均不具有NBS-LRR抗病基因保守結構域(表4,圖3-a),在R候選基因區(qū)段內,有73個候選基因存在(—)。
利用大白麻×南農1138-2的F2作圖群體,Wang等[34]將大白麻攜帶的抗病基因R精細定位在大豆第14染色體上,緊密連鎖的2個基因組SSR標記BARCSOYSSR_14_1413和BARCSOYSSR_14_1416與R的遺傳距離分別為0.17和0.27 cM(表1),物理距離63.4 kb(表1,圖3-b)。參考大豆Willimas82的基因組序列(http://www.phytozome.net/soybean,Wm82.a2.v1),結合R和定位結果分析可以發(fā)現,在大豆第14染色體46 852 283—47 135 871 bp共有17個可能的候選基因(表5),其中有5個候選基因具NBS-LRR保守結構域。從圖3-b還可以發(fā)現,2個SMV抗病基因位點R和的候選基因位置具有重疊的物理區(qū)段,推測R與可能是等位或緊密連鎖的。
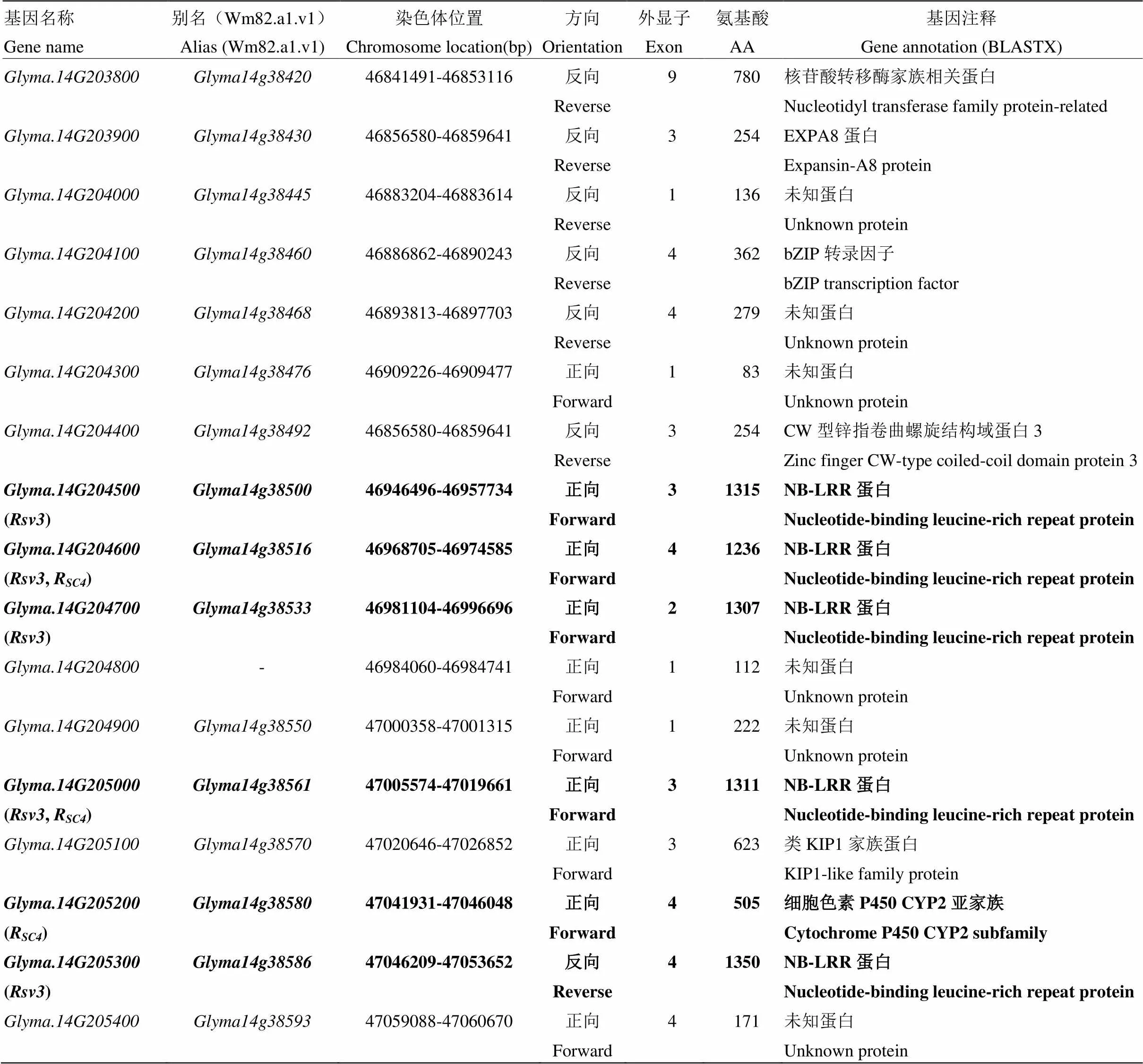
表5 抗病基因位點Rsv3和RSC4在大豆第14染色體上可能的候選基因
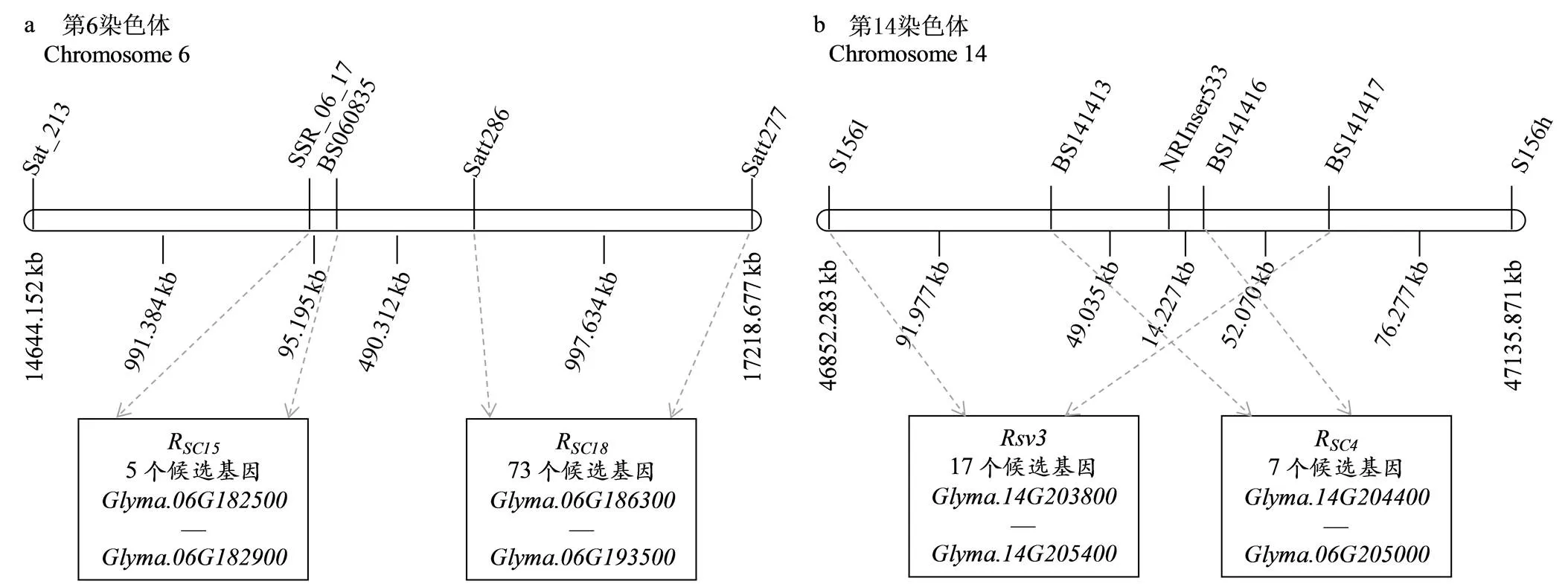
圖3 大豆抗SMV基因在第6和14染色體上的遺傳、物理圖譜及抗病候選基因
2 SMV抗病候選基因預測
隨著大豆對SMV抗病基因不斷地發(fā)現、標記和精細定位,大豆對SMV候選抗病基因保守結構域的生物信息學分析、qRT-PCR(quantitative real time polymerase chain reaction,qRT-PCR)表達分析及克隆測序分析等也在不斷地拓展和深入,極大地縮小了候選基因的篩選范圍。
在第2染色體上,Saghai Maroof等[51]對的候選基因序列(http://www.phytozome.net/soybean,Wm82.a1.v1)進行分析發(fā)現,這些基因均不具有NBS-LRR結構域(圖1,表2),推測屬于一種新類型的抗病基因,其可能是通過干擾病毒感染和細胞間運動,并延遲病毒在維管束間移動,從而達到抗病的目的。Ilut等[27]通過對SMV株系G1和G7具有不同抗性反應的6個野生大豆和13個栽培大豆位點所在的120 kb的區(qū)域進行深度測序分析發(fā)現,這一區(qū)域至少存在2個單倍型,其中最可能的一個單倍型包含4個候選基因:1個脅迫響應基因、2個RD類受體蛋白酶基因(和)和1個銅轉運蛋白基因()(表2)。從基因結構可以看出,這幾個候選基因均不具有經典的NBS-LRR型抗病基因結構,驗證了可能屬于一種新類型抗病基因的推測[27,50]。作者對該區(qū)域的單倍型系統發(fā)育分析發(fā)現位點是最新才從野生大豆進化到栽培大豆中的[27]。不過,Klepadlo等[28]通過對10個大豆品種的測序卻證實可能是的候選基因(表2)。
Adhimoolam等[44]將SNP標記及抗性鑒定表型數據進行關聯分析和qRT-PCR表達分析發(fā)現,科豐1號攜帶對SC5最有可能的抗病候選基因是()(表2)。
根據候選基因的結構分析,Yan等[41]推測具NBS-LRR結構域的基因,具有熱休克蛋白40(HSP40)結構的基因()和具有絲氨酸羧肽酶型(Serine carboxypeptidase-type)的基因可能是R的候選基因,不過基因和在最新的大豆參考基因組序列中已經被重新進行整合(http://www.phytozome.net/soybean,Wm82.a2.v1,2018年3月12日)。CHE等[57]通過GWAS(genome wide association study,GWAS)和qRT-PCR技術研究發(fā)現,與擬南芥轉錄調控因子、真核生物延伸因子和同源的3個大豆基因(、和)可能參與了大豆抗SC7株系的過程。
Wang等[33]運用qRT-PCR技術對精細定位的R抗病候選基因分析發(fā)現,其中5個候選基因的表達量在抗感品種間的響應模式或時間上有很大的差異,推測這些基因可能參與了大豆對SMV株系的抗性反應。Zhao等[43]進一步將R的候選基因確定為具MADS-box保守結構域的基因和(表2)。
通過對SC15的抗病品種RN-9和感病品種7605的5個候選基因(—)DNA和cDNA進行測序和qRT-PCR分析,Ren等[46]推測過氧化物膜蛋白基因()是R最有可能的候選基因[46]。根據測序和表達分析,Li等[40]研究發(fā)現、和可能是科豐1號對SMV株系SC18的抗病候選基因(表4)。
通過分析大豆抗病基因位點定位區(qū)域的NBS-LRR基因簇,Hayes等[58]從抗SMV品種PI96983中克隆到一個在結構上具有non-TIR(toll interleukin1 receptor,TIR)-NBS分子特征的抗病候選基因(),該基因全長3 390 bp,包含一個完整的編碼框,SHAO等[59]從抗病基因進化的角度證實了()是的一個候選基因。Ma等[48]對抗病基因的8個候選基因序列進行分析發(fā)現,和具有CC(coiled-coil,CC)-NBS-LRR抗病基因的保守結構,推測這兩個基因可能是的最佳候選基因(表3)。
在精細定位的基礎上,Zheng等[39]通過qRT-PCR分析發(fā)現,、()、()、()和()等5個基因可能是R的候選基因(表3)。Li等[42]進一步通過轉錄組和qRT-PCR研究表明齊黃1號攜帶對SC3抗病基因R可能的候選基因為()、()、()、()等4個具NBS-LRR抗病基因保守結構域的基因(表3),其中有3個候選基因與Zheng等[39]的研究結果相一致。
Adhimoolam等[45]綜合候選基因的測序結果和qRT-PCR表達分析發(fā)現,齊黃1號攜帶對SC20最有可能的抗病候選基因是和(表2)。
在大豆的第14染色體上,基于抗病基因R精細定位的結果,Wang等[34]參考Willimas82的參考基因組序列和qRT-PCR技術發(fā)現3個抗病候選基因()、()和()在抗感品種間不同時間點的表達量具有差異,初步推測這些基因可能參與了大豆對SC4株系的抗性反應(表5)。
對抗病基因所在的154 kb區(qū)段進行分析,Suh等[26]發(fā)現這個區(qū)段是一個富集多個抗病基因的基因簇,其中有5個抗病候選基因()、()、()、()和()編碼NB-LRR結構的抗病蛋白(表5),作者認為可能是NB-LRR家族中的一員[26]。值得注意的是,SMV抗病基因R和均在這個區(qū)段內,說明R和屬于同一個抗病基因簇。Suh等[26]推測的基因和與Wang等[34]通過qRT-PCR獲得的抗病候選基因是一樣的,不過2個基因位點是不是同一個基因仍需進一步的研究分析。
通過分析抗病品種L29接種SMV株系G7后1—3 h的表達量,Redekar等[60]發(fā)現,基因()是5個候選基因中表達量最大的一個基因。比較測序研究表明,在抗病基因型(型)和感病基因型(3型)間有150個SNP和6個Indel差異,而且這種差異主要集中在LRR編碼區(qū)。因此,作者推測最有可能是的候選基因[60](表5)。
3 SMV抗病相關基因篩選
大豆對SMV抗病基因的發(fā)掘除了利用精細定位圖位克隆外,還有通過同源克隆等方法獲得了一系列抗SMV相關基因的報道。
賀超英等[61]和Wang等[62]利用同源克隆的方法從科豐1號中獲得2個SMV抗性相關基因和,它們都具有NBS和LRR等抗病基因的結構特征,RT-PCR分析表明二者均受SMV株系Sa接種和水楊酸處理的影響,推測和可能參與了大豆抗SMV的過程[62-63]。此外,Wang等[64]研究發(fā)現大豆同源異型-亮氨酸拉鏈蛋白基因具有HD-Zip結構域,是植物特有的轉錄因子。接種SMV株系N3后,該基因在感病品種中的表達量上調,而在抗病品種中則下調,初步證實可能作為轉錄激活因子參與大豆對SMV的抗性(表6)。
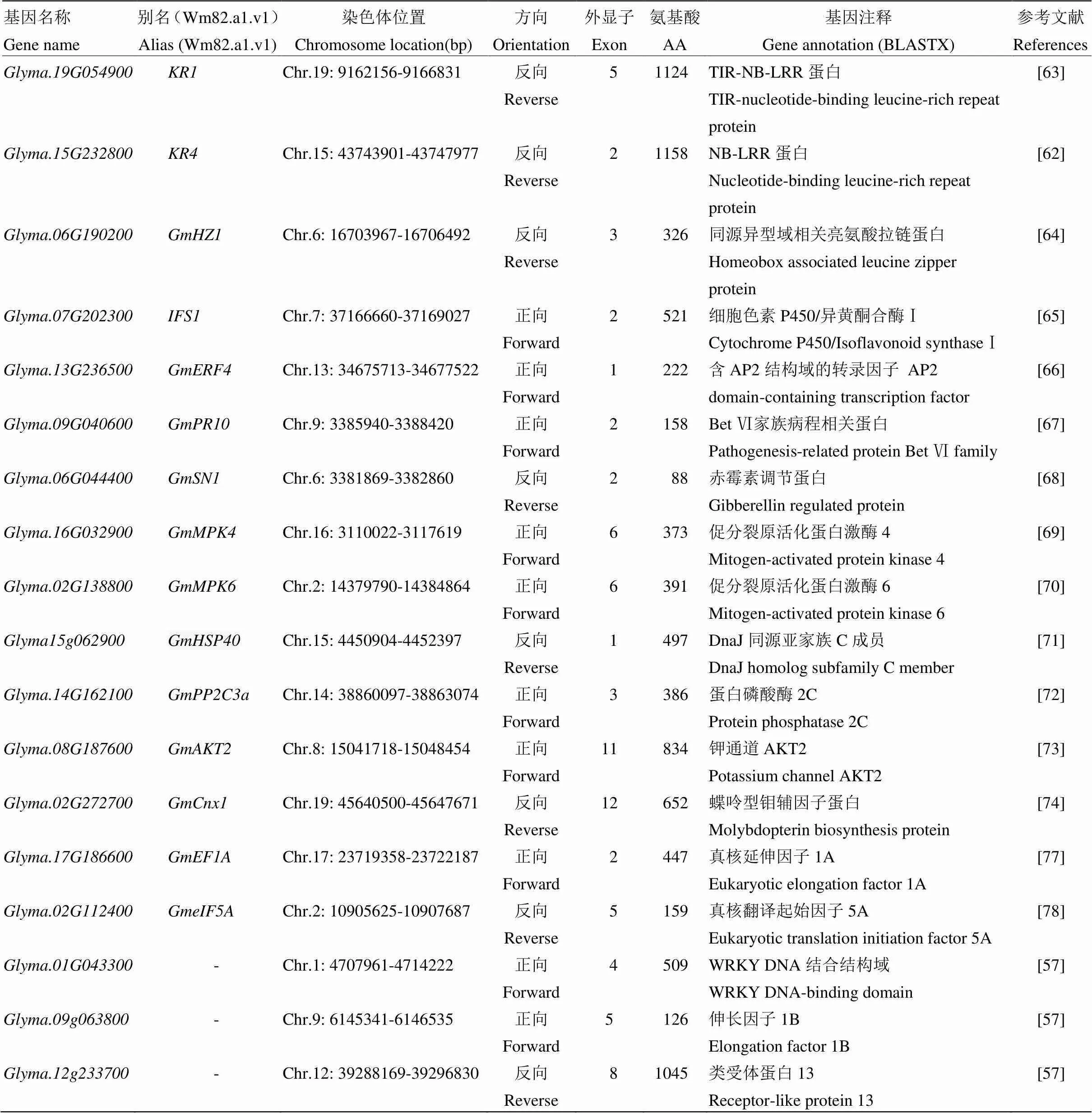
表6 參與大豆抗大豆花葉病毒可能的相關基因
關聯分析研究發(fā)現,大豆異黃酮合成酶基因與大豆對SC3的抗性顯著相關[65]。表達分析發(fā)現接種SC3后7 d,基因在抗病品種中的表達量顯著上調,而在感病品種中沒有變化,推測該基因可能參與了大豆抗SMV侵染的過程[65]。Zhang等[66]對大豆中一個耐脅迫基因研究發(fā)現,其能夠被SMV誘導表達,推測該基因可能是大豆對SMV抗性反應的一個相關因子。是大豆中的一個PR10蛋白基因,RT-PCR分析表明,大豆接種SMV后該基因在葉片中被強烈誘導并高效表達,推測其可能在病毒誘導下使大豆獲得系統抗性以抵抗外來病原菌的侵襲[67]。
通過轉錄組分析,He等[68]發(fā)現GAST(GA- stimulated transcript,GAST)基因亞家族成員在接種SMV后,在感病品種(感病型:)中的表達量下調,而在抗病品種(抗病型:)中沒變化。進一步測序和聚類比較分析表明,GAST家族成員之一基因與土豆中的抗病基因同源,可能參與了大豆抗SMV的過程[68]。
4 大豆抗SMV相關基因的功能鑒定
在利用生物信息學、qRT-PCR及克隆測序等一系列技術對SMV抗病候選基因進行預測和抗SMV相關基因進行篩選的基礎上,相關學者進一步通過病毒誘導的基因沉默VIGS(virus induced gene silencing,VIGS)、轉基因操作等技術對抗SMV相關基因的功能進行了鑒定和研究。
與空載對照相比,Liu等[69-70]研究發(fā)現沉默大豆蛋白激酶()和()的植株均對SMV表現更好的抗性;雙分子熒光互補(bimolecular fluorescence complementation,BiFC)試驗表明,和與相互作用,同時和在的上游起作用;與相互作用,在的上游起作用[70](表6)。
熱激蛋白在植物防御生物及非生物脅迫中具有至關重要的作用,Liu等[71]研究發(fā)現沉默具有DnaJ結構域的大豆熱激蛋白基因Seo等[72]通過大豆與SMV互作研究表明大豆2C型蛋白磷酸酶基因()是介導的對SMV抗性中的一個調控因子。
Zhou等[73]研究表明在多施鉀肥的情況下能夠提高大豆對SMV的抗性,過表達大豆鉀離子通道基因發(fā)現,轉基因陽性植株對SMV的抗性明顯增強。其可能的原因是大豆的過量表達導致其韌皮部發(fā)生改變,進而不利于外部病毒的侵染、移動和復制[73]。轉基因研究發(fā)現SMV對過表達大豆硝酸還原酶及黃嘌呤脫氫酶輔助因子轉基因植株的侵染率顯著低于對照品種,轉基因植株顯著增強大豆對SMV株系的抗性[74]。
、和在抗感品種間表達量有顯著差異且具有NB-LRR抗病基因保守結構域,它們被作為抗病基因R的3個候選基因。Li等[75]研究發(fā)現,沉默3個基因后的抗病品種顯著感病,3個基因在抗感品種間的序列比對發(fā)現它們在抗感品種間均有氨基酸上的差異,推測這3個基因與大豆對SMV的抗病均有關系[75]。
Tran等[76]對的候選基因進行研究發(fā)現,在缺失基因型的大豆品種中過量表達基因后SMV的侵染被明顯抑制;而對攜帶基因型的大豆品種L29進行瞬時沉默后發(fā)現,原來對L29不致病的病毒株系表現較多的SMV積累[76]。將SMV抗病候選基因過表達轉化到擬南芥和大豆中,He等[68]發(fā)現與對照相比,過表達轉基因擬南芥植株能夠增強對蘿卜花葉病毒的抗性,轉基因大豆植株能夠增強對SMV的抗性。
通過酵母雙雜交和VIGS研究發(fā)現,大豆轉錄延伸因子家族()蛋白基因能夠增進SMV的復制,調控病原侵染;沉默該基因可降低SMV的積累,提高大豆對SMV的抗性[77]。敲除大豆真核翻譯起始因子(),Chen等[78]研究發(fā)現植株減少了系統過敏性壞死反應,病毒積累增強,說明在介導的對SMV株系G7的系統過敏性壞死反應信號通路中起重要作用。
5 SMV抗病候選基因的調控網絡
對介導的極端抗性(extreme resistance,ER)候選基因、和進行沉默,Zhang等[79]研究發(fā)現沉默后的植株表現感病,由介導的ER喪失。同時,通過VIGS研究發(fā)現基因、、、、和2個WRKY轉錄因子(和)都參與了介導的抗性[79]。基于上述研究結果并結合以前的研究,Zhang等[79]形成了一個由介導的ER調控網絡:其中non-TIR-NBS-LRR基因及熱激蛋白基因和伴侶蛋白基因和位于調控網絡頂端,和脂肪酶位于的下游但在轉錄因子的上游,和最有可能在與和到轉錄因子的通路中是平行起作用的,最終表現對SMV的極端抗性(圖4-a)。
Seo等[72]構建了一個介導對SMV的ER調控模型:在大豆最初感染的細胞中,SMV復制并產生效應因子。通過R蛋白識別,從而導致激素脫落酸(abscisic acid,ABA)在大豆體內的積累,進而激活ABA信號通路。ABA信號通路與介導的極端抗性相關聯,通過上調信號傳導正調控因子,進而刺激胼胝質沉積。最終,在胞間連絲處的胼胝質沉積抑制病毒從最初侵染的細胞向健康細胞的移動并限制病毒的積累[72,81](圖4-b)。
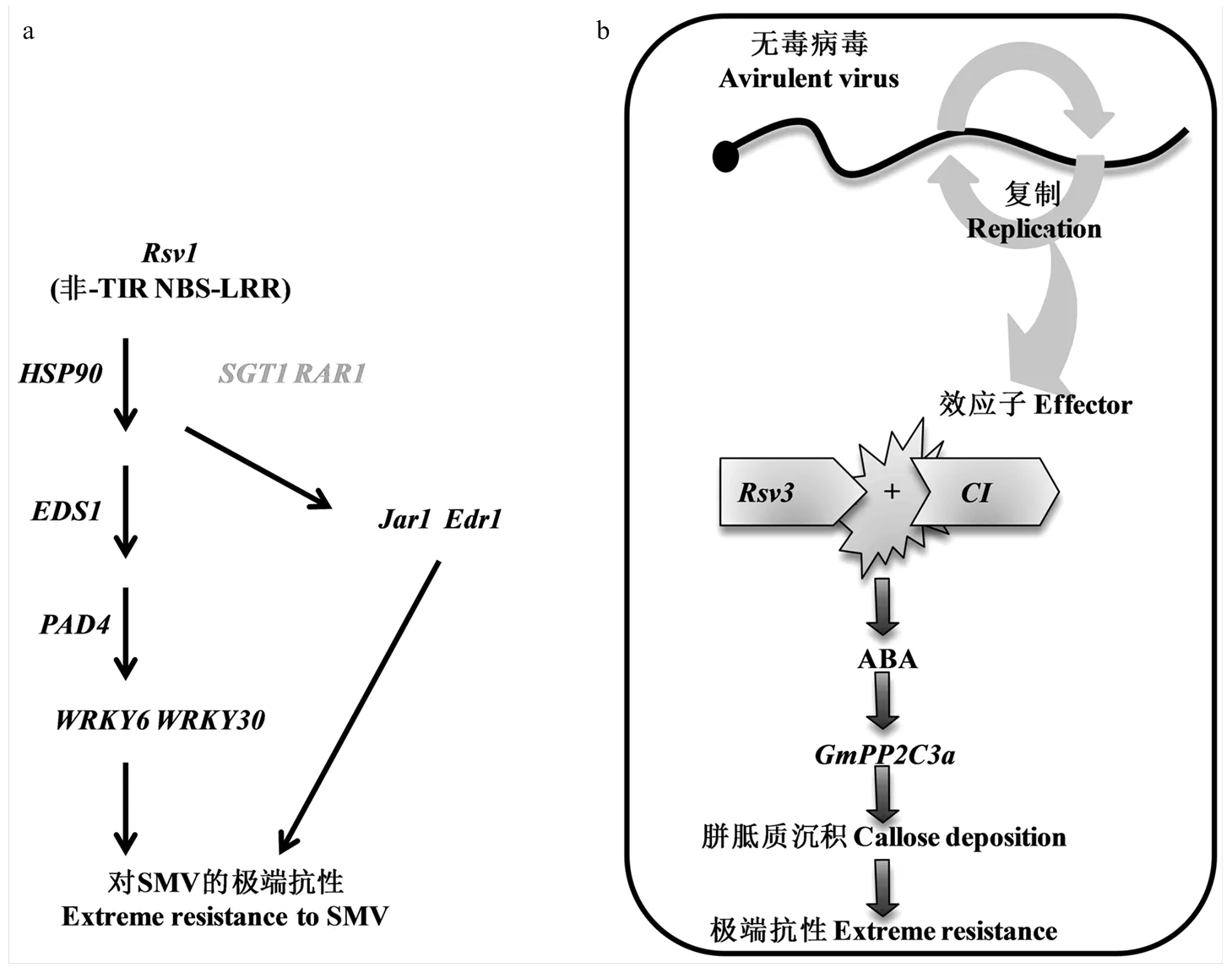
灰色字體的基因由Fu等[80]鑒定 Genes indicated in gray were identified by Fu et al.[80]
通過對轉基因擬南芥進行轉錄組表達分析,He等[68]研究發(fā)現,該基因主要參與植物抗病的信號傳導和免疫反應,而且在轉基因大豆中發(fā)現另外一個與SMV抗性相關的基因也能夠上調表達,表明這兩個基因協同提高大豆對SMV的抗性。
6 展望
近年來,隨著對大豆抗SMV研究的不斷深入,在抗病基因的分子標記及候選基因的鑒定方面取得很大的進展。在大豆第2、6、13和14染色體上研究發(fā)現的SSR、SNP、Indel等分子標記與SMV抗病基因之間均緊密連鎖或共分離,為分子標記輔助選擇及分子設計育種奠定了良好的基礎。Ma等[31]研究表明:單獨使用標記Satt334或Sct_033對R在齊黃22×南農1138-2的3個世代以及對R在齊黃1號×南農1138的2個世代分子標記輔助選擇的準確率均在85%以上,同時使用2個標記的準確率達95%以上。Saghai Maroof等[82]、Shi等[83]和Wang等[84]利用與SMV抗病基因緊密連鎖的分子標記均成功地將分布在3條不同染色體上抗病基因精準地聚合在同一個大豆品系中,為廣譜高抗SMV育種提供了優(yōu)異的抗源材料。
齊黃1號對中國的18個SMV株系和美國的7個株系均具有抗性[53-55],其所攜帶的單顯性抗病基因均定位在第13染色體上[11,32,39-40,45]。鄭桂杰[85]收集整理骨干親本齊黃1號的衍生品種(系)發(fā)現,齊黃1號在全國11個省市衍生出92個高產優(yōu)質品種和優(yōu)異品系,其中黃淮海地區(qū)衍生出87個品種(系)[86]。通過對90個衍生品種(系)接種SC3和SC7發(fā)現,生產上大面積推廣的品種徐豆1號、躍進5號、中黃19、豫豆19和齊黃22等對兩個株系均表現抗病[85]。此外,對SMV具有廣譜抗性的科豐1號及大白麻的衍生品種在生產上的推廣也對控制SMV的危害起到了關鍵的作用[87]。
然而,在日本和韓國相繼發(fā)現了使具有單個抗病基因的品種失去抗性的SMV毒株[88-90],特別是在加拿大發(fā)現的SMV分離物L-RB能夠攻克對美國所有7個株系都表現抗病的基因[91]。最近,WANG等[92]研究發(fā)現大豆花葉病毒P3蛋白單個氨基酸的改變就能使無毒株系攻克的抗性而成為致病株系。此外,中國報道的強致病株系及一些新發(fā)現的株系和重組株系也預示著SMV與大豆寄主之間一直處在動態(tài)平衡中[53-54,93-95]。因此,持續(xù)尋找新的抗病材料并發(fā)掘新的抗病基因是抗SMV育種的一個長期目標。
中國是大豆的起源地,不僅具有大量的栽培大豆類型,而且野生資源豐富多樣,目前發(fā)現的野生大豆占世界90%以上[96]。Wen等[97]通過野生大豆與地方品種的遺傳多樣性比較發(fā)現,野生大豆中僅有51%的等位基因在地方大豆品種中保留下來,大豆的重測序研究結果也印證了這一發(fā)現[98]。可見,經過多次馴化和人工選擇,野生大豆中大量優(yōu)異的遺傳變異沒有保留在栽培大豆中[97-98]。因此,利用野生大豆資源,發(fā)掘新的SMV抗病基因,對減少SMV危害,提高抗SMV育種成效具有關鍵性的作用。
大豆抗SMV的研究為確定SMV抗病基因和闡明大豆抗SMV調控網絡奠定了扎實的基礎[27-28,42-46]。以往由于大豆遺傳轉化及再生比較困難,對SMV抗病基因的功能驗證研究較少[68,73,76]。目前,隨著現代生物技術的發(fā)展,利用不斷完善的大豆轉基因技術、候選基因沉默技術及CRISPR/Cas9基因編輯技術等,對標記定位的SMV抗病候選基因進行功能驗證,對盡快確定抗SMV過程中的關鍵基因將發(fā)揮重要作用。此外,從SMV侵染大豆的角度出發(fā),研究者篩選出一些能夠協助SMV侵染大豆的基因,通過沉默這些基因發(fā)現,大豆增強了對SMV的抗性[77-78]。因此,后期如果能夠通過CRISPR/Cas9基因編輯技術對研究發(fā)現的這些基因或一些新發(fā)現的負調控關鍵因子進行改造,將是培育高效廣譜抗SMV大豆品種的一個新途徑。
解決抗SMV育種最根本的方法是在明確SMV抗病基因功能的基礎上,闡明大豆抗SMV過程中調控的信號網絡。SMV抗病基因的標記定位及分析對理解大豆抗SMV的調控信號網絡以及發(fā)掘新的大豆抗病基因具有重要價值。Zhang等[79]和Seo等[72]對SMV抗病基因和在抗病過程中的調控網絡進行了構建和模擬,為進一步驗證提供了重要線索。因此,探索大豆抗SMV的調控網絡及對大豆的抗病性進行改良也是一個很重要的研究方向。
[1] 李凱, 劉志濤, 李海朝, 張鍇, 王成坤, 任銳, 盧為國, 智海劍. 國家大豆區(qū)域試驗品種對SMV和SCN的抗性分析. 大豆科學, 2013, 32(5): 670-675.
LI K, LIU Z T, Li H C, ZHANG K, WANG C K, REN R, LU W G, ZHI H J. Resistance to soybean mosaic virus and soybean cyst nematode of soybean cultivars from China national soybean uniform trials., 2013, 32(5): 670-675. (in Chinese)
[2] 王大剛, 智海劍, 張磊. 大豆抗大豆花葉病毒研究進展. 中國油料作物學報, 2013, 35(3): 341-348.
WANG D G, ZHI H J, ZHANG L. Review on resistance to soybean mosaic virus in soybean., 2013, 35(3): 341-348. (in Chinese)
[3] 李凱, 智海劍. 大豆對大豆花葉病毒病抗性的研究進展. 大豆科學, 2016, 35(4): 525-530.
LI K, ZHI H J. Advances in resistance to soybean mosaic virus disease in soybean., 2016, 35(4): 525-530. (in Chinese)
[4] 智海劍, 蓋鈞鎰. 大豆對SMV數量抗性的表現形式與種質鑒定. 中國農業(yè)科學, 2004, 37(10): 1422-1427.
ZHI H J, GAI J Y. Performances and germplasm evaluation of quantitative resistance to soybean mosaic virus in soybeans., 2004, 37(10): 1422-1427. (in Chinese)
[5] 智海劍, 蓋鈞鎰, 何小紅. 大豆對SMV數量(程度)抗性的綜合分級方法研究. 大豆科學, 2005, 24(2): 5-11.
ZHI H J, GAI J Y, HE X H. Study on methods of classification of quantitative resistance to soybean mosaic virus in soybean., 2005, 24(2): 5-11. (in Chinese)
[6] 郭丹丹, 陳海峰, 楊中路, 單志慧, 朱曉玲, 陳水蓮, 周新安, 周蓉. 大豆對SMV SC-3株系的抗性遺傳和QTL分析. 大豆科學, 2012, 31(4): 511-516.
GUO D D, CHEN H F, YANG Z L, SHAN Z H, ZHU X L, CHEN S L, ZHOU X A, ZHOU R. Analysis of resistance inheritance to soybean mosaic virus (SMV) strain SC-3 and mapping related QTL in soybean., 2012, 31(4): 511-516. (in Chinese)
[7] KIIHL R A S, HARTWIG E E. Inheritance of reaction to a soybean mosaic virus in soybeans., 1979, 19(3): 372-375.
[8] LIM S M. Resistance to soybean mosaic virus in soybeans., 1985, 75(2): 199-201.
[9] 陳怡, 杜維廣, 欒曉燕, 張桂茹, 黃承運, 滿為群, 谷秀芝, 王彬如. 大豆對兩個大豆花葉病毒株系的抗性遺傳研究. 黑龍江農業(yè)科學, 1991(5): 21-24.
CHEN Y, DU W G, LUAN X Y, ZHANG G R, HUANG C Y, MAN W Q, GU X Z, WANG B R. Inheritance of resistance to two soybean mosaic virus strains in soybean., 1991(5): 21-24. (in Chinese)
[10] 李海朝, 智海劍, 白麗, 楊華, 馬瑩, 劉寧, 王大剛. 大豆對SMV株系SC-11的抗性遺傳及抗性基因的等位性研究. 大豆科學, 2006, 25(4): 365-368.
LI H C, ZHI H J, BAI L, YANG H, MA Y, LIU N, WANG D G. Studies on inheritance and allelism of resistance genes to SMV strain SC-11 in soybean., 2006, 25(4): 365-368. (in Chinese)
[11] 白麗, 李海朝, 馬瑩, 王大剛, 劉寧, 智海劍. 大豆對大豆花葉病毒SC11株系抗性的遺傳及基因定位. 大豆科學, 2009, 28(1): 1-6.
BAI L, LI H C, MA Y, WANG D G, LIU N, ZHI H J. Inheritance and gene mapping of resistance to soybean mosaic virus strain SC11 in soybean., 2009, 28(1): 1-6. (in Chinese)
[12] 王大剛, 馬瑩, 劉寧, 鄭桂杰, 楊中路, 楊永慶, 智海劍. 大豆花葉病毒(SMV)株系SC4和SC8的抗性遺傳分析. 作物學報, 2012, 38(2): 202-209.
WANG D G, MA Y, LIU N, ZHENG G J, YANG Z L, YANG Y Q, ZHI H J. Inheritance of resistances to soybean mosaic virus strains SC4 and SC8 in soybean., 2012, 38(2): 202-209. (in Chinese)
[13] BUZZELL R I, TU J C. Inheritance of soybean resistance to soybean mosaic virus., 1984, 75(1): 82.
[14] CHEN P Y, BUSS G R, TOLIN S A. Resistance to soybean mosaic virus conferred by two independent dominant genes in PI486355., 1993, 84(1): 25-28.
[15] Yu Y G, SAGHAI MAROOF M A, BUSS G R, MAUGHAN P J, TOLIN S A. RFLP and microsatellite mapping of a gene for soybean mosaic virus resistance., 1994, 84(1): 60-64.
[16] GUNDUZ I, BUSS G R, MA G, CHEN P Y, TOLIN S A. Genetic analysis of resistance to soybean mosaic virus in OX670 and Harosoy soybean., 2001, 41(6): 1785-1791.
[17] GUNDUZ I, BUSS G R, CHEN P Y, TOLIN S A. Genetic and phenotypic analysis of soybean mosaic virus resistance in PI88788 soybean., 2004, 94(7): 687-692.
[18] GORE M A, HAYES A J, JEONG S C, YUE Y G, BUSS G R, SAGHAI MAROOF M A. Mapping tightly linked genes controllinginfection at theandregion in soybean., 2002, 45(3): 592-599.
[19] JEONG S C, KRISTIPATI S, HAYES A J, MAUGHAN P J, NOFFSINGER S L, GUNDUZ I, BUSS G R, SAGHAI MAROOF M A. Genetic and sequence analysis of markers tightly linked to the soybean mosaic virus resistance gene,., 2002, 42(1): 265-270.
[20] CHEN P Y, BUSS G R, TOLIN S A, GUNDUZ I, CICEK M. A valuable gene in Suweon 97 soybean for resistance to soybean mosaic virus., 2002, 42(2): 333-337.
[21] WANG Y, NELSON R L, HU Y. Genetic analysis of resistance to soybean mosaic virus in four soybean cultivars from China., 1998, 38(4): 922-925.
[22] CHEN P Y, MA G, BUSS G R, GUNDUZ I, ROANE C W, TOLIN S A. Inheritance and allelism tests of Raiden soybean for resistance to soybean mosaic virus., 2001, 92(1): 51-55.
[23] GUNDUZ I, BUSS G R, MA G, CHEN P Y, TOLIN S A. Genetic and phenotypic analysis of soybean mosaic virus resistance in PI88788 soybean.,2004, 94(7): 687-692.
[24] JEONG S C, SAGHAI MAROOGF M A. Detection and genotyping of SNPs tightly linked to two disease resistance loci,and, of soybean., 2004, 123(4): 305-310.
[25] SHI A N, CHEN P Y, LI D X, ZHENG C M, HOU A F, ZHANG B. Genetic confirmation of two independent genes for resistance to soybean mosaic virus in J05 soybean using SSR markers., 2008, 99(6): 598-603.
[26] SUH S J, BOWMAN B C, JEONG N, YANG K, KASTL C, TOLIN S A, SAGHAI MAROOF M A, JEONG S C. Thelocus conferring resistance to soybean mosaic virus is associated with a cluster of coiled-coil nucleotide-binding leucine-rich repeat genes., 2011, 4(1): 55-64.
[27] ILUT D C, LIPKA A E, JEONG N, BAE D N, KIM D H, KIM J H, REDEKAR N, YANG K, PARK W, KANG S T, KIM N, MOON J K, SAGHAI MAROOF M A, GORE M A, JEONG S C. Identification of haplotypes at thegenomic region in soybean associated with durable resistance to soybean mosaic virus., 2016, 129(3): 453-468.
[28] KLEPADLO M, CHEN P Y, SHI A N, MASON R E, KORTH K L, SRIVASTAVA V S, WU C J. Two tightly linked genes for soybean mosaic virus resistance in soybean., 2017, 57(4): 1-10.
[29] FU S X, ZHAN Y, ZHI H J, GAI Y J, YU D Y. Mapping of SMV resistance geneby SSR markers in soybean., 2006, 128(1/3): 63-69.
[30] LI H C, ZHI H J, GAI J Y, GUO D Q, WANG Y W, LI K, LI B, YANG H. Inheritance and gene mapping of resistance to soybean mosaic virus strain SC14 in soybean., 2006, 48(12): 1466-1472.
[31] MA Y, LI H C, WANG D G, LIU N, ZHI H J. Molecular mapping and marker assisted selection of soybean mosaic virus resistance geneRLegume Genomics & Genetics, 2010, 1(8): 41-46.
[32] MA Y, WANG D G, LI H C, ZHENG G J, YANG Y Q, LI H W, ZHI H J. Fine mapping of theRlocus for resistance to soybean mosaic virus in soybean., 2011, 181(1): 127-135.
[33] WANG D G, MA Y, YANG Y Q, LIU N, LI C Y, SONG Y P, ZHI H J. Fine mapping and analyses ofRresistance candidate genes to soybean mosaic virus in soybean.&, 2011, 122(3): 555-565.
[34] WANG D G, MA Y, LIU N, YANG Z L, ZHENG G J, ZHI H J. Fine mapping and identification of the soybeanRresistance candidate gene to soybean mosaic virus., 2011, 130(6): 653-659.
[35] YANG Q H, GAI J Y. Identification, inheritance and gene mapping of resistance to a virulent soybean mosaic virus strain SC15 in soybean., 2011, 130(2): 128-132.
[36] 李春燕, 楊永慶, 王大剛, 李華偉, 鄭桂杰, 王濤, 智海劍. 大豆對SMV株系SC10的抗性遺傳及抗病基因的定位研究. 中國農業(yè)科學, 2012, 45(21): 4335-4342.
LI C Y, YANG Y Q, WANG D G, LI H W, ZHENG G J, WANG T, ZHI H J. Studies on mapping and inheritance of resistance genes to SMV strain SC10 in soybean., 2012, 45(21): 4335-4342. (in Chinese)
[37] YANG Y Q, ZHENG G J, HAN L, WANG D G, YANG X F, YUAN Y, HUANG S H, ZHI H J. Genetic analysis and mapping of genes for resistance to multiple strains of soybean mosaic virus in a single resistant soybean accession PI96983.&, 2013, 126(7): 1783-1791.
[38] 陽小鳳, 楊永慶, 鄭桂杰, 智海劍, 李曉紅. 大豆對大豆花葉病毒株系SC6和SC17抗病基因的精細定位. 作物學報, 2013, 39(2): 216-221.
YANG X F, YANG Y Q, ZHENG G J, ZHI H J, LI X H. Fine mapping of resistance genes to SMV strains SC6 and SC17 in soybean., 2013, 39(2): 216-221. (in Chinese)
[39] ZHENG G J, YANG Y Q, MA Y, YANG X F, CHEN S Y, REN R, WANG D G, YANG Z L, ZHI H J. Fine mapping and candidate gene analysis of resistance geneRto soybean mosaic virus in Qihuang 1., 2014, 13(12): 2608-2615.
[40] LI K, REN R, ADHIMOOLAM K, GAO L, YUAN Y, LIU Z T, ZHONG Y K, ZHI H J. Genetic analysis and identification of two soybean mosaic virus resistance genes in soybean [(L.) Merr]., 2015, 134(6): 684-695.
[41] YAN H L, WANG H, CHENG H, HU Z B, CHU S S, ZHANG G Z, YU D Y. Detection and fine-mapping of SC7 resistance genes via linkage and association analysis in soybean.2015, 57(8): 722-729.
[42] LI C, ADHIMOOLAM K, YUAN Y, YIN J L, REN R, YANG Y Q, ZHI H J. Identification of candidate genes for resistance to soybean mosaic virus strain SC3by using fine mapping and transcriptome analyses., 2017, 68(2): 156-166.
[43] ZHAO L, WANG D G, ZHANG H Y, SHEN Y C, YANG Y Q, LI K, WANG L Q, YANG Y H, ZHI H J. Fine mapping of theRlocus and expression analysis of candidate SMV resistance genes in soybean., 2016, 135(6): 701-706.
[44] ADHIMOOLAM K, LI K, JIANG H, REN R, LI G, ZHI H J, CHEN S Y, GAI J Y. Inheritance, fine-mapping, and candidate gene analyses of resistance to soybean mosaic virus strain SC5in soybean., 2017, 292(4): 811-822.
[45] ADHIMOOLAM K, LI K, LI C, YIN J L, LI N, YANG Y H, SONG Y P, REN R, ZHI H J, GAI J Y. Fine-mapping and identifying candidate genes conferring resistance to soybean mosaic virus strain SC20 in soybean., 2018, 131(2): 461-476.
[46] REN R, LIU S C, ADHIMOOLAM K, WANG T, NIU H P, YIN J L, YANG Y H, WANG L Q, YANG Q H, ZHI H J, LI K. Fine-mapping and identification of a novel locusRscunderlying soybean resistance to soybean mosaic virus., 2017, 130(11): 2395-2410.
[47] 李凱, 任銳, 王濤, 高樂, 落金艷, 劉士超, 智海劍, 蓋鈞鎰. 大豆對大豆花葉病毒SC18株系的抗性遺傳和基因定位. 大豆科學, 2017, 36(2): 187-192.
LI K, REN R, WANG T, GAO L, LUO J Y, LIU S C, ZHI H J, GAI J Y. Genetic analysis and mapping of soybean mosaic virus resistance genes to SC18 in soybean., 2017, 36(2): 187-192. (in Chinese)
[48] MA F F, WU X Y, CHEN Y X, LIU Y N, SHAO Z Q, WU P, WU M, LIU C C, WU W P, YANG J Y, LI D X, CHEN J Q, WANG B. Fine mapping of thegene in the soybean cultivar Suweon 97 that confers resistance to two Chinese strains of the soybean mosaic virus., 2016, 129(11): 2227-2236.
[49] HAYES A J, MA G, BUSS G R, SAGHAI MAROOF A S. Molecular marker mapping of, a gene conferring resistance to all known strains of soybean mosaic virus., 2000, 40(5): 1434-1437.
[50] HWANG T Y, MOON J K, YU S, YANG K, MOHANKUMAR S, YU Y H, LEE Y H, KIM S H, KIM H M, SAGHAI MAROOF M A, JEONG S C. Application of comparative genomics in developing molecular markers tightly linked to the virus resistance gene, 2006, 49(4): 380-388.
[51] SAGHAI MAROOF M A, TUCKER D M, SKONECZKA J A, BOWMEN B C, TRIPATHY S, TOLIN S A. Fine mapping and candidate gene discovery of the soybean mosaic virus resistance gene,., 2010, 3(1): 14-22.
[52] CHEN P Y, BUSS G R, ROANE C W, TOLIN S A. Allelism among genes for resistance to soybean mosaic virus in strain-differential soybean cultivars., 1991, 31(2): 305-309.
[53] LI K, YANG Q H, ZHI H J, GAI J Y. Identification and distribution of soybean mosaic virus strains in Southern China., 2010, 94(3): 351-357.
[54] 王大剛, 田震, 李凱, 李華偉, 黃志平, 胡國玉, 張磊, 智海劍. 魯豫皖大豆產區(qū)大豆花葉病毒株系的鑒定及動態(tài)變化分析. 大豆科學, 2013, 32(6): 806-809.
WANG D G, TIAN Z, LI K, LI H W, HUANG Z P, HU G Y, ZHANG L, ZHI H J. Identification and variation analysis of soybean mosaic virus strains in Shandong, Henan and Anhui provinces of China., 2013, 32(6): 806-809. (in Chinese)
[55] 蔡春梅, 姜驍, 趙春梅, 馬建新. 中國大豆花葉病毒SC7外殼蛋白基因的序列測定及其與美國株系的比較. 病毒學報, 2014, 30(5): 489-494.
CAI C M, JIANG X, ZHAO C M, MA J X. Sequence analysis of the coat protein gene of Chinese soybean mosaic virus strain SC7and comparison with those of SMV strains from the USA., 2014, 30(5): 489-494. (in Chinese)
[56] 郭東全, 王延偉, 智海劍, 蓋鈞鎰, 李海朝, 李凱. 大豆對SMV SC-13株系群的抗性遺傳及基因定位的研究. 大豆科學, 2007, 26(1): 21-24.
GUO D Q, WANG Y W, ZHI H J, GAI J Y, LI H C, LI K. Inheritance and gene mapping of resistance to SMV strain group SC-13in soybean., 2007, 26(1): 21-24. (in Chinese)
[57] CHE Z J, LIU H L, YI F L, CHENG H, YANG Y M, WANG L, DU J Y, ZHANG P P, WANG J, YU D Y. Genome-wide association study reveals novel loci for SC7 resistance in a soybean mutant panel., 2017, 8: 1-12.
[58] HAYES A J, JEONG S C, GORE M A, YU Y G, BUSS G R, TOLIN S A, SAGHAI MAROOF M A. Recombination within a nucleotide- binding-site/leucine-rich-repeat gene cluster produces new variants conditioning resistance to soybean mosaic virus in soybeans.,2004, 166(1): 493-503.
[59] SHAO Z Q, ZHANG Y M, HANG Y Y, XUE J Y, ZHOU G C, WU P, WU X Y, WU X Z, WANG Q, WANG B, CHEN J Q. Long-term evolution of nucleotide-binding site-leucine-rich repeat genes: understanding gained from and beyond the legume family., 2014, 166(1): 217-234.
[60] REDEKAR N R, CLEBINGER E M, LASKAR M A, BIYASHEV R M, ASHFIELD T, JENSEN R V, JEONG S C, TOLIN S A, SAGHAI MAROOF M A. Candidate gene sequence analyses toward identifying-type resistance to soybean mosaic virus., 2016, 9(2): 1-12.
[61] 賀超英, 張志永, 陳受宜. 大豆中NBS類抗性基因同源序列的分離與鑒定. 科學通報, 2001, 46(12): 1017-1021.
HE C Y, ZHANG Z Y, CHEN S Y. Isolation and identification of NBS type resistance gene analogs in soybean., 2001, 46(12): 1017-1021. (in Chinese)
[62] WANG B J, WANG Y J, WANG Q, LUO G Z, ZHANG Z G, HE C Y, HE S J, ZHANG J S, GAI J Y, CHEN S Y. Characterization of an NBS-LRR resistance gene homologue from soybean., 2004, 161(7): 815-822.
[63] HE C Y, TIAN A G, ZHANG J S, ZHANG Z Y, GAI J Y, CHEN S Y. Isolation and characterization of a full-length resistance gene homolog from soybean., 2003, 106(5): 786-793.
[64] WANG Y J, LI Y D, TIAN A G, WANG H WM ZHANG J S, CHR S Y. Cloning and characterization of an HDZip I genefrom soybean., 2005, 221(6): 831-843.
[65] CHENG H, YANG H, ZHANG D, GAI J Y, YU D Y. Polymorphisms of soybean isoflavone synthase and flavanone 3-hydroxylase genes are associated with soybean mosaic virus resistance., 2010, 25(1): 13-24.
[66] ZHANG G Y, CHEN M, CHEN X P, XU Z S, LI L C, GUO J M, MA Y Z. Isolation and characterization of a novel EAR-motif-containing genefrom soybean (L.)., 2010, 37(2): 809-818.
[67] 陳華濤, 陳新, 顧和平, 張紅梅, 袁星星, 崔曉燕. 大豆GmPR10基因克隆與植物表達載體的構建. 江蘇農業(yè)學報, 2011, 27(3): 494-499.
CHEN H T, CHEN X, GU H P, ZHANG H M, YUAN X X, CUI X Y. Cloning and plant expression vector construction of GmPR10 in soybean., 2011, 27(3): 494-499. (in Chinese)
[68] HE H L, YANG X D, XUN H W, LOU X , LI S Z, ZHANG Z B, JIANG L L, DONG Y S, WANG S C, PANG J S, LIU B. Over-expression ofenhances virus resistance inand soybean., 2017, 36(9): 1441-1455.
[69] LIU J Z, HORSTMAN H D, BRAUM E, GRAHAM M A, ZHANG C, NAVARRE D, QIU L W, LEE Y, NETTLETON D, HILL J H, WHITHAM S A. Soybean homologs ofnegatively regulate defense responses and positively regulate growth and development.,2011, 157(3): 1363-1378.
[70] LIU J Z, BRAUN E, QIU W L, SHI Y F, MARCELINO- GUIMARAES F C, NAVARRE D, HILL J H, WHITHAM S A. Positive, and negative roles for soybean MPK6 in regulating defense responses., 2014, 27(8): 824-834.
[71] LIU J Z, WHITHAM S A. Overexpression of a soybean nuclear localized type-III DnaJ domain-containing HSP40 reveals its roles in cell death and disease resistance., 2013, 74(1): 110-121.
[72] SEO J K, KWON S J, CHO W K, CHOI H S, KIM K H. Type 2C protein phosphatase is a key regulator of antiviral extreme resistance limiting virus spread., 2014, 4: 5905-5913.
[73] ZHOU L, HE H L, LIU R F, HAN Q, SHOU H X, LIU B. Overexpression ofpotassium channel enhances resistance to soybean mosaic virus., 2014, 14(1): 154-165.
[74] ZHOU Z, HE H L, MA L P, YU X Q, MI Q, PANG J S, TANG G X, LIU B. Overexpression of agene enhanced activity of nitrate reductase and aldehyde oxidase, and boosted mosaic virus resistance in soybean., 2015, 10(4): 1-15.
[75] LI N, YIN J L, LI C, WANG D G, YANG Y Q, ADHIMOOLAM K, LUAN H X, ZHI H J. NB-LRR gene family required for- mediated resistance to soybean mosaic virus., 2016, 67(5): 541-552.
[76] TRAN P T, WIDYASARI K, SEO J K, KIM K H. Isolation and validation of a candidategene from a soybean genotype that confers strain-specific resistance to soybean mosaic virus., 2017, 513: 153-159.
[77] LUAN H X, SHINE M B, CUI X Y, CHEN X, MA N, KACHROO P, ZHI H J, KARCHROO A. The potyviral P3 protein targets eukaryotic elongation factor 1A to promote the unfolded protein response and viral pathogenesis., 2016, 172(1): 221-234.
[78] CHEN H, ARSOVSKI A A, Yu K F, Wang A M. Deep sequencing leads to the identification of eukaryotic translation initiation factor 5a as a key element in-mediated lethal systemic hypersensitive response to soybean mosaic virus infection in soybean., 2016, 18(3): 391-404.
[79] ZHANG C Q, GROSIC S, WHITHAM S A, HILL J H. The requirement of multiple defense genes in soybean-mediated extreme resistance to soybean mosaic virus., 2012, 25(10): 1307-1313.
[80] FU D Q, GHABRIAL S, KACHROO A.andare required for basal, R gene-mediated and systemic acquired resistance in soybean., 2009, 22(1): 86-95.
[81] LI W L, ZHAO Y S, LIU C J, YAO G B, WU S S, HOU C Y, ZHANG M C, WANG D M. Callose deposition at plasmodesmata is a critical factor in restricting the cell-to-cell movement of soybean mosaic virus., 2012, 31(5): 905-916.
[82] SAGHAI MAROOF M A, JEONG S C, GUNDUZ I, TOLIN S. Pyramiding of soybean mosaic virus resistance genes by marker-assisted selection., 2008, 48(2): 517-526.
[83] SHI A N, CHEN P Y, LI D X, ZHENG C M, ZHANG B, HOU A F. Pyramiding multiple genes for resistance to soybean mosaic virus in soybean using molecular markers., 2009, 23(1): 113-124.
[84] WANG D G, ZHAO L, LI K, MA Y, WANG L Q, YANG Y Q, YANG Q H, ZHI H J. Marker-assisted pyramiding of soybean resistance genesR,R, andRto soybean mosaic virus., 2017, 16(11): 2413-2420.
[85] 鄭桂杰. 齊黃1號對大豆花葉病毒抗性基因的精細定位、育種應用及候選基因表達分析[D]. 南京: 南京農業(yè)大學, 2012.
ZHENG G J. Fine mapping, expression analysis and breeding application of resistance genes to soybean mosaic virus in Qihuang No.1[D]. Nanjing: Nanjing Agriculture University, 2012. (in Chinese)
[86] 徐冉, 石傳娥, 張禮鳳, 王彩潔, 聶翠琴, 李建和. 黃淮海大豆優(yōu)異種質齊黃1號的育種應用. 植物遺傳資源學報, 2004, 5(2): 170-175.
XU R, SHI C E, ZHANG L F, WANG C J, NIE C Q, LI J H. Utilization of Qihuang 1 in soybean breeding in the Huanghuaihai region., 2004, 5(2): 170-175. (in Chinese)
[87] 張孟臣, 張磊, 劉學義, 李衛(wèi)東. 黃淮海大豆改良種質. 北京: 中國農業(yè)出版社, 2014.
ZHANG M C, ZHANG L, LIU X Y, LI W D.. Beijing: China Agriculture Press, 2014. (in Chinese)
[88] CHOI B K, KOO J M, AHN H J, YUM H J, CHOI C W, RYU K H, CHEN P Y, TOLIN S A. Emergence of-resistance breaking soybean mosaic virus isolates from Korean soybean cultivars., 2005, 112(1): 42-51.
[89] KOO J M, CHOI B K, AHN H J, YUM H J, CHOI C W. First report of anresistance-breaking isolate of soybean mosaic virus in Korea., 2005, 54(4): 573.
[90] SARUTA M, KIKUCHI A, OKABE A, SASAYA T. Molecular characterization of A2 and D strains of soybean mosaic virus, which caused a recent virus outbreak in soybean cultivar Sachiyutaka in Chugoku and Shikoku regions of Japan., 2005, 71(6): 431-435.
[91] GAGARINOVA A G, BABU M, POYSA V, HILL J H, WANG A M. Identification and molecular characterization of two naturally occurring soybean mosaic virus isolates that are closely related but differ in their ability to overcomeresistance., 2008, 138(1/2): 50-56.
[92] WANG Y, HAJIMORAD M R. Gain of virulence by soybean mosaic virus on-genotype soybeans is associated with a relative fitness loss in a susceptible host., 2016, 17(7): 1154-1159.
[93] YANG Y Q, LIN J, ZHENG G J, ZHANG M C, ZHI H J. Recombinant soybean mosaic virus is prevalent in Chinese soybean fields., 159(7): 1793-1796.
[94] YANG Y Q, GONG J W, LI H W, LI C Y, WANG D G, LI K, ZHI H J. Identification of a novel soybean mosaic virus isolate in China that contains a unique5' terminus sharing high sequence homology with bean common mosaic virus., 2011, 157(1): 13-18.
[95] ZHOU G C, SHAO Z Q, MA F F, WU P, WU X Y, XIA Z Y, YU D Y, CHENG H, LIU Z H, JIANG Z F, CHEN Q S, WANG B, CHEN J Q. The evolution of soybean mosaic virus: An updated analysis by obtaining 18 new genomic sequences of Chinese strains/isolates., 2015, 208: 189-198.
[96] 董英山. 中國野生大豆研究進展. 吉林農業(yè)大學學報, 2008, 30(4): 394-400.
DONG Y S. Advances of research on wild soybean in China., 2008, 30(4): 394-400. (in Chinese)
[97] WEN Z X, DING Y L, ZHAO T J, GAI J Y. Genetic diversity and peculiarity of annual wild soybean (Sieb. et Zucc.) from various eco-regions in China., 2009, 119(2): 371-381.
[98] ZHOU Z K, JIANG Y, WANG Z, GOU Z H, YU J L, LI W Y, YU Y J, SHU L P, ZHAO Y J, MA Y M, FANG C, SHEN Y T, LIU T F, LI C C, GAO Y D, XIANG H, ZHU B G, LEE S H, WANG W, TIAN Z X. Resequencing 302 wild and cultivated accessions identifies genes related to domestication and improvement in soybean., 2015, 33(4): 408-414.
(責任編輯 李莉)
Progresses of Resistance on Soybean Mosaic Virus in Soybean
WANG DaGang1, LI Kai2, ZHI HaiJian2
(1Crop Institute of Anhui Academy of Agricultural Sciences/Key Laboratory of Crop Quality Improvement of Anhui Province, Hefei 230031;2Soybean Research Institute of Nanjing Agriculture University/National Center for Soybean Improvement/ National Key Laboratory for Crop Genetics and Germplasm Enhancement, Nanjing 210095)
Soybean mosaic virus disease, caused by soybean mosaic virus (SMV), is one of the most serous pathogens of soybean ((L.) Merr.), which give rise to the loss of yield and quality in soybean production worldwide. During the recent decade, the studies on soybean against SMV stress have made some progress, includes the mapping of SMV resistant genes, the functional analysis of candidate resistance genes, and the progress in dissecting the SMV resistant signaling pathways in soybean. There are two kinds of resistance to SMV in soybean, quantitative resistance and qualitative resistance. The quantitative resistance to SMV is controlled by an additive major gene plus additive-dominant polygenes, and the qualitative resistance is mainly controlled by a dominant resistance gene. Research showed that the quantitative trait loci (QTL) were mainly on chromosome 6, 10 and 13. So far, 22 SMV qualitative resistance loci have been successively mapped on soybean chromosome 2, 6, 13 and 14. And most of the physical distance between the markers on both sides of the SMV resistance gene loci is within 1 Mb. Among them, there are 8 gene loci (,R,R,RandRetc.) located on chromosome 2, 10 loci (,,R,RandRetc.) on chromosome 13, 2 loci on chromosome 6 (RandR) and 14 (andR), respectively. According to the Williams 82 whole genomic sequence (http://www.phytozome.net/soybean), the putative candidate genes of SMV qualitative resistance loci were subsequently narrowed down based on their predicted functions, expression patterns and sequence comparison etc. Among these genes,,,,,,andet al. eight genes were considered potential resistance candidate genes to SMV on chromosome 2;as the most promising candidate gene on chromosome 6;,,,,,,,,et al. nine genes and,,,,,et al. six genes were the putative candidate genes to SMV on chromosome 13 and 14, respectively. Using virus induced gene silencing (VIGS) and transgenic technology etc. analyzed the functions of the candidate genes. The results showed genes,andcan participate to SMV resistance in soybean as positive regulator. After knocked downand, viral accumulation level of SMV and pathogenicity increased. They were considered negative regulator to SMV resistance. Based on the research results of SMV resistance gene, regulated model were built forandmediating extreme resistance (ER) against SMV.-mediated ER has provided new insight into the soybean signaling network required for ER against SMV. The primary mechanism of-mediated ER against viruses was the inhibition of viral cell-to-cell movement by callose deposition in an ABA signaling-dependent manner. In this report, the research progress on the mapping and function analysis of SMV resistance genes and the future research directions of SMV resistance in soybean are summarized. It will provide a basis for molecular design breeding and mechanism research of resistance genes to SMV in soybean.
soybean; soybean mosaic virus; resistance gene; marker location; functional research
2018-04-07;
2018-06-05
國家自然科學基金(31571687、31571690和31201235)、安徽省自然科學基金(1708085MC69)、國家大豆產業(yè)技術體系建設專項資金(CARS-04)、江蘇省現代作物生產協同創(chuàng)新中心(JCIC-MCP)
王大剛,Tel:0551-65149851;E-mail:smvwang@163.com。
智海劍,Tel:025-84396463;E-mail:zhj@njau.edu.cn
10.3864/j.issn.0578-1752.2018.16.002
