柑橘CitCEP基因家族的鑒定及對逆境和激素的響應
潘小婷,張靜,葛廷,馬巖巖,鄧烈,何紹蘭,易時來,鄭永強,呂強,謝讓金
?
柑橘CitCEP基因家族的鑒定及對逆境和激素的響應
潘小婷,張靜,葛廷,馬巖巖,鄧烈,何紹蘭,易時來,鄭永強,呂強,謝讓金
(西南大學柑桔研究所/中國農業科學院柑桔研究所/國家柑桔工程技術研究中心,重慶 400712)
【目的】基于全基因組鑒定分析柑橘CEP基因(CitCEPs)家族成員,了解各成員的分類進化關系,研究各基因成員在不同組織中的表達特異性以及對激素和非生物脅迫的響應,為進一步研究CEP基因家族的生物學功能打下基礎。【方法】利用BLASTp基于Phytozome數據庫鑒定柑橘CEP家族成員,采用GSDS、ProtScale Tool、EXPASY、CLUSTALX、MEGA6.0、plantCARE、Cello等軟件繪制家族成員基因結構圖;分析預測蛋白的相對分子質量與等電點等理化性質;構建系統進化樹及亞細胞定位預測等,利用實時熒光定量PCR技術(qRT-PCR)檢測柑橘CEP基因家族各成員在‘資陽香橙’(‘Ziyang’)不同處理下的瞬時表達情況。【結果】柑橘CEP基因家族由11個成員組成,其中和含1個內含子,其余成員均無內含子;該基因家族成員均含有SPGV/IGH保守結構域序列,其預測蛋白均為親水性蛋白,親水性最強的氨基酸其值為3.711,親水性最弱的氨基酸其值為-2.778;亞細胞定位預測結果顯示該基因家族成員位于細胞不同位置,其中CitCEP1、CitCEP2、CitCEP4、CitCEP5、CitCEP6、CitCEP8定位于細胞外,CitCEP3和CitCEP7在細胞外和線粒體中均存在,CitCEP9位于細胞核中,CitCEP10在細胞質膜上,而CitCEP11位于線粒體和細胞核中;聚類分析發現,該家族成員分布于不同的分支中,分別與擬南芥CEP不同成員聚在一起,表明柑橘CEP成員間具有不同生物功能。表達分析顯示,主要在莖、葉和子葉中表達,主要在根和子葉中表達,而和主要在果皮中表達,其余成員在上述組織中表達極低或不表達,體現了不同成員間組織特異性的差異。在干旱條件下,表達下調,、和的相對表達量均逐漸上調;而在鹽脅迫下,的響應模式與其所對應的干旱脅迫類似,和則呈現先升后降趨勢。在乙烯(ETH)、脫落酸(ABA)處理下,的表達量明顯受到抑制,而在茉莉酸甲酯(MeJA)、水楊酸(SA)、生長素(IAA)、赤霉素(GA3)處理下均表現為先上升、后下降的趨勢;在6-芐氨基腺嘌呤(6-BA)、IAA、GA3、ABA處理下也表現出類似趨勢。在ETH處理下表達量顯著上升,在6-BA、SA、ABA處理下表達量呈現先下降后上升趨勢,而在MeJA、IAA、GA3處理中無明顯規律。在ETH、MeJA、SA、IAA、GA3、ABA處理下表達均呈現下調趨勢,與在ETH、SA處理下的表達模式類似。在6-BA處理下,表達呈現先下降后上升趨勢。【結論】從柑橘全基因組上鑒定出了11個CitCEP基因成員,各成員均為親水蛋白,并含有SPGV/IGH保守結構域,位于細胞中不同位置。在不同逆境和激素處理下,、、和呈現不同程度的響應,而其余成員響應不明顯或未響應。推測、與在柑橘生長發育和逆境響應過程中可能起著重要作用。
柑橘;CitCEP基因家族;基因表達;激素;脅迫
0 引言
【研究意義】柑橘是世界范圍內栽培面積最大的果樹作物之一,但由于高溫、伏旱等原因導致了柑橘大量異常落花落果,嚴重影響了產量。植物CEP基因可參與逆境響應,并對激素處理也具有不同的響應模式。因此,本研究基于柑橘全基因組對CEP基因家族進行全面分析,深入了解柑橘生長發育和逆境響應的分子基礎,研究結果對進一步培育或改良柑橘抗性品種具有重要意義。【前人研究進展】許多植物多肽(多肽激素)具有類似生長素(IAA)、乙烯(ETH)和赤霉素(GA3)等激素的調節功能,廣泛參與植物的一系列生長發育過程、花粉與柱頭識別、防御反應、介導植物細胞間信號的轉導和傳遞等[1-9]。多肽激素以配基的形式與細胞膜表面的相應受體激酶互作,從而激活下游基因、啟動相關信號轉導[10]。編碼植物多肽激素的基因家族主要包括CLE家族、RGF家族、ERF家族和CEP家族等[10-16]。其中CEP(-TERMINALLYNCODEDEPTIDE)基因廣泛存在于種子植物中,在進化上比較保守。CEP蛋白含一個或者多個CEP結構域[16],在CEP結構域的C-末端存在保守的蛋白序列(SPGV/IGH)[17-19]。在擬南芥中,Roberts等[19]根據結構域中脯氨酸數目的不同將CEP家族分為Group I和Group II,其中,Group I的CEP結構域含有3個脯氨酸,包括AtCEP1—AtCEP12,Group II的CEP結構域含有2個脯氨酸,包括AtCEP13、AtCEP14、AtCEP15。單子葉植物和雙子葉植物的CEP結構域不同,其中單子葉植物的CEP結構域中有2個位點缺少苯丙氨酸殘基,然而,這2個位點在雙子葉植物中卻非常保守,并且所有雙子葉植物的CEP結構域的最后一個氨基酸均為組氨酸,而單子葉植物的CEP結構域末端氨基酸為組氨酸或天冬氨酸[18]。Ogilvie等[17]研究發現,禾本科植物的CEP基因堿基序列中GC含量明顯高于其他植物種類。在擬南芥中,與受體XYLEM INTERMINXED WITH PHLOEM 1(XIP1)/CEP RECEPTOR 1(CEPR1)作用,從而調控主根長度和側根密度[20-21]。的超表達使得根系變短,同時出現開花期延遲、葉片數目減少,其他CEP基因成員也呈現類似功能,但也存在差異[18-19]。在其他植物中,CEPs也參與植物根/莖生長、側根發育以及根瘤分化等生長發育過程[16,18-19,22]。Roberts等通過在線數據庫eFP[23]和Genevestigator[24]分析了擬南芥CEP基因在不同器官組織中的表達,如主要在頂端分生組織,和主要在芽頂端分生組織中表達,其中也在莖尖分生組織中表達,和在主根的根尖分生組織中表達,和主要在子葉或葉中表達,在側根形成和發育過程中,這些CEP基因均可轉錄表達[19]。進一步探索發現CEP家族基因在同一組織器官的不同位置其表達模式也有差異,例如在5日齡幼苗的子葉中,在葉脈中表達,在葉尖端表達,在兩周齡的葉中,在葉緣中表達,和在葉柄中表達[19]。此外,已有研究表明CEP基因可參與逆境響應,例如擬南芥和在水楊酸(SA)處理的條件下表達量降低;而赤霉素能誘導擬南芥和的表達;脫落酸(ABA)能使表達下調;茉莉酸能使表達上調等;AtCEPs家族成員間對生長素的應答模式存在差異,如該激素可誘導和表達升高,卻抑制和的表達;同樣,油菜素內酯可以使的表達降低而誘導的表達;另外,乙烯、內脂、細胞分裂素等對AtCEPs家族基因的表達沒有顯著的影響[19],超表達能夠顯著抑制側根數目,RNA干擾能夠促進側根生長[6,10,22,25]。【本研究切入點】CEP肽激素廣泛參與植物根系在逆境下的生長發育調控,因此受到眾多研究者的關注,但是目前對CEP家族的研究主要集中在模式植物如擬南芥、玉米、水稻和苜蓿,在柑橘中對該基因家族進行鑒定和分析尚未見報道。【擬解決的關鍵問題】基于柑橘全基因組鑒定柑橘CEP家族成員,并對該基因家族進行生物信息學分析,同時利用實時熒光定量PCR技術(qRT-PCR)檢測該基因在不同組織中以及多種脅迫條件下的表達模式,解析CEPs調控柑橘根系生長發育及其逆境響應的分子基礎。
1 材料與方法
1.1 試材與處理
選用‘資陽香橙’(‘Ziyang’)為試材,其種子為多胚,苗由珠心胚發育而來,具有一致的遺傳背景。2016年11月在中國農業科學院柑桔研究所國家柑橘種質資源圃采集‘資陽香橙’種子,去除內外種皮,放在濕潤的紗布上,塑料膜保濕;然后放入28℃的培養箱中暗培養10 d,進行催芽。當根長至2 cm左右時將幼苗轉入營養液(250 μmol·L-1KH2PO4,3 000 μmol·L-1KNO3,2 000 μmol·L-1Ca(NO3)2,250 μmol·L-1MgSO4,25 μmol·L-1MgCl2,12.5 μmol·L-1H3BO3,1 μmol·L-1MnSO4,1 μmol·L-1ZnSO4,0.25 μmol·L-1CuSO4,0.25 μmol·L-1(NH4)6Mo7O24,25 μmol·L-1Fe-Na-EDTA,pH 5.8)中進行水培[26],當第一片真葉充分展開后,選取長勢一致的幼苗進行激素和非生物脅迫處理。激素包括ABA(100 μmol·L-1)、SA(100 μmol·L-1)、MeJA(茉莉酸甲酯,100 μmol·L-1)、IAA(20 μmol·L-1)、GA3(0.5 m mol·L-1)、ETH(1.5 mmol·L-1)、6-BA(6-芐氨基腺嘌呤,0.05 mmol·L-1),具體步驟如下:在室溫下,將幼苗洗凈,放入盛有激素或純凈水(平行對照)的滅菌瓶中搖勻,使根系充分接觸液體。在處理后的0(對照)、3、6、12、24 h分別收集幼苗根系,立刻放入液氮中進行速凍,-80℃貯藏備用。高鹽NaCl(300 mmol·L-1)處理及取樣方法與激素處理相同[27]。干旱脅迫采用紙巾包裹幼苗根系,放置室溫下使其脫水,分別在0、1、3、6和12 h收集根系,液氮速凍,-80℃貯藏備用。每個處理至少5株幼苗。
采集‘資陽香橙’實生幼苗的根、莖、葉、子葉,及其成熟果子的果皮和種子,液氮速凍,-80℃貯藏,用于分析柑橘CEP家族成員表達的組織特異性。
1.2 總RNA提取與cDNA合成
植物RNA提取試劑盒(RNA prep pure Kit)購自天根生化科技有限公司,用于抽提總RNA。使用TaKaRa公司的反轉錄試劑盒(Prime ScriptTMRT Reagent Kit)合成第一條cDNA鏈。上述試驗均按照試劑盒說明書進行。
1.3 基因的結構與序列分析
以擬南芥CEP蛋白為參考序列,采用BLASTp在phytozome數據庫(www.phytozome.net)中進行比對,鑒定出柑橘CEP基因(CitCEPs)成員;用EXPASY軟件(http://web.expasy.org/protparam/)分析蛋白編碼的氨基酸數目、相對分子量以及等電點等理化性質[28];ProtScale Tool軟件(http://web.expasy.org/protscale/)分析蛋白質的親水性/疏水性[29],基因結構采用GSDS軟件(http://gsds.cbi.pku.edu.cn/)進行分析并繪制結構圖。用CLUSTALX對蛋白多序列進行比對,采用MEGA6.0軟件鄰接法構建NJ系統進化樹。采用MBC(http://cello.life.nctu.edu.tw)軟件對CitCEP蛋白進行亞細胞定位預測。采用在線軟件plantCARE(http:// bioinformatics.psb.ugent.be/webtools/plantcare/html/)對啟動子順式元件進行分析。
1.4 實時熒光定量分析
引物由Primer5.0設計,引物信息見表1。以柑橘為內參,在Bio-Rad CFX3.1熒光定量PCR儀上對CEP基因各成員進行定量分析。反應體系為10 μL:5 μL熒光染料(Bio-Rad公司)、3 μL無菌水、0.5 μL正向引物、0.5 μL反向引物、1 μL cDNA。反應程序為:95℃ 30 s;95℃ 5 s,55℃ 5 s,39個循環。每個處理3次重復。相對表達量采用2-ΔΔCt法計算,用spss19.0軟件對數據進行統計分析。

表1 本試驗所用引物
2 結果
2.1 CitCEP基因家族成員的鑒定與生物信息學分析
2.1.1 柑橘CitCEP基因的鑒定 以擬南芥CEP蛋白序列為參考,基于Phytozome數據庫采用BLASTp從柑橘全基因組中鑒定出11個CitCEP成員(表2),其中只存在于克林曼丁橘的有4個,即、、、;甜橙的有2個,和,而、、、、在甜橙和克林曼丁橘基因組上均含有。通過蛋白序列比對發現,C-末端均存在該家族的特征結構域(SPGV/IGH)(圖1)。
2.1.2 基因結構與預測蛋白特性分析 基于柑橘全基因組,分別獲得柑橘CitCEP成員的mRNA及其基因組DNA序列,利用GSDS在線軟件對各成員的基因結構進行了分析。結果表明,—均無內含子,而和含有1個內含子(圖2)。采用EXPASY軟件對預測蛋白的氨基酸數目、等電點、分子量、不穩定系數、脂肪系數、平均疏水指數進行分析(表2)。結果顯示,柑橘CEP家族成員均為親水性蛋白,CitCEP5的第12位氨基酸的親水性最強,其值為3.711,CitCEP4的第32位氨基酸疏水性最強,其值為-2.778。從表2可看出,CitCEP成員之間基因組長度變化明顯,其中最短,只含有255個堿基,可編碼84個氨基酸,而最長,具有865個堿基,可編碼141個氨基酸,但其分子量最高,為15 528.08 kD。該基因家族成員的等電點介于6.04—10.43,不穩定指數介于30.70—57.86,脂肪系數介于59.12—98.23,平均疏水指數介于-0.811—0.198。
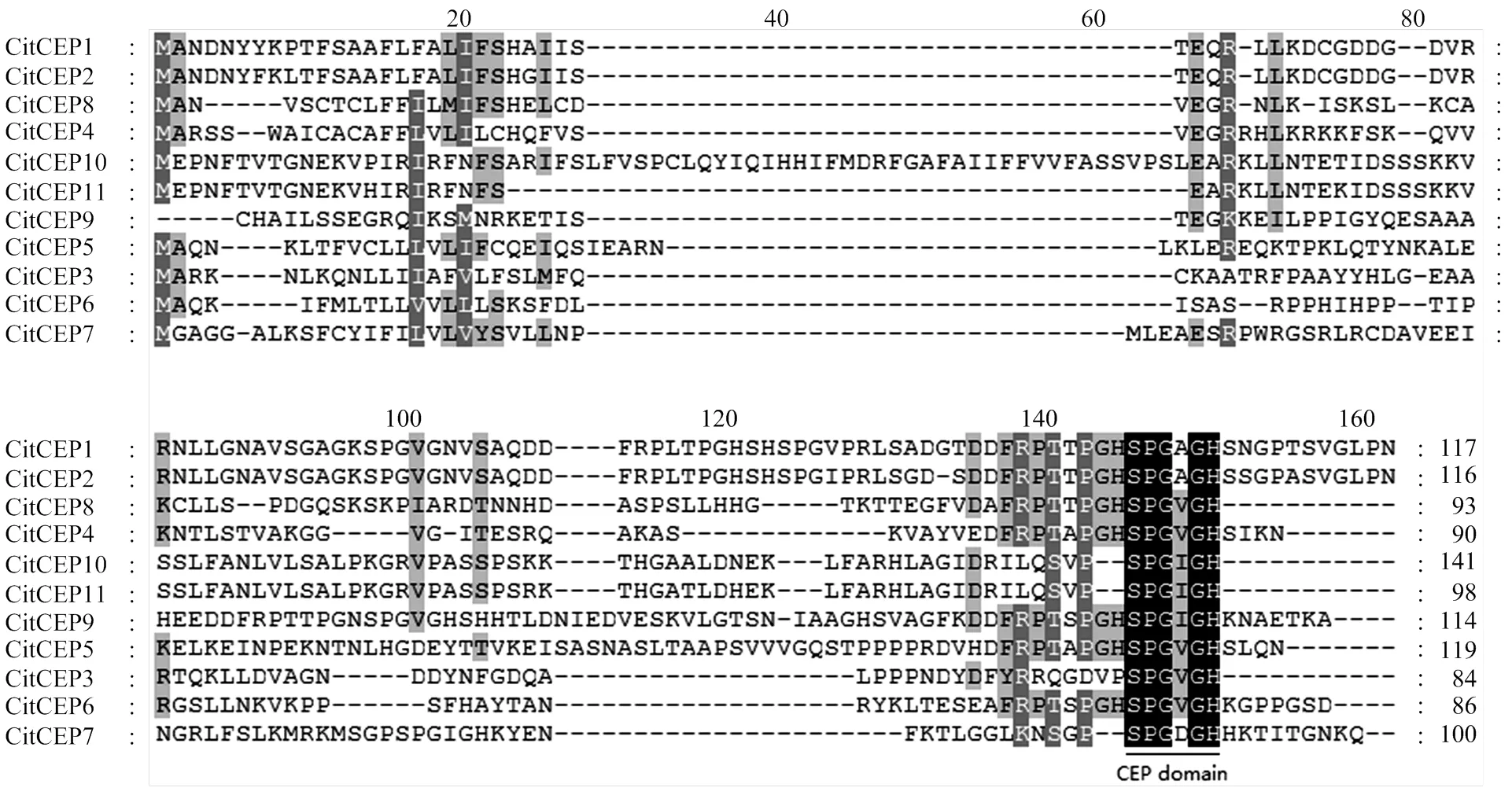
圖1 CitCEP家族蛋白序列的氨基酸聚類分析
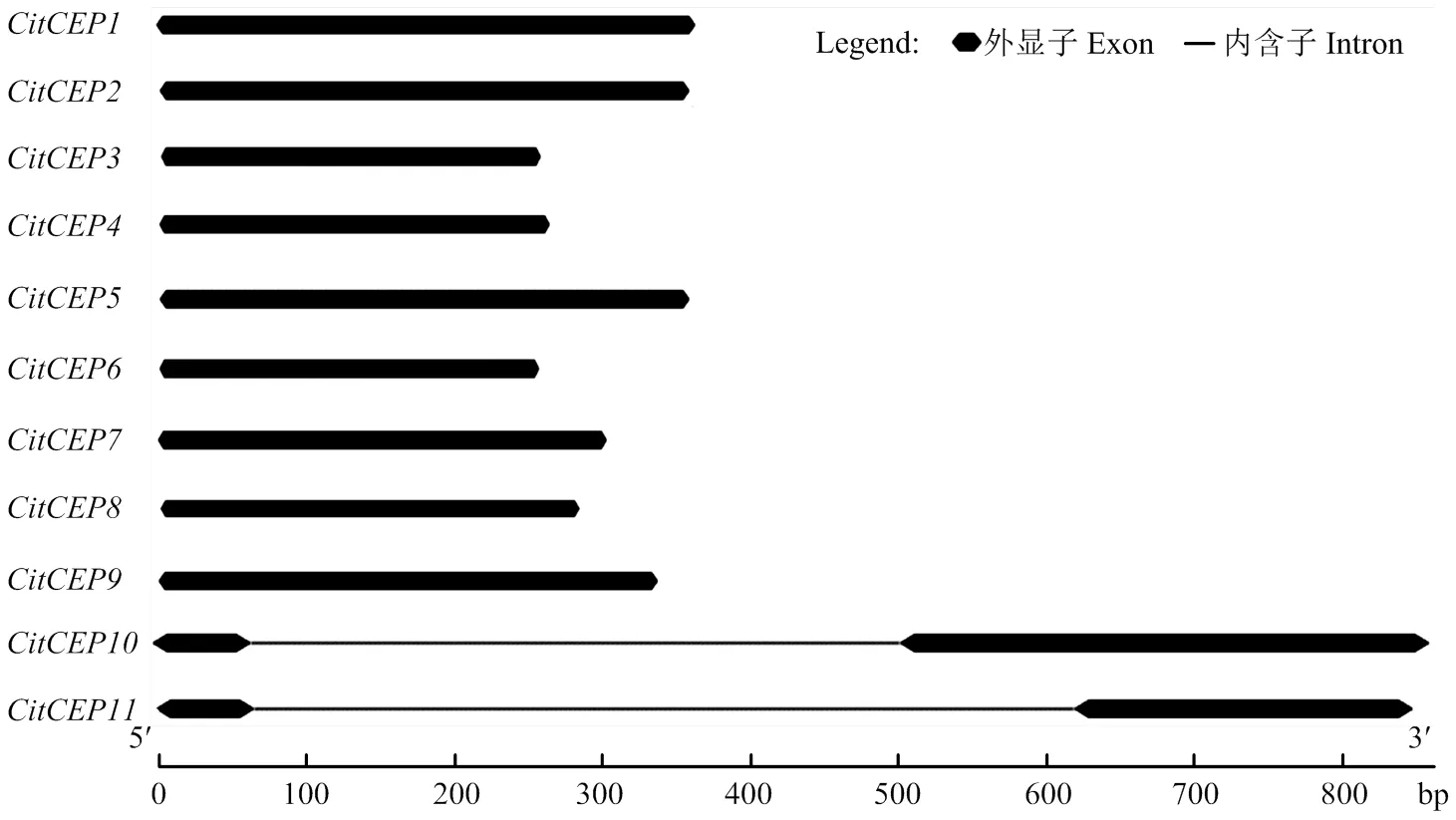
圖2 CitCEP成員的基因結構

表2 CitCEP家族成員蛋白質的理化性質
2.1.3 CitCEP蛋白的亞細胞定位預測與親緣關系 利用Cello軟件對柑橘CEP家族成員的亞細胞定位進行了分析,結果表明CitCEP1、CitCEP2、CitCEP4、CitCEP5、CitCEP6、CitCEP8定位在細胞外,CitCEP3和CitCEP7在細胞外和線粒體中均存在,CitCEP9定位在細胞核中,CitCEP10定位于細胞質膜上,而CitCEP11存在于線粒體和細胞核中。
為了解柑橘CEP成員間的親緣關系,推測其生物功能,利用Mega6.0軟件構建了擬南芥和柑橘CEP蛋白成員的NJ進化樹(圖3)。從圖3可以看出,擬南芥CEP成員與柑橘CEP成員相互嵌合在一起,形成3大分支(G1、G2和G3),表明植物CEP基因在進化過程中相對保守。G1含有成員最多共計17個,包括4個柑橘CitCEPs和13個AtCEPs;G2由CitCEP1、CitCEP2、CitCEP3和AtCEP5組成;G3有CitCEP8、CitCEP9、CitCEP10、CitCEP11和AtCEP3組成。在這些成員中CitCEP5與AtCEP7,CitCEP4與AtCEP1,CitCEP1、CitCEP2與AtCEP5,CitCEP9與AtCEP3的親緣關系較近,推測這些CEP具有類似的生物功能;其他柑橘CitCEP成員與擬南芥成員的親緣關系較遠,表明這些成員之間的生物功能可能差異較大。
2.2 CitCEP家族基因的表達分析
2.2.1 組織特異性 柑橘CitCEP成員間具有明顯不同的組織特異性。其中,、、、、和在各組織中均未檢測到明顯的表達信號。在莖中表達量最高,葉中次之,在根和子葉中較低,果皮和種子中幾乎不表達。主要在根和子葉中表達,在莖、葉、種子中表達很低,而在果皮中幾乎不表達;和均顯著在果皮中表達,而在莖、果皮、種子和葉中表達極低或不表達(圖4)。
2.2.2 非生物脅迫與激素對CitCEP表達的影響、、、、、和對干旱、鹽脅迫、ABA等各處理響應不顯著,其表達很低或不表達。而、、和對各處理表現出不同的表達模式。在鹽脅迫和干旱條件下,的表達量均受到抑制,而在鹽脅迫條件下表達量先上調后下降,最后恢復至對照水平,在干旱條件下其表達量顯著上調;在干旱處理下,和的表達量均顯著上調,呈現相似趨勢,而在鹽脅迫下,表達顯著受到誘導,卻在處理3 h后出現顯著上調后恢復到對照水平。上述結果表明,、、和參與了柑橘的非生物脅迫應答(圖5)。在ETH、ABA處理下,的表達量明顯受到抑制,而在MeJA、SA、IAA、GA3處理下均表現為先上升、后下降的趨勢;在6-BA、IAA、GA3、ABA處理下也表現出類似趨勢。在ETH條件下表達顯著上升,在6-BA、SA、ABA處理條件表達呈現先下降后上升的趨勢,在MeJA、IAA、GA3處理下無明顯規律。在ETH、MeJA、SA、IAA、GA3、ABA處理下均呈現表達下調趨勢,與在ETH、SA處理下的表達模式類似。在6-BA條件下,表達呈現先下降后上升趨勢(圖6)。綜上所述,、、和可能與柑橘的生長發育和脅迫響應有關。

圖3 柑橘與擬南芥CEP家族蛋白的系統進化樹(鄰接法)
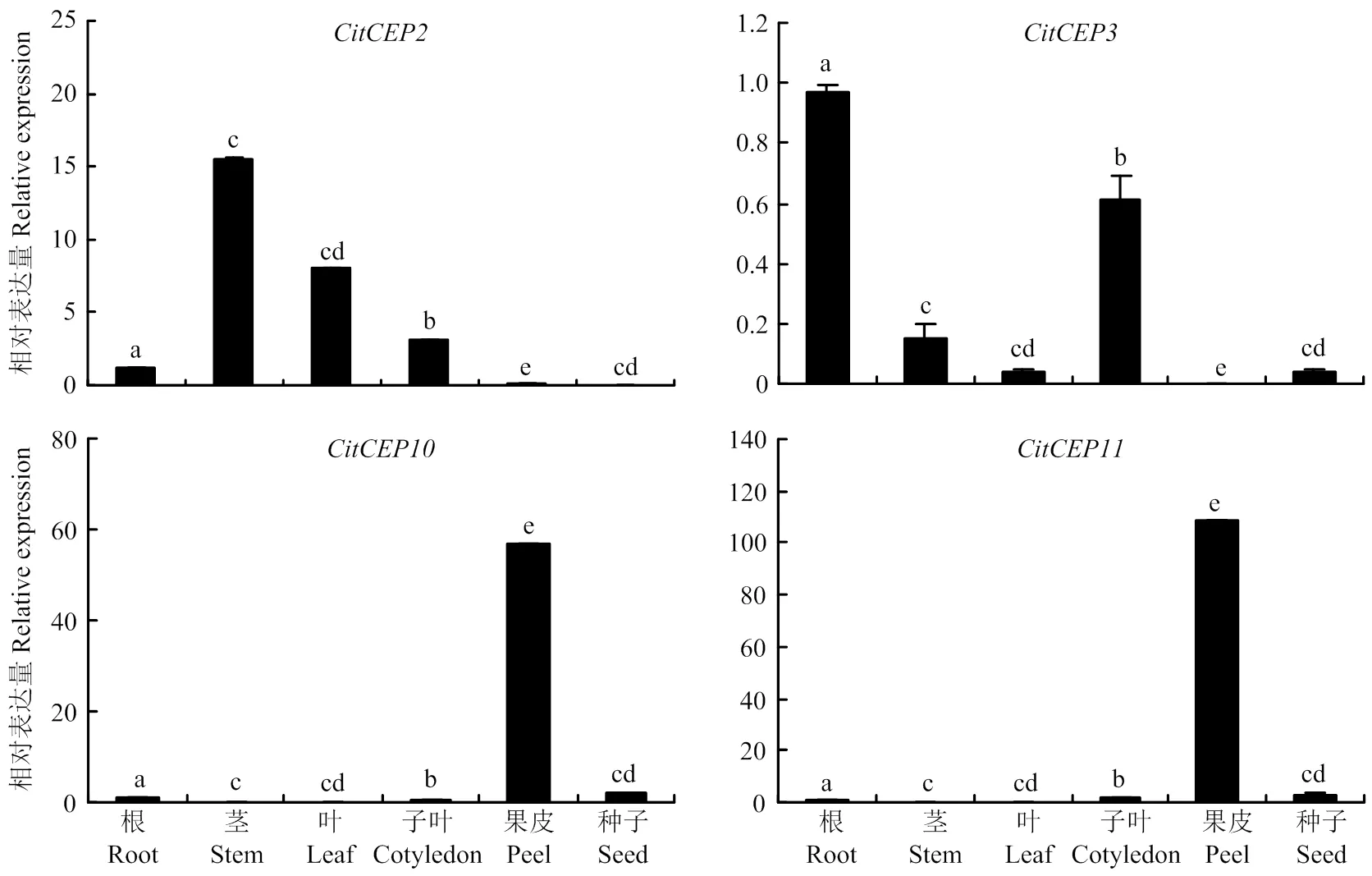
圖4 CitCEP2、CitCEP3、CitCEP10、CitCEP11在不同組織中的相對表達量
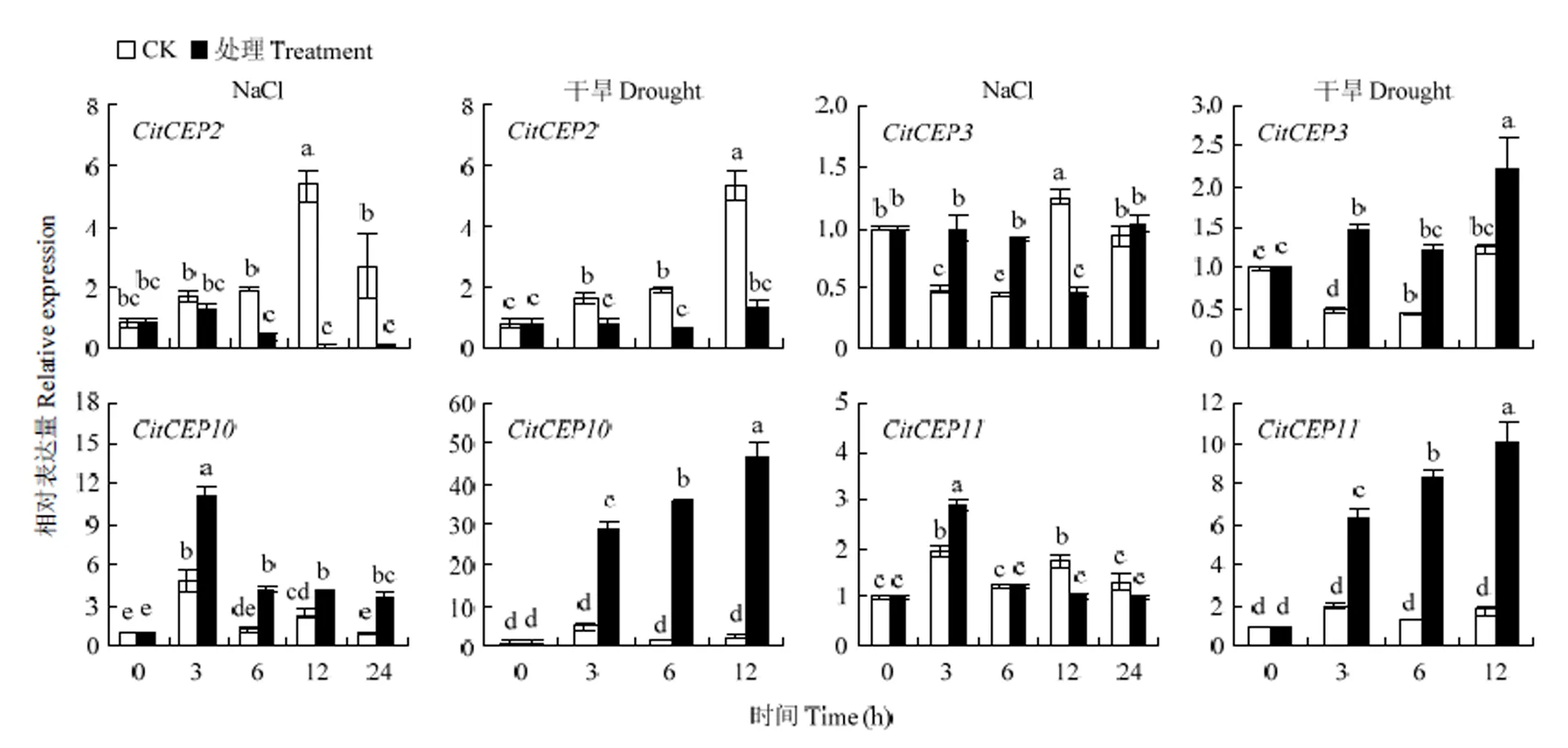
圖5 非生物脅迫下CitCEP2、CitCEP3、CitCEP10、CitCEP11的相對表達量
為了進一步說明、和對各處理的應答機制,利用柑橘全基因組分別獲得各基因起始密碼子前2 000 bp的啟動子序列。采用plantCARE在線軟件對順式作用元件分別進行了預測分析,結果顯示、和的啟動子中包含了多個與逆境和激素相關的順式作用元件(表3)。其中,、和均包含光響應元件(ATCT-motif、G-box)熱應激響應元件(HSE)和水楊酸響應元件(TCA-element)。另外,、和還含有ACE光響應元件脫落酸響應元件(ABRE)和低溫響應元件(LTR),而赤霉素響應元件(GARE-motif)只存在于、啟動子中。和啟動子含有與干旱相關的MBS元件。此外,啟動子還含有冷和脫水脅迫相關的響應元件(C-reapt/DRE),和含有MeJA響應元件(TGACG-motif)。

表3 PlantCARE預測CitCEP2、CitCEP3、CitCEP10、CitCEP11啟動子區順式作用元件

圖6 不同外源激素處理條件下CitCEP2、CitCEP3、CitCEP10、CitCEP11的相對表達量
3 討論
植物多肽激素大約包含20個氨基酸,是一類經過剪切后形成的具有特殊功能的、成熟的多肽[30]。自1991年在番茄中發現了第一個多肽激素系統素,隨后植物多肽激素受到越來越多的關注[31],CEP蛋白是由N末端的分泌信號、變異結構、一個或者多個CEP結構域和C-末端序列組成[16]。目前在植物基因組中鑒定出的CEP多肽主要存在于種子植物中,其中在擬南芥基因組共鑒定出15個,但有4個AtCEP家族基因(、、和)未被注釋[16-19,22,32];水稻中鑒定出15個,玉米中鑒定到12個[10],苜蓿中鑒定出9個,蘋果中迄今只報道了1個[33]。本研究從柑橘全基因組數據庫中獲得了11個CEP基因成員,與玉米中CEP成員數相當,而少于水稻和擬南芥中的成員數,表明不同物種的CEP成員數變化較大。該基因家族成員均為親水性蛋白,且亞細胞定位預測結果顯示主要分布于細胞外、線粒體、細胞質膜、細胞核中。序列分析發現,CitCEP10與CitCEP11的序列基本一致,且數據表達顯示對各激素處理以及非生物脅迫的結果響應趨勢相似度很高,但來源于不同的柑橘品種,因此推測CitCEP10與CitCEP11為不同等位變異類型,此種變異類型在水稻OsCEP中也有報道[10]。
聚類分析發現,柑橘與擬南芥CEP成員相互嵌合分布于不同分支中,表明CEP在進化過程中保守性較強,這與Roberts等[19]的研究結果一致,即CEP家族在被子植物中的進化較為保守。柑橘、與擬南芥位于一個亞分支中,具有較近的親緣關系。在擬南芥中敲除導致植物生長勢有所增加;相反,過量表達致使轉基因植株發育遲緩且不表現出向地性[19],由此推測、可能也與柑橘的生長發育和向地性有關。已有研究表明在滲透強度增加以及鹽水平升高的條件下使得擬南芥芽中的表達量上調,但在根中卻無響應[18],過量表達可抑制轉基因植株的主根生長[16]。本試驗中與具有較高的相似度,表明可能具有調控柑橘主根生長的生物功能。與相似度最高,但目前擬南芥功能還沒有得到驗證,有待于進一步研究。與擬南芥相似性最高,已有研究表明高氮、鉀水平能使表達下調[19],超表達擬南芥可抑制根生長、導致葉片變小及黃化[18-19],由此可見可能與柑橘的營養調控有關,從而影響葉片和根系生長。其余柑橘CEP成員如與擬南芥CEP各成員的親緣關系較遠,表明它們之間的生物功能差異較大,有待于進一步研究。
本試驗中,、、、、、和在所采集的幼苗時期的根、莖、葉、子葉以及果皮和種子中均無表達。這些成員可能為假基因或在其他特定組織和時期中表達[25]。已有研究表明,CEP基因參與了根、莖、芽、花的生長發育[10,16,18,22,32]。本試驗中主要在莖、葉、子葉中表達,主要在根和子葉中表達,而和均在果皮中顯著表達,表明CEP基因除了調控根、莖、芽、花的生長發育,同時可能也參與了葉、子葉和果皮發育。
本研究中,、、、、、和對各處理均無響應,此現象在其他基因家族中也存在[26]。擬南芥中,的轉錄水平受逆境、營養水平和激素等調控,Delay等對擬南芥9個()在不同濃度的N含量、鹽脅迫及CO2處理條件下的表達進行了分析,表明除外其余8個基因均有不同程度的響應[18]。另外,這些基因的表達水平受水楊酸、赤霉素、脫落酸、生長素等激素處理影響[19]。在本研究中,、與對激素、干旱和鹽脅迫均有響應,與前人研究結果類似[19]。由此推測、與可能參與了植物生長發育以及逆境響應調控。對、與的啟動子分析發現,其啟動子均含有逆境、激素、溫度等響應順式作用元件如G-box光響應元件、TCA水楊酸響應元件、HSE熱應激響應元件等,該結果為柑橘CEP基因對逆境和激素的應答機制提供了進一步證明。
4 結論
從柑橘全基因組上鑒定出了11個CitCEP基因成員,其蛋白均為親水蛋白且含有SPGV/IGH保守結構域。除和含有1個內含子外,其余成員均無內含子,亞細胞定位預測顯示柑橘CEP家族基因成員在細胞外、線粒體、細胞核、細胞質膜中均存在,通過實時熒光定量PCR技術對柑橘CEP基因家族進行表達分析,結果表明、、、、、和在‘資陽香橙’的根、葉、子葉、果皮、種子中幾乎不表達,對激素與非生物脅迫均不響應。、、的組織特異性差異明顯,且對激素和非生物脅迫有不同程度的響應。
[1] Czyzewicz N, Yue K, Beeckman T, De SMET I. Message in a bottle: Small signalling peptide outputs during growth and development., 2013, 64(17): 5281-5296.
[2] 周兆禧, 杜中軍, 陳業淵, 趙家桔, 王文林. 多肽在農作物生長發育中的作用研究進展. 廣東農業科學, 2008(11): 145-147.
Zhou Z X, Du Z J, Chen Y Y, Zhao J J, Wang W L. The role of polypeptides in crop growth and development., 2008(11): 145-147. (in Chinese)
[3] Rowe M H, Bergmann D C. Complex signals for simple cells: The expanding ranks of signals and receptors guiding stomatal development., 2010, 13(5): 548-555.
[4] Butenko M A, Vie A K, Brembu T, Aalen R B, Bones A M. Plant peptides in signalling: Looking for new partners., 2009, 14(5): 255-263.
[5] Ryan C A, Pearce G, SCHEER J, MOURA D S. Polypeptide hormones., 2002, 14(Suppl.): S251-S264.
[6] Mohd-Radzman N A, Binos S, Truong T T, Imin N, Mariani M, Djordjevic M A. Novel MtCEP1 peptides produceddifferentially regulate root development in., 2015, 66(17): 5289-5300.
[7] Katsir L, Davies K A, Bergmann D C, Laux T. Peptide signaling in plant development., 2011, 21(9): R356-R364.
[8] 熊國勝, 李家洋, 王永紅. 植物激素調控研究進展. 科學通報, 2009, 54(18): 2718-2733.
Xiong G S, Li J Y, Wang Y H. Advances in the regulation and crosstalks of phytohomones., 2009, 54(18): 2718-2733. (in Chinese)
[9] Malamy J E, Benfey P N. Organization and cell differentiation in lateral roots of., 1997, 124(1): 33-44.
[10] 隋志鵬. 水稻和玉米多肽激素基因CEP家族的鑒定及功能分析[D]. 北京: 中國農業大學, 2016.
Sui Z P. Identification and functional analysis of CEP peptide gene family in rice () and maize (L.)[D]. Beijing: China Agricultural University, 2016. (in Chinese)
[11] Kondo T, Nakamura T, YOKOMINE K, Sakagami Y. Dual assay for MCLV3 activity reveals structure-activity relationship of CLE peptides., 2008, 377(1): 312-316.
[12] Fiers M, Golemiec E, Schors R V D, Geest L V D, Li K W, Stiekema W J, Liu C M. The CLAVATA3/ESR motif of Clavata3 is functionally independent from the nonconserved flanking sequences., 2006, 141(4): 1284-1292.
[13] Matsuzaki Y, Ogawa-ohnishi M, Mori A, MatsubayashiY. Secreted peptide signals required for maintenance of root stem cell niche in., 2010, 329(5995): 1065-1067.
[14] Kondo T, Sawa S, Kinoshita A, Mizuno S, Kakimoto T, Fukuda H, Sakagami Y. A plant peptide encoded byidentified byMaldi-Tof Ms analysis., 2006, 313(5788): 845-848.
[15] Whitford R, Fernandez A, De Groodt R, Ortega E, Hilson P. Plant CLE peptides from two distinct functional classes synergistically induce division of vascular cells., 2008, 105(47): 18625-18630.
[16] Ohyama K, Ogawa M, Matsubayashi Y. Identification of a biologically active, small, secreted peptide inbygene screening, followed by Lc-Ms-based structure analysis., 2008, 55(1): 152-160.
[17] Ogilvie H A, Imin N, Djordjevic M A. Diversification of the C-terminally encoded peptide (CEP) gene family in angiosperms, and evolution of plant-family specific CEP genes., 2014, 15: 870.
[18] Delay C, Imin N, Djordjevic M A.genes regulate root and shoot development in response to environmental cues and are specific to seed plants., 2013, 64(17): 5383-5394.
[19] Roberts I, Smith S, DE Rybel B, Van Den Broeke J, Smet W, De Cokere S, Mispelaere M, De Smet I, Beeckman T. The CEP family in land plants: Evolutionary analyses, expression studies, and role inshoot development., 2013, 64(17): 5371-5381.
[20] Roberts I, Smith S, Stes E, DE Rybel B, Staes A, Van de Cotte B, Njo M F, Dedeyne L, Demol H, Lavenus J, Audenaert D, Gevaert K, Beeckman T, De Smet I. CEP5 and XIP1/CEPR1 regulate lateral root initiation in., 2016, 67(16): 4889-4899.
[21] Taleski M, Imin N, Djordjevic M A. New role for a CEP peptide and its receptor: Complex control of lateral roots., 2016, 67(16): 4797-4799.
[22] Imin N, Mohd-radzman N A, Ogilvie H A, Djordjevic M A. The peptide-encodinggene modulates lateral root and nodule numbers in., 2013, 64(17): 5395-5409.
[23] Winter D, Vinegar B, Nahal H, Ammar R, Wilson G V, Provart N J. An “electronic fluorescent pictograph” browser for exploring and analyzing large-scale biological data sets., 2007, 2(8): e718.
[24] Hruz T, Laule O, Szabo G,Wessendorp F, Bleuler S, Oertle L, Widmayer P, Gruissem W, Zimmermann P. Genevestigator V3: A reference expression database for the Meta- analysis of transcriptomes., 2008, 2008: 420747.
[25] Ishimaru K, Hirotsu N, Madoka Y, Murakami N, Hara N, Onodera H, Kashiwagi T, Ujiie K, Shimizu B, Onishi A, Miyagawa H, Katoh E. Loss of function of the Iaa-glucose hydrolase geneenhances rice grain weight and increases yield., 2013, 45(6): 707-711.
[26] Xie R, Li Y, He S, Zheng Y, Yi S, Lv Q, Deng L. Genome-wide analysis of citrusgenes and their spatiotemporal expression under stresses and hormone treatments., 2014, 9(12): e113971.
[27] Aguilar-rangel M R, Montes R A C, González- segovia E, Ross-ibarra J, Simpson J K, Sawers R J H. Allele specific expression analysis identifies regulatory variation associated with stress-related genes in the Mexican highland maize landrace Palomero Toluque?o., 2017, 5(6): e3737.
[28] Artimo P, Jonnalagedda M, Arnold K, Baratin D,Csardi G,Castro E D, Duvaud S,Flegel V, Fortier A, Gasteiger E, Grosdidier A, Hernandez C, Ioannidis V, Kuznetsov D, Liechti R, Moretti S, Mostaguir K, Redaschi N, Rossier G, Xenarios I, Stockinger H. Expasy: SIB bioinformatics resource portal., 2012, 40 (Web Server issue): W597-W603.
[29] Kyte J, Doolittle R F. A simple method for displaying the hydropathic character of a protein., 1982, 157(1): 105-132.
[30] Murphy E, Smith S, DE Smet I. Small signaling peptides indevelopment: How cells communicate over a short distance., 2012, 24(8): 3198-3217.
[31] Pearce G, Strydom D, Johnson S, Ryan C A. A polypeptide from tomato leaves induces wound-inducible proteinase inhibitor proteins., 1991, 253(5022): 895-897.
[32] Tabata R, Sumida K, Yoshii T,Ohyama K,Shinohara H,Matsubayashi Y. Perception of root-derived peptides by shoot Lrr-Rks mediates systemic N-demand signaling., 2014, 346(6207): 343-346.
[33] 李睿, 李浩浩, 安建平, 由春香, 王小非, 郝玉金. 蘋果多肽激素及其編碼基因調控擬南芥根系發育. 園藝學報, 2017, 44(7): 1225-1234.
Li R, Li H H, An J P, You C X, Wang X F, Hao Y J.Apple peptide hormone and its coding gene MdCEP1regulate root development in Arabidopsis thaliana. Acta Horticulturae Sinica, 2017, 44(7): 1225-1234. (in Chinese)
(責任編輯 岳梅)
Identification of citrus CitCEP genes and their transcriptional response to stress and hormone treatments
Pan Xiaoting, Zhang Jing, Ge Ting, Ma Yanyan, Deng Lie, He Shaolan, Yi Shilai, Zheng Yongqiang, lü Qiang, Xie Rangjin
(Citrus Research Institute, Southwest University-Chinese Academy of Agricultural Sciences/National Engineering Research Center for Citrus Technology, chongqing 400712)
【Objective】 The objectives of this study are to identify citrus CEP genes () based on citrus whole genome sequences, understand the phylogenetic relationship among CitCEPs as well as their expression specificity in different tissues and response to hormone and abiotic stress, and to lay a foundation for further study on the biological function of.【Method】The citrus CEP genes were identified by BLASTp based on Phytozome database. Gene structure, phylogenetic tree, relative molecular mass, isoelectric point and other physical and chemical properties of CitCEP members were analyzed using GSDS, ProtScale Tool, EXPASY, CLUSTALX, MEGA6.0, plant CARE and Cello. Real-time quantitative PCR (qRT-PCR) was used to detect the expression levels ofunder different treatments of ‘Ziyang’.【Result】theconsists of 11 members, of whichandcontain 1 intron, and the other members have no intron. All CitCEPs contain conserved domain sequences (SPGV/IGH), and belong to hydrophilic protein. The magnitude of the most hydrophilic amino acid is 3.711 and that of the weakest hydrophilic amino acid is -2.778. Subcellular localization prediction showed that 11 CitCEPs were located in different positions of the cell. Among them, CitCEP1, CitCEP2, CitCEP4, CitCEP5 CitCEP6 and CitCEP8 were all located in extracellular. CitCEP3 and CitCEP7 existed in both extracellular and mitochondria. CitCEP9 and CitCEP10 were located in nucleus and plasma membrane, respectively. CitCEP11 was located in both mitochondria and nucleus. The phylogenetic analysis showed that the CitCEP members together withCEPs were clustered into 3 groups, which indicated that the CitCEP members had different biological functions. Expression analysis showed thatwas mainly expressed in stem, leaf and cotyledon,was mainly expressed in root and cotyledon, whileandwere mainly expressed in pericarp, and the other members expressed very low or no expression in the above tissues, reflecting the difference in tissue specificity among different members. Under drought conditions, the expression ofwas down-regulated, while that of,andwas gradually increased. The expression ofandunder salt stress was similar to that under drought conditions, respectively, while the expression ofandincreased first and then decreased. The expression ofwas significantly inhibited under ethylene (ETH) and abscisic acid (ABA) treatments, but the expressionincreased firstly and then decreased undermethyl jasmonate (MeJA), salicylic acid (SA), indole acetic acid (IAA) and gibberellin (GA3) treatments.showed a similar trend under the treatments of 6-benzylamino adenine (6-BA), IAA, GA3and ABA.The expression ofwas significantly up-regulated under ETHtreatment, and decreased firstly and then increased under 6-BA, SAand ABA treatments, but there was no obvious regularity in the treatment of MeJA, IAA, GA3. The expression ofshowed a downward trend under the treatments of ETH, SA, MeJA, IAA, GA3and ABA, which was similar to that ofunder ETH and SA treatments. Under 6-BA treatment, theexpression ofshowed a trend of decreasing firstly and then increasing.【Conclusion】A total of 11members were identified from citrus whole genome sequences, and all of them are hydrophilic proteins which contain a conserved SPGV/IGH domain. The subcellular positions were varied among CitCEP members. Under different stress and hormonal treatments,,,andexhibited different degrees of response, while the response of the other member was not obvious or unresponsive. It is inferred that,,andmay play an important role in citrus growth and development as well as stress response.
citrus; CitCEP genes; gene expression; hormone; stress
2018-02-06;
2018-04-02
“十二五”國家科技計劃(2014BAD16B02-1)、中央高校基本科研業務費專項資金(XDJK2016B022)
潘小婷,Tel:023-68349726;E-mail:panxiaoting-sunny@foxmail.com。
謝讓金,Tel:023-68349726;E-mail:xierangjin@cric.cn
10.3864/j.issn.0578-1752.2018.16.010
