菜粉蝶HSP20基因的克隆及溫度脅迫下表達分析
袁遠 張連根 高熹
摘要:【目的】克隆分析菜粉蝶(Pieris rapae)熱激蛋白20基因(HSP20),并檢測其在溫度脅迫下的表達情況,為探析菜粉蝶適應溫度脅迫機制提供參考。【方法】以菜粉蝶為試驗材料,采用RT-PCR和RACE克隆獲得菜粉蝶HSP20基因cDNA全長,并通過生物學軟件分析其序列,明確其基因結構特征和親緣關系;采用實時熒光定量PCR檢測菜粉蝶HSP20基因在低溫(-20~0 ℃)和高溫(5~45 ℃)脅迫下的表達響應。【結果】菜粉蝶HSP20基因全長725 bp,5'-端非編碼區105 bp,3'-端非編碼區109 bp,開放閱讀框531 bp,編碼176個氨基酸,預測蛋白分子量19.5 kD,理論等電點6.61;其編碼氨基酸序列中含有1個典型的HSP20家族結構域(位于第60~142位氨基酸)、1個α-晶體蛋白(位于第60~157位氨基酸)和1個Allato allatostatin(位于第14~24位氨基酸);基序列相似度分析其與二化螟、家蠶和甘藍夜蛾HSP20蛋白氨基酸相似度較高并聚為一族。該基因隨環境溫度的升高或降低發生變化,在-5和40 ℃誘發該基因高效表達。【結論】低溫和高溫均能誘導菜粉蝶HSP20基因高效表達。
關鍵詞: 菜粉蝶;熱激蛋白;HSP20;基因克隆;序列分析;溫度脅迫;表達分析
中圖分類號: S433.4? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2019)02-0286-06
Abstract:【Objective】Heat shock protein 20 gene(HSP20) from Pieris rapae was cloned and its expression under temperature stress was detected. The result was helpful for further investigation of the adaptation mechanisms of P. rapae to high and low temperature stresses. 【Method】Full length cDNA of HSP20 gene of P. rapae was cloned by RT-PCR and RACE with P. rapae as materials. Its sequence was analyzed by biology software, and its gene structural sequence and genetic relationship were also studied. Response of HSP20 gene to low(-20-0 ℃) and high (5-45 ℃) temperatures were ana-lyzed by fluorescence quantitative PCR. 【Result】The full-length of P. rapae HSP20 was 725 bp, and contained 105 bp 5'-noncoding region, 109 bp 3'-noncoding region, a 531 bp open reading frame(ORF),encoded 176 amino acids, with an estimated molecular mass of 19.5 kD and an theoretical isoelectric point(pI) of 6.61. In the encoded amino acid sequences, there was one typical HSP20 family domain(located at 60-142 amino acids), one α-crystallin(located at 60-157 amino acids) and a Allato allatostatin(located at 14-24 amino acids). The sequence similarities between P. rapae HSP20 and that of? Chilo suppressalis, Bombyx mori, Mamestra brassicae were high and they were clustered into one group. The HSP20 expressions changed with temperature, and high expressions of this gene was observed at -5 and 40 ℃. 【Conclusion】Low and high temperatures can lead to the high expression of P. rapae HSP20.
Key words: Pieris rapae; heat shock protein; HSP20; gene cloning; sequence analysis; temperature stress; expression analysis
0 引言
【研究意義】熱激蛋白(Heat shock protein,HSP)又稱應激蛋白或熱休克蛋白,是生物有機體遇到高溫等逆境刺激后大量產生的一類特定的應急蛋白,是生物體對逆境脅迫適應的重要組成成分,其普遍參與生物的各種生理代謝途徑,對減輕逆境脅迫對自身的傷害起重要作用。HSP普遍存在于各種生物體內,對生物個體的生長、發育和生存發揮著重要作用(韓政等,2010;代玲玲等,2011;徐劍文等,2017;張響英等,2017)。隨著全球氣溫的持續升高,高溫脅迫對昆蟲及其他生物的影響也越來越大,通過研究熱激蛋白調控細胞使昆蟲適應外界脅迫的機制,對揭示生物應對全球溫室效應具有重要意義。【前人研究進展】目前,對于HSP尚無明確的分類標準,普遍認同Morimoto的劃分方法,他將主要的HSP分為4個家族:HSP90(83~90 kD)、HSP70(66~78 KD)、HSP60和小分子HSP(15~30 kD)家族(Lindquist,1986)。近年來,已有系列文獻報道各種昆蟲HSP基因的克隆、表達分析及其功能鑒定研究,但這些研究主要集中在熱休克蛋白家族的HSP70和HSP90,而針對小分子熱休克蛋白的研究相對較少。HSP20是生物體應激反應誘導產生的主要蛋白質之一。多數應激反應使蛋白質結構受損,HSP20能夠結合損傷的蛋白質,并預防胞漿內的蛋白質發生聚集,同時能遷移到核仁中,與細胞內的核糖體建立聯系,達到保護細胞核蛋白質的作用。不同物種的HSP20基因具有高度保守的氨基酸序列,該特征亦為生物進化研究提供了一定的科學依據(Mehlen et al.,1996)。目前,已報道在家蠶(Bombyx mori)、小菜蛾(Plutella xylostella)、斜紋夜蛾(Spodoptera litura)和二化螟(Chilo suppressalis)等昆蟲中分離克隆出許多小分子HSP基因,這些基因在昆蟲生長發育、生殖和應對環境脅迫中發揮重要作用(Li et al.,2009;Shen et al.,2011;夏曉峰等,2013;張青等,2014;Liu et al.,2018)。【本研究切入點】菜粉蝶(Pieris rapae)隸屬鱗翅目(Lepioloptera)粉蝶科(Pierididae),分布于世界各地,主要危害甘藍和白菜等十字花科植物。菜粉蝶對環境的適應能力極強,無論是在炎熱的夏季還是寒冷的冬季均能生存。目前,有關菜粉蝶HSP20基因的研究鮮見報道。【擬解決的關鍵問題】采用RACE-PCR從菜粉蝶中克隆HSP20基因,運用生物信息學方法對獲得的菜粉蝶HSP20基因序列特征進行分析,同時采用實時熒光定量PCR檢測在不同溫度下該基因的表達情況,為深入揭示菜粉蝶對溫度脅迫適應機制提供參考。
1 材料與方法
1. 1 試驗材料
供試菜粉蝶幼蟲采集于云南農業大學后山農場甘藍蔬菜地。主要試劑和藥品:TRIzol試劑購自Invitrogen公司,Advantage 2 PCR Kit(Clontech)和Premix Ex TaqTM購自TaKaRa公司,pGEM?-T載體購自Promega公司,Taqman-MGB探針購自上海GeneCore生物技術公司。
1. 2 RNA抽提
采用TRIzol試劑提取菜粉蝶總RNA,分別用紫外分光光度計和甲醛變性瓊脂糖凝膠電泳檢測其質量(Tachibana et al.,2005)。
1. 3 HSP20基因克隆
以提取的菜粉蝶總RNA為模板,用Smarttm RACE cDNA Amplification Kit(Clontech)合成3'-和5'-RACE cDNA模板。根據對菜粉蝶cDNA文庫測序得到HSP20基因片段序列,設計3'-RACE(5'-GGTG AATTTGGATGTCCAGCATTTCACT-3')和5'-RACE(5'-CCTAGGAGCAATGACGGTCAGAACGCCAT-3')引物。以RACE cDNA為模板,擴增菜粉蝶HSP20基因的中間片段序列。擴增程序:94 ℃預變性3 min;94 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 1 min,進行30個循環;72 ℃延伸10 min,16 ℃保存。
根據擴增獲得的菜粉蝶HSP20基因中間片段序列,設計其3'-RACE和5'-RACE引物,結合RACE試劑盒中的UPM引物(Universal Primer A Mix,UPM),PCR擴增獲得該基因的3'-和5'-端序列。擴增程序:94 ℃預變性3 min;94 ℃ 30 s,64 ℃ 30 s,72 ℃ 1 min,進行30個循環;72 ℃延伸10 min,16 ℃保存。采用Advantage 2 PCR Kit(Clontech)進行PCR。
PCR產物經1%瓊脂凝膠電泳檢測,切膠并用AxyAprep DNA凝膠回收試劑盒(Biosciences)回收,將回收產物連接到pGEM?-T載體,轉化大腸桿菌DH5α感受態細胞后進行藍白斑篩選(Sonoda et al.,2006a)。將得到的陽性克隆菌液送金思特科技(南京)有限公司測序。
1. 4 基因序列分析
用Genetyx Ver. 8.0分析菜粉蝶HSP20基因核序列酸,推導氨基酸序列。用ClustalX(v1.83)程序進行多序列對比,用Motif scan對該序列中的蛋白質二級結構域進行預測(Rinehart et al.,2000)。
1. 5 溫度脅迫下HSP20基因表達分析
將菜粉蝶蟲體分別置于低溫(-20、-15、-5和0 ℃)和高溫(5、25、30、35、40和45 ℃)下1 h,每個溫度值作為一個處理,其中室溫25 ℃作為對照。合成的菜粉蝶cDNA作實時熒光定量PCR檢測模板。用Primer 5.0設計定量引物(5'-ACTCGTGACCTTG GCTCCA-3'和5'-CCCTCTACCACGATGTATC-3'),以18S RNA為內參(5'-CAGTGATGGGATGAGTGC TTT-3',R:5'-TACGCTATTGGAGCTGGAATT-3'),參照SYBR Premix Ex TaqTM II(Tli RNaseH Plus)說明進行定量擴增。擴增程序:95 ℃預變性10 s;95 ℃ 5 s,60 ℃ 34 s,72 ℃ 30 s,進行40個循環。所有樣品進行3次生物學重復。實時熒光定量PCR數據用 2-△△Ct方法進行分析(Livak and Schmittgen, 2001),基因表達量差異采用SPSS 20.0中的最小顯著差法(LSD)檢驗,顯著性水平P<0.05。
2 結果與分析
2. 1 菜粉蝶HSP20基因序列分析結果
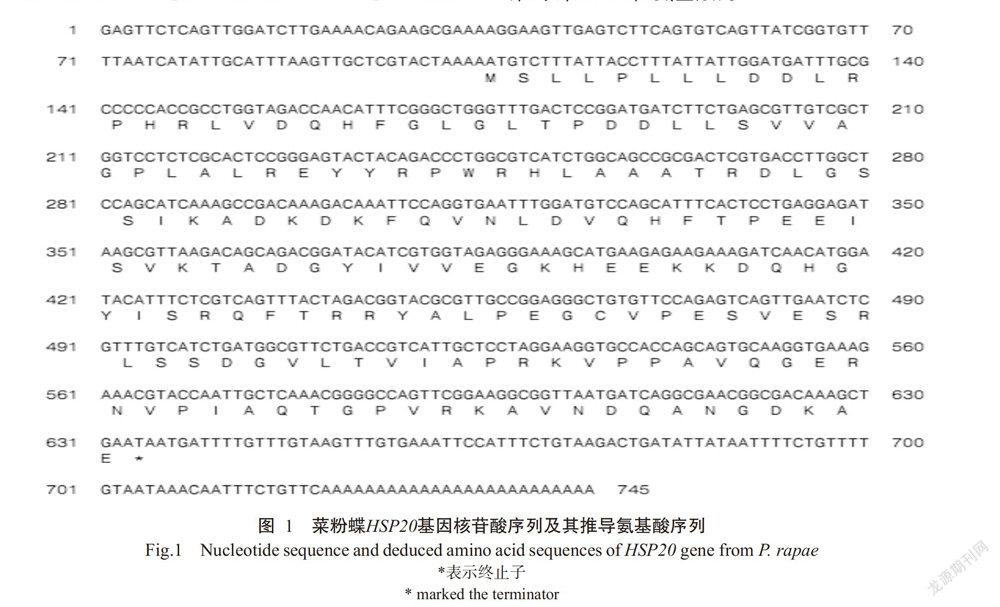
圖1結果顯示,菜粉蝶HSP20基因全長725 bp,5'-端非編碼區105 bp,3'-端非編碼區109 bp,開放閱讀框(Open reading frame,ORF)531 bp,編碼176個氨基酸,預測蛋白分子量19.5 kD,理論等電點(pI)6.61。該基因具有多聚腺苷酸信號(AATAAA),出現在核苷酸序列終止密碼子下游68 bp處,且有ploy(A)尾巴。
通過Motif sacn分析(Sonoda et al.,2006b)發現,該蛋白中存在3個蛋白激酶C磷酸化位點(SIK60~62、SVK83~85和TRR112~114)、2個酪蛋白激酶Ⅱ磷酸化位點(TPDD26~29和TPEE78~81)、1個典型的HSP20家族結構域(位于第60~142位氨基酸)、1個a-晶體蛋白(位于第60~157位氨基酸)和1個Allato Allatostatin(位于第14~24位氨基酸)。
2. 2 菜粉蝶HSP20蛋白同源性分析結果
菜粉蝶與其他昆蟲的HSP20蛋白氨基酸多序列比對結果見圖2。菜粉蝶與其他昆蟲HSP20蛋白的氨基酸相似度比較結果(表1)顯示,菜粉蝶與二化螟(C. suppressalis)、家蠶(B. mori)、甘藍夜蛾(Mamestra brassicae)、云杉卷葉蛾(Choristoneura fumiferana)、小菜蛾(P. xylostella)、東亞飛蝗(Locusta migratoria)、赤擬谷盜(Tribolium castaneum)、腰帶長體繭蜂(Macrocentrus cingulum)和美洲斑潛蠅(Liriomyza sativae)的HSP20蛋白氨基酸相似度分別為85%、84%、84%、72%、57%、48%、51%、46%和43%。
應用MEGA 4.0鄰接法(NJ)對菜粉蝶和其他昆蟲的HSP20蛋白氨基酸序列進行分析,并構建系統發育進化樹,結果(圖3)顯示,菜粉蝶HSP20蛋白氨基酸序列以95%的支持率與家蠶、二化螟和甘藍夜蛾聚為一族。
2. 3 溫度脅迫下菜粉蝶HSP20基因的表達分析結果
通過實時熒光定量PCR分析菜粉蝶HSP20基因在低溫和高溫脅迫下的表達情況,結果(圖4)顯示,低溫條件下菜粉蝶HSP20基因的表達量明顯升高,在-5 ℃時達峰值,是對照組的1.76倍;高溫條件下,在40 ℃時表達量明顯升高,是對照組的2.75倍,差異達顯著水平(P<0.05)。表明低溫和高溫脅迫均誘導菜粉蝶體內HSP20基因的高效表達。
3 討論
HSP普遍存在于自然界的原核生物和真核生物中,其進化上相對保守。適宜條件下,其多數是作為分子伴侶幫助蛋白質正確折疊、移位、維持特定構象或參與蛋白質降解(任莉萍和曹小漢, 2006)。當機體受到外界環境刺激時,HSP基因的表達量常會大幅度提升,參與生物的細胞結構維持、更新、修復和免疫等環節,以提高生物體抵抗外來刺激的能力。鑒于HSP的免疫保護作用對生物有重要意義,因此一直是研究的熱點(李斌,2001;申建茹等,2011;張青等,2014)。目前,已有系列文獻報道各種昆蟲HSP基因克隆、表達分析及初步功能鑒定研究(Multhoff,2007),但這些研究主要集中在HSP家族的HSP70和HSP90,而對小分子HSP的研究相對較少,已報道的僅有果蠅、家蠶和斜紋夜蛾等(寇利花等,2015)。隨著測序技術的不斷發展及測序成本的降低,將鑒定出越來越多的昆蟲小分子HSP基因序列,而這些序列的鑒定將揭示該類蛋白在昆蟲中的進化過程,為昆蟲的系統進化研究提供新視野,同時為后續研究昆蟲小分子HSP的功能提供必要基礎。
本研究以其他昆蟲HSP20基因保守區設計簡并引物擴增菜粉蝶HSP20基因的中間片段,然后利用RACE技術獲得全長cDNA。克隆得到的菜粉蝶HSP20基因結構分析結果表明,菜粉蝶HSP20基因全長725 bp,5'-端非編碼區105 bp,3'-端非編碼區109 bp,開放性閱讀框531 bp,編碼176個氨基酸,預測蛋白分子量19.5 kD,理論等電點6.61。經結構域預測分析發現,菜粉蝶HSP20蛋白中具有1個典型的HSP20家族結構域,可判斷其屬于HSP20家族成員。此外,Motif scan分析發現該菜粉蝶HSP20基因存在3個蛋白激酶C磷酸化位點和2個酪蛋白激酶Ⅱ磷酸化位點,而蛋白質的磷酸化和脫磷酸化在細胞信號轉導中起主導作用,推測這幾個位點可能是HSP20基因完成其生理功能的結構基礎;同源性分析結果顯示,菜粉蝶HSP20蛋白以95%的支持率與家蠶、二化螟和甘藍夜蛾聚為一族,說明HSP20基因在進化中具有高度的保守性;通過氨基酸序列比對得知,該基因5'-端在物種進化過程中保守性低。這些特征均與迄今已克隆獲得的其他昆蟲HSP20基因結構特征相似(Mehlen et al.,1996;Wang et al.,2012)。通過實時熒光定量PCR分析菜粉蝶HSP20基因在低溫脅迫下的表達量發現,隨著溫度的下降表達量明顯升高,在-5 ℃時菜粉蝶HSP20基因的表達量明顯升高至峰值,隨后開始降低;高溫脅迫下,隨著溫度的升高表達量明顯提高,在40 ℃時菜粉蝶HSP20基因的轉錄水平達峰值,隨后開始降低。菜粉蝶HSP20基因的克隆分析為后續該蟲的熱激機制及對溫度的脅迫適應機制研究提供分子生物學依據,并為進一步開展相關后續研究打下理論基礎。
4 結論
菜粉蝶HSP20基因具有1個典型的HSP20家族結構域,存在3個蛋白激酶C磷酸化位點和2個酪蛋白激酶Ⅱ磷酸化位點,與家蠶、二化螟和甘藍夜蛾的HSP20基因有較高的同源性。環境溫度脅迫能誘導HSP20基因大量表達。
參考文獻:
代玲玲,彭德良,黃文坤,彭煥,龍海波,王高峰. 2011. 松材線蟲及其近緣種熱激蛋白90基因(hsp90)克隆與序列分析[J]. 農業生物技術學報,19(5): 916-923. [Dai L L, Peng D L,Huang W K,Peng H,Long H B,Wang G F. 2011. Cloning and sequence analysis of three new heat shock protein 90 gene(hsp90) from Bursaphelenchus xylophilus,B. mucronatus and B. doui[J]. Journal of agricultural Biotechnology,19(5): 916-923.]
韓政,趙龍龍,陳光,馬瑞燕. 2010. 熱激蛋白與昆蟲的耐熱性關系研究進展[J]. 山西農業科學,38(8):92-94. [Han Z,Zhao L L,Chen G,Ma R Y. 2010. Research advances of the relationship between heat shock proteins and Insects heat tolerance[J]. Journal of Shanxi Agricultural Sciences,38(8): 92-94.]
寇利花, 吳海花, 劉耀明, 張育平, 張建珍, 郭亞平, 馬恩波. 2015. 鎘急性染毒對中華稻蝗精卵巢小分子熱休克蛋白基因表達的影響[J]. 農業環境科學學報,34(1): 7-14. [Kou L H,Wu H H,Liu Y M,Zhang Y P,Zhang J Z,Guo Y P,Ma E B. 2015. Expression analysis of OcsHSP genes in testis and ovary of Oxya chinensis exposed to acute cadmium[J]. Journal of Agro-Environment Science, 34(1): 7-14.]
李斌. 2001. 家蠶分子連鎖圖譜的構建和低分子量熱激蛋白基因的克隆與特異表達[D]. 重慶:西南農業大學學報.[Li B. 2001. Constructin of the molecular linkage map and isolation,identification and specific expression of small heat-shock genes in silkworm, Bombyx mori[D].Chonɡqing:Southwest Agricultural University.]
任莉萍,曹小漢. 2006. 熱休克蛋白研究進展[J]. 安徽農業科學, 34(10): 2040-2042. [Ren L P,Cao X H. 2006. Study on heat shock protein[J]. Journal of Anhui Agricultural Sciences, 34(10):2040-2042.]
申建茹, 李明福, 陳乃中, 王進軍,萬方浩. 2011. 蘋果蠹蛾熱激蛋白Hsp90基因的克隆及熱脅迫下的表達分析[J]. 昆蟲學報, 54(11):1236-1248. [Shen J R,Li M F,Chen N Z,Wang J J,Wan F H. 2011. Cloning and analysis of the expression characteristics under heat stress of Hsp90 gene in the codling moth,Cydia pomonella(Lepidoptera: Tortricidae)[J]. Acta Entomologica Sinica,54(11):1236-1248.]
夏曉峰, 林海蘭, 鄭丹丹, 楊廣,尤民生. 2013. 小菜蛾熱休克蛋白基因的鑒定及其表達模式分析[J]. 昆蟲學報, 56(5):457-464. [Xia X F,Lin H L,Zhen D D,Yang G,You M S. 2013. Identification and expression patterns of heat shock protein genes in the diamondback moth,Plute-lla xylostella(Lepidoptera:Yponomeutidae)[J]. Acta Entomologica Sinica,56(5):457-464.]
徐劍文,趙君,劉劍光,肖松華. 2017. 芝麻全基因組Hsp70基因的鑒定與生物信息學分析[J]. 江蘇農業學報,33(3):493-502. [Xu J W,Zhao J,Liu J G,Xiao S H. 2017. Genome-wide identification and bioinformatics analysis of Hsp70 gene family in Sesamum indicum[J]. Jiangsu Journal of Agricultural Sciences,33(3):493-502.]
張青, 陸明星, 祝樹德. 2014. 灰飛虱熱激蛋白Hsp90基因的克隆、序列分析與表達模式[J]. 昆蟲學報, 57(7):777-786. [Zhang Q,Lu M X,Zhu S D. 2014. Cloning, sequence analysis and expression profiling of heat shock protein 90 gene in the small brown plant hopper, Laodelphax striatellus(Hemiptera: Delphacidae)[J]. Acta Entomologica Sinica, 57(7):777-786.]
張響英,唐現文,陳靜波,李騰騰. 2017. 熱應激誘導奶牛乳腺上皮細胞凋亡及其對HSP70基因表達的影響[J]. 河南農業科學,46(8):142-146. [Zhang X Y,Tang X W,Chen J B,Li T T. 2017. Effects of heat stress on apoptosis and expression of HSP70 mRNA in bovine mammary epithelial cells[J]. Journal of Henan Agricultural Sciences,46(8):142-146.]
Li Z W,Li X,Yu Q Y. 2009. The small heat shock protein (sHSP)genes in silkworm,Bombyx mori and comparative analysis withother insect sHSP genes[J]. BMC Evolutionary Biology,9:215.
Lindquist S. 1986. The heat-shock response[J]. Annual Review of Biochemistry, 55(1):1151-1191.
Liu Q N,Liu Y,Zhao Z X,Zhu X Y,Ge B M,Li C F,Wang D,Bian X G,Yang L,Chen L,Tian J W,Zhou C L, Tang B P. 2018. A small heat shock protein 21(sHSP21) mediates immune responses in Chinese oak silkworm Antheraea pernyi[J]. International Journal of Biological Macromolecules,111:1027-1031.
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ct method[J]. Methods,25:402-408.
Mehlen P,Schulze-Osthoff K,Arrigo A P. 1996. Small stress proteins as novel regulators of apoptosis. Heat shock protein 27 blocks Fas/APO-1-and staurosporine-induced cell death[J]. Journal of Biological Chemistry,271(28):16510-16514.
Multhoff G. 2007. Heat shock protein 70(Hsp70):Membrane Iloca-tion,export and immunological relevance[J].Me-thods,43: 229-237.
Rinehart J P, Yocum G D, Denlinger D L. 2000. Developmental upregulation of inducible hsp70 transcripts, but not the cognate form, during pupal diapause in the flesh fly, Ssarcophaga crassipalpis[J]. Insect Biochemistry & Molecular Biology, 30(6):515-21.
Shen Y,Gu J,Huang L H,Zheng S C,Liu L,Xu W H,Feng Q L,Kang L. 2011. Cloning and expression analysis of six small heat shock protein genes in the common cutworm, Spodoptera litura[J]. Journal of Insect Physiology,57(7):908-914.
Sonoda S,Ashfaq M,Tsumuki H. 2006a. Cloning and nucleotide sequencing of three heat shock protein genes(hsp90,hsc70,and hsp19.5) from the diamondback moth, Plutella xylostella(L.) and their expression in relation to deve-lopmental stage and temperature[J]. Archives of Insect Biochemistry & Physiology, 62(2):80-90.
Sonoda S,Fukumoto K,Izumi Y,Ashfaq M,Yoshida H,Tsumuki H. 2006b. A small HSP gene is not responsible for diapause and cold tolerance acquisition in Chilo supp-ressalis[J]. Journal of Applied Entomology,130(5): 309-313.
Tachibana S,Numata H,Goto S G. 2005. Gene expression of heat-shock proteins(Hsp23,Hsp70 and Hsp90) during and after larval diapause in the blow fly Lucilia sericata[J]. Journal of Insect Physiology, 51(6):641-647.
Wang H, Li K, Zhu J Y, Fang Q, Ye G Y. 2012. Cloning and expression pattern of heat shock protein genes from the endoparasitoid wasp, pteromalus puparum in response to environmental stresses[J]. Archives of Insect Bioche-mistry and Physiology,79(4-5):247-263.
(責任編輯 麻小燕)

