桑樹NBS.LRR類基因家族的全基因組鑒定及其調(diào)控microRNAs分析
劉潮 褚洪龍 韓利紅 楊云錦 高永 唐利洲



摘要:利用生物信息學(xué)方法,全面分析了桑樹NBS-LRR類基因家族組成、結(jié)構(gòu)、進(jìn)化、組織表達(dá),并對該家族基因的調(diào)控microRNA( miRNA)進(jìn)行了預(yù)測。共篩選到112個桑樹NBS-LRR類基因,根據(jù)功能域主要分為NBS、CC-NBS、CC-NBS-LRR、NBS-LRR 4種類型。內(nèi)含子數(shù)和相位分析結(jié)果顯示,基因結(jié)構(gòu)類型多樣。聚類分析結(jié)果顯示不同聚類組間存在較多的類型交叉現(xiàn)象。該家族基因存在組織表達(dá)特異性。大部分桑樹NBS-LRR類基因均具有被miRNAs調(diào)控的可能性,miR472b和miR482b在調(diào)控該家族基因表達(dá)中可能發(fā)揮了主要作用。桑樹NBS-LRR家族基因結(jié)構(gòu)和進(jìn)化的復(fù)雜性決定了其功能的多樣性,miRNA在控制該家族基因表達(dá)的適應(yīng)性成本中發(fā)揮作用。
關(guān)鍵詞: 桑樹;NBS-LRR;生物信息學(xué);miRNA
中圖分類號: S792.99
文獻(xiàn)標(biāo)識碼:A
文章編號: 1000-4440( 2019) 03-0544-10
植物在與病原的長期協(xié)同進(jìn)化過程中,發(fā)展出了一整套高度復(fù)雜的免疫系統(tǒng),以識別并對抗病原的侵襲。植物先天免疫系統(tǒng)包括2層防御系統(tǒng):第1層是植物通過細(xì)胞表面的模式識別受體( Patternrecognition receptors,PRRs)對保守的病原/微生物相關(guān)分子模式( Pathogen/microbe associated molecularpattern,P/MAMP)的識別,稱為病原相關(guān)分子模式引發(fā)的免疫(PAMP-triggered immunity,PTI)[1-3];第2層的免疫一般發(fā)生在細(xì)胞內(nèi)部,主要依靠抗性基因( Re-sistance gene)編碼的多態(tài)性抗病蛋白識別無毒因子而實(shí)現(xiàn),這些蛋白質(zhì)多數(shù)是胞內(nèi)富含亮氨酸重復(fù)序列和核苷酸結(jié)合位點(diǎn)蛋白( Nucleotide binding site andleucine-rich repeat proteins, NBS-LRR proteins),能直接或間接識別病原特異性效應(yīng)蛋白,而激發(fā)相似的防御反應(yīng),稱為效應(yīng)子引發(fā)的免疫(Effector-triggeredimmunity,ETI)[1]。與PRRs相比,R蛋白在進(jìn)化上出現(xiàn)相對較晚,在與病原互作中持續(xù)出現(xiàn)了大量新成員。典型的NBS-LRR類基因編碼的蛋白質(zhì)具有一些共同的結(jié)構(gòu)域:位于中心區(qū)的NBS結(jié)構(gòu)域、C端的LRR結(jié)構(gòu)域、N端與Toll/白細(xì)胞介素受體(Toll-inter-leukin-l receptor,TIR)同源的結(jié)構(gòu)域或卷曲螺旋( Coiled coil,CC)結(jié)構(gòu)域。其中中心區(qū)的NBS作為一個信號轉(zhuǎn)換器,通過與ADP或ATP的結(jié)合而改變蛋白質(zhì)的鈍化或激活狀態(tài),使信號關(guān)閉或開放;C端的LRR結(jié)構(gòu)域是植物R蛋白中最具多態(tài)性的部分,與其識別效應(yīng)子的特異性相關(guān)[4];N端的CC或TIR結(jié)構(gòu)域則在下游信號的激活中發(fā)揮關(guān)鍵作用[5-6]。NB-ARC結(jié)構(gòu)域是在細(xì)菌和真核生物中發(fā)現(xiàn)的信號基序,存在于植物抗病基因和動物細(xì)胞凋亡調(diào)節(jié)基因中[7],這一結(jié)構(gòu)域無論是在植物還是動物的NBS-LRR蛋白中都十分保守[4],并成為克隆各種作物抗病基因類似物( Resistance gene analogs,RGAs)的NBS區(qū)域重要的序列依據(jù)。
有研究結(jié)果表明,小RNA在植物的先天免疫中發(fā)揮作用[8]。植物抗病基因存在適應(yīng)性成本,NBS-LRR的高表達(dá)對植物的生長是不利的[9]。MicroRNA是一類21 nt的小分子RNA.miRNA通過轉(zhuǎn)錄或轉(zhuǎn)錄后水平調(diào)控NBS-LRR類基因的表達(dá)[1O-11]。在進(jìn)化上,NBS-LRR類基因與其調(diào)控的miRNA在基因組水平上保持動態(tài)平衡,從而控制了NBS-LRR的潛在適應(yīng)性成本[12]。同一家族miRNA往往有靶向保守區(qū)域,因此一個miRNA可靶向多個NBS-LRR類基因[13-14]。
川桑(Morus notabilis)是桑科桑屬落葉喬木,分布于中國中部和北部各省。桑葉養(yǎng)蠶在中國有著悠久的歷史,是中國絲綢文化的基礎(chǔ)。桑樹木材可用于制作器具,樹皮可作為造紙原料,桑椹可供食用、釀酒。田間栽培中,經(jīng)常出現(xiàn)桑萎縮病、疫病、褐斑病、赤銹病和青枯病等多種病害,往往造成植株發(fā)育不良、桑葉產(chǎn)量降低和品質(zhì)變劣等問題,嚴(yán)重影響蠶桑產(chǎn)業(yè)的健康發(fā)展。目前,隨著川桑基因組數(shù)據(jù)的公布[15],大量桑樹抗病基因的研究結(jié)果先后發(fā)表[16-19],然而關(guān)于桑樹NBS-LRR家族基因的研究鮮見報道。本研究利用生物信息學(xué)方法對桑樹基因組中NBS-LRR類基因進(jìn)行鑒定,并分析該家族基因結(jié)構(gòu)、系統(tǒng)發(fā)育、組織表達(dá)及其調(diào)控miRNA特征,為NBS-LRR類基因功能的揭示和開發(fā)利用提供借鑒。
1 材料與方法
1.1 桑樹NBS-LRR類基因的獲取
以擬南芥NBS-LRR類基因序列為查詢序列,搜索桑樹基因組數(shù)據(jù)庫( https://morus. swu. edu. cn/morusdb/),下載桑樹NBS-LRR類基因序列。
1.2 桑樹NBS-LRR類基因的鑒定與分類
通過GenBank數(shù)據(jù)庫在線工具CDD( https://www. ncbi. nlm. nih. gov/Structure/cdd/wrpsb. cgi)鑒定蛋白質(zhì)結(jié)構(gòu)域。使用Pfam( http://pfam. xfam. org/)和COILS Server(http://www. ch. embnet. org/software/COILS_form. html)對候選NBS-LRR類基因編碼蛋白質(zhì)結(jié)構(gòu)域進(jìn)行確認(rèn),包括NBS、TIR、LRR、CC等,排除不含NBS結(jié)構(gòu)域的基因。根據(jù)蛋白質(zhì)結(jié)構(gòu)域類型,將NBS-LRR類基因進(jìn)行分類。
1.3 桑樹NBS-LRR類基因和編碼蛋白質(zhì)結(jié)構(gòu)分析
所有桑樹NBS-LRR類基因堿基序列和編碼的氨基酸序列均從桑樹基因組數(shù)據(jù)庫下載。使用GSDS在線軟件( http://gsds. cbi. pku. edu. cn/index. php)繪制基因結(jié)構(gòu)示意圖。通過MEME SUITE在線工具(ht-tp://meme-suite. org/tools/meme)預(yù)測桑樹NBS-LRR蛋白質(zhì)氨基酸序列的保守Motif,參數(shù)為默認(rèn)設(shè)置。
1.4 桑樹NBS-LRR蛋白氨基酸序列比對與系統(tǒng)發(fā)育分析
對所有蛋白質(zhì)使用ClustalX進(jìn)行氨基酸序列比對。應(yīng)用MEGA 5.0軟件,采用鄰接法(Neighbor-Joining,NJ)構(gòu)建系統(tǒng)進(jìn)化樹。
1.5 桑樹NBS-LRR類基因表達(dá)分析
通過桑樹基因組數(shù)據(jù)庫下載獲得候選NBS-LRR類基因轉(zhuǎn)錄組數(shù)據(jù),分別對其在根、樹皮、冬芽、雄花和葉中表達(dá)進(jìn)行分析。
1.6 miRNA靶標(biāo)NBS-LRR類基因預(yù)測
根據(jù)序列互補(bǔ)原則對NBS-LRR類基因的調(diào)控miRNA進(jìn)行預(yù)測。使用psRNATarget在線軟件(ht-tp://plantgrn. noble. org/psRNATarget/)對文獻(xiàn)中111個桑樹miRNA[ 20-21]的靶標(biāo)NBS-LRR類基因進(jìn)行預(yù)測。
2 結(jié)果與分析
2.1 桑樹NBS-LRR類基因的鑒定與分類
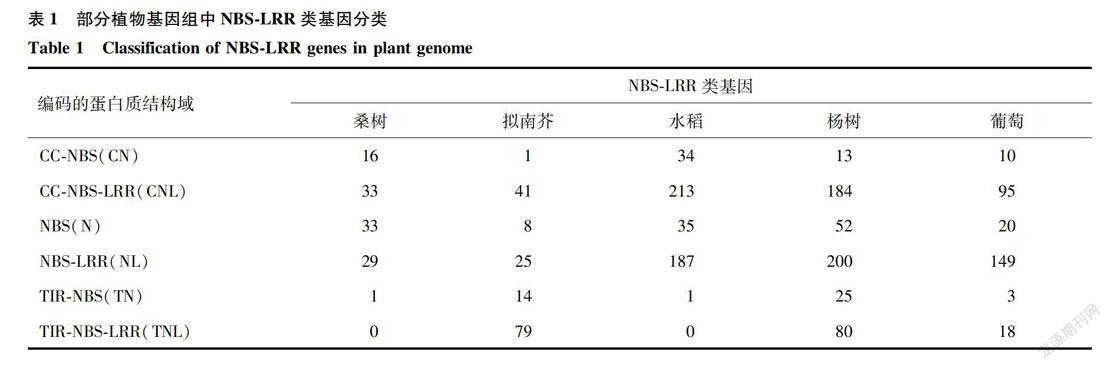
通過搜索桑樹基因組數(shù)據(jù)庫,共獲得含有NBS核心結(jié)構(gòu)的桑樹NBS-LRR類候選基因112個,占桑樹基因組總基因數(shù)(29 261個)的0.4%,其中編碼的蛋白質(zhì)具有NBS、LRR和CC完整結(jié)構(gòu)域的基因共33個,占NBS-LRR類基因的29.5%(表1)。編碼的蛋白質(zhì)含有LRR結(jié)構(gòu)域的有63個,不含LRR結(jié)構(gòu)域的基因有49個。桑樹NBS-LRR類基因比例與擬南芥比較接近,而明顯少于其他一些物種[12];物種間NBS-IRR類基因數(shù)量存在較大差異,這可能與植物在進(jìn)化過程中NBS-IRR類基因經(jīng)受的選擇壓力有關(guān)。
研究發(fā)現(xiàn),桑樹NBS-LRR類基因主要分為NBS、CC-NBS、CC-NBS-LRR、NBS-LRR4種結(jié)構(gòu)類型。除了具有NBS、TIR、LRR、CC結(jié)構(gòu)域之外,還包含DUF、P-loop、zf-RVT和RPW8等結(jié)構(gòu)域類型。發(fā)現(xiàn)L848_014445、L848_013225、L848_013226含有RPW8結(jié)構(gòu)域,RPW8為擬南芥白粉病廣譜抗性蛋白家族功能域,這些基因可能與植物的白粉病抗性有關(guān)。功能注釋分析結(jié)果顯示,編碼抗病蛋白RPM1基因34個,編碼抗病蛋白RGA基因24個,編碼抗病蛋白RPP13基因12個,此外還有非活性疾病易感蛋白LOVI、晚疫抗性蛋白、TMV抗性蛋白N等。
2.2 桑樹NBS-LRR類基因結(jié)構(gòu)與系統(tǒng)發(fā)育分析
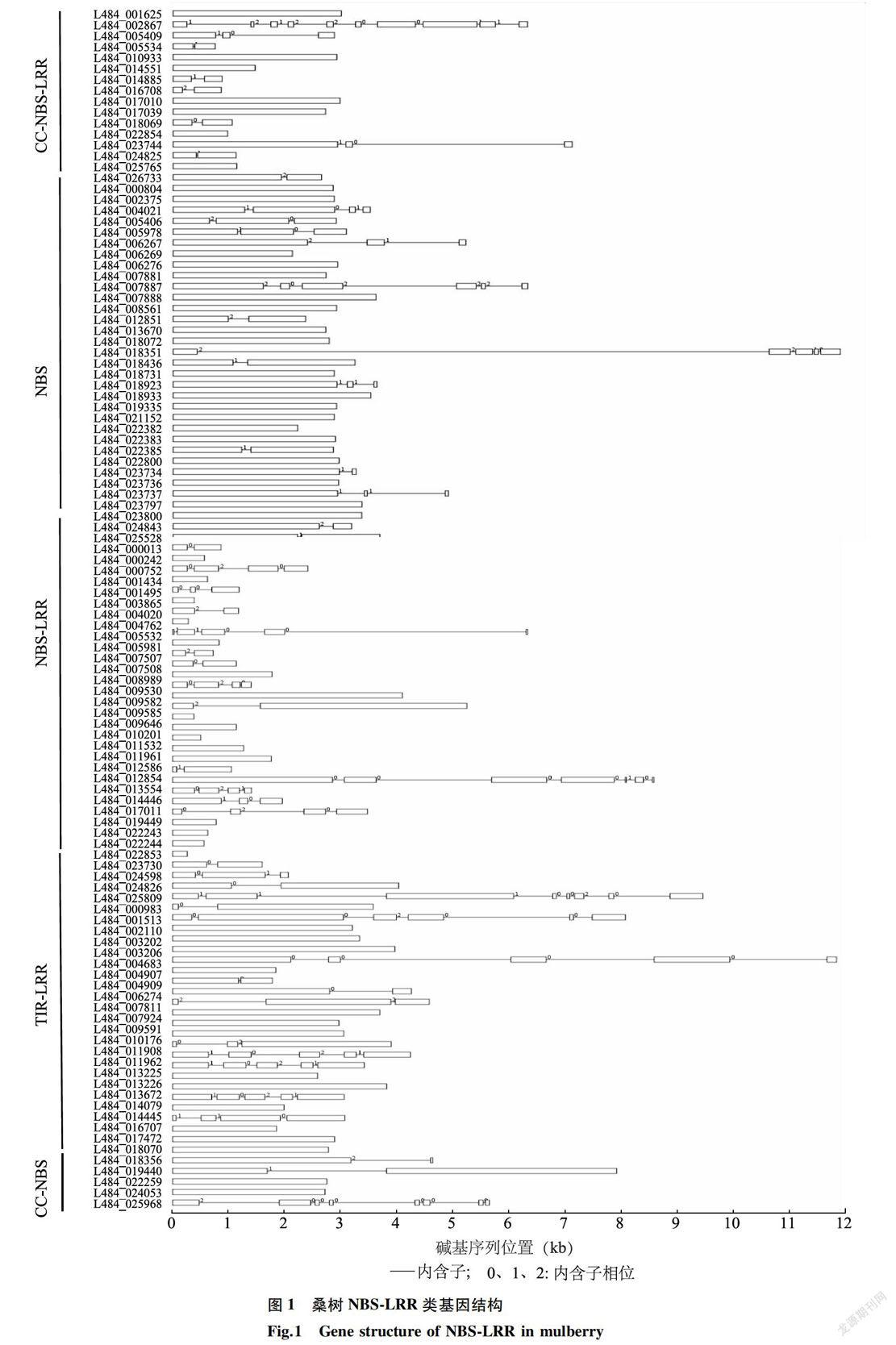
利用基因結(jié)構(gòu)顯示系統(tǒng)對桑樹NBS-LRR類基因結(jié)構(gòu)進(jìn)行分析,發(fā)現(xiàn)該家族基因結(jié)構(gòu)類型多樣。大部分CC-NBS-LRR類型基因只含有1個外顯子,編碼序列長度一致性較高,而NBS類型基因內(nèi)含子數(shù)和內(nèi)含子相位類型較復(fù)雜,編碼序列長度差異較大(圖1)。
使用MEME在線軟件對桑樹NBS-LRR蛋白質(zhì)基序進(jìn)行分析(表2)。Motif 1位于NBS功能域N末端,也是AAA功能域(ATPases associated witha variety of cellular activities)的一部分,參與膜融合、蛋白質(zhì)水解和DNA復(fù)制等多種細(xì)胞過程;Motif 2- Motif 5和Motif 10位于NBS功能域中間部分,也是AAA功能域的組成部分;Motif 7位于NBS功能域的C端,大部分桑樹NBS-LRR蛋白均含有這些Motif,也有少部分桑樹缺少該類Moti;Motif 6位于LRR功能域內(nèi),也是SCOP dlfqva2功能域的一部分,屬于RNI-like超家族,由相似重復(fù)構(gòu)成的規(guī)則結(jié)構(gòu),形成右手盧-a超螺旋的LRR功能域,NBS-LRR蛋白質(zhì)中含有O至多個Motif 6,最多的IA84_025528蛋白質(zhì)含有8個;Motif 8和Motif 9分別位于CC結(jié)構(gòu)的N端和中部,在下游信號的激活中發(fā)揮作用,CC-NBS和CC-NBS-LRR類蛋白質(zhì)含有至少1個該類Motif。
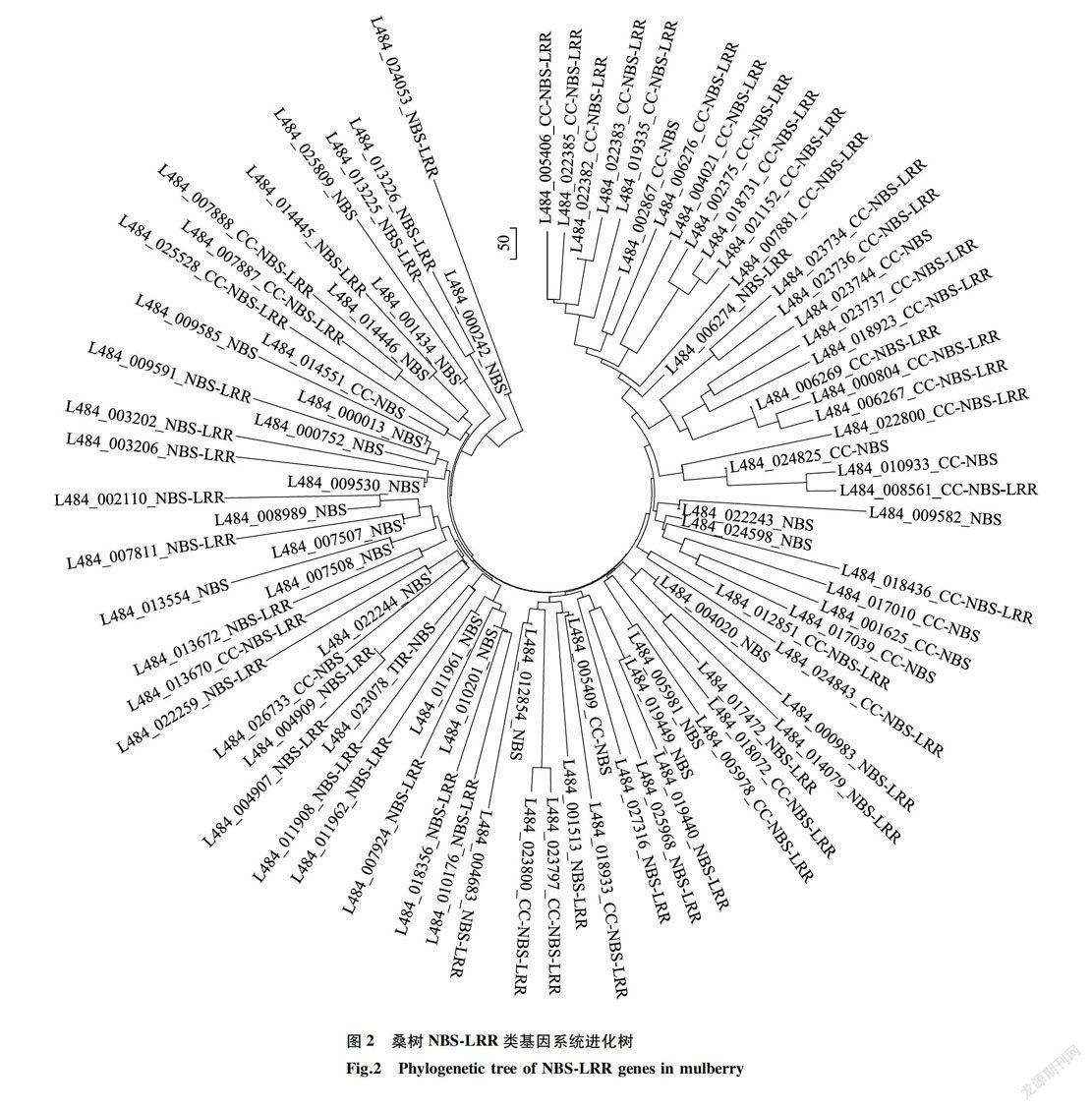
去掉部分相似度較低的序列,選擇92個桑樹NBS-LRR類基因構(gòu)建NJ系統(tǒng)進(jìn)化樹,對基因進(jìn)化關(guān)系進(jìn)行分析。結(jié)果(圖2)顯示,桑樹NBS-LRR類基因主要分為4個大的聚類組,4種主要NBS-LRR類基因聚類存在交叉現(xiàn)象,部分CC-NBS和NBS-LRR類基因聚類到CC-NBS-LRR組中,部分CC-NBS-LRR和NBS類基因聚到CC-NBS組中,部分NBS、CC-NBS-LRR和CC-NBS類基因聚到NBS-LRR組中,NBS組中含有多個其他3種基因類型成員。說明進(jìn)化過程中不同類型的NBS-LRR類基因間可能存在遺傳信息的交換或共進(jìn)化現(xiàn)象。每個小聚類組中,NBS類型基因的進(jìn)化枝長要短于同組其他類型基因,表明該組基因可能來源于該NBS類型基因。
2.3 桑樹NBS-LRR類基因的組織表達(dá)
為了解桑樹NBS-LRR類基因的組織表達(dá)特征,下載并分析了桑樹基因組數(shù)據(jù)庫中轉(zhuǎn)錄組數(shù)據(jù)。共檢測到93個基因在不同組織中有表達(dá)(圖3)。其中基因L484_013225和L484_ 014445在所有檢測組織中均有較高表達(dá),這2個基因均含有RPW8結(jié)構(gòu)域,屬于RPW8-NBS-LRR結(jié)構(gòu)域類型,可能在桑樹基礎(chǔ)抗病中發(fā)揮主要作用。L484一022383、L484一013226、L484_017039、LA84_ 025528、LA84一018436、LA84_017010、LA84一010201等基因在樹枝樹皮、雄花和葉中表達(dá)量較高,而在根(除L484一022383和LA84_ 013226外)和冬芽中表達(dá)量稍低。基因L484一013672僅在根和樹枝樹皮中檢測到較高表達(dá)。部分基因僅在特定組織中檢測到較低表達(dá)。總體而言,較多的NBS-LRR類基因在樹枝樹皮、雄花和葉等組織中表達(dá)值較高,其次是根,而冬芽中多數(shù)NBS-LRR類基因的表達(dá)較低或未檢測到表達(dá)。說明不同的NBS-LRR類基因在桑樹的不同組織中發(fā)揮作用,部分基因存在組織表達(dá)特異性。
2.4 miRNA對NBS-LRR類基因的調(diào)控分析
根據(jù)序列互補(bǔ)原則,利用psRNATarget在線軟件對桑樹miRNA與NBS-LRR類基因的調(diào)控關(guān)系進(jìn)行序列比對分析,篩選到614個miRNA與NBS-LRR類基因存在調(diào)控關(guān)系,發(fā)現(xiàn)101個桑樹miRNA可調(diào)控106個NBS-LRR類基因。較低的期望值表示miRNA與靶基因序列匹配較好。非配對靶標(biāo)位點(diǎn)最大能量( UPE)為解開靶基因mRNA靶位點(diǎn)二級結(jié)構(gòu)所需的能量,較低的UPE值表示miRNA結(jié)合或裂解靶基因的可能性較高。本研究選擇期望值小于3的部分比對結(jié)果進(jìn)行展示(表3)。miR472b和miR482b可對多個NBS-LRR類基因靶標(biāo)位點(diǎn)進(jìn)行結(jié)合,LA84一022259同時被miR472b和miR482a-3p
2個miRNA識別。miRNA對靶標(biāo)NBS-LRR類基因的調(diào)控有轉(zhuǎn)錄抑制和序列裂解2種方式,這2種抑制基因表達(dá)的方式比例相當(dāng),但不同類型miRNA的調(diào)控方式不同,miR472b可通過轉(zhuǎn)錄抑制和裂解2種方式調(diào)控不同靶基因的表達(dá),miR482主要通過轉(zhuǎn)錄抑制方式調(diào)控靶基因表達(dá)(表3)。
3 討論
植物NBS-LRRS存在群體水平上的多態(tài)性,其在直系同源基因的序列組成和旁系同源基因的總體數(shù)量上都有差異,宿主中NBS-LRRS基因的數(shù)量與物種病原效應(yīng)子的多態(tài)性有關(guān)[22]。自1994年第一個NBS-LRR類基因被成功克隆后[23],大量NBS-LRR類基因陸續(xù)被研究和報道,研究集中在NBS-LRR類基因的多樣性及進(jìn)化上[24]。本研究通過搜索桑樹基因組數(shù)據(jù)庫,共獲得112個具有NBS核心結(jié)構(gòu)域的桑樹NBS-LRR類候選基因,其中63個同時具有NBS和LRR功能域。NBS結(jié)構(gòu)域參與信號轉(zhuǎn)導(dǎo),具有高度保守和序列一致的基序,而LRR結(jié)構(gòu)域參與病原體配體的識別,通常是高度可變的[25]。進(jìn)化分析結(jié)果顯示,桑樹NBS-LRR類基因聚類存在類型間的交叉,成員較少的聚類組中基因類型一致性較高,而成員較多的聚類組中往往有多個類型的基因。聚類組中類型和結(jié)構(gòu)一致的基因可能是由共同的NBS-LRR類祖先基因復(fù)制而來,而同一聚類組中不同類型的基因可能是由異位重組而來,異位重組導(dǎo)致該聚類組基因的擴(kuò)張[24]。有些抗病基因需要2個NBS-LRR類基因共同介導(dǎo),例如水稻中Pikml TS和Pikm2 TS共同介導(dǎo)抗病基因Pikm[26] ,RRS1和RPS4在十字花科植物多種細(xì)菌性病害抗性中共同發(fā)揮作用[27]。這可能與NBS-LRR類基因的適應(yīng)性成本有關(guān),也是該家族基因進(jìn)化復(fù)雜性的體現(xiàn)。
多個NBS-LRR類基因被鑒定為廣譜抗性基因,過表達(dá)NBS-LRR類基因,顯著增強(qiáng)了植物的抗病性[28]。本研究中,NBS-LRR類基因的組織表達(dá)存在差異,基因L484一013225和IA84一014445在多個組織中均有較高表達(dá),這2個基因編碼的蛋白質(zhì)均含有白粉病抗性蛋白功能域RPW8,可能在桑樹的基礎(chǔ)抗病中起作用,而L484一022383等基因在部分組織中表達(dá)較高,可能在桑樹的特定組織器官或特定時期生長發(fā)育或抗病中發(fā)揮作用,也有些基因組織表達(dá)水平較低或未檢測到表達(dá),這些基因可能參與植物特定時期的抗病過程或者處于非激活狀態(tài)。
miRNA作為調(diào)節(jié)子在NBS-LRR類基因的表達(dá)調(diào)控中發(fā)揮作用[24]。植物NBS-LRR類基因的高表達(dá)導(dǎo)致能量損耗而影響植物生長,植物進(jìn)化出miR-NA-NBS-LRR系統(tǒng)來調(diào)控NBS-LRR的表達(dá)[12]。Lu等[29]首次報道了miRNA對NBS-LRR類基因的調(diào)控,隨后大量miNRA與其靶NBS-LRR類基因的作用關(guān)系被明確[8]。番茄miR482通過靶NBS-IRR受體的P-loop,與168個抗性基因中的58個發(fā)生作用30]。在番茄、馬鈴薯和煙草中至少搜索到10個miRNA家族及其靶抗性基因之間的作用關(guān)系,說明miRNA調(diào)節(jié)抗性基因表達(dá)在植物基因組中普遍存在[31]。通過對桑樹miRNA與NBS-LRR類基因調(diào)控關(guān)系的分析,發(fā)現(xiàn)大部分桑樹NBS-LRR類基因均具有被miRNA調(diào)控的潛在可能,有17個miRNA與靶標(biāo)序列匹配期望值小于3,其中miR472b和miR482b均有5個靶NBS-LRR預(yù)測結(jié)合位點(diǎn),這些miRNA在桑樹生長發(fā)育和抗病中的調(diào)控作用值得進(jìn)一步深入研究。
參考文獻(xiàn)
[1]JONES J D G, DANGL J L. The plant immune system[ J] . Nature,2006. 444 ( 7117 ) : 323-329.
[2]BOLLER T,HE S Y. Innate immunity in plants: an arms race be-tween pattern recognition receptors in plants and effectors in micro-bial pathogens [ J] . Science , 2009 , 324( 5928) : 742.
[3]SANABRIA N M, HUANG J C, DUBERY I A. Sejf/non-self per-ception in plants in innate immunity and defense [J]. Self/nonself, 2010, 1( 1) : 40-54.
[4]QI D , INNES R W. Recent advances in plant NLR structure , func-tion , localization , and signaling [J] . NLR-protein functions in im-munity, 2015 ,4 :348.
[5] BERNOUX M . VE T, WILLIAMS S, et al. Structural and functionalanalysis of a plant resistance protein TIR domain reveals interfacesfor self-association, signaling, and autoregulation [J] . Cell HostMicrobe. 2011, 9 : 200-211.
[6]MAEKAWA T, CHENC W. SPIRIDON L N, et al. Coiled-coil do-main-dependent homodimerization of intracellular barley immunereceptors defines a minimal functional module for triggering cejldeath[J] . Cejl Host Microbe , 2011, 9 : 187-199.
[7]VAN DER BIEZEN E A,JONES J D C. The NB-ARC domain: anovel signalling motif shared by plant resistance gene products andregulators of cell death in animals [J] . Current Biology, 1998, 8(7) : 226-228.
[8]FEI Q,ZHANG Y, XIA R, et al. Small RNAs add zing to the Zig-Zag-Zig model of plant defenses [J] . Molecular Plant-microbe In-teractions, 2016, 29(3) : 165-169.
[9]STOKES T L, KUNKEL B N, RICHARDS E J. Epigenetic variationin Arabidopsis disease resistance [J]. Genes & Development,2002, 16(2) : 171.
[10]HA M , KIM V N. Regulation of microRNA biogenesis [J] . NatureReviews Molecular Cejl Biology, 2014, 15( 8) : 509-524.
[11] MA W, CHEN C. LIU Y, et al. Coupling of micro RNA-directedphased small interfering RNA generation from long noncodinggenes with alternative splicing and alternative polyadenylation insmall RNA - mediated gene silencing [J]. New Phytologist,2018 , 217 (4) : 1535-1550.
[12] ZHANG Y.XIA R, KUANG H, et al. The diversification of plantNBS-LRR defense genes directs the evolution of microRNAs thattarget them[ J] . Molecular Biology and Evolution . 2016. 33 ( 10) :2692-2705.
[13] ZHAI J,JEONC D H.DE P E.et al. MicroRNAs as master regula-tors of the plant NB-LRR defense gene family via the production of phased, trans-acting siRNAs [J] . Cenes & Development, 2011,25( 23) : 2540-2553.
[14]KHALFALLAH Y. BOUKTILA D, HABACHI-HOUIMLI Y . et al.Regulation of NBS-LRR genes by microRNAs in wheat: Computa-tional identification of candidate MIR-2118 genes and evidence offlexibility [J] . Cereal Research Communications , 2017 , 45 (1) :1-10.
[15]HE N,ZHANC C , QI X, et al. Draft genome sequence of the mul-berry tree Morus notabilis [J] . Nature Communications, 2013,4: 2445.
[16] HUANC Y, ZOU Q, WANC Z B. Computational identification ofmiRNA genes and their targets in mulberry[ J] . Russian Journal ofPlant Physiology , 2014 . 61(4) : 537-542.
[17]BARANWAL V K.NEGI N .KHURANA P. Genome-wide identifi-cation and structural. functional and evolutionary analysis ofWRKY components of mulberry [J] . Scientific Reports, 2016,6: 30794.
[18]劉潮 .韓利紅 ,宋培兵 ,等.桑樹 WRKY轉(zhuǎn)錄因子的全基因組鑒定及生物信息學(xué)分析 [J] .南方農(nóng)業(yè)學(xué)報 . 2017 . 48 (9) :1691-1699.
[19]GAI Y P, ZHAO H N, ZHAO Y N, et al. MiRNA-seq-basedprofiles of miRNAs in mulberry phloem sap provide insight into thepathogenic mechanisms of mulberry yellow dwarf disease [J] . Sci-entific Reports, 2018, 8( 1) : 812.
[20]WU P , HAN S.ZHAO W . et al. Genome-wide identification of abi-otic stress-regulated and novel microRNAs in mulberry leaf [J] .Plant Physiology & Biochemistry. 2015 , 95 : 75-82.
[21] JIA L.ZHANC D,QI X,et al. Identification of the conserved andnovel miRNAs in Mulberry by high-throughput sequencing [J] .PLoS ONE. 2014. 9( 8) : e104409.
[22] KUANC H, WOO S S, MEYERS B C. et al. Multiple geneticprocesses result in heterogeneous rates of evolution within the majorcluster disease resistance genes in lettuce [J] . Plant Cell , 2004 ,16(11) : 2870-2894.
[23]WHITHAM S.DINESH-KUMAR S P , CHOI D.et al. The productof the tobacco mosaic virus resistance gene N : similarity to toLl andthe interleukin-l receptor[ J] . Cell, 1994 , 78( 6) : 1101-1115.
[24]YANC X . WANC J. Cenome-wide analysis of NBS-LRR genes insorghum genome revealed several events contributing to NBS-LRRgene evolution in grass species [J] . Evolutionary Bioinformatics,2016, 12 : 36433.
[25]MACE E,TAI S.INNES D. et al. The plasticity of NBS resistancegenes in sorghum is driven by multiple evolutionary processes [J] .BMC Plant Biology, 2014, 14(1) : 253.
[26]ASHIKAWA I.HAYASHI N . YAMANE H , et al. Two adjacent nu-cleotide-binding site-leucine-rich repeat class genes are required toconfer Pikm-specific rice blast resistance [J] . Cenetics, 2008,180(4) : 2267-2276.
[27]NARUSAKA M.SHIRASU K.NOUTOSHI Y.et al. RRSI and RPS4provide a dual resistance-gene system against fungal and bacterialpathogens[ J] . The Plant Journal, 2009 , 60( 2) : 218-226.
[28] ZHANC C. CHEN H.CAI T, et al. Overexpression of a novelpeanut NBS-LRR gene AhRRS5 enhances disease resistance toRalstonia soLanacearum in tobacco [J]. Plant BiotechnologyJournal, 2017, 15(1) : 39-55.
[29]LU C, KULKARNI K. SOURET F F. et al. MicroRNAs and othersmall RNAs enriched in the Arabidopsis RNA-dependent RNA pol-ymerase-2 mutant [J]. Cenome Research. 2006. 16 (10) :1276-1288.
[30]SHIVAPRASAD P V.CHEN H M.PATEL K. et al. A microRNAsuperfamily regulates nucleotide binding site-leucine-rich repeatsand other mRNAs [J] . The Plant Cell. 2012 . 24( 3) : 859-874.
[31] 11 F, PIGNA'ITA D. BENDIX C. et al. MicroRNA regulation ofplant innate immune receptors [ J] . Proceedings of the National A-cademy of Sciences. 2012. 109(5) : 1790-1795.

