禾生指葡孢霉的遺傳多樣性及對苗期青稞的致病性
陳琳藺瑞明王鳳濤龐云星李雪趙愛平張艷霞張金玲李文興何蘇琴馮晶李赟文才藝徐世昌
禾生指葡孢霉的遺傳多樣性及對苗期青稞的致病性
陳琳1,藺瑞明1,王鳳濤1,龐云星1,李雪1,趙愛平1,張艷霞2,張金玲1,3,李文興4,何蘇琴5,馮晶1,李赟1,6,文才藝6,徐世昌1
(1中國農業科學院植物保護研究所植物病蟲害生物學國家重點實驗室,北京 100193;2青海海北藏族自治州農業科學研究所,青海海北 810299;3西南科技大學生命科學與工程學院,四川綿陽 621010;4西藏日喀則市農業科學研究所,西藏日喀則 857000;5甘肅省農業科學院植物保護研究所,蘭州 730070;6河南農業大學植物保護學院,鄭州 450001)
在青藏高原及周邊高海拔地區,青稞是當地唯一的小禾谷類糧食作物,也是主要飼草來源。穗腐病是近年來僅在青藏高原青稞種植區發現的一種新真菌病害,病原菌為禾生指葡孢霉(),它能侵染多種麥類作物及禾本科雜草,給青稞安全生產帶來嚴重的挑戰。【】研究禾生指葡孢霉遺傳多樣性、系統進化以及對寄主種子萌發的影響,揭示該病原菌的流行傳播、與寄主植物互作關系以及初侵染菌源,為病害防治提供參考。從病害流行區采集病株標本,并從典型病穗組織分離禾生指葡孢霉菌株27個,依據無性世代形態特征加以鑒定,分離獲得菌株的單孢培養物,提取基因組DNA。利用SRAP分子標記分析禾生指葡孢霉的全基因組多態性,獲得菌株特異性分子標記;利用LSU和保守遺傳位點比較禾生指葡孢霉與其近緣種屬的進化關系;將禾生指葡孢霉分生孢子懸浮液與未萌發或發芽后的青稞種子共培養,分析其對青稞種子萌發及幼苗的影響。選用14對SRAP引物組合進行穗腐菌遺傳多樣性分析,平均一對引物擴增產生90個多態性條帶;平均每個穗腐病菌株1.4個特異分子性標記,分離自小麥的菌株Z 13008特異性標記最多(7個),而對照菌株禾谷鐮孢()具有20個特異性標記。禾生指葡孢霉的地域分布與其遺傳多樣性缺乏明顯相關性。在鑒定的菌株中,僅有分離自小麥(Z 13008)、黑麥(Z 13024)和2個分離自青稞的菌株(Z 13013和Z 13006)的遺傳多樣性與其他菌株差異較大,說明病原菌群體的遺傳變異水平較低。利用20個分子標記編制DNA指紋二叉式分類檢索表,可鑒定21個禾指葡孢霉菌株。系統進化分析揭示禾生指葡孢霉與叢赤殼屬真菌遺傳距離較近。青稞種子萌發前接種處理對種子萌發、幼苗生長均無明顯的抑制作用,在萌發后共培養處理能明顯抑制幼苗根系生長,導致根系變為淺褐色,但對幼苗植株生長無明顯影響。與近緣種禾谷鐮孢相比,禾生指葡孢霉群體內基因組DNA多態性較低,而且其遺傳變異與寄主種類密切相關。禾生指葡孢霉致病性較弱,對青稞種子萌發和幼苗生長無明顯的抑制作用。
大麥;青稞;禾生指葡孢霉;穗腐病;SRAP;遺傳多樣性;指紋檢索表
0 引言
【研究意義】大麥()種植分布區域非常廣泛,是世界上第四大禾谷類作物(http:// faostat.fao.org)。根據播種面積和產量,大麥也是我國第四大禾谷類作物。大麥是有稃大麥(又稱皮大麥)和裸大麥的總稱,在我國青藏高原地區裸大麥被稱作青稞(var.)。青稞是青藏高原及周邊高海拔地區的唯一的小禾谷類糧食作物,也是主要飼草來源。青稞在我國各生態區域均有不同規模種植,但集中分布于青海全省、西藏自治區、四川西北部、甘肅甘南州和云南西北部如迪慶等青藏高原及其周邊高海拔冷涼地帶。穗腐病是近年來在青藏高原及其周邊麥區發現的一種嚴重危害青稞穗部的真菌新病害,給該地區青稞安全生產帶來嚴重的挑戰。研究穗腐病病原菌群體的遺傳多樣性、系統進化以及對寄主種子萌發及幼苗生長的影響,可以揭示病原菌的流行傳播、與寄主植物互作關系以及初侵染菌源,為病害防治提供依據。【前人研究進展】2009年在甘肅省甘南藏族自治州局部青稞地塊發現了穗腐病[1],也稱鞘腐病,其病原菌無性世代鑒定為禾生指葡孢霉()[2]。2012年筆者在青海省主要青稞種植區域也發現了這種病害。根據筆者2011—2018年系統跟蹤調查結果,目前已在西藏日喀則、青海全省、甘肅甘南州、四川甘孜州青稞種植區域發現了穗腐病,危害程度和流行區域總體呈不斷加重和擴散蔓延態勢。在病害流行區,一般青稞田塊病穗率為1%—5%,局部病穗率高達10%—15%。同時發現,青藏高原青稞田主要禾本科雜草之一野燕麥()是該病原菌另一個重要自然寄主植物。此外,已發現禾谷類作物如普通小麥()、栽培燕麥(.)、黑麥()以及其他禾本科雜草如冰草()也是其自然寄主。復雜的寄主范圍給研究病害傳播途徑和制定防治技術帶來挑戰。【本研究切入點】禾生指葡孢霉引起的穗腐病對青稞等小禾谷類作物造成嚴重經濟損失,但對該病原菌的遺傳變異以及對寄主植物種子萌發、幼苗生長的影響等情況了解甚少。【擬解決的關鍵問題】對禾生指葡孢霉菌株進行分離純化培養,采用SRAP技術分析其遺傳多樣性,揭示禾生指葡孢霉遺傳變異和不同病害流行區域病原菌群體間的關系,進一步分析病原菌群體遺傳特征和不同地區間病害傳播及流行規律;利用共培養接種試驗分析禾生指葡孢霉對青稞種子萌發及幼苗生長的影響,為明確穗腐病初侵染菌源及制定防治策略提供依據。
1 材料與方法
試驗于2017—2019年在中國農業科學院植物保護研究所完成。
1.1 病原菌材料
采集新鮮典型標本共27份,包括3份黑麥、8份野燕麥、14份青稞、1份小麥病穗和1份青稞麥粒(表1),將病穗標本快速脫水干燥后室溫保存,用于病原菌分離;以遺傳距離較近的小麥赤霉病菌——禾谷鐮孢()作為遺傳多樣性分析的對照菌株。
1.2 病原菌分離純化與鑒定
采用常規組織分離法對所采集的典型病穗標本進行病原菌分離與純化[3]。剝開未抽穗病株葉鞘,選取病變穗部麥芒、小穗穗梗或穗軸,切取小塊病樣組織(0.5—1.0 cm),經75%乙醇(v/v)消毒3—5 s,在稀釋4倍次氯酸鈉溶液(活性氯含量7.5%)消毒0.5—1 min后,用無菌水漂洗3遍,每次漂洗10 min,然后移至PDA(馬鈴薯200 g,葡萄糖20 g,瓊脂粉15 g,蒸餾水定容至1 L)培養基平板上,置于23—25℃恒溫箱中培養3—5 d,在菌落邊緣挑取小塊培養基,轉移至另一個PDA培養基平板中進行純化培養,培養基內放置8—10個無菌濾紙片(1.0 cm×1.0 cm),培養7 d后收菌濾紙片,真空干燥后于-20℃長期保存。為抑制細菌污染,可以在PDA培養基中添加乳酸(3—5滴/100 ml)。收集純化后的病原菌菌株的分生孢子,采用稀釋法獲得單孢培養物。
1.3 病原菌形態鑒定
將分離所得單孢菌株置于23—25℃下培養6—7 d,待產孢后于光學顯微鏡下觀察鑒定病原菌分生孢子和分生孢子梗的形態特征,同時觀察記錄菌落形態特征,確定分離獲得菌株為禾生指葡孢霉。
1.4 禾生指葡孢霉對種子萌發和幼苗根系的影響
分離自青海門源縣青稞禾生指葡孢霉Z 15403-3-1單孢菌株培養6—7 d后收集分生孢子,用無菌水配制1×103、1×105孢子/ml的孢子懸浮液。將青稞品種‘柴青1號’種子先用70%乙醇消毒5 min,然后在稀釋1倍次氯酸鈉溶液(活性氯含量7.5%)消毒30 min,無菌水漂洗3次,每次10 min。將消毒后的種子在23—25℃條件下無菌水中浸泡24 h,去除剩余的水繼續培養約1 d,待種子萌發后(即胚根突破種皮)用不同濃度的孢子懸浮液浸泡24 h,然后轉移至水瓊脂(0.5%,w/v)平板上,置于培養箱中繼續培養;或將消毒后的種子用滅菌的濾紙吸干多余水分,浸泡在不同濃度的孢子懸浮液24 h,然后轉移至水瓊脂(0.5%,w/v)平板上在同樣條件下繼續培養。接種共培養溫度為25℃晝/23℃夜,光照時間為16 h·d-1,光照強度為5 000—6 000 lx。每個處理培養3皿,3.0 ml孢子懸浮液浸泡90粒種子,每皿培養30粒種子。發芽率=(發芽種子數/供檢測種子總數)×100%,出苗率=(長出幼苗種子數/供檢測種子總數)×100%。以滅菌水浸泡已消毒的種子作為空白對照處理(Mock)。培養5 d后調查,采用SPSS Statistics v20(IBM,美國)進行方差齊性檢驗和單因素方差分析,采用Duncan’s multiple range test(<0.05)分析處理間差異顯著性。試驗獨立重復2次。
1.5 SRAP遺傳多樣性分析
每個菌株在PDA培養基上培養3皿(直徑9 cm),收集培養7 d的菌絲,CTAB(cetyltrimethylammonium bromide)法提取基因組DNA。檢測DNA濃度及純度,并稀釋至標準工作液50 ng·μL-1備用。選取16條已公布的SRAP標準引物(表2)[4],由北京三博遠志生物技術有限責任公司合成。以禾生指葡孢霉菌株和禾谷鐮孢對照菌株DNA為模板,進行特異PCR引物篩選及遺傳多樣性分析。PCR反應體系包括Taq酶(5 U·μl-1)0.2 μl,10×PCR緩沖液2.0 μl,dNTPs 0.8 μl,上游引物和下游引物各1.0 μl,DNA模板2.0 μl,加ddH2O至20.0 μl。SRAP-PCR反應體系采用復性變溫法,反應程序設定為兩個階段:第一階段為94℃預變性5 min,94℃變性1 min,35℃退火1 min,72℃延伸2 min,5次循環;第二階段為94℃變性1 min,50℃退火1 min,72℃延伸2 min,35次循環,72℃延伸5 min,擴增產物置于4℃下保存。制備6%聚丙烯酰胺凝膠,電泳分離檢測擴增產物[5]。
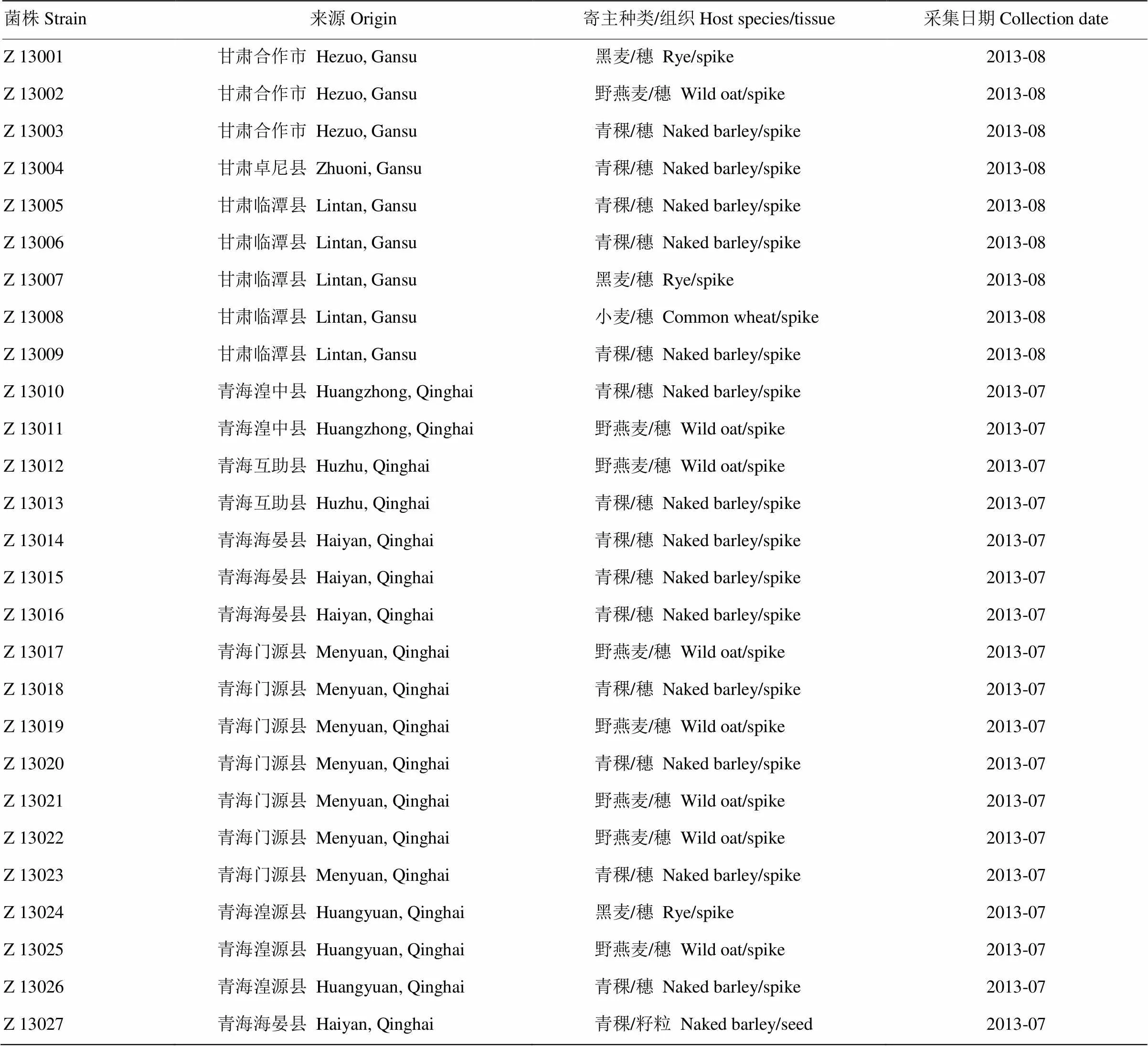
表1 禾生指葡孢霉菌株來源、寄主種類及分離的病變組織
1.6 進化樹分析和DNA指紋檢索表構建
以禾生指葡孢霉Z 13005菌株DNA為模板,選用LSU(LRORf:GTACCCGCTGAACTTAAGC,LR5r:ATCCTGAGGGAAACTTC)[6]和(RPB2-f5F:GAYGAYMGWGATCAYTTYGG,Rpb2-7cR:CCCAT RGCTTGYTTRCCCAT)[7]保守基因位點引物進行PCR擴增,分別獲得DNA片段克隆到T載體(Takara,大連)中測序。分別以LSU、部分序列在NCBI數據庫中BLAST分析,獲得與禾生指葡孢霉的保守基因具有較高相似性的真菌DNA序列,構建進化樹。采用MEGA5.05軟件進行序列比對處理,NJ(neighbor-joining)法構建系統發育樹,進行1 000次Bootstrap檢驗。基于SRAP分析方法獲得的多態性DNA條帶結果構建禾生指葡孢霉菌株的二叉式分子檢索表,具體方法參考Shan等[8]。
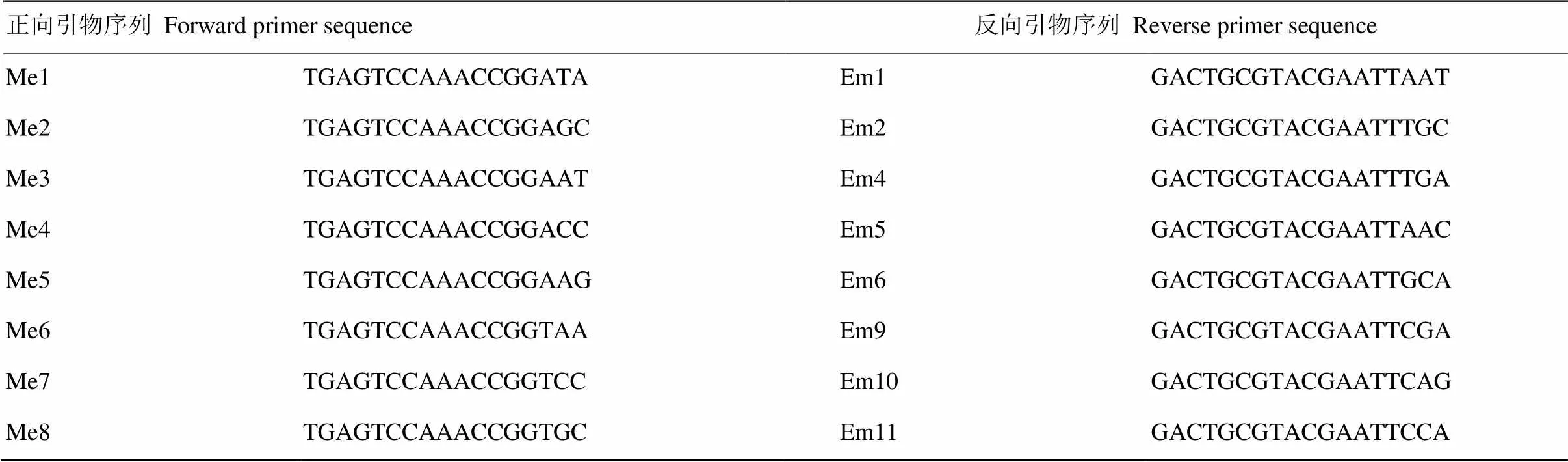
表2 SRAP分析的引物序列
1.7 SRAP數據處理
以Marker ?X174Ⅲ酶切產物和DL500 DNAmarker條帶為標準,確定PCR產物片段大小(bp),同時采用二進位制記錄電泳后DNA條帶數據,在擴增產物相同位點處依據條帶有和無分別記錄為‘1’和‘0’,用于SRAP遺傳多樣性分析,并利用NTSYS軟件的非加權組平均法(UPGMA,Unweighted Pair Group Method with Arithmetic Mean)進行聚類分析。
2 結果
2.1 禾生指葡孢霉分離純化
對27份穗腐病標本(表1)進行病原菌常規組織分離,經培養菌落形態和分生孢子及孢子梗形態特征鑒定,均為目的病原菌的純培養物,同時獲得27個菌株的單孢培養物65份,選用其中21個菌株的單孢培養物進行遺傳多樣性分析。
2.2 禾生指葡孢霉對青稞種子萌發及幼苗生長的影響
將初萌發的青稞種子與禾生指葡孢霉分生孢子懸浮液共培養24 h,然后在水瓊脂平板上繼續培養5 d,共培養處理能明顯抑制幼苗根系生長(圖1-A、1-C)。處理后幼苗根系長度與對照(Mock)相比差異顯著(=0.009<0.05),能造成幼苗根系變為淺褐色(圖1-A),而不同濃度孢子懸浮液處理間差異均不顯著;孢子懸浮液處理后出苗率(=0.822>0.05)和幼苗平均株高(=0.206>0.05)與對照相比差異不顯著(圖1-C)。另外,將表面消毒的未萌發種子與禾生指葡孢霉分生孢子懸浮液共培養24 h,在水瓊脂培養基上繼續培養5 d后與對照相比較,共培養處理后種子發芽率(=0.675>0.05)和出苗率(=0.992>0.05)均差異不顯著,青稞幼苗株高(=0.369>0.05)和幼苗根系長度(=0.203>0.05)與對照相比也無顯著差異(圖1-D),幼苗生長沒有受到明顯的抑制,無明顯的褪綠或萎蔫癥狀(圖1-B)。
2.3 SRAP引物PCR擴增
利用優化PCR體系擴增反應程序進行引物篩選,選用14對較高多態性的特異性引物組合(表3),共獲得具有多態性DNA條帶1 255條,平均一對SRAP引物能對21個禾生指葡孢霉菌株和1個禾谷鐮孢對照菌株PCR擴增產生90個條帶,其中Me6-Em5引物組合擴增產生的多態性條帶最多,其次是為Me6-Em10 引物組合。PCR擴增DNA片段大小分布于100—700 bp。統計發現,其中11個禾生指葡孢霉菌株具有1—7個菌株特異性分子標記,分離自小麥菌株Z 13008的特異性標記最多(7個),其次是分離自黑麥(Z 13024)和野燕麥(Z 13002)菌株具有5個特異性標記,而分離自青稞的菌株特異性標記較少(1—4個);未發現其余10個菌株的特異性標記。平均每個禾生指葡孢霉菌株僅有1.4個特異性標記,而對照禾谷鐮孢菌株特異性標記多達20個(表4)。
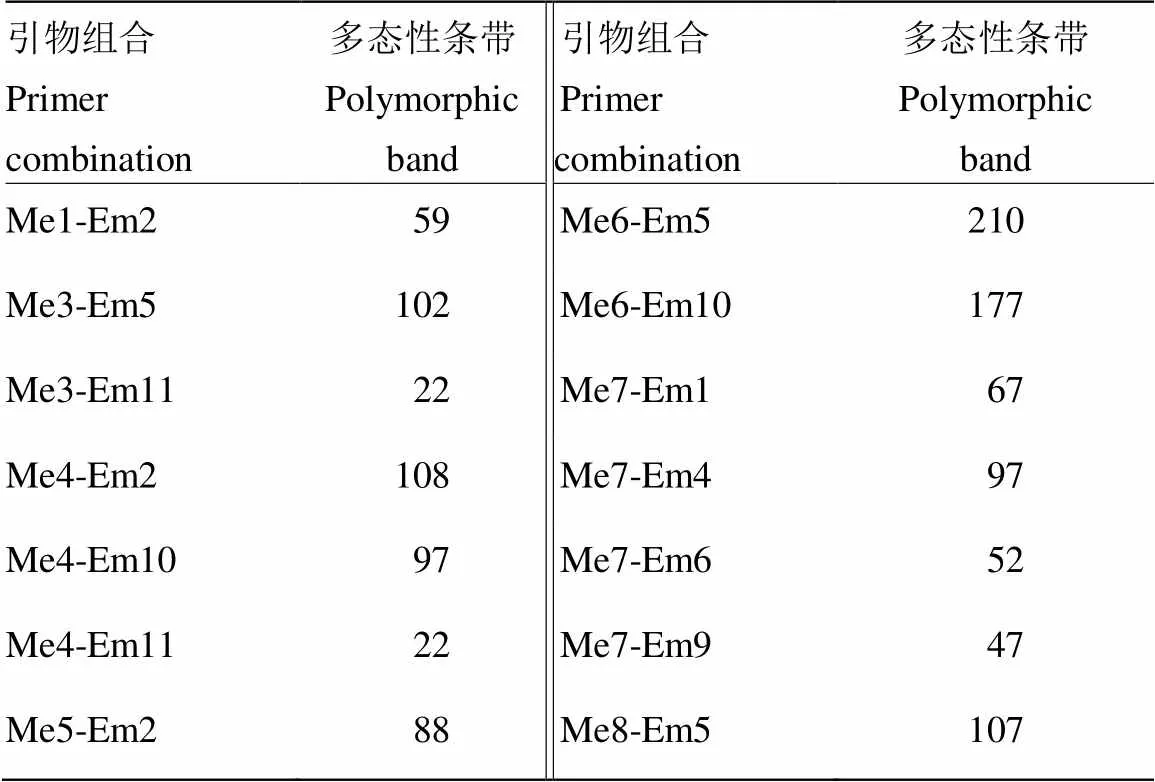
表3 利用14對SRAP引物組合擴增禾生指葡孢霉菌株獲得的多態性DNA條帶
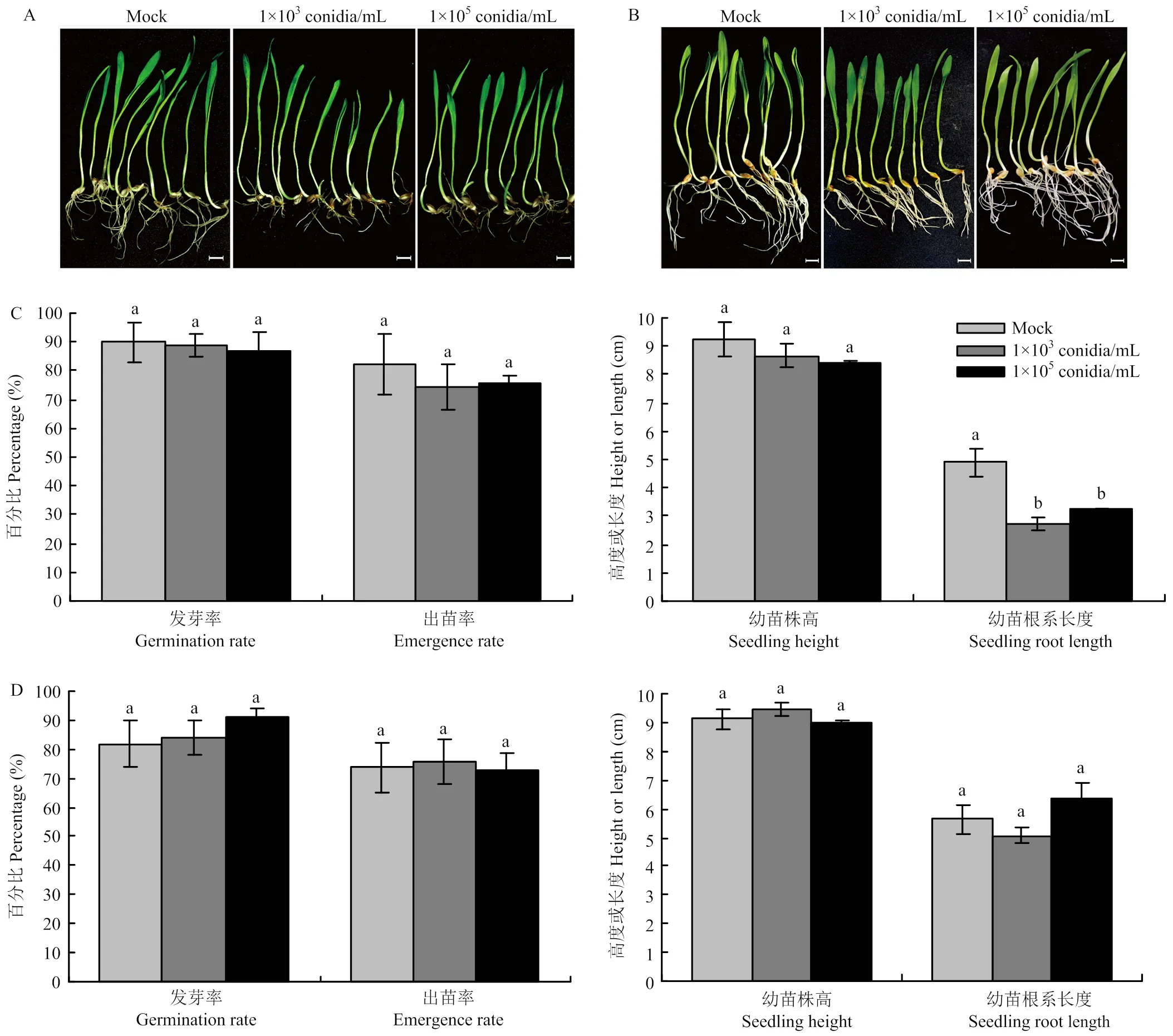
標尺Scale bar =1.0 cm
2.4 禾生指葡孢霉的遺傳聚類分析
根據對不同SRAP引物組合產生的PCR條帶的統計結果,利用軟件NTSYS pc 2.10對試驗數據進行非加權組平均法(UPGMA)聚類分析,根據遺傳相似系數(GS)法對禾生指葡孢霉21個菌株及對照禾谷鐮孢菌株遺傳多樣性進行聚類(圖2)。對照禾谷鐮孢菌株與禾生指葡孢霉菌株間菌遺傳距離較大,說明SRAP方法可以用于分析禾生指葡孢霉群體中不同菌株間的遺傳多樣性。分析21份禾生指葡孢霉菌株的遺傳距離系數范圍0.70—0.99,具有一定的多態性,而與禾谷鐮孢遺傳距離系數<0.70。在遺傳距離0.82水平上,可將21個禾生指葡孢霉菌株劃分為4個類群,類群Ⅰ包含17個菌株,來源于甘肅甘南州以及青海省多個地區,分離自寄主植物青稞和野燕麥,類群Ⅱ包括Z 13008和Z 13013,分別從甘肅甘南州臨潭縣小麥和青海互助縣青稞病穗,類群Ⅲ僅包括分離自甘肅甘南州臨潭縣寄主青稞的菌株Z 13006,而類群Ⅳ僅包括菌株Z 13024,來自青海湟源縣寄主黑麥病穗。類群Ⅰ中17個成員與類群Ⅱ、Ⅲ和Ⅳ中的4個菌株遺傳背景差異較大。類群Ⅰ中菌株來源地較多樣化,病原菌地域分布與其遺傳多樣性缺乏明顯相關性,青海與甘肅流行區間穗腐病原菌交流頻繁。根據類群Ⅰ菌株的寄主種類,可以確定青稞和野燕麥對病原菌遺傳多樣性的選擇作用較小;但類群Ⅱ、Ⅲ和Ⅳ中的4個菌株分別分離自小麥(Z 13008)、黑麥(Z 13024)和青稞(Z 13013,Z 13006),其遺傳多樣性與其他菌株差異較大,說明病原菌群體中存在一定程度的遺傳變異,寄主小麥和黑麥可能比青稞和野燕麥對禾生指葡孢霉遺傳變異具有更大的選擇作用。

表4 禾生指葡孢霉菌株特異性SRAP分子標記
a分子標記以SRAP引物組合及DNA片段大小(bp)表示Markers are indicated by SRAP primer combinations, and the marker size shown as base pairs;b禾谷鐮孢菌株作為對照,其他菌株均為禾生指葡孢霉的單孢培養物The strain of.is used as control, the others are.strains propagated from a single conidium
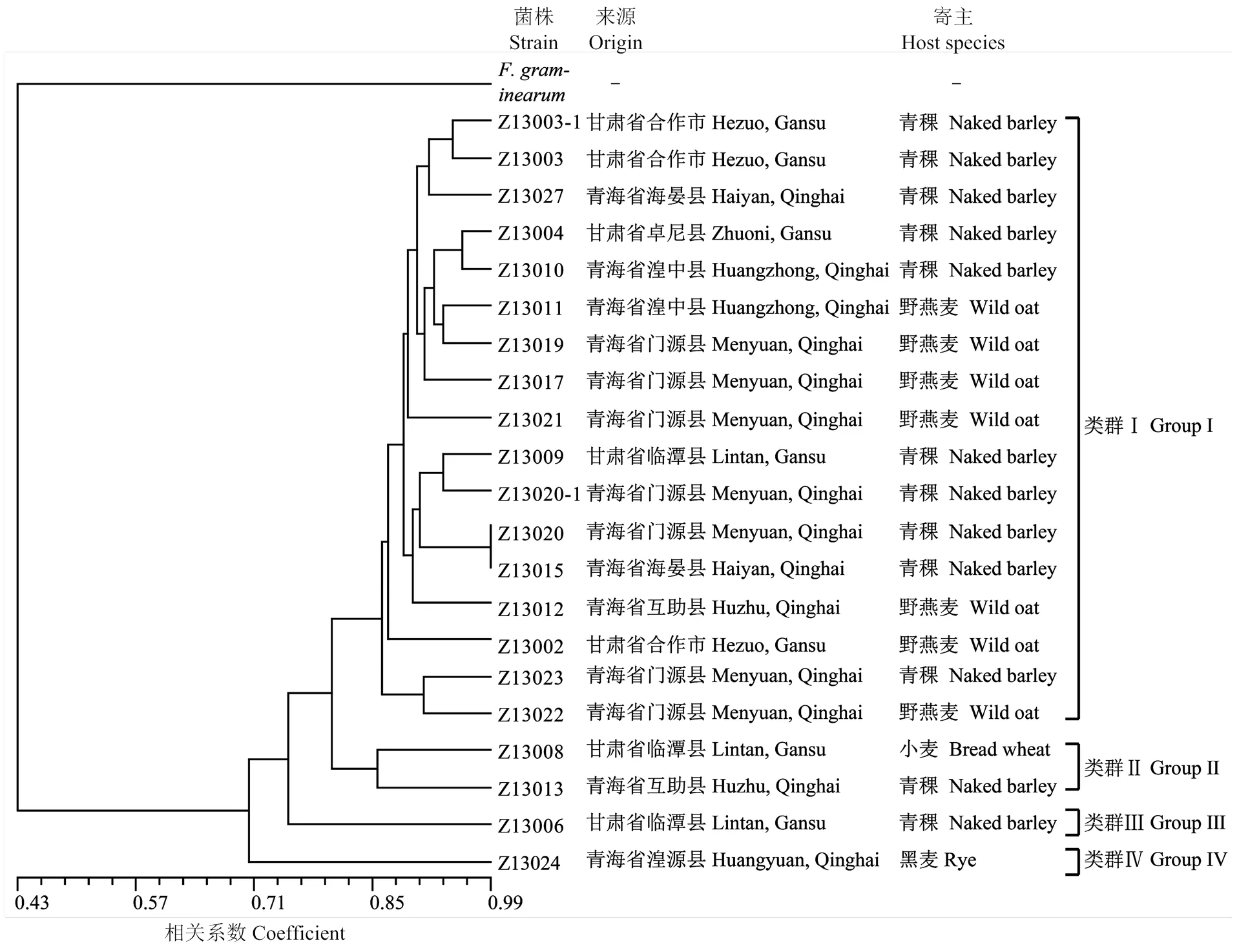
圖2 基于SRAP遺傳多樣性鑒定及UPGMA方法分析21個禾生指葡孢霉菌株及對照禾谷鐮孢菌株的遺傳變異
2.5 DNA指紋檢索表的制定
從14對多態性SRAP引物組合中選擇9對引物組合(Me1-Em2、Me3-Em2、Me3-Em5、Me4-Em2、Me6-Em5、Me6-Em10、Me7-Em4、Me7-Em6和Me7-Em9)擴增所得的20個分子標記,編制21個禾生指葡孢霉菌株的DNA指紋二叉式分類檢索表,根據檢索表可鑒定21個禾生指葡孢霉菌株(表5)。
2.6 禾生指葡孢霉的進化關系
以核糖體大亞基(LSU)和RNA聚合酶Ⅱ第2大亞基()基因的兩個保守位點序列(LSU+)構建進化樹,作為外群對照菌株。禾生指葡孢霉與肉座菌目(Hypocreales)叢赤殼科(Nectriaceae)的叢赤殼屬(sp.)真菌遺傳距離最近,其次是鐮孢菌屬(sp.)真菌,而與肉座菌科(Hypocreaceae)真菌(如sp.、sp.和sp.)遺傳距離較遠(圖3)。此外,利用其他保守位點序列組合(LSU+TUB和RPB2+TUB)構建進化樹,也驗證了禾生指葡孢霉與叢赤殼屬真菌遺傳距離較近。
3 討論
3.1 禾生指葡孢霉群體的遺傳多樣性及其系統發育
依據ITS1-5.8S-ITS2序列分析結果,禾生指葡孢霉與肉座菌目叢赤殼科一些屬種(如sp.、和.)的遺傳距離比與其他屬種真菌的遺傳距離近[2]。在此基礎上,利用兩個保守基因位點(LSU+)序列組合進一步構建精簡進化樹,發現禾生指葡孢霉與叢赤殼屬真菌遺傳距離最近,其次是鐮孢菌屬真菌(圖3)。因此,可以初步確定穗腐病原菌屬于叢赤殼科,該結果還需要從有性世代的形態特征以及多個保守基因位點的系統發育樹構建分析加以驗證。SRAP標記技術非常適合用于生物遺傳多樣性的分析和基因標記定位研究[9-10],如用于金針菇[11]、西葫蘆[12]種質資源鑒定以及遺傳圖譜構建[4,13-14]。本研究中采用SRAP技術分析得出21個禾生指葡孢霉菌株的遺傳距離相關系數范圍為0.70—0.99,群體內具有一定水平的遺傳變異,但與禾谷鐮孢遺傳背景差異較大(圖2),進一步驗證了利用保守基因位點構建的系統發育樹的分析結果。僅獲得11個禾生指葡孢霉菌株的特異性分子標記,平均每個菌株僅有1.4個特異性標記,而對照禾谷鐮孢菌株特異性標記多達20個(表4),說明禾生指葡孢霉群體中遺傳多樣性水平明顯低于作為多種禾谷類作物重要病原真菌的禾谷鐮孢。
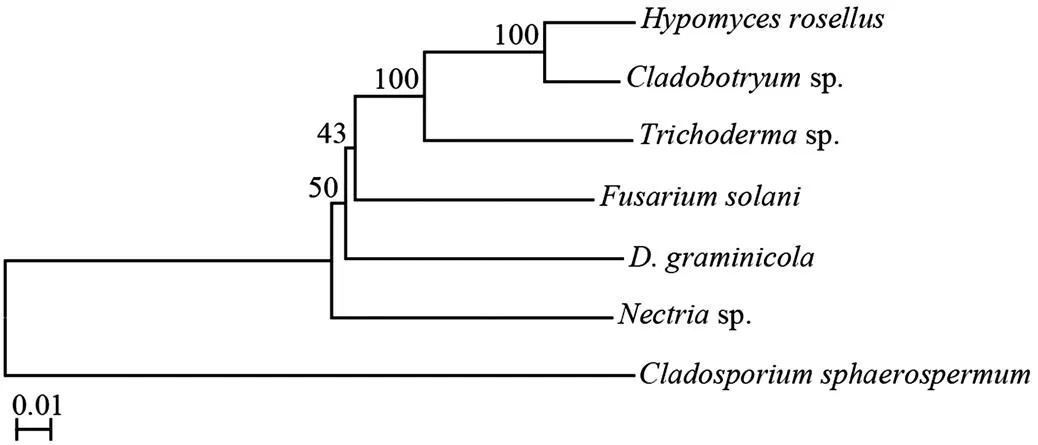
菌株LSU和Rpb2序列GenBank序列號GenBank accession numbers of LSU and Rpb2 sequences of fungal species:Cladobotryum sp.(MH871991,FN868654);Hypomyces rosellus(FN859443,EU710774);Trichoderma sp.(MH333256,FJ860531);Fusarium solani(AY097317,AB986567);Nectria sp.(JN939838,HM534886);Cladosporium sphaerospermum(KY873376,KJ443161);D. graminicola(菌株Z 13005,本研究測序結果strain Z 13005, sequences in this study)
3.2 禾生指葡孢霉的分子標記
類似于植物分類檢索表,基于基因組DNA分子標記編制的二分式檢索表首先用于區別蘿卜品種[15],該方法已用于構建花椰菜品種、野生鷹嘴豆及窄葉羽扇扁豆的指紋圖譜[8,16-17],利用MFLP分子標記編制了遺傳多樣性較低的栽培鷹嘴豆品種分子檢索表[18]。在本研究中利用20個SRAP分子標記編制了21個禾生指葡孢霉菌株的分子檢索表(表5),便于禾生指葡孢霉菌株的分子鑒定。
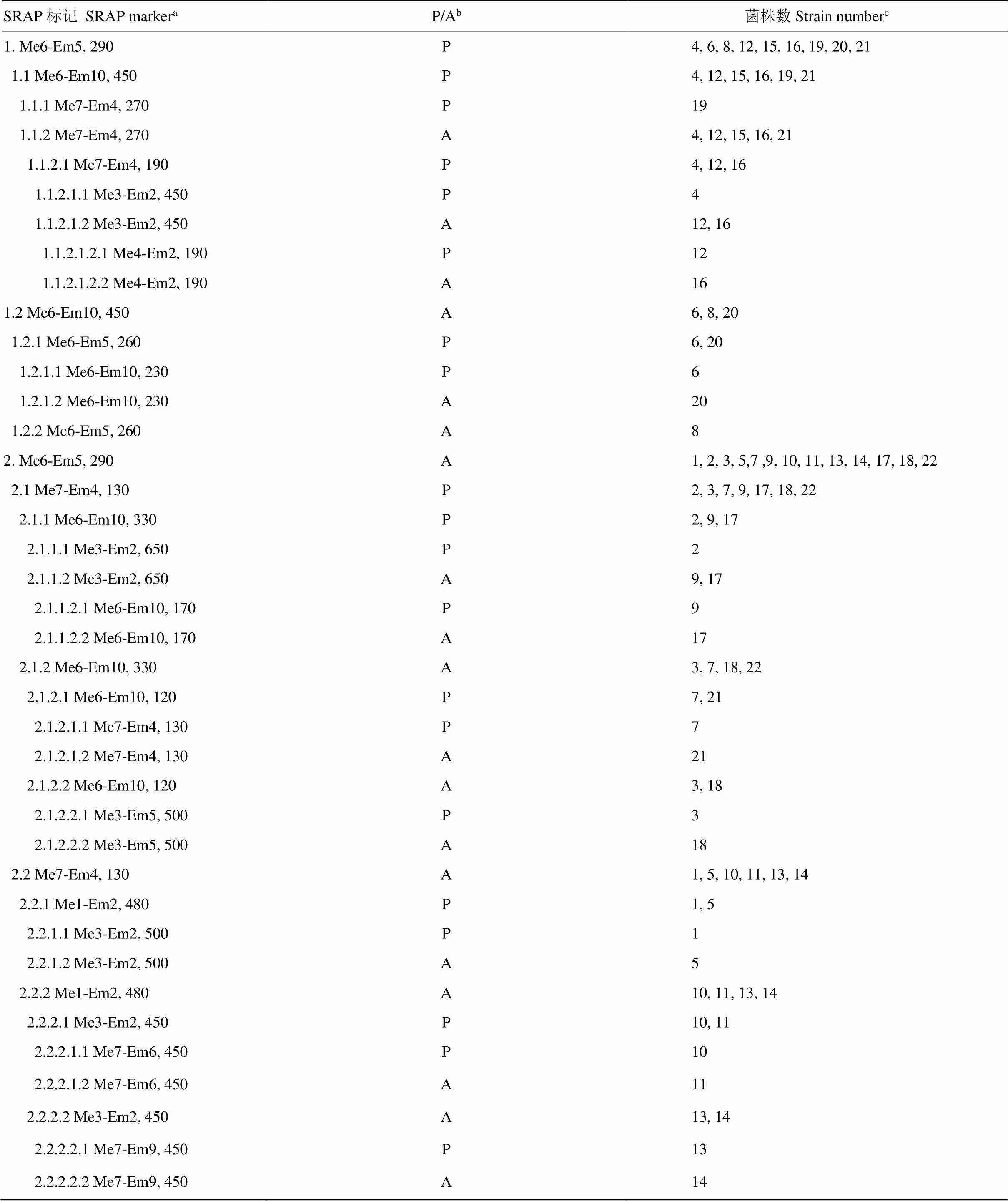
表5 利用SRAP分子標記構建禾生指葡孢霉及對照禾谷鐮孢菌株二叉式分類檢索表
a分子標記以SRAP引物組合及DNA片段大小(bp)表示Markers are expressed as SRAP primer combinations and marker size in base pairs;bP:有分子標記presence;A:無分子標記Absence;c菌株號1代表對照禾谷鐮孢菌株,其他菌株號分別代表表1中列出的菌株The strain number 1 represents.and other strain numbers represent the strains listed in Table 1, respectively. 2: Z 13003-1; 3: Z 13003; 4: Z 13002; 5: Z 13008; 6: Z 13006; 7: Z 13004; 8: Z 13009; 9: Z 13027; 10: Z 13010; 11: Z 13011; 12: Z 13012; 13: Z 13013; 14: Z 13019; 15: Z 13020; 16: Z 13020-1; 17: Z 13021; 18: Z 13023; 19: Z 13022; 20: Z 13024; 21: Z 13015; 22: Z 13017
3.3 寄主植物對病原菌遺傳變異的選擇
在青藏高原及周邊麥區發生的禾谷類作物穗腐病的地域分布具有一定的局限性,目前僅在青海各州、甘肅甘南高海拔地區以及西藏日喀則青稞、燕麥以及野燕麥、冰草等禾本科雜草上發現了該病害(表1)。在自然條件下禾生指葡孢霉僅侵染寄主幼穗引起穗腐病,同時侵染包裹幼穗的旗葉葉鞘或倒二葉葉鞘形成梭形褐色病斑,而不能侵染葉片組織或從葉鞘外表面直接侵入幼穗。因此該病原菌的致病性較弱,僅適合在氣候冷涼的青藏高原獨特生態環境中進化成為青稞和燕麥等麥類作物及多種禾本科雜草的病原菌。病原菌在寄主-病原菌互作識別、相互適應過程中建立寄生關系,二者協同進化能導致寄主抗病等位基因及病原菌毒性等位基因和遺傳結構的高度多樣化[19-21]。因此,寄主對病原菌基因型具有關鍵的選擇作用,也是維持病原菌遺傳多樣性及致病性變異的一種機制,如小眼夜蛾核多角體病毒(nucleopolyhedrovirus,PaflNPV)能侵染多種昆蟲寄主,從而改變了田間病毒遺傳多樣性[22];膠孢炭疽菌()遺傳多樣性及其致病性變異才能適應寄主毛薯()數量抗病性[23]。在本研究中,禾生指葡孢霉的最初主要寄主可能是青藏高原普遍分布的野燕麥及本地唯一的麥類糧食作物青稞,二者對病原菌的遺傳變異具有相似的選擇作用,而在自然界中野燕麥可能是穗腐病流行與病原菌傳播途徑中的重要‘橋梁’寄主。與青稞不同,小麥和黑麥僅在低海拔或溫度較高的局部區域內種植,要克服小麥和黑麥寄主作物抗病性及逆境脅迫,禾生指葡孢霉發生了更明顯的遺傳變異(圖2)。另外,依據穗腐病自然發生危害情況調查結果,常見的主要危害對象是青稞、栽培燕麥和野燕麥,而采集到的小麥、黑麥及冰草病害標本較少。因此,青稞和野燕麥是禾生指葡孢霉更適宜的寄主植物,然后寄主范圍逐漸擴大。因標本來源不均勻,還需要更多菌株驗證不同寄主對禾生指葡孢霉遺傳變異的選擇效應的差異。同時,在分離自青稞和野燕麥的菌株中,個別菌株與其他菌株的遺傳相似性存在較大的變異,如Z 13013和Z 13006。甘肅甘南州和青海省是相對獨立的穗腐病流行區域,但在遺傳多樣性聚類分析中菌株地域分布與其遺傳多樣性缺乏明顯相關性(圖2)。因此,青藏高原地區禾生指葡孢霉受到相似的環境選擇壓力,病原菌可能隨帶菌種子等病原載體在不同區域傳播擴散,導致來自地理距離較近的甘肅和青海禾生指葡孢霉菌株的遺傳變異不具有明顯的地域分布特征。
3.4 禾生指葡孢霉與近緣屬種病原真菌的致病性比較
叢赤殼科包括150個屬(http://www.mycobank. org/),多數是腐生菌,其中一些屬種是重要的農作物病原真菌。叢赤殼屬真菌能侵染多種作物或樹木,如(異名)的無性世代是爪哇鐮孢(),侵染番茄引起莖腐病[24];.(異名,.)的無性世代為,能侵染蘋果等多種果樹引起潰瘍病[25];赤霉屬(sp.)的無性世代大多屬于鐮孢屬,如.(無性世代)能侵染多種禾本科植物,引起麥類作物赤霉病[26]及玉米苗枯、根腐、莖基腐和穗腐等病害[27];麥類雪腐菌(,異名,.)的無性世代為雪腐鐮孢(),能造成麥類作物雪腐病和赤霉病[28]。禾生指葡孢霉作為叢赤殼科中新成員,目前發現該病原菌僅危害麥類作物及禾本科雜草,引起穗腐病,寄生能力較弱,與叢赤殼科其他植物病原菌的營養方式和侵染特點高度相似。
3.5 禾生指葡孢霉對青稞種子萌發和幼苗生長的影響
被病原菌侵染的種子是重要的初侵染菌源,降低了種子發芽率和種子活力,引起苗期病害。例如麥根腐平臍蠕孢()或spp.侵染大麥或小麥灌漿期種子引起黑胚病[29],能嚴重降低種子發芽率[30]。麥根腐平臍蠕孢、黃色鐮孢()和禾谷鐮孢侵染大麥根系和幼苗引起普通根腐病和立枯病[29]。大麥條紋病菌()引起的大麥條紋病是典型的種傳病害,種子帶菌幾乎不影響種子萌發及出苗[29]。然而禾生指葡孢霉僅侵染麥類作物及野燕麥等禾本科雜草植物穗部,因此攜帶病原菌的種子是重要的初侵染源之一,也是該病害跨區域傳播的主要途徑。利用禾生指葡孢霉分生孢子懸浮液與已發芽或未發芽的青稞種子進行共培養試驗(圖1),結果表明其對青稞種子萌發及對幼苗植株生長無明顯影響,僅高濃度的孢子懸浮液與已發芽的青稞種子共培養24 h能明顯抑制幼苗根系生長,根系顏色變為淺褐色,對幼嫩根系能產生致病作用。因此禾生指葡孢霉對青稞種子萌發以及幼苗生長期間的致病作用微弱,它是一種弱致病性植物病原真菌,種子攜帶的病原菌不能對青稞種子萌發和幼苗正常生長產生明顯的致病作用。而該病原菌可能在青稞或其他禾本科雜草寄主根部或表層土壤中植物枯殘組織上生長擴繁,為在青稞生育后期侵染穗部提供初始菌源。另外,在青稞收獲時節,青稞或其他禾本科雜草的病穗組織極易破碎而遺落在田間地表或表層土壤中,這些病殘體也能在青稞生育中后期產生和積累大量的分生孢子,是穗腐病的主要初侵染菌源。
4 結論
由禾生指葡孢霉侵染麥類作物以及多種禾本科雜草的幼穗引起穗腐病,它是僅在我國青藏高原及其周邊的部分青稞種植區域發生的一種真菌新病害。禾生指葡孢霉是弱致病性病原菌,對青稞種子萌發及幼苗生長無明顯的抑制作用,其與叢赤殼屬真菌遺傳距離較近,是叢赤殼科新成員。禾生指葡孢霉基因組多態性較低,其遺傳變異與寄主種類及其所處的環境條件密切相關。
[1] 何蘇琴, 王三喜, 趙桂琴, 文朝慧, 劉永剛, 馬福全. 燕麥和青稞的一種新病害——鞘腐病. 甘肅農業科技, 2010(10): 3-4.
HE S Q, WANG S X, ZHAO G Q, WEN Z H, LIU Y G, MA F Q. A new disease of oat and hulless barley—sheath rot disease., 2010(10): 3-4. (in Chinese)
[2] 何蘇琴, 文朝慧, 王生榮, 趙桂琴, 王三喜, 劉永剛, 荊卓瓊, 馬永強. 引起青稞和燕麥鞘腐病的無性型菌物新屬種——禾生指葡孢霉. 菌物學報, 2015, 34(3): 331-340.
HE S Q, WEN Z H, WANG S R, ZHAO G Q, WANG S X, LIU Y G, JING Z Q, MA Y Q.: a new phytopathogen causing sheath rot of hulless barley and oats., 2015, 34(3): 331-340. (in Chinese)
[3] 方中達. 植病研究方法. 3版. 北京: 中國農業出版社, 1998: 122-125.
FANG Z D.. Beijing: China Agriculture Press, 1998: 122-125. (in Chinese)
[4] LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP) a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in., 2001, 103(2/3): 455-461.
[5] 李登輝, 郭煥強, 馬麗, 馬東方, 王鳳濤, 馮晶, 藺瑞明, 徐世昌. 我國大麥條紋病菌()群體遺傳多樣性AFLP分析. 植物保護, 2016, 42(4): 71-76.
LI D F, GUO H Q, MA L, MA D F, WANG F T, FENG J, LIN R M, XU S C. Genetic diversity analysis ofpopulations in China, the causal organism of barley leaf stripe by using AFLP method., 2016, 42(4): 71-76. (in Chinese)
[6] VILGALYS R, HESTER M. Rapid genetic identification and mapping of enzymatically amplified ribosomal DNA from severalspecies., 1990, 172(8): 4238-4246.
[7] LIU Y J, WHELEN S, HALL B D. Phylogenetic relationships among Ascomycetes: evidence from an RNA polymerase II subunit., 1999, 16(12): 1799-1808.
[8] SHAN F, CLARKE H, YAN G, PLUMMER J A, SIDDIQUE K H M. Development of DNA fingerprinting keys for discrimination of(P.H. Davis) accessions using AFLP markers., 2004, 55(9): 947-952.
[9] 柳李旺, 龔義勤, 黃浩, 朱獻文. 新型分子標記——SRAP與TRAP及其應用. 遺傳, 2004, 26(5): 777-781.
LIU L W, GONG Y Q, HUANG H, ZHU X W. Novel molecular marker systems—SRAP and TRAP and their application., 2004, 26(5): 777-781. (in Chinese)
[10] ROBARTS D W H, WOLFE A D. Sequence-related amplified polymorphism (SRAP) markers: A potential resource for studies in plant molecular biology.,2014, 2(7): 1400017.
[11] 朱堅, 高巍, 林伯德, 孫淑靜, 鄭昭, 楊潔, 謝寶貴. 金針菇種質資源的SRAP分析. 福建農林大學學報(自然科學版), 2007, 36(2): 154-158.
ZHU J, GAO W, LIN B D, SUN S J, ZHENG Z, YANG J, XIE B G. SRAP analysis ofgermplasm resources., 2007, 36(2): 154-158. (in Chinese)
[12] FERRIOL M, PICO B, NUEZ F. Genetic diversity of a germplasm collection ofusing SRAP and AFLP markers., 2003, 107(2): 271-282.
[13] LI G, GAO M, YANG B, QUIROS C F. Gene for gene alignment between theandgenomes by direct transcriptome mapping., 2003, 107(1): 168-180.
[14] 林忠旭, 張獻龍, 聶以春. 新型標記SRAP在棉花F2分離群體及遺傳多樣性評價中的適用性分析. 遺傳學報, 2004, 31(6): 622-626.
LIN Z X, ZHANG X L, NIE Y C. Evaluation of application of a new molecular marker SRAP on analysis of F2segregation population and genetic diversity in cotton., 2004, 31(6): 622-626. (in Chinese)
[15] PRADHAN A, YAN G, PLUMMER J A. Development of DNA fingerprinting keys for the identification of radish cultivars., 2004, 44(1): 95-102.
[16] ASTARINI I A, PLUMMER J A, LANCASTER R A, YAN G. Fingerprinting of cauliflower cultivars using RAPD markers., 2004, 55(2): 117-124.
[17] YUAN H, YAN G, SIDDIQUE KHM, YANG H. RAMP based fingerprinting and assessment of relationships among Australian narrow-leafed lupin (L.) cultivars., 2005, 56(12): 1339-1346.
[18] LIN R, YANG H, KHAN T N, SIDDIQUE K H M, YAN G. Characterization of genetic diversity and DNA fingerprinting of Australian chickpea (L.) cultivars using MFLP markers., 2008, 59(8): 707-713.
[19] CARROLL S P, FOX C W.:. Oxford UK: Oxford University Press, 2008: 380.
[20] M?LLER M, STUKENBROCK E H. Evolution and genome architecture in fungal plant pathogens., 2017, 15(12): 756-771.
[21] GARCíA-ARENAL F, FRAILE A, MALPICA J M. Variability and genetic structure of plant virus populations., 2001, 39: 157-186.
[22] HITCHMAN R B, HODGSON D J, KING L A, HAILS R S, CORY J S, POSSEE R D. Host mediated selection of pathogen genotypes as a mechanism for the maintenance of baculovirus diversity in the field., 2007, 94(3): 153-162.
[23] FREZAL L, JACQUA G, NEEMA C. Adaptation of a fungal pathogen to host quantitative resistance., 2018, 9: 1554.
[24] KWON J H, CHOI O, KANG B, LEE Y, PARK J, KANG D W, HAN I, PARK E J, KIM J. Identification ofcausing tomato stem rot in Korea., 2017, 12: 34.
[25] XU X M, ROBINSON J D. Effects of fruit maturity and wetness on the infection of apple fruit by., 2010, 59(3): 542-547.
[26] GOSWAMI R S, CORBY KISTLER H. Heading for disaster:on cereal crops., 2004, 5(6): 515-525.
[27] GAI X T, XUAN Y H, GAO Z G. Diversity and pathogenicity ofspecies complex from maize stalk and ear rot strains in northeast China., 2017, 66(8): 1267-1275.
[28] Inglis D A, Cook R J.causes scab in the Pacific Northwest., 1981, 65(11): 923-924.
[29] Mathre D E. 大麥病害概略. 2版. 藺瑞明, 馮晶, 陳萬權, 徐世昌, 譯. 北京: 中國農業科學技術出版社, 2014: 66-69, 150-153.
Mathre D E..LIN R M, FENG J, CHEN W Q, XU S C, trans.Beijing: China Agricultural Science and Technology Press, 2014: 66-69,150-153. (in Chinese)
[30] LI Q Y, QIN Z, JIANG Y M, SHEN C C, DUAN Z B, NIU J S. Screening wheat genotypes for resistance to black point and the effects of diseased kernels on seed germination., 2014, 121(2): 79-88.
Genetic Diversity ofand its Pathogenicity tovar.Seedlings
CHEN Lin1, LIN RuiMing1, WANG FengTao1, PANG YunXing1, Li Xue1, ZHAO AiPing1, ZHANG YanXia2, ZHANG JinLing1,3, LI WenXing4, HE SuQin5, FENG Jing1, LI Yun1, 6, WEN CaiYi6, XU ShiChang1
(1State Key Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural Sciences, Beijing 100193;2Institute of Agricultural Sciences, Haibei Tibetan Autonomous Prefecture of Qinghai, Haibei 810299, Qinghai;3School of Life Science and Engineering, Southwest University of Science and Technology, Mianyang 621010, Sichuan;4Shigatse Institute of Agricultural Sciences of Tibet, Shigatse 857000, Tibet;5Institute of Plant Protection, Gansu Academy of Agricultural Sciences, Lanzhou 730070;6College of Plant Protection, Henan Agricultural University, Zhengzhou 450001)
【】In the high-latitude agricultural areas of Qinghai-Tibet Plateau and its peripheral regions, hulless or naked barley (var.) is the only small grain cereal crop in the local area, and it is also the major forage. Dactylobotrys spike blight, caused by, is a new fungal disease only found in the hulless barley growing areas in Qinghai-Tibet Plateau in recent years, which can attack several triticeae crops and cereal grasses and make a great challenge to security of hulless barley production.【】The objective of this study is to research the genetic diversity, systemic evolution ofthe fungal pathogen of.and its effects on the host seed germination, reveal the pathogen epidemics and spread ways, host-pathogen interrelationship and the primary inoculum, and to provide valuable information for disease control.【】The diseased plant samples were collected from the disease epidemic areas, and 27 strains were isolated from the typical infected host spike tissues. After identification based on its biological traits of anamorph, the single-conidium strains were cultivated and used for genomic DNA extraction. The whole genomic diversity was evaluated with SRAP markers and strain-specific markers were developed. The evolution relationship between.and its related genera or species was analyzed using the two conserved loci of LSUand. The conidium suspension was co-cultivated with the germinated or ungerminated hulless barely seeds, and its effect on barley seed germination and seedling growth of hulless barely was analyzed.【】Fourteen SRAP primer combinations were selected to detect genetic diversity of.strains, and on average, 90 polymorphic DNA bands were produced per primer combination. For.strains, only 1.4 strain-specific markers were developed averagely. The strain of Z 13008 derived from common wheat possessed the most 7 SRAP markers. Forused as a control strain, 20 specific markers were identified. It was confirmed that there is no close relationship between strain origins of.and their genetic diversity. Among the identified strains, only those like Z 13008 from common wheat, Z 13024 from rye and another two (Z 13013 and Z 13006) from hulless barley showed higher differences in genetic diversity compared with the others, indicating that the genetic variation level of.population is low. Besides, it is feasible to characterize the 21.strains with a DNA dichotomous fingerprinting key constructed with 20 SRAP markers.is genetically closely related to the fungal species of genusthrough phylogenetic analysis. When inoculating seeds with conidial suspensions before germination, the pathogen had no significant suppression on seed germination and growth of seedlings and their roots. While it could infect and restrain seedling root growth when co-cultivating the germinated seeds with conidial suspensions, and resulted in the infected young roots changing into light brown color, but had no significant effect on seeding growth.【】.showed lower genetic diversity compared with its relative species of., and its genetic variation is closely related to its host plant species.the pathogenicity of the pathogen is weak, and it has no obvious inhibiting effect on the germination and seedling growth of hulless barley.
;var.;; Dactylobotrysspike blight; SRAP; genetic diversity; fingerprinting key
10.3864/j.issn.0578-1752.2020.01.020

2019-06-18;
2019-08-16
國家現代農業產業技術體系建設專項(CARS-05)
陳琳,E-mail:lin170521@163.com。通信作者藺瑞明,E-mail:linruiming@caas.cn
(責任編輯 岳梅)

