大青楊PubZIP1基因的克隆及亞細胞定位與抗旱表達特性分析
劉 曉 楊 佳 張 馨 馬苗苗 楊靜莉
(東北林業大學林木遺傳育種國家重點實驗室,哈爾濱 150040)
大青楊(PopulusussuriensisKom.)屬楊柳科(Salicaceae)是一種速生落葉喬木,廣泛分布于中國北方、朝鮮和西伯利亞。大青楊材質韌性較好,抗腐蝕性強,木質緊密凈白,是造紙業和建筑業的優質原材料。隨著自然環境的日漸惡化,大青楊表現出多種優良性狀,比如其育種周期短,適應性強,因此廣泛應用于人工造林和退耕還林。楊樹在三北地區是主要的經濟樹種與綠化樹種,但是其生長發育經常受到干旱、鹽堿等逆境脅迫的限制[1]。因此對于研究大青楊抗逆的基因工程會越來越受到重視。
由于植物的固著性,它們無法移動以避免不利的環境,因此它們不得不應對各種惡劣的環境因素。為了在這些應激源中生存,植物發展出各種自我保護機制,例如合成具有不同功能的功能蛋白。功能蛋白的表達主要受特異性轉錄因子的控制。轉錄因子編碼基因被認為是導致植物多樣性和進化的重要原因之一。轉錄因子及其同源轉錄因子結合位點的鑒定對于操縱調控網絡以獲得目標分子所需的特性是十分必要的[2]。此外,轉錄因子通過對轉錄起始率的控制來調節基因表達,進而調節機體的生長發育[3]。
基本區域亮氨酸拉鏈(bZIP)家族是最保守的轉錄因子家族之一,并廣泛分布于多數真核生物中。迄今為止,它們已被發現廣泛存在擬南芥、水稻、番茄、玉米、高粱、胡蘿卜等多種植物[4~10]。bZIP轉錄因子根據bZIP保守域命名,該域由60~80個氨基酸組成,包含兩個功能區域:基本區域和亮氨酸拉鏈[11]。根據堿性結構域以及其他保守的結構域,同時依照擬南芥的77個bZIP類轉錄因子基因家族成員劃分的A、B、C、D、E、F、G、H、I和S類10個亞家族[12],有報道將214條楊樹bZIP轉錄因子根據其保守域氨基酸序列劃分為A、B、C、D、E、F、G、H、I、S及其他等11個亞族[1]。
bZIP轉錄因子作為關鍵的調控因子在植物的各種生物過程和應激反應中起作用[13]。GEN研究發現bZIP轉錄因子參與了植物對生物脅迫和非生物脅迫的應激反應,包括病原體防御[14~15],激素和糖信號[16~17],光響應[18~19],耐鹽耐旱[8,20]等。例如,在擬南芥(A.thaliana)中,轉錄因子AtbZIP1-AtbZIP75可以調控多種生物過程,如脅迫信號,光信號轉導[21]。玉米ZmbZIP72基因在擬南芥中過表達提高了非生物脅迫的耐受性[22]。在大豆中GmbZIPs,表現出明顯的抗鹽性和抗旱性[23]。此外,bZIP轉錄因子家族bZIP73的提高了水稻在寒冷的氣候條件下的適應性[24]。bZIP家族的FmABI5基因參與了水曲柳對高鹽、干旱、低溫與甘露醇的響應[25]。BebZIP2和BebZIP6基因參與了慈竹對冷和干旱等非生物脅迫的響應[26]。
近年來,關于bZIP轉錄因子的研究主要集中在農作物和草本植物中,但是對于木本植物的研究較少[1]。本文以木本植物大青楊為對象,利用生物信息學技術分析預測bZIP1基因的功能及結構,并分析了該基因在滲透脅迫下的表達模式。
1 材料與方法
1.1 植物材料
采用本實驗室保存的無菌的大青楊組培苗,放置在25℃組培室,光周期為16 h/8 h,培養基為1/2MS培養基,直至株高8~10 cm用于以下試驗。
1.2 實驗方法
1.2.1 大青楊RNA的提取及cDNA的合成
將植物材料放入液氮中研磨,利用北京諾博萊德科技有限公司供應的QIAGEN RNeasy?Plant Mini Kit(50),提取大青楊總RNA,使用南京諾唯贊生物生產的HiScript? Ⅱ Q RT SuperMix for qPCR(+gDNA wiper)將其反轉錄成cDNA。反轉錄成功后的cDNA放置在-20℃冰箱內保存備用。
1.2.2 大青楊PubZIP1基因的克隆
根據本實驗室前期對干旱脅迫下大青楊轉錄組測序分析,篩選獲得多個干旱響應的bZIP轉錄因子成員。本研究對其中與毛果楊基因Potri.005G170500.1同源的一個成員做進一步的功能研究,并將其命名為PubZIP1(accession number:MG387125)。
在PubZIP1基因序列兩端分別設計引物,由庫美生物公司合成正向引物F:5′-GAGATTCTTTTAGAGCATG-3′以及反向引物R:5′-GACTAAGTGGGTTCACGAGG-3′。以提取的大青楊RNA反轉錄的cDNA為模版,利用購自北京全式金生物技術有限公司2×Easy Taq PCR SuperMix克隆試劑盒進行PCR,克隆出基因的全長片段。PCR總反應體系為20 μL,其中cDNA模版1 μL、正反向引物各1 μL、2×Easy Taq PCR SuperMix酶10 μL、去離子水7 μL。PCR反應條件為:94℃預變性2 min,94℃變性30 s,60℃退火30 s,72℃延伸30 s,共38個循環,72℃延伸2 min。電泳檢測目的條帶正確后,使用購自康為世紀的快速瓊脂糖凝膠DNA回收試劑盒對PCR擴增產物進行膠回收,具體步驟見試劑盒說明書。
PCR產物經過1.2%瓊脂糖凝膠電泳,使用DL2000 DNA Marker目的片段比對,利用購自北京康為世紀生物科技有限公司的快速瓊脂糖凝膠DNA回收試劑盒膠回收目的片段。連接到PEASY-T1載體,利用熱激法轉化大腸桿菌,菌液涂板,次日挑取單克隆,搖菌送測序。
1.2.3 植物表達載體pCAMBIA1300-PubZIP1-GFP的構建
1.2.3.1 設計載體的雙酶切引物
通過分析楊樹PubZIP1基因全長序列及pCAMBIA1300-sGFP載體圖譜設計帶有酶切位點的引物。在載體圖譜的多克隆位點中找出載體全部可用的酶切位點,以確定可供選擇的限制性內切酶,然后將楊樹PubZIP1基因全長CDS序列輸入到BioEdit軟件中,通過軟件分析的結果得出該基因不可用的酶切位點,選取二者共有的限制性內切酶作為最終酶切位點。本實驗選取BamHⅠ和SalⅠ兩個酶切位點,正向酶切引物F:GCGGATCCATGAGCTCTACGTCAACTC,反向酶切引物R:GCGTCGACTAGTGGGTTCACGAGGACAAG。
1.2.3.2 目的基因PCR擴增及膠回收
酶切引物合成后,以連接PEASY-T1-PubZIP1陽性質粒為模板PCR擴增。在120 V電壓下,電泳檢測目的條帶是否正確,若條帶正確則利用購自北京全式金生物技術有限公司2×Easy Taq PCR SuperMix克隆試劑盒進行PCR擴增,然后使用快速瓊脂糖凝膠回收試劑盒對其進行膠回收,并按照康為世紀公司的快速DNA產物純化試劑盒說明書的操作步驟進行純化。
1.2.3.3 pCAMBIA1300-GFP質粒的提取
在無菌的150 mL小瓶中加入20 mL含50 mg·mL-1的卡那霉素(Kan)LB液體培養基,再加入15 μL轉入pCAMBIA1300-GFP質粒的大腸桿菌菌液,放置于37℃搖床上160 r·min-1震蕩培養14 h,使用北京康為世紀生物科技有限公司的質粒小提試劑盒提取pCAMBIA1300-GFP質粒。
1.2.3.4 目的基因與載體的雙酶切反應及純化
將膠回收產物和pCAMBIA1300-GFP質粒同時進行BamH/SalⅠ雙酶切,酶切體系如下表4(100 μL):反應條件為37℃,酶切反應時間為2~3 h。最后使用康為世紀公司的快速DNA產物純化試劑盒對酶切產物分別進行純化。雙酶切總反應體系為100 μL,其中pCAMBIA1300-GFP質粒50 μL、0×T Buffer 10 μL、bamH 5 μL、SalⅠ 5 μL、去離子水30 μL。
1.2.3.5 大腸桿菌的轉化及PCR檢測
將連接產物通過熱激法轉化到大腸桿菌DH5α中,抽取200 μL菌液到無菌的含50 mg·L-1Kan的LB固體培養基平板中,用酒精燈燒過的無菌涂棒涂至均勻。在37℃搖床中倒置過夜培養后,用無菌的鑷子夾取無菌的小槍頭挑取單克隆菌落搖菌,然后進行菌液PCR及電泳檢測,并將陽性的菌液抽取500 μL送去公司測序,剩余菌液和50%甘油按1∶1比例混勻加入無菌的1.5 mL離心管中,充分混勻放入液氮快速冷凝,-80℃冷凍保存。測序比對正確后的菌液使用高純度質粒小提試劑盒提取質粒pCAMBIA1300-PubZIP1-GFP。
1.2.4 大青楊PubZIP1基因生物信息學及進化樹分析
利用NCBI(https://www.ncbi.nlm.nih.gov/orffinder/)在線軟件分析大青楊PubZIP1基因的開放閱讀框;通過http://www.bio-soft.net/sms/index.html查詢該基因的同源核苷酸及氨基酸序列;利用ExPASy服務器中的Protparam(https://web.expasy.org/protparam/)工具分析蛋白的理化性質;根據ExPASy(https://web.expasy.org/protscale/)服務器中的ProtScale程序分析大青楊PubZIP1蛋白的疏水性;通過在線軟件NPS@(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_gor4.pl)預測蛋白的二級結構。通過TMHMM Server v.2.0對蛋白進行跨膜區分析;使用SignalP 3.0 server(http://www.cbs.dtu.dk/services/SignalP-3.0/)對蛋白進行信號肽預測,并預測該蛋白質N端信號肽的有無及其酶切位點;使用COILS Server對肝蛋白卷曲螺旋預測分析;NCBI在線預測蛋白結構域;通過http://multalin.toulouse.inra.fr/multalin/multalin.html進行氨基酸多序列比對;通過http://multalin.toulouse.inra.fr/multalin/cgi-bin/multalin.pl網址進行氨基酸結構比對;利用MEGA5.05軟件采用鄰接法構建系統進化樹。
1.2.5 亞細胞定位
構建PubZIP1基因融合綠色熒光蛋白(GFP)瞬時表達載體(pBI121-PubZIP1-GFP),并利用氯化銫—溴化乙錠超高速梯度密度離心方法進行質粒提取。將提取的質粒包裹金粉,用基因槍(PDS-1000/He Particle Delivery System)轟擊洋蔥表皮,并用激光共聚焦顯微鏡(Olympus-FV1000MPE)觀察亞細胞定位情況。
1.2.6 大青楊PubZIP1基因的表達模式分析
采用1/2MS固體培養基,將大青楊幼苗培養24 d左右,選擇生長良好且一致的5 cm高的組培苗作為試驗材料。
分別提取3株未受脅迫的生長狀況良好的大青楊的根莖葉的RNA,反轉錄成cDNA,以根為對照組,使用大青楊PtrActin基因作為內參基因,設計內參引物由庫美生物公司合成正向引物PtrActin-F:5′-TGTTGCCCTTGACTATGAGCAGGA-3′以及反向引物PtrActin-R:5′-ACGGAATCTCTCAGCTCCAATGGT-3′,長度為165 bp。
將生長良好、狀態相似的大青楊組培苗插入添加7% PEG6000的1/2MS固體培養基中模擬干旱脅迫,在脅迫0、6、12、24 h時分別提取根莖葉部位的RNA,反轉錄成cDNA(每個脅迫時間段選取3個獨立植株)。以未處理的根部的表達量為對照組,分別用提取的各個脅迫時間段的cDNA為模版,通過熒光定量PCR儀(Applied Biosystems,Foster City,CA,USA)使用南京諾唯贊生物生產的ChamQTMUniversal SYBR? qPCR Master Mix進行qRT-PCR分析,用2-ΔΔCT計算法計算其相對表達量。PCR總反應體系為20 μL,其中cDNA模版1 μL、正反向引物各1 μL、2×Easy Taq PCR SuperMix酶10 μL、去離子水7 μL。
2 結果與分析
2.1 大青楊PubZIP1基因的克隆
以大青楊為材料分別提取根、莖、葉和總RNA(圖1)。利用總RNA反轉錄成cDNA,以2000 Marker為對照,擴增該基因的CDS序列,擴增產物長度為1 083 bp(圖2)。
圖1 大青楊RNA的提取 M. DNA Mareker DL2000;1~5.RNA產物Fig.1 The RNA production of P.ussuriensis Kom. M.DNA Mareker DL2000;1-5.RNA production
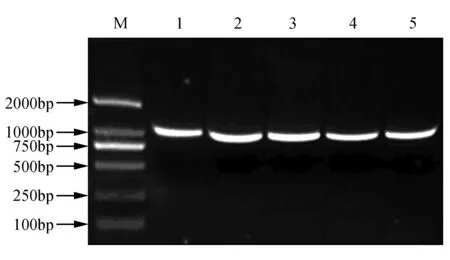
圖2 大青楊PubZIP1基因的克隆 M. DNA Mareker DL2000;1~5.PubZIP1克隆產物Fig.2 PCR production of PubZIP1 M.DNA Mareker DL2000;1-5.PCR production of PubZIP1
2.2 大青楊PubZIP1基因編碼的氨基酸序列及其理化特性
2.2.1 大青楊PubZIP1基因編碼的氨基酸序列
通過在線查詢得知大青楊PubZIP1基因共編碼360個氨基酸(圖3)。利用ExPASy服務器中的Protparam工具分析蛋白的理化性質,發現氨基酸的數量為360,分子質量為40 645.82,理論等電子點為6.68,總共包括5 670個原子,分子式為C1772H2812N530O543S13,在組成蛋白的20種氨基酸中,亮氨酸(Leu)所占比例最高,占10.8%,(Cys)所占比例最低,占0.3%,不包括含硒半胱氨酸(Sec)和吡咯賴氨酸(Pyl)。蛋白的不穩定指數為47.52,脂肪指數為81.67,總平均親水性為-0.531。
2.2.2 大青楊PubZIP1基因的理化特性2.2.2.1 大青楊PubZIP1基因編碼的蛋白疏水性
根據ExPASy服務器中的ProtScale程序分析大青楊PubZIP1蛋白的疏水性,計算基于K-D法的蛋白質疏水性。分析結果發現,第285位的氨基酸為纈氨酸(Val),疏水性最大,分值為1.833;第70位的氨基酸為谷氨酸(Glu),親水性最大,分值為-2.522。根據圖4中數據,分值為負值的氨基酸數量大于分值為正值的氨基酸數量,表示親水性大于疏水性,因而推測該蛋白為親水性蛋白。
2.2.2.2 大青楊PubZIP1基因編碼的蛋白的二級結構及功能結構域預測
通過在線預測蛋白的二級結構,如圖5所示該蛋白的二級結構主要是由α-螺旋(62.50%),無規卷曲(29.72%),延伸鏈(5.56%),β-折疊(2.22%)組成。
2.2.2.3 對蛋白跨膜區進行分析
通過TMHMM Server v.2.0對蛋白進行跨膜區分析(圖6)。發現細胞膜表面并沒有典型的跨膜螺旋區,也沒有氨基酸位于細胞膜內,參考該蛋白的疏水性區域分析結果,表明該蛋白并不是一個與細胞信號傳導有關的膜受體蛋白。

圖3 大青楊PubZIP1基因序列及其推導的氨基酸序列Fig.3 Nucleotide and deduced polypeptide sequences of PubZIP1

圖4 PubZIP1蛋白的疏水性預測Fig.4 Hydrophobicity analysis prediction PubZIP1 protein
2.2.2.4 蛋白信號肽及蛋白卷曲螺旋預測分析
使用SignalP 3.0 server(http://www.cbs.dtu.dk/services/SignalP-3.0/)對蛋白進行信號肽預測,預測蛋白質N端沒有信號肽。預測結果如圖7所示。使用COILS Server對蛋白卷曲螺旋預測分析,分析結果如圖8所示,檢測不到該蛋白具有卷曲螺旋結構。
2.2.2.5 蛋白結構域預測
通過NCBI在線分析蛋白的功能結構域,分析結果如圖9所示。經過分析得出該蛋白包括bZIP結構域和DOG1結構域。bZIP結構域由第163到239的77個氨基酸構成bZIP結構域,bZIP結構域廣泛存在于植物蛋白中。它們在人屬和異質二聚體的網絡中起作用,調節一系列不同的細胞過程,包括細胞存活、學習和記憶、脂質代謝和癌癥進展等。它們在對刺激或壓力信號的反應中扮演重要角色,如細胞因子、基因毒性藥物或生理應激。DOG1結構域由第77到129的53個氨基酸組成。
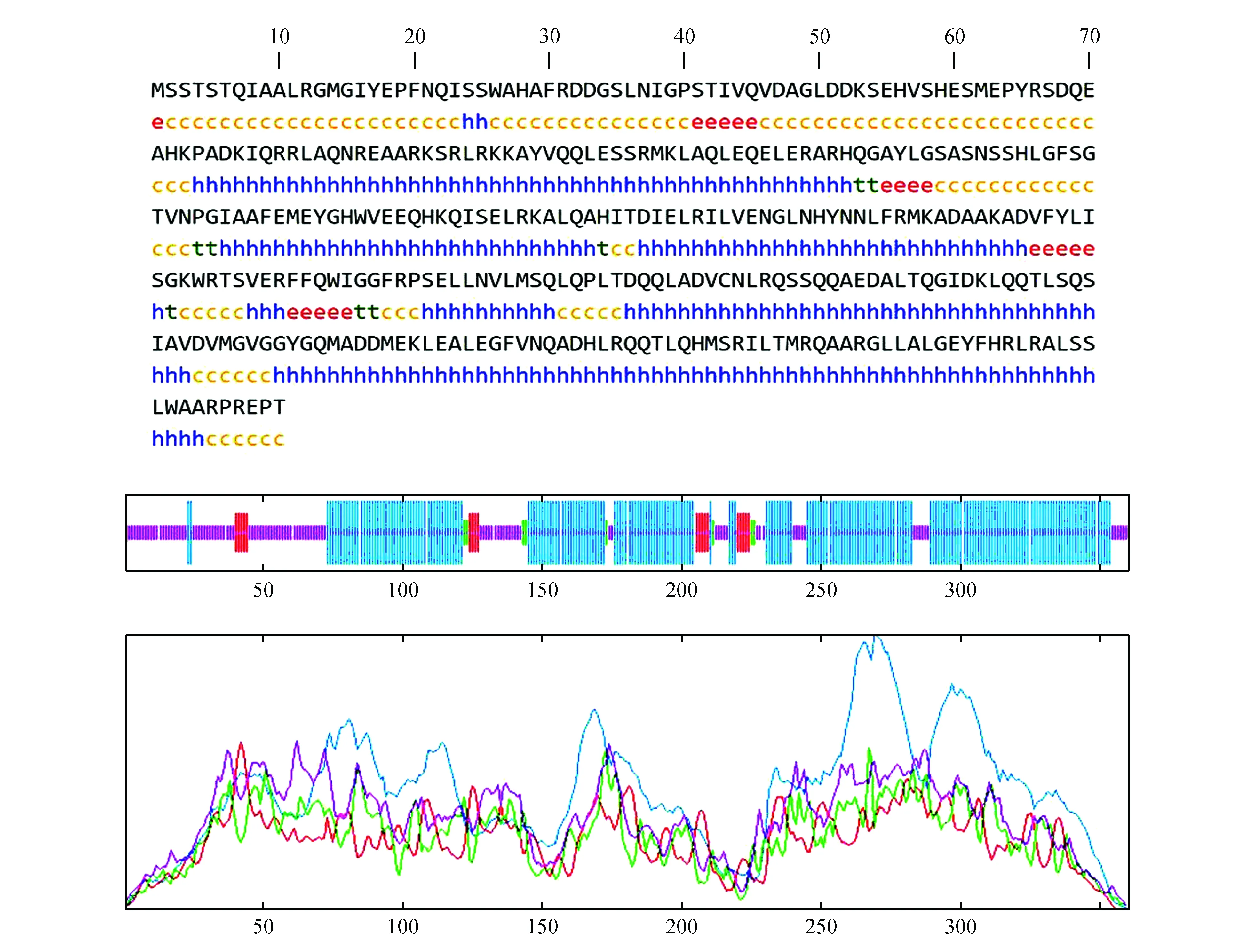
圖5 PubZIP1蛋白的二級結構預測Fig.5 Predicted secondary structure prediction of PubZIP1 protein
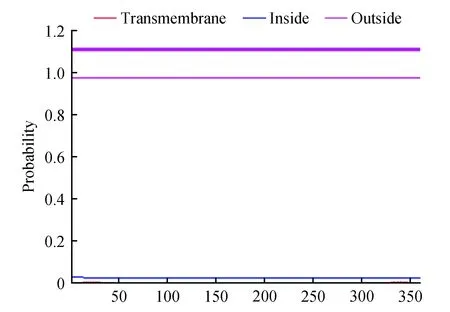
圖6 PubZIP1蛋白的跨膜區分析Fig.6 Transmembrane region prediction of PubZIP1 protein
2.2.2.6 氨基酸序列比對及系統進化樹的構建
通過在線比對氨基酸序列,將大青楊PubZIP1基因推導的氨基酸序列與其他植物相同基因的氨基酸序列進行同源性比對,結果顯示,大青楊(Populusussuriensis)PubZIP1與棉花(Gossypiumhirsutum)、煙草(Nicotianatabacum)、甜椒(Capsicumannuum)、擬南芥(Arabidopsisthaliana)等物種同源蛋白序列有高度保守性(圖10)。通過MEGA5.05軟件進行系統進化樹的構建(圖11),大青楊(Populusussuriensis)與蘿藦(Ricinuscommunis)、紅茄苳(Rhizophoramucronata)有較近的遺傳距離,所以預測他們有更近的親緣關系。而與蒺藜苜蓿(Medicagotruncatula)、大豆(Glycinesoja)、桑樹(Morusnotabilis)、煙草(Nicotianatabacum)、甜椒(Capsicumannuum)遺傳距離較遠,因此預測他們親緣關系更遠。
2.2.2.7 大青楊PubZIP1基因的亞細胞定位實驗結果
利用基因槍分別將金粉包裹的pCAMBIA1300-PubZIP1-GFP質粒和pCAMBIA1300-GFP質粒通過基因槍轟擊洋蔥內表皮,將洋蔥內表皮放置在1/2MS培養基上,暗培養24 h后,通過激光共聚焦顯微鏡觀察發現(圖12),對照pCAMBIA1300-GFP空載體在整個洋蔥細胞均能看到綠色熒光,為組成型表達;pCAMBIA1300-PubZIP1-GFP的融合表達載體在35S啟動下PubZIP1-GFP融合蛋白發出熒光,這進一步證明PubZIP1定位在細胞核中,具有轉錄因子的一般特征。
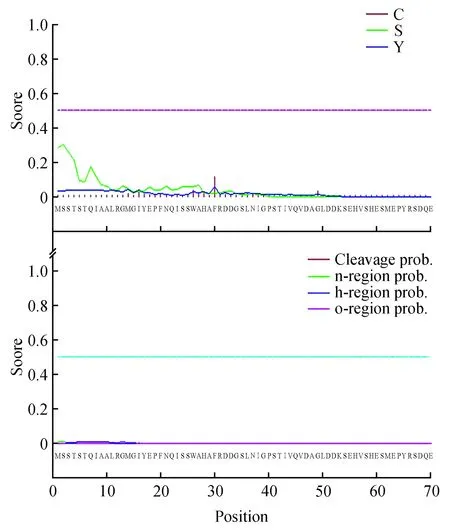
圖7 PubZIP1蛋白的信號肽預測Fig.7 Signal peptide prediction of the PubZIP1 protein
圖8 PubZIP1蛋白的卷曲螺旋預測Fig.8 Coiled coils prediction of PubZIP1 protein

圖10 大青楊PubZIP1氨基酸與其他植物氨基酸序列比對(框選為保守區域)Fig.10 P.ussuriensis alignment analysis of PubZIP1 conservative structure domain in different plants(boxes selected as conservative areas)
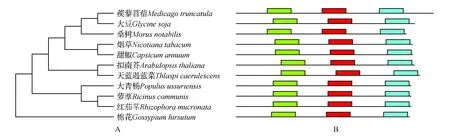
圖11 不同植物PubZIP1基因的系統進化樹(鄰接法)及保守區域 A.系統進化樹;B.氨基酸序列保守區域Fig.11 Molecular phylogenetic tree analysis of PubZIP1 in plant species A.Molecular phylogenetic tree; B.The conserved region of amino acid sequence
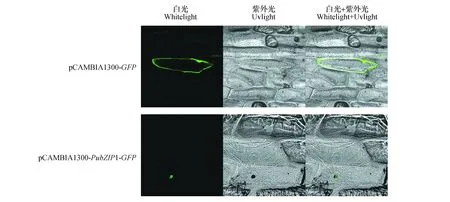
圖12 大青楊PubZIP1基因的亞細胞定位Fig.12 Subcellular localization analysis of the PubZIP1 gene
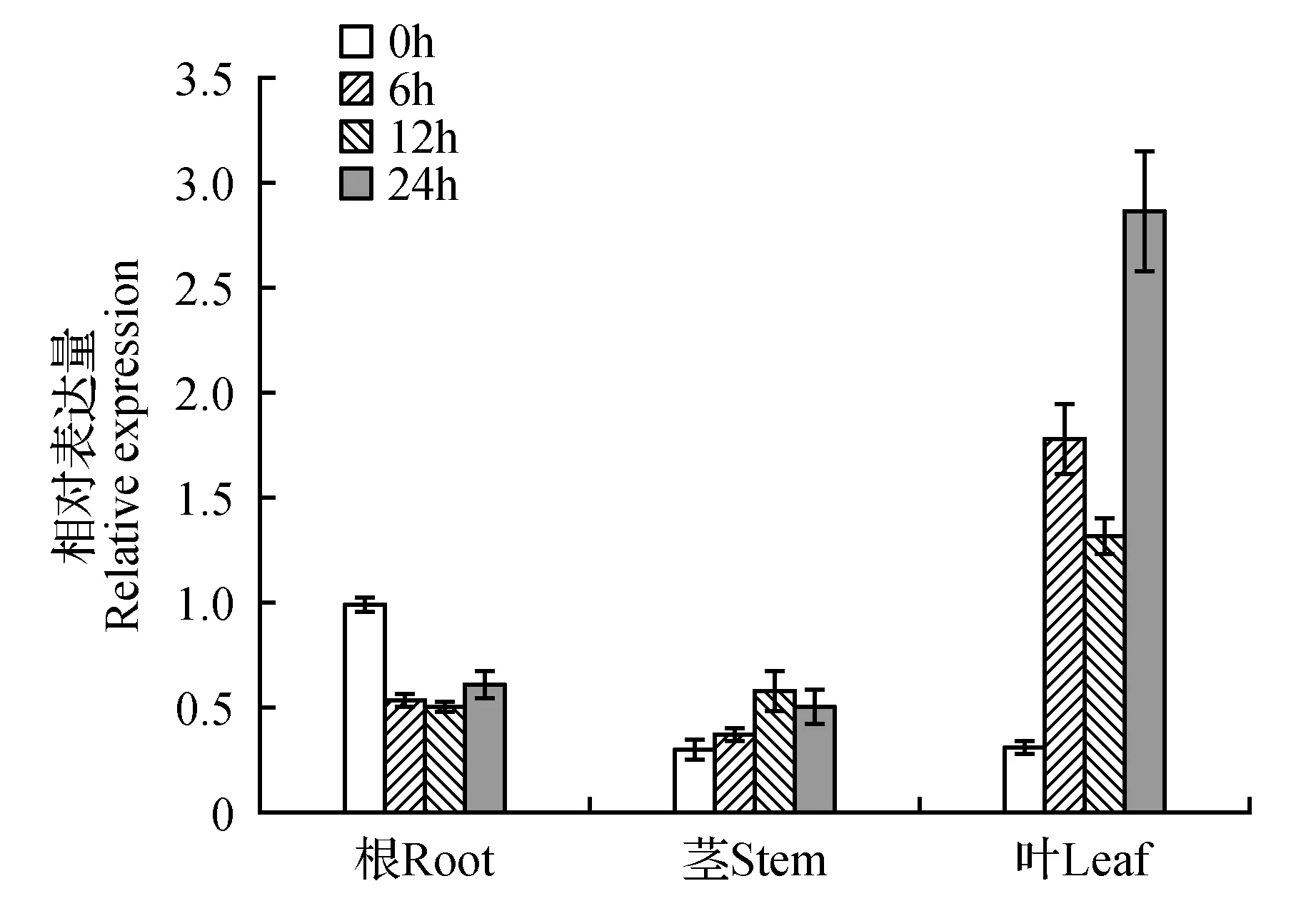
圖13 大青楊PubZIP1基因在PEG6000處理下不同時間段的組織特異性表達情況Fig.13 Quantitative real-time PCR analysis of the expression pattern of PubZIP1 gene in different tissues after 7% PEG6000 treatment
2.2.2.8 大青楊PubZIP1基因的表達模式分析
分別檢測PubZIP1基因在不同PEG6000脅迫時間下大青楊根、莖、葉中的表達量(圖13),以脅迫0 h的PubZIP1基因在大青楊根部中的表達量為對照組,分析結果表明脅迫后PubZIP1基因在大青楊根中的表達量呈下降趨勢。而在大青楊莖和葉片中的表達量呈上升趨勢,尤其是在葉片中明顯被誘導表達。預測該基因可能主要在葉片中表達并行使功能。
3 討論
植物的生長和發育經常受到各種不利的生物和非生物脅迫(如干旱、高鹽和低溫)的影響。植物本質上是固著的,因此它們發展出一系列有效的機制來感知并對這些環境壓力做出充分的反應,例如,通過轉錄因子與它們的啟動因子中存在的特定順式作用元件結合,對應激反應基因進行轉錄調控。本研究從大青楊中克隆獲得PubZIP1轉錄因子,該基因包括有1 083 bp的CDS序列,共編碼360個氨基酸。通過對PubZIP1氨基酸序列進行二級結構預測及保守區域可知,分析得出該蛋白包括bZIP結構域和DOG1結構域。通過預測不含有指導蛋白轉移的信號肽和跨膜運輸結構。蛋白的二級結構主要是由α-螺旋(62.50%)、無規卷曲(29.72%),構成,其次還有少量延伸鏈(5.56%),β-折疊(2.22%)。通過亞細胞定位技術,證明該基因位于細胞核中。
研究發現,bZIP在不同植物中參與抗旱脅迫,例如:小麥中bZIP類轉錄因子編碼的基因Wa-bi5,其表達量在干旱脅迫下明顯上調,轉Wa-bi5基因的煙草對干旱的耐受性也顯著增強[27];在擬南芥中bZIP轉錄因子ABF3/ABF4可以提高植株的抗旱性[28];在大豆中克隆的GmbZIP-32基因在干旱和高鹽脅迫下表現出不同程度的響應應答[29];桑樹MnbZIP基因屬于bZIP轉錄因子,通過熒光定量PCR法對MnbZIP在干旱、高鹽和低溫條件下的表達量進行分析,結果表明,該基因表達量在這幾種脅迫下都有上調[30]。我們對生長狀態相同的大青楊分別使用模擬抗旱的7% PEG6000處理不同時間,通過實時熒光定量PCR法分析PubZIP1在PEG6000脅迫下的表達情況。結果表明,在PEG處理下PubZIP1在葉片部位和莖部的表達量明顯被誘導上調,在根部的表達量明顯被誘導下調。表明PubZIP1可能參與了大青楊對滲透脅迫的信號傳導,屬于逆境脅迫下的調控因子。
本研究初步證明大青楊PubZIP1基因具有一定的抗旱能力,但是關于其信號傳導與調控機制的研究需進一步的研究。由于大青楊物種的基因組測序并未完成,所以需要進一步的研究發現更多的基因組信息及其結構和功能。本文從大青楊中克隆出PubZIP1基因,并通過生物信息學和非生物因素脅迫等方法預測基因結構和功能。不僅為大青楊基因組的測序完成提供更多的數據,同時為大青楊bZIP1基因的抗旱研究提供理論基礎。

