山茱萸bHLH2基因的克隆與分析
李志紅 楊萌萌 徐靜雅 凡貞潔 王瑤瑤 王龍 侯典云
摘要?目的:獲得山茱萸轉錄因子CobHLH2的cDNA序列。方法:以山茱萸轉錄組測序數據中的c101916_g1序列為參考,利用Primer Premier 5.0軟件在其開放閱讀框兩端設計特異性引物,把山茱萸果實總RNA反轉錄獲得的cDNA作模板,通過PCR擴增、純化回收和克隆測序,獲得CobHLH2序列,并經生物信息學分析初步了解其基本結構與功能。結果:CobHLH2的ORF長度為795 bp,共編碼264個氨基酸,為親水性蛋白。CobHLH2蛋白的二級結構以無規卷曲和α-螺旋為主,有少量的β-轉角和延伸鏈。多序列比對和系統進化樹分析結果表明,CobHLH2的氨基酸序列與獼猴桃和茶樹的bHLH類轉錄因子同源性較高。結論:首次獲得山茱萸CobHLH2轉錄因子的cDNA序列,并進行了生物信息學分析,為深入研究bHLH類轉錄因子的結構和功能奠定基礎。
關鍵詞?山茱萸;轉錄組;轉錄因子;CobHLH2;基因;克隆;生物信息學;分析
Cloning and Analysis of Transcription CobHLH2 Genes in Cornus officinalis
LI Zhihong1, 2, YANG Mengmeng1, 2, XU Jingya1, 2, FAN Zhenjie1, 2, WANG Yaoyao1, 2, WANG Long1, 2, HOU Dianyun1, 2
(1 College of Agricultural, Henan University of Science and Technology, Luoyang 471003, China; 2 The Luoyang Engineering Research Center of Breeding and Utilization of Dao-di Herbs, Luoyang 471023, China)
Abstract?Objective:To obtain the cDNA sequence of CobHLH2 transcription factor in Cornus officinalis.Methods:Based on the c101916_g1 sequence from the transcriptome data of C.officinalis as the reference, a pair of specific primers were designed at the 2 ends of its open reading frame using Primer Premier 5.0 software.The total RNA of Cornus officinalis fruits was extracted and reversed transcribed into cDNA as the model.The CobHLH2 sequence was obtained by PCR amplification, purification recovery and clone sequencing.Its basic structure and function through bioinformatics analysis were understood.Results:The ORF of CobHLH2 was 795 bp in length, encoding 264 amino acids and it was a hydrophilic protein.The secondary structure of CobHLH2 protein mainly was Random curl and α-helix, and β-turns and extended chains were less.The analysis of multiple sequence alignments and phylogenetic trees showed that the amino acids sequence of CobHLH2 shared high homology with bHLH transcription factors of Camellia sinensis and Actinidia chinensis.Conclusion:The cDNA sequence of CobHLH2 from C.officinalis was cloned for the first time and bioinformatics analysis was performed, which would lay the foundation for further study on the structure and function of bHLH transcription factors.
Keywords?Cornus officinalis; Transcriptome; Transcription factors; CobHLH2; Gene; Cloning; Bioinformatics; Analysis
中圖分類號:R284文獻標識碼:Adoi:10.3969/j.issn.1673-7202.2020.05.007
山茱萸(Cornus officinalis)為山茱萸科多年生的木本植物,《中華人民共和國藥典》規定山茱萸以干燥的成熟果肉入藥,具有補益肝腎的功效[1]。山茱萸廣泛分布在中國的秦嶺、伏牛山等地[2]。山茱萸的果實中含有多種活性物質,如多糖、環烯醚萜苷類、黃酮類和鞣質等[3]。研究表明,山茱萸具有廣泛的藥理活性,如保護肝腎、抗糖尿病、抗氧化、抗腫瘤、抗炎、抗衰老等[4-7]。現今山茱萸在臨床上的應用已十分廣泛。
bHLH(basic Helix-loop-helix)類轉錄因子的C端含有α-螺旋-環-α-螺旋結構,N端則由堿性氨基酸組成,因此得名[8]。HLH區結構富含疏水性氨基酸,主要是與DNA結合從而發揮功能[9]。bHLH類轉錄因子廣泛存在于真核生物,是植物中最大的轉錄因子家族之一。bHLH轉錄因子家族成員對植物生長過程中起著重要的調控作用[10],如調節花青素的物質合成[11]、植物抗逆性[12]、腺毛或根毛的形成以及表皮細胞建成[13-14]等生理作用。MYC蛋白的主要是由2個bHLH蛋白聚合形成的[15-16],2個結構域共同參與了活性DNA結合復合物的形成[17]。有研究表明,bHLH轉錄因子在植物體內還可與MYB轉錄因子協同發揮作用,可調控黃酮類物質的合成[18]。完整的bHLH蛋白還可與缺少堿性區域的bHLH蛋白結合形成二聚體,從而抑制目的基因的表達[19]。
眾多研究發現bHLH類轉錄因子對黃酮類、生物堿類、萜類等物質的合成有重要的調控作用。CsbHLHs和CsMYBs轉錄因子在山茶生長過程中有特定的表達模式用以調節重要的類黃酮物質的合成,有助于山茶形成獨特的風味[20]。丹參SmbHLH3的過表達植株使得丹參酮等活性物質含量減少,功能鑒定結果表明SmbHLH3可通過抑制丹參酮合成關鍵酶的轉錄而下調其在植物體內的積累[21]。PbbHLH4過表達可顯著增加蝴蝶蘭中揮發性單萜物質的合成和積累,顯著增加了該蘭花的香氣[22]。
本研究根據山茱萸轉錄組測序結果[23],成功克隆了山茱萸CobHLH2基因,并利用生物信息學分析該基因序列及其編碼蛋白,為研究轉錄因子bHLH轉錄因子在山茱萸生長代謝過程中的功能奠定了基礎。
1?材料與方法
1.1?材料
試驗材料選自河南省洛陽市隋唐城遺址植物園,用蒸餾水清洗果實,快速分離果肉置于離心管中,液氮速凍后儲存在-70 ℃超低溫冰箱內。
1.2?試劑與儀器
EASYspinPlus多糖多酚植物總RNA快速提取試劑盒(RN37)、TRUEscript 1st Strand cDNA Synthesis Kit(PC1802)、2×long Taq PCR Master Mix(PC1501)、瓊脂糖凝膠純化回收試劑盒(DR01)與Zero Background pTOPO-TA Cloning Kit(CV2101)均購買自艾德萊生物科技;DH5α感受態細胞(上海昂羽生物公司,型號:AYBIO-G6016);氨芐青霉素(北京索萊寶科技有限公司,型號:A7490)。Thermo MultiskanGO酶標儀(Thermo公司,美國,型號:MultiskanGO)、PCR儀(Biometra公司,德國,型號:Tpersonal)、臺式高速冷凍離心機(Sigma公司,德國,型號:3K15)、電泳儀(BIO-RAD公司,美國,型號:PowerPac Basic)。
1.3?引物設計
以山茱萸轉錄組數據篩選得到的Unigene c101916_g1序列為參考,利用Primer Premier 5.0軟件設計特異引物,引物序列為CobHLH2F:5′-TCGGTGCTTATTTCGGTTGG-3′ 和CobHLH2R:5′-CTCCCTACTACTTCCCCACT-3′,引物送華大基因有限公司合成。
1.4?總RNA提取與cDNA合成
將試驗材料放置于液氮中并充分研磨,根據RN35-EASYspinPlus多糖多酚植物總RNA快速提取試劑盒的說明提取山茱萸的總RNA。取適量總RNA,用1%的瓊脂糖凝膠檢測其完整性,并利用MultiskanGO酶標儀檢測其純度和濃度。按照TRUEscript 1st Strand cDNA Synthesis Kit的說明書將總RNA逆轉錄成cDNA,-20 ℃保存備用。
1.5?CobHLH2基因克隆與測序
CobHLH2基因的PCR體系是25 μL,2×long Taq Master Mix 12.5 μL,上下游引物終濃度為0.1 μM,cDNA模板為1.5 μL,ddH2O補足體積。設定的程序為94 ℃預變性5 min,設置32個循環,95 ℃變性30 s,53 ℃退火30 s,72 ℃延伸1 min,循環結束后,72 ℃延伸5 min。待PCR結束后電泳檢測,回收純化目的條帶,與T載體連接后轉化E.coli DH5α感受態細胞,在含有氨芐青霉素(Amp)抗性的LB固體平板上過夜培養,經菌液PCR檢測后送至上海美吉生物公司測序。
1.6?CobHLH2的生物信息學分析
利用美國生物技術信息中心數據庫(NCBI)提供的ORFfinfer和Conserved Domains-search在線分析其開放讀碼框(ORF)及保守結構域;使用ProtParam tool和ProtScale在線分析CobHLH2編碼蛋白的理化性質與親/疏水性;利用SOPMA網站在線分析CobHLH2的二級結構,并利用SWISS-MODEL工具構建其三維模型;利用BLAST搜索與CobHLH2相似性較高的氨基酸序列,并利用DANMAN 8.0進行多序列比對,基于多序列比對結果使用MEGA 7.0構建NJ樹分析其同源性。
2?結果與分析
2.1?山茱萸果實總RNA提取
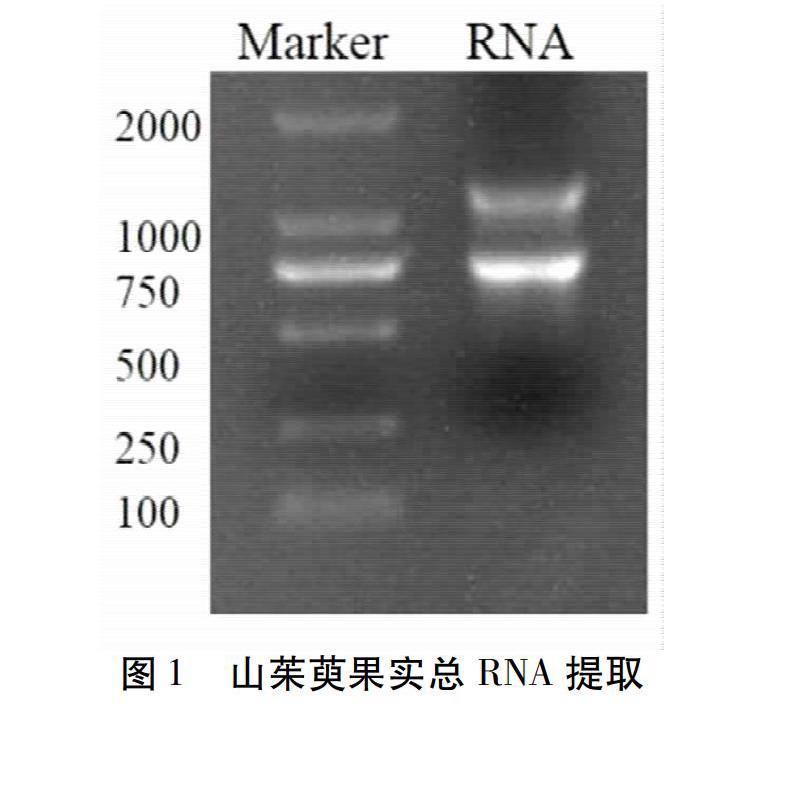
取若干無酶的裝有3個小鋼珠的1.5 mL離心管,向其中加入約100 mg的山茱萸果肉,利用球磨儀將其研磨成粉狀,按照植物總RNA提取試劑盒提取其總RNA。1.2%瓊脂糖凝膠電泳檢測結果見圖1,RNA的條帶清晰,可進行cDNA第一鏈的合成。
2.2?CobHLH2克隆與測序
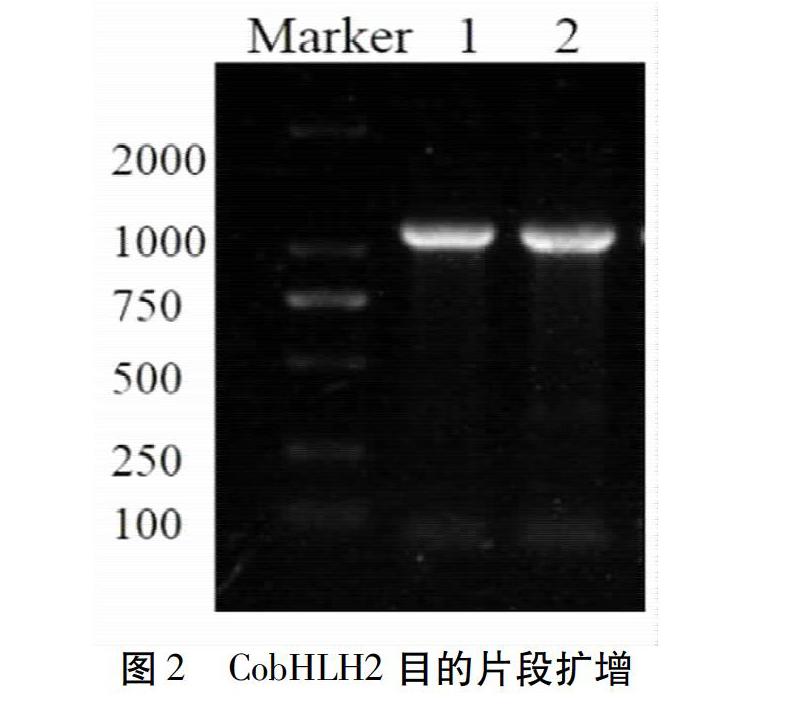
利用Primer Premier 5.0軟件,在轉錄本c101916_g1序列的ORF兩端設計特異性引物(CobHLH2-F和CobHLH2-R)。以山茱萸的第一鏈cDNA為模板,RT-PCR擴增后經1.0%的凝膠電泳檢測,結果見圖2所示。回收純化該亮帶,與載體pTOPO-T連接,轉化感受態DH5α,經菌液PCR鑒定,挑選陽性菌液測序。測序結果去除污染載體序列,獲得長度為1 047 bp的CobHLH2目的片段。
2.3?CobHLH2編碼蛋白的特征分析
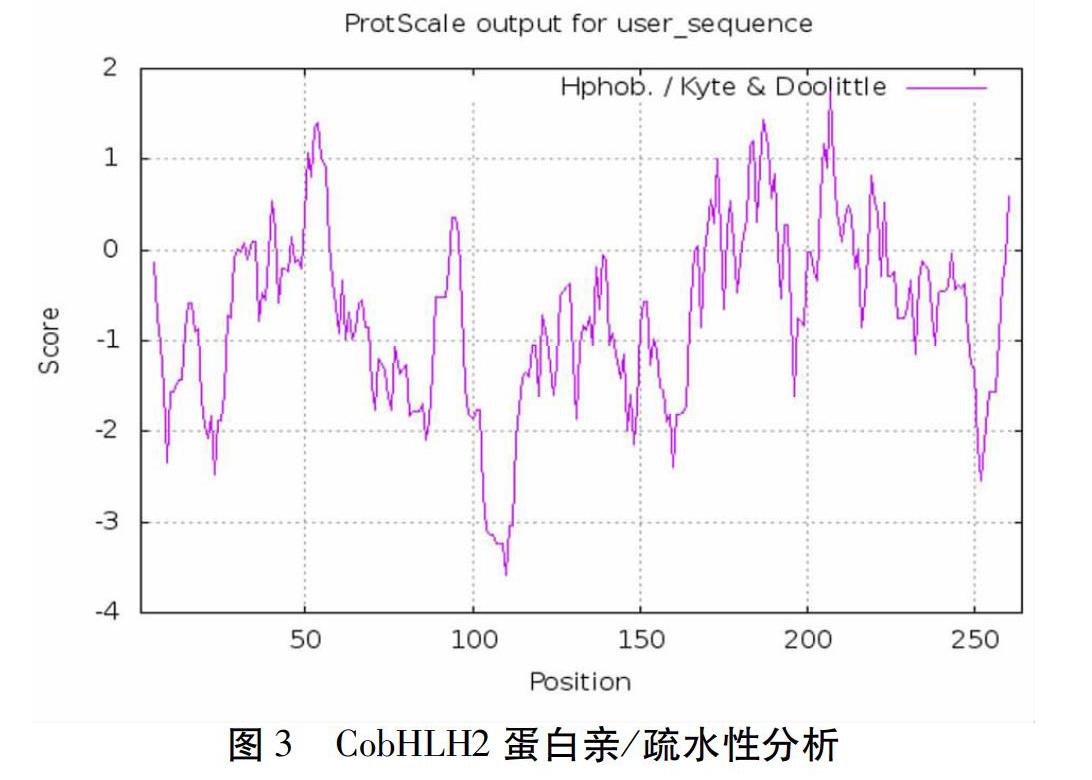
利用NCBI提供的ORFfinder工具預測,CobHLH2序列含有一個完整的ORF,長795 bp,共編碼264個氨基酸。ProtParam分析表明,CobHLH2蛋白分子式為C1268H2030N370O411S8,相對分子質量為29.29 kDa,其理論pI值8.77。利用ProtScale工具分析CobHLH2蛋白的親/疏水性,結果見圖3,總體來看,該蛋白整體的親水性氨基酸數量要多于疏水性氨基酸,因此初步推定此蛋白為親水性蛋白。
利用Conserved Domain-search工具分析CobHLH2蛋白的保守結構域,結果見圖4,CobHLH2蛋白具有HLH結構域和特定的DNA結合位點,表明該編碼蛋白屬于bHLH轉錄因子超家族。
2.4?CobHLH2二級、三級結構預測
利用SOPMA工具在線預測CobHLH2蛋白的二級結構,結果見圖5。CobHLH2蛋白的二級結構共有4種,無規卷曲(紫色)、α-螺旋(藍色)、延伸鏈(紅色)與β-轉角(綠色)所占比例分別為48.48%、33.33%、12.12%、6.06%。
利用SWISS-MODEL工具在線預測構建CobHLH2蛋白的三維結構見圖6。其三維結構主要是α-螺旋與無規卷曲,這與預測的CobHLH2蛋白的二級結構一致。
2.5?CobHLH2的同源性分析
利用NCBI數據庫的BLASTp工具搜索CobHLH2蛋白的相似性較高的序列,序列下載后用DANMAN 8.0軟件進行多序列比對,結果如圖7所示,CobHLH2與藍果樹(Nyssa sinensis,KAA8517024)、葡萄(Vitis vinifera,XP_002282897)、菜豆(Phaseolus vulgaris,XP_007162273)、胡桃(Juglans regia,XP_018858223)、獼猴桃(Actinidia chinensis,PSS36323)、番木瓜(Carica papaya,XP_021894046)、豇豆(Vigna unguiculata,QCD97477)、茶樹(Camellia sinensis,XP_028114859)等序列相似性較高,表明植物bHLH類轉錄因子在進化過程中相對保守。
基于CobHLH2的多序列比對結果,利用MEGA 7.0軟件構建CobHLH2的NJ樹,并通過1 000次Bootstrap重復檢驗系統樹各分支的可信度。結果見圖8,山茱萸CobHLH2序列與獼猴桃(Actinidia chinensis,PSS36323)和茶樹(Camellia sinensis,XP_028114859)的親緣關系較近,而與幾個豆科植物的親緣關系較遠。
3?討論
藥用植物的提取物或其衍生物是許多化學藥物的重要原料,其中多數已是臨床用藥[24],但關于藥用植物活性物質的生物合成途徑及其調控機制的研究較為緩慢。隨著本草基因組學的提出和快速發展[25],利用全基因組學和轉錄組學測序技術獲得大量的基因組數據,是研究藥用植物功能基因組學的重要基礎[26]。鐵皮石斛[27]、人參[28]等藥用植物活性物質合成的基因組研究已取得了較大的進展。
本研究基于對山茱萸轉錄組測序數據分析的基礎上[23],通過基因克隆,獲得了CobHLH2序列,并進行相關分析。結果表明,CobHLH2序列含有HLH保守結構域和相應的保守序列,與已報道文獻一致[29],屬于bHLH轉錄因子家族。bHLH轉錄因子對藥用植物的有效成分含量具有重要的調節作用,有研究表明MYC2過表達可產生較多的花青素,且轉基因擬南芥植株對茉莉酸甲酯的敏感性高于野生型[30]。白樺BpbHLH4[31]和烏拉爾甘草GubHLH3[32]的表達可影響三萜類及其皂苷等物質的生物合成。長春花CrBIS1亦是bHLH轉錄因子家族中的成員,與單萜類物質吲哚生物堿的生物合成途徑有關[33]。當前,利用高通量轉錄組測序[34]和基因工程等技術,改進藥用植物有效成分合成過程中的調控元件,可極大地提高活性物質的含量,有助于提高藥用植物的經濟價值。
現今,山茱萸的基因組數據較為匱乏,其次生代謝物的生物合成的分子機制與轉錄調控機理的研究也較少。本研究已獲得了山茱萸的CobHLH2序列,并進行了結構特征和同源性分析,對于其功能分析還需深入研究,進一步豐富山茱萸基因組學數據,從而發現山茱萸生長發育過程與次生代謝產物積累的之間的分子機理,為山茱萸活性物質的大量合成積累奠定基礎。
參考文獻
[1]中華人民共和國藥典[S].北京:中國醫藥出版社,2015:27-28.
[2]Bai CK,Cao B,Li GH,et al.Ecological effects on phenotypic,cytological and biochemical diversity of Cornus officinalis germplasm resources in China and USA[J].Biochem Syst Ecol,2014,55:241-248.
[3]Ma W,Wang KJ,Cheng CS,et al.Bioactive compounds from Cornus officinalis fruits and their effects on diabetic nephropathy[J].J Ethnopharmacol,2014,153(3):840-845.
[4]丁霞,王明艷,余宗亮,等.山茱萸中5-羥甲基糠醛的分離鑒定及生物活性研究[J].2008,33(4),392-396,484.
[5]Lee KY,Sung SH,Kim SH,et al.Cognitive-enhancing activity of loganin isolated from C.of-cinalis in scopolamine-induced amnesic mice[J].Arch Pharm Res,2009,32(5):677-683.
[6]周瑞,唐志書,武婧,等.基于抗類風濕關節炎作用評價膜分離技術富集山茱萸抗炎組分的適用性[J].中草藥,2019,50(5):1182-1188.
[7]Huang J,Zhang Y,Dong L,et al.Ethnopharmacology,phytochemistry,and pharmacology of Cornus officinalis Sieb.et Zucc[J].J Ethnopharmacol,2018,213:280-301.
[8]Atchley WR,Fitch WM.A natural classification of the basic helix-loop-helix class of transcription factors[J].Proc Natl Acad Sci U S A,1997,94(10):5172-5176.
[9]Pires N,Dolan L.Origin and diversification of basic-helix-loop-helix proteins in plants[J].Mol Biol Evol,2010,27(4):862-874.
[10]Sonnenfeld MJ,Delvecchio C,Sun X.Analysis of the transcriptional activation domain of the Drosophila tango bHLH-PAS transcription factor[J].Dev Genes Evol,2005,215(5):221-229.
[11]Feng S,Wang Y,Yang S,et al.Anthocyanin biosynthesis in pears is regulated by a R2R3-MYB transcription factor PyMYB10[J].Planta,2010,232(1):245-255.
[12]Anjum SA,Wang L,Farooq M,et al.Methyl jasmonate-induced alteration in lipid peroxidation,antioxidative defence system and yield in soybean under drought[J].J Agron Crop Sci,2011,197(4):296-301.
[13]Fan M,Bai MY,Kim JG,et al.The bHLH transcription factor HBI1 mediates the trade-off between growth and pathogen-associated molecular pattern-triggered immunity in Arabidopsis[J].Plant Cell,2014,26(2):828-841.
[14]Hichri I,Barrieu F,Bogs J,et al.Recent advances in the transcriptional regulation of the flavonoid biosynthetic pathway[J].J Exp Bot,2011,62(8):2465-2483.
[15]Ferré-D′amaré AR,Prendergast GC,Ziff EB,et al.Recognition by Max of its cognate DNA through a dimeric b/HLH/Z domain[J].Nature,1993,363(6424):38-45.
[16]Ferré-D′amaré AR,Pognonec P,Roeder RG,et al.Structure and function of the b/HLH/Z domain of USF[J].EMBO J,1994,13(1):180-189.
[17]Chang WH,LAI AG.Genome-wide analyses of the bHLH superfamily in crustaceans:reappraisal of higher-order groupings and evidence for lineage-specific duplications[J].R Soc Open Sci,2018,5(3):172433.
[18]Nakatsuka T,Haruta KS,Pitaksutheepong C,et al.Identification and Characterization of R2R3-MYB and bHLH Transcription Factors Regulating Anthocyanin Biosynthesis in Gentiana Flowers[J].Plant Cell Physiol,2008,49(12):1818-1829.
[19]Sun H,Fan HJ,Ling HQ.Genome-wide identification and characterization of the bHLH gene family in tomato[J].BMC Genomics,2015,16(1):9.
[20]Zhu J,Xu Q,Zhao S,et al.Comprehensive co-expression analysis provides novel insights into temporal variation of flavonoids in fresh leaves of the tea plant(Camellia sinensis)[J].Plant Sci,2020,290:110306.
[21]Zhang C,Xing B,Yang D,et al.SmbHLH3 acts as a transcription repressor for both phenolic acids and tanshinone biosynthesis in Salvia miltiorrhiza hairy roots[J].Phytochemistry,2020,169:112183.
[22]Chuang YC,Hung YC,Tsai WC,et al.PbbHLH4 regulates floral monoterpene biosynthesis in Phalaenopsis orchids[J].J Exp Bot,2018,69(18):4363-4377.
[23]Hou DY,Shi LC,Yang MM,et al.De novo transcriptomic analysis of leaf and fruit tissue of Cornus officinalis using Illumina platform[J].PloS One,2018,13(2):e0192610.
[24]陳士林,孫永珍,徐江,等.本草基因組計劃研究策略[J].藥學學報,2010,45(7):807-812.
[25]宋經元,徐志超,陳士林.本草基因組學專輯簡介[J].中國科學:生命科學,2018,48(4):349-351.
[26]宋經元,羅紅梅,李春芳,等.丹參藥用模式植物研究探討[J].藥學學報,2013,48(7):1099-1106.
[27]Yan L,Wang X,Liu H,et al.The genome of Dendrobium officinale illuminates the biology of the important traditional Chinese orchid herb[J].Mol Plant,2015,8(6):922-934.
[28]Xu J,Chu Y,Liao B,et al.Panax ginseng genome examination for ginsenoside biosynthesis[J].Gigascience,2017,6(11):1-15.
[29]Zhang X,Luo H,Xu Z,et al.Genome-wide characterisation and analysis of bHLH transcription factors related to tanshinone biosynthesis in Salvia miltiorrhiza[J].Sci Rep,2015,5:11244.
[30]An JP,Li HH,Song LQ,et al.The molecular cloning and functional characterization of MdMYC2,a bHLH transcription factor in apple[J].Plant Physiol Biochem,2016,108:24-31.
[31]Yin J,Li X,Zhan Y,et al.Cloning and expression of BpMYC4 and BpbHLH9 genes and the role of BpbHLH9 in triterpenoid synthesis in birch[J].BMC Plant Biol,2017,17(1):214.
[32]Tamura K,Yoshida K,Hiraoka Y,et al.The basic helix-loop-helix transcription factor GubHLH3 positively regulates soyasaponin biosynthetic genes in Glycyrrhiza uralensis[J].Plant Cell Physiol,2018,59(4):778-791.
[33]Van Moerkercke A,Steensma P,Schweizer F,et al.The bHLH transcription factor BIS1 controls the iridoid branch of the monoterpenoid indole alkaloid pathway in Catharanthus roseus[J].Proc Natl Acad Sci U S A,2015,112(26):8130-8135.
[34]陳延清,胡志剛,劉大會,等.藥用植物冬凌草高通量轉錄組測序與分析[J].中國現代中藥,2018,20(12):1476-1482.
(2020-02-10收稿?責任編輯:徐穎)

