微流控芯片液滴數字化分析用于快速定量檢測細菌
何浩延 黃恩奇 黎柱均 舒博文 徐邦牢 劉大漁
摘要現有細菌定量檢測多依賴專業實驗室,檢測周期較長。針對此問題,本研究基于微流控芯片液滴數字化分析,建立了一種細菌定量檢測方法。采用具有平行液滴分析單元的微流控芯片,其特點在于使用了注射器真空驅動液滴產生方法。借助刃天青顯色反應引起的熒光強度改變,可指示液滴內活性細菌的存在。通過計算細菌陽性液滴的比例,采用泊松分布算法,計算出原始樣品中的細菌密度。實驗結果表明,本方法可在3.5 h內完成細菌定量分析,動態檢測范圍為105~108 CFU/mL,相對標準偏差(RSD)低于5%。本方法具有操作簡便和分析速度快的優勢,有望廣泛用于細菌快速定量檢測。
關鍵詞微流控芯片; 液滴數字化分析; 注射器真空; 細菌; 定量檢測
1引 言
細菌定量檢測對于食品安全、環境監測和疾病診斷均具有重要作用。由于細菌定量檢測經常在資源有限的情況下進行,而且對于檢測時間要求很高,因此需要簡便快速的檢測方法。目前,用于細菌定量檢測的方法主要包括培養計數法[1,2]、熒光定量分析PCR[3]、流式細胞術[4,5]等,以及一些基于新型生物傳感器的檢測方法[6]。這些技術仍存在一定的局限性,如檢測周期較長、操作步驟繁瑣、依賴于大型設備和專業技術人員[7,8]。因此,迫切需要建立操作簡便、分析快速的細菌定量檢測方法。
液滴數字化分析是近年發展起來的一種高精度定量分析方法。液滴數字化分析的原理是利用液滴技術將待測靶標隨機分配到大量相互獨立的微滴中,每個液滴中最多含有一個靶細胞,每個微滴相當于一個獨立的微反應器; 繼而針對待測靶標在微液滴中的直接或間接信號,區分陽性和陰性液滴。陽性微滴判讀為1,陰性微滴判讀為0[9,10],可根據泊松分布[11]原理進行計算,實現待測靶標的絕對定量分析。與傳統的分析技術相比,液滴數字化分析最突出的特點是其具有不依賴標準品的絕對定量分析能力。此外,基于大規模液滴分散體系的數字化分析在檢測靈敏度和定量分析精度等方面也顯著優于傳統定量分析方法。鑒于微流控技術是大規模液滴操控的有利工具,微流控芯片成為數字化液滴分析的主要平臺[11]。
自液滴數字化分析提出以來,此技術被廣泛用于多種類型的定量生物分析中, 包括核酸[12~16]、蛋白[17~19]和細胞分析[20~23],以及數字化細菌分析。得益于微液滴對于反應信號的相對富集作用,以及數字化分析方法的高檢測精度,數字化細菌分析在分析時間和定量分析精度等方面均顯著優于傳統的細菌定量分析。 Boedicker等[24]利用阿爾瑪藍指示細菌活性,結合液滴數字化分析技術,最快可在2 h內檢測出樣品中的活性細菌,并且能夠在7 h內獲得細菌的耐藥性信息。Kang等[25]結合DNA酶傳感器與液滴數字化分析技術,能夠在3 h內得到血液樣品中的細菌定量分析信息,其實際密度與理論值的相關性良好(R2=0.999),測定下限達到1 CFU/mL。Kaushik等[26]結合刃天青顯色原理與液滴數字化分析技術,將細菌與顯色體系限制在約20 pL的液滴中,能夠在1 h內得到細菌的耐藥性信息。Scheler等[27]用含綠色熒光蛋白的大腸桿菌驗證了液滴數字化分析的定量分析能力,與平板計數法結果的相關性良好(R2=0.9964),結合刃天青顯色法原理,在3~4 h得到樣品中細菌的耐藥性信息。
液滴數字化分析不依賴標準品的絕對定量分析能力,以及在靈敏度和定量分析精度方面的優勢,非常有利于現場快速細菌檢測,然而,此技術在該領域的應用還鮮有報道。其主要原因在于微流控芯片上的液滴發生多依賴基于氣壓或注射泵等流體驅動裝置, 這類裝置體積較大且操作繁瑣,難以應用于現場快速檢測[26,28]。相比較而言,正壓驅動液滴發生方式使用廣泛,文獻已經報導了基于負壓的液滴發生方法[29~32]。在負壓驅動的液滴發生體系中,僅需要一個簡單的注射器即可驅動油水兩相液體流動,并發生液滴。這種液滴發生方法操作簡便,無需復雜結構流體控制設備,因而有望將液滴數字化細菌分析拓展到現場快速檢測領域。
微流控芯片技術在生物樣品分析中得到廣泛的應用[33]。 本研究基于負壓驅動液滴微流控芯片,建立了一種細菌定量檢測方法。采用具有平行液滴分析單元的微流控芯片,其特點在于使用了注射器真空驅動的流動聚焦式液滴發生方法[34]。利用單個注射器,即可實現多個分析單元中的快速平行液滴發生和捕獲。以大腸桿菌為模式分析對象,借助刃天青顯色反應,實現數字化液滴細菌定量分析。依據顯色反應陽性液滴的比例,使用泊松分布原理計算原始樣品中的大腸桿菌密度。實驗結果表明,本方法具有操作簡便和分析速度快的優勢,有望用于細菌的現場快速定量檢測。
2實驗部分
2.1儀器與試劑
IX71倒置熒光顯微鏡(日本Olympus公司); 麥氏比濁儀(梅里埃公司); PH030A型干燥箱(上海一恒公司); URE2000/35深紫外光刻機(中國科學院光電技術研究所); KS180EI 超聲波清洗機(寧波海曙科生公司); Spin Master51 甩膠機(上海凱美特公司); PDCM等離子體清洗機(成都銘恒公司)。
H2SO4(98%)、H2O2(30%)(廣州化學試劑廠); SU8 3025光刻膠及顯影液(美國Microchem公司); 聚二甲基硅氧烷(PDMS)前體和引發劑(美國 Dow Corning公司); 十六烷和礦物油(上海生工公司); ABIL EM90 表面活性劑(德國Degussa AG公司);
2.2微流控芯片
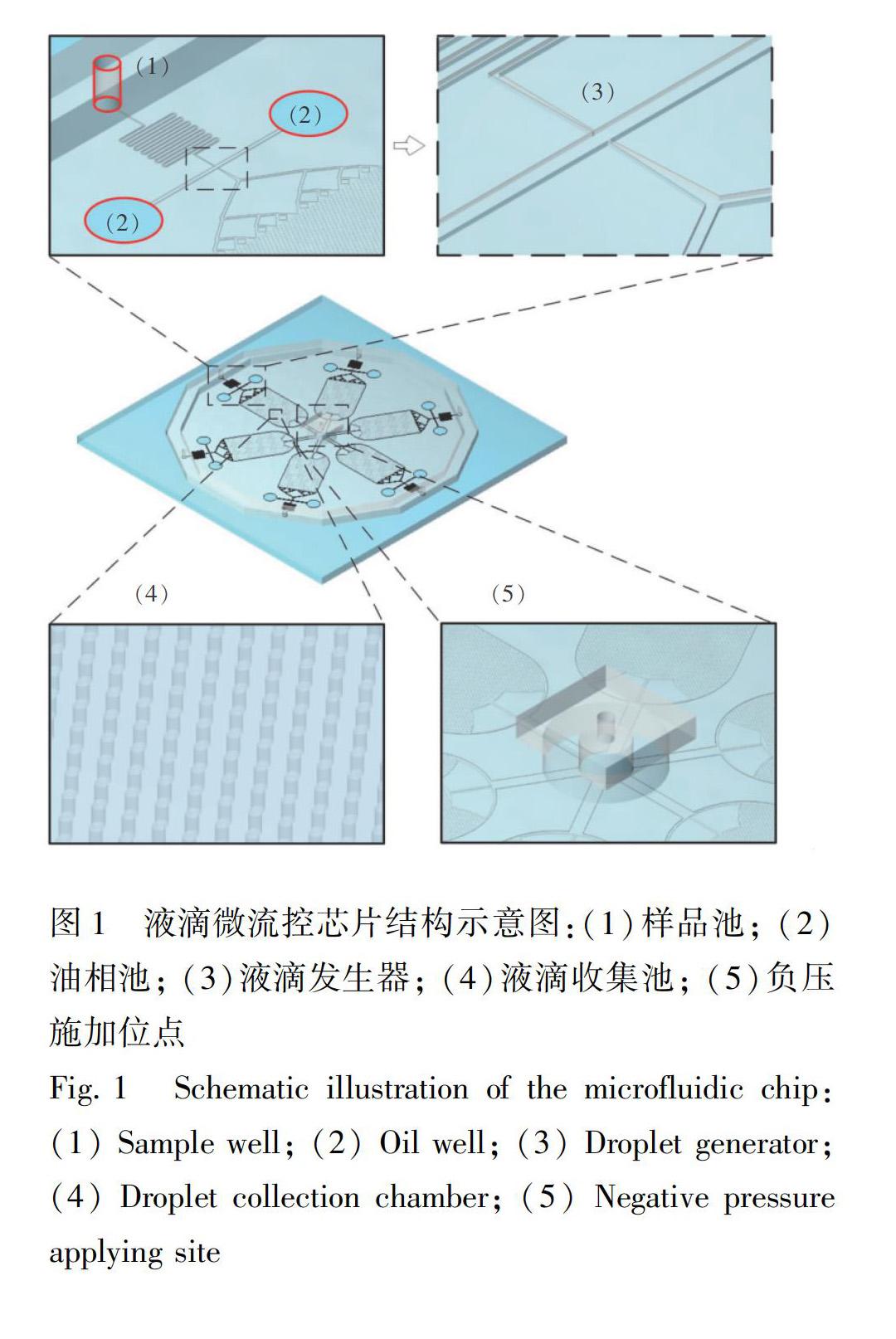
微流控芯片包含具有圖形化結構的PDMS頂層和無結構的玻璃底層(圖1)。PDMS層包含一組6個輻射狀排列分析單元,匯聚于同一出口,即負壓施加點。每個單元包括上游的液滴發生器以及下游的液滴儲存池。PDMS層使用標準光刻倒模工藝加工[35],等離子處理后,與玻璃底層封接。加工完成的芯片經紫外線照射1 h,并于使用之前保存1周。與芯片連接的特氟龍毛細管經高壓消毒后保存備用。
2.3實驗方法
2.3.1樣品準備將細菌懸液與MH肉湯以1∶9 (V/V)比例加入無菌試管,獲得梯度密度細菌懸液。將107 CFU/mL菌液5倍倍比稀釋,獲得一組5個梯度密度細菌懸液。實驗前,向待測樣品溶液中加入刃天青儲備液至終濃度為400 μmol/L,另外以1∶50 (V/V) 比例加入ProLong Live抗淬滅劑。在3組平行實驗中,對這5個梯度密度細菌懸液進行同步液滴數字化顯色分析。
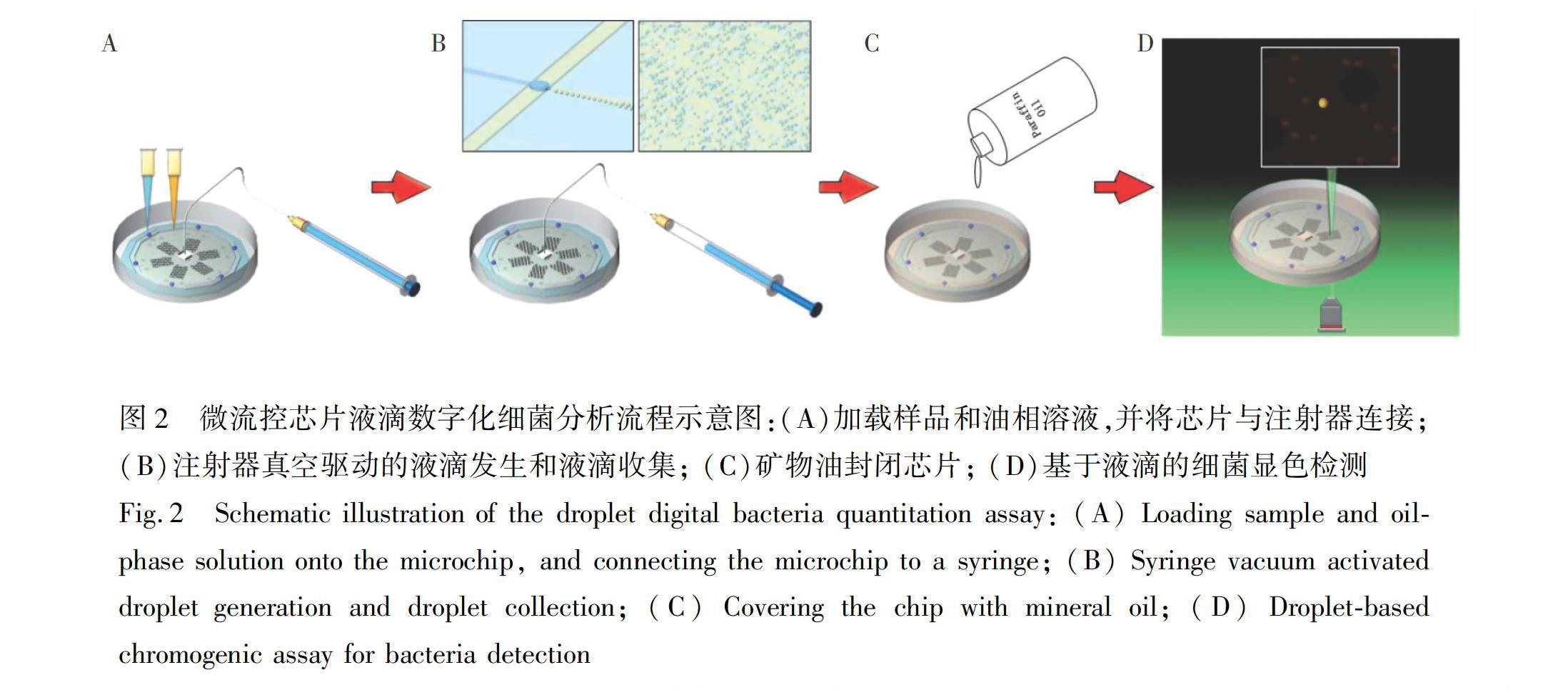
2.3.2液滴數字化顯色分析液滴數字化顯色分析流程如圖2所示,具體操作如下:(1)使用特氟龍毛細管將注射器與芯片出口連接,將樣品溶液和油相溶液分別加載到芯片的樣品池和油相池; (2)拉動注射器針柄,利用注射器真空產生的負壓驅動油相和水相溶液流動,引發液滴發生。待液滴充滿液滴收集池,于芯片出口端剪斷特氟龍管; (3)將芯片轉入培養皿,用石蠟油淹沒,37℃下孵育一定時間; (4)用熒光顯微鏡觀察芯片并成像,使用Image J軟件計數刃天青顯色反應陽性液滴數(n1)和陰性液滴數(n2)。樣品中細菌密度D(CFU/mL)=
2.3.3平板計數用生理鹽水將2.3.1節制備的5個梯度密度細菌懸液分別10倍倍比稀釋7次。每份細菌懸液用平板計數法平行檢測3次,每次取1 mL樣品加入到培養皿中。培養皿中加入適量溶解的營養瓊脂,搖勻后,室溫下固化,37℃培養24 h。依據GB4789.22016《食品安全國家標準,食品微生物學檢驗,菌落總數測定》的原則,選取各組中具有適宜菌落數目的平板進行計數,并計算平均值,再根據稀釋倍數推算出原始樣品的細菌密度。
3結果與討論
3.1注射器真空驅動的液滴發生
在注射器真空作用下,液滴分析單元的末端壓力接近于0,而入口端液面壓力為1個大氣壓,這種壓力差存在于每個分析單元。因此,利用一個注射器可實現多個分析單元內的同步液滴發生。在大氣壓驅動下,水相和油相共同進入芯片通道。在十字型通道結構液滴發生器處,油相剪切力作用于水相,因而將水相切割為液滴。液滴的尺寸由液滴發生器處通道尺寸以及油相和水相的流速決定。對于注射器真空驅動的液滴發生,由于壓力是恒定的,液滴發生頻率和液滴尺寸主要通過調節通道尺寸實現。
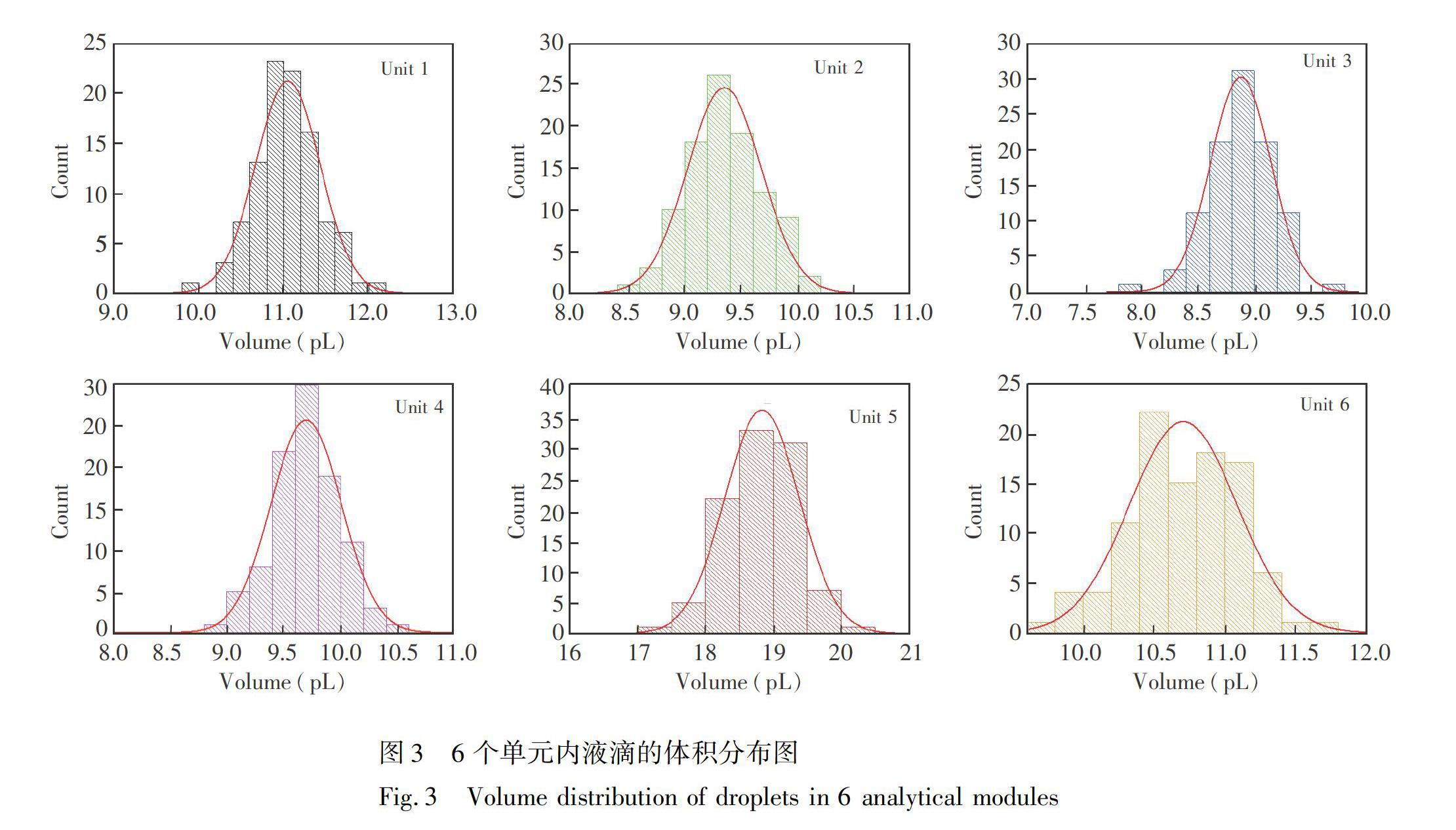
本實驗中,液滴發生器十字型通道的尺寸為:水相孔徑20 μm,油相孔徑175 μm,深度40 μm。在注射器真空驅動下,6個平行單元的液滴發生頻率介于180~193 Hz之間,生成的液滴可在3 min內充滿液滴收集池。成像分析(圖3)顯示,收集池內液滴變化范圍介于25.69~33.01 μm之間,每個單元內的液滴體積相對標準偏差(RSD)<2%,均符合正態分布(p>0.05)。由此可見,這種注射器真空驅動發生的液滴均一性較好,滿足數字化分析要求。不同分析單元的液滴體積偏差的原因是單元內流動阻力的細微差異造成的流速差異。由于液滴數字化分析引入了體積校正,不同單元內液滴體積的偏差并不影響定量分析結果。
3.2液滴反應體系的穩定性
PDMS材質芯片在長時間培養中存在水分遷移問題[36]。如圖4所示,原生PDMS芯片中的液滴在連續培養中體積持續降低,3 h內液滴體積可減少95%以上,提示存在水相油相PDMS的水蒸汽遷移途徑。本研究從兩個方面采取措施,有效控制水分遷移問題:一是芯片底層采用密閉的玻璃材料,部分降低蒸發效應; 二是在PDMS頂層覆蓋礦物油,降低蒸發。如圖4所示,在優化的芯片反應條件下,37℃孵育3 h后,液滴體積只降低了13.54% ± 5.65%; 孵育6 h后,液滴體積也只降低了20.79% ± 4.68%。
實驗中還發現了油水界面成分串擾問題。在使用7500氟化油、十六烷和石蠟油的液滴體系中,均發現細菌穿梭于油水界面(圖5)以及液滴內刃天青的油相擴散(圖6),明顯影響了液滴顯色實驗的穩定性和靈敏度。這些現象的產生可能是由于油水界面的屏蔽作用不足。本研究通過調整表面活性劑濃度改善液滴穩定性。結果表明,即使增加表面活性劑濃度,也不能完全消除7500氟化油、十六烷液滴體系存在的成分串擾,但添加3% ABIL EM90的石蠟油則可完全杜絕此問題。這是因為外表面活性劑增強了油水界面的穩定性,同時石蠟油具有較高的粘度,遏制了細菌游出。
3.3液滴內的細菌指示顯色反應
液滴細菌顯色反應以刃天為指示劑。刃天青指示細菌活性的原理是:在活性細菌的多種還原酶作用下,無熒光的刃天青轉變為有熒光的試鹵靈。鑒于液滴內細菌數量對于顯色反應信號強度的影響,顯色反應條件的優化均使用單細菌分散體系。本研究借助Syto 9染色,確定了適宜的輸入細菌密度為1.5×106 CFU/mL。 在此輸入密度下,理論上,100%的液滴內細菌數≤1,即每個液滴最多包裹1個細菌。
測定了刃天青顯色信噪比相對孵育時間的變化,由圖7A可見,培養2 h后,開始出現顯著區別于噪聲的熒光(+)信號。根據圖7A的結果分析培養5 h的陽性液滴數量穩定時間,如圖7B所示,3.5 h后,陽性液滴數不再增加,因此,后續實驗均采用3.5 h作為反應終點時間。相較于傳統試管方法12 h左右的顯色反應時間,液滴顯色反應所需時間大幅縮短,這得益于液滴分散對于樣品和顯色信號的相對富集效應。在液滴分散體系內只有部分液滴內包裹細菌,相應地液滴顯色反應結果表現為“全”或“無”的形式,而宏觀反應體系呈現的是平均反應強度。對于包裹細菌的液滴,液滴內細菌密度較宏觀體系提高。例如,直徑30 μm的液滴中包含單個細菌情況下,液滴內細菌密度約為9×106 CFU/mL,其密度相對于106 CFU/mL密度的細菌懸液提升9倍,而相對于105 CFU/mL的細菌懸液提升了90倍。由于液滴的封閉效應,液滴內的顯色信號快速蓄積,因而可在較短時間內達到檢測限。由于不同種類細菌存在代謝活性差異,這種液滴數字化顯色分析方法的普適性需要通過測試更多種類細菌進行驗證。
為了驗證液滴刃天青顯色方法對于細菌的特異性指示效果,借助圖像指示,在1個液滴細菌分散體系內隨機抽取100個不含菌的液滴和100個含活性菌液滴,使用非參數秩和檢驗分析了兩組液滴內熒光強度的差異。結果表明,兩組細菌的顯色信號具有顯著性差異(p<0.05),提示液滴刃天青顯色法可特異性指示活性細菌 。
3.4液滴數字化分析對細菌定量分析效果的評價
選取5倍系列稀釋獲得的一系列梯度密度細菌懸液進行液滴數字化分析,并與平板接種計數方法進行了相關性分析。由圖8可見,液滴數字化分析獲得的系列細菌密度與理論濃度具有良好的線性關系(R2=0.99)。相關性分析結果表明,在105 ~108 CFU/mL范圍內,液滴數字化方法測定的每個樣品密度的RSD均小于5%,表明本方法具有良好的定量分析精度,液滴數字化分析與平板接種計數所獲得的定量分析結果一致。
液滴數字化分析的定量分析效果取決于分散體系的規模,分散單元的數目越多,定量分析范圍越寬,分析精度也越高。為了分析單元數目和每個單元內液滴數目,本研究發展的液滴數字化分析使用了約4×104左右的液滴數,實驗結果表明,已經可獲得足夠的定量分析精度。如引入在線或離線樣品預富集方法,采用本方法可望分析更低密度的細菌樣品,。后續將進行更多種類細菌的測試,以評價方法在細菌分析中應用的普適性。
4結 論
建立了一種基于微流控液滴數字化顯色分析的細菌快速定量分析方法。利用注射器真空發生液滴,操作簡便可靠; 芯片采用平行分析單元設計,可提供適宜的測試通量。本方法定量分析精度高,定量分析RSD低于5%; 分析時間短,可在3.5 h內獲得細菌定量分析信息。上述特點使得這種細菌定量分析方法非常適用于資源有限條件下的細菌快速檢測。
References
1Austin B.Antonie Van Leeuwenhoek, 2017, 110(10): 1247-1256
2Wong I, Atsumi S, Huang W C, Wu T Y, Hanai T, Lam M L, Tang P, Yang J, Liao J C, Ho C M.Lab Chip, 2010,? 10(20): 2710-2719
3Salipante S J, Jerome K R.Clin. Chem., 2019,? 66(1): 117-123
4Adan A, Alizada G, Kiraz Y, Baran Y, Nalbant A.Crit. Rev. Biotechnol., 2017,? 37(2): 163-176
5FANG HanShu, LANG MingFei, SUN Jing. Chinese J. Anal. Chem., 2019,? 47(9): 1293-1301
方涵書, 郎明非, 孫 晶. 分析化學, 2019,? 47(9): 1293-1301
6Behera B, Vishnu G K A, Chatterjee S, Sitaramgupta V V, Sreekumar N, Nagabhushan A, Rajendran N, Prathik B H, Pandya H J.Biosens. Bioelectron., 2019,? 142: 111552
7Kumar S, Nehra M, Mehta J, Dilbaghi N, Marrazza G, Kaushik A.Sensors (Basel), 2019,? 19(20): 4476-4498
8Rajapaksha P, Elbourne A, Gangadoo S, Brown R, Cozzolino D, Chapman J.Analyst, 2019,? 144(2): 396-411
9Boedicker J Q, Li L, Kline T R, Ismagilov R F.Lab Chip, 2008,? 8(8): 1265-1272
10Kaminski T S, Scheler O, Garstecki P.Lab Chip, 2016,? 16(12): 2168-2187
11Basu A S.SLAS Technol, 2017,? 22(4): 369-386
12Men Y, Fu Y, Chen Z, Sims P A, Greenleaf W J, Huang Y.Anal. Chem., 2012,? 84(10): 4262-4266
13Bahder B W, Soto N, Komondy L, Mou D F, Humphries A R, Helmick E E.Plant Dis., 2019,? 103(8): 1918-1922
14Sanmiguel J, Gao G, Vandenberghe L H.Methods Mol. Biol., 2019,? 1950: 51-83
15Nie M, Zheng M, Li C, Shen F, Liu M, Luo H, Song X, Lan Y, Pan J Z, Du W.Anal. Chem., 2019,? 91(3): 1779-1784
16Hu Y, Xu P, Luo J, He H, Du W.Anal. Chem., 2017,? 89(1): 745-750
17Lee S, Lee W, Kim H, Bae P K, Park J, Kim J.Biosens. Bioelectron., 2019,? 131: 280-286
18Ng A, Fobel R, Fobel C, Lamanna J, Rackus D G, Summers A, Dixon C, Dryden M, Lam C, Ho M, Mufti N S, Lee V, Asri M, Sykes E A, Chamberlain M D, Joseph R, Ope M, Scobie H M, Knipes A, Rota P A, Marano N, Chege P M, Njuguna M, Nzunza R, Kisangau N, Kiogora J, Karuingi M, Burton J W, Borus P, Lam E, Wheeler A R.Sci. Transl. Med., 2018,? 10(438): 438-449
19Li N, Men W, Zheng Y, Wang H, Meng X.Molecules, 2019,? 24(23): 4384-4398
20Beneyton T, Krafft D, Bednarz C, Kleineberg C, Woelfer C, Ivanov I, VidakovicKoch T, Sundmacher K, Baret J C.Nat. Commun., 2018,? 9: 2391
21 LIN DongGuo, LIN JinQiong, LI PeiWen, YANG Na, XU BangLao, LIU DaYu. Chinese J. Anal. Chem., 2018, 46(1): 1-10
林冬果, 林錦瓊, 李佩文, 楊 娜, 徐邦牢, 劉大漁. 分析化學, 2018, 46(1): 1-10
22Dolega M E, Abeille F, PicolletD'Hahan N, Gidrol X.Biomaterials, 2015,? 52: 347-357
23Thibault D, Jensen P A, Wood S, Qabar C, Clark S, Shainheit M G, Isberg R R, van Opijnen T.Nat. Commun., 2019,? 10: 5729
24Boedicker J Q, Li L, Kline T R, Ismagilov R F.Lab Chip, 2008,? 8(8): 1265-1272
25Kang D K, Ali M M, Zhang K, Huang S S, Peterson E, Digman M A, Gratton E, Zhao W.Nat. Commun., 2014,? 5: 5427
26Kaushik A M, Hsieh K, Chen L, Shin D J, Liao J C, Wang T H.Biosens. Bioelectron., 2017,? 97: 260-266
27Scheler O, Pacocha N, Debski P R, Ruszczak A, Kaminski T S, Garstecki P.Lab Chip, 2017,? 17(11): 1980-1987
28Ma Y D, Luo K, Chang W H, Lee G B.Lab Chip, 2018,? 18(2): 296-303
29Zhang Q, Zeng S, Qin J, Lin B.Electrophoresis, 2009,? 30(18): 3181-3188
30Hsieh K, Zec H C, Chen L, Kaushik A M, Mach K E, Liao J C, Wang T H.Anal. Chem., 2018,? 90(15): 9449-9456
31Teo A, Li K H, Nguyen N T, Guo W, Heere N, Xi H D, Tsao C W, Li W, Tan S H.Anal. Chem., 2017,? 89(8): 4387-4391
32Li Y, Jiang Y, Wang K, Wu W.Anal. Chem., 2018,? 90(20): 11925-11932
32WANG ZhiLe, WANG ZhuYuan, ZONG ShenFei, CUI YiPing.Chinese Optics, 2018,? 11(3): 513-530
王志樂, 王著元, 宗慎飛, 崔一平. 中國光學, 2018,? 11(3): 513-530
33SHI Yang, SHAO XiaoGuang. Chinese Journal of Chromatography, 2019, 37(9): 89-94
石 楊, 邵小光. 色譜, 2019, 37(9): 89-94
34Xu B, Du Y, Lin J, Qi M, Shu B, Wen X, Liang G, Chen B, Liu D.Anal. Chem., 2016,? 88(23): 11593-11600
35Chuang Y C, Chu C K, Lin S Y, Chen L J.Soft Mat., 2014,? 10(19): 3394-3403

