甘薯羽狀斑駁病毒O株系和RC株系中國分離物全基因組序列分析及其遺傳特征
秦艷紅,王永江,王爽,喬奇,田雨婷,張德勝,張振臣
甘薯羽狀斑駁病毒O株系和RC株系中國分離物全基因組序列分析及其遺傳特征
秦艷紅,王永江,王爽,喬奇,田雨婷,張德勝,張振臣
(河南省農業科學院植物保護研究所/河南省農作物病蟲害防治重點實驗室/農業農村部華北南部作物有害生物綜合治理重點實驗室,鄭州 450002)
【】對甘薯羽狀斑駁病毒(,SPFMV)O株系中國分離物(SPFMV-O-Ch1)和RC株系中國分離物(SPFMV-RC-Ch1)的基因組全序列進行克隆,明確SPFMV-O-Ch1和SPFMV-RC-Ch1的基因組結構特征及其遺傳變異情況,為研究甘薯羽狀斑駁病毒的致病機制打下基礎。根據GenBank中登錄的SPFMV基因組全序列設計2對簡并引物和3對特異性引物,利用RT-PCR方法,從感染SPFMV的甘薯葉片中擴增SPFMV O株系和RC株系中國分離物的基因組全長序列,將目的片段分別克隆到pMD19-T載體上,經序列測定、分析和拼接,獲得SPFMV-O-Ch1和SPFMV-RC-Ch1的全序列,利用DNAMAN和MEGA7對SPFMV基因組全序列及不同編碼區序列進行遺傳變異和系統進化樹分析,利用RDP軟件分析SPFMV基因組重組情況。經序列測定和拼接,結果表明SPFMV-O-Ch1和SPFMV-RC-Ch1基因組分別包含10 922和10 851 nt,均包含一個開放閱讀框,分別由10 557和10 482 nt組成,編碼一個多聚蛋白,分別由3 518和3 493個氨基酸殘基組成。兩個分離物均在P1蛋白內編碼一個P1N-PISPO蛋白,在P3蛋白內編碼一個P3N-PIPO蛋白。基因組全序列核苷酸一致性分析表明,SPFMV-O-Ch1與SPFMV-RC-Ch1的一致性為87.3%,與GenBank登錄的其他分離物基因組全序列一致性為86.0%—95.8%,與Ruk73分離物的一致性最高,為95.8%,與11-1分離物的一致性最低,為86.0%。SPFMV-RC-Ch1與GenBank登錄的其他分離物基因組全序列一致性為85.9%—98.7%,與IS90分離物的一致性最高,為98.7%,與Aus1-2B分離物的一致性最低,為85.9%。基于多聚蛋白基因核苷酸序列的遺傳進化樹分析表明,SPFMV-O-Ch1與Ordinary、10-O和17-O等O株系的分離物形成一個分支,SPFMV-RC-Ch1與S、IS90和CW137等RC株系的分離物形成一個分支。重組分析結果表明,O-Ch1分離物中發現3個重組事件,分別發生在7 731—9 710、135—10 012和4 825—6 948 nt,RC-Ch1沒有發現重組事件。我國的SPFMV-O-Ch1和SPFMV-RC-Ch1分離物的基因組結構與其他分離物相同,O-Ch1與O株系分離物一致性較高,RC-Ch1與RC株系分離物一致性較高,O-Ch1分離物檢測到3個重組事件,RC-Ch1未發現重組事件。
甘薯羽狀斑駁病毒;株系;全基因組序列;遺傳變異;重組分析
0 引言
【研究意義】甘薯羽狀斑駁病毒(,SPFMV)屬于馬鈴薯Y病毒科()馬鈴薯Y病毒屬()病毒,是侵染甘薯的重要病毒之一,在全世界甘薯產區和主要甘薯品種上均有發生[1]。SPFMV的寄主范圍較窄,僅侵染旋花科植物[2-3],其傳播介體為蚜蟲,靠蚜蟲以非持久方式進行傳播[4]。SPFMV單獨侵染甘薯時一般產生輕微花葉癥狀或無癥狀表現,對甘薯產量影響較小[5],但其在田間常與甘薯褪綠矮化病毒(,SPCSV)混合侵染甘薯,產生甘薯復合病毒病(sweet potato virus disease,SPVD),表現為植株嚴重矮化,葉片畸形、花葉、褪綠、明脈等癥狀,SPFMV的含量比單獨侵染時增加600倍,對甘薯造成嚴重危害[5-6]。根據核苷酸序列及在寄主上的癥狀類型,SPFMV可劃分為SPFMV普通株系(Ordinary,SPFMV-O)、褐裂株系(Russet Crack,SPFMV-RC)和東非株系(East Africa,SPFMV-EA)3個株系[1,6-7]。獲得SPFMV中國主要株系類型的基因組全序列,對該病毒的遺傳進化及分子變異研究具有重要意義。【前人研究進展】1990年,李汝剛等[8]利用血清學方法首次從我國北京、江蘇、四川和山東等地區的甘薯樣品上檢測到SPFMV。目前,SPFMV在我國發生比較普遍,在南方、北方和長江中下游三大甘薯產區的多個省(直轄市)均有分布[9-10]。Qin等[10-11]對SPFMV的CP基因進行了分子變異分析,明確了我國SPFMV的株系類型,目前我國甘薯上存在O、RC和EA株系,O株系是我國的優勢株系,分布最為廣泛,其次是RC株系。1997年,Sakai等[12]利用RT-PCR方法從日本甘薯上克隆了SPFMV RC株系S分離物的基因組全序列,對該分離物的基因組結構和編碼蛋白進行了分析,SPFMV與馬鈴薯Y病毒屬其他成員的結構相似,包含一個開放閱讀框(open reading frame,ORF),經切割加工產生P1、HC-Pro、P3、6K1、CI、6K2、VPg、NIa-Pro、NIb和CP 10個成熟蛋白,是基因組最大的馬鈴薯Y病毒屬成員。2009年,Kreuze等[13]利用深度測序結合RT-PCR方法從秘魯獲得了EA株系Piu3分離物的基因組全序列。2010年,Yamasaki等[14]從日本獲得了O株系的兩個分離物Ordinary和10-O的基因組全序列,并對Ordinary和10-O分離物及RC株系的代表性分離物S和EA株系的代表性分離物EA-Piu3等的基因組結構、序列相似性等進行了比較分析。隨后,韓國、西班牙、澳大利亞、東帝汶、美國、日本、巴西、南非、阿根廷、秘魯、肯尼亞和烏干達等多個國家和地區的不同分離物基因組全序列陸續被報道[15-18]。與其他馬鈴薯Y病毒屬病毒一樣,SPFMV在P3內部通過移碼策略編碼一個P3N-PIPO蛋白。近年來的研究表明,SPFMV等4種侵染甘薯的馬鈴薯Y病毒屬病毒在P1蛋白內通過移碼還編碼一個具有基因沉默抑制子功能的P1N-PISPO蛋白[16,19-20]。【本研究切入點】目前,SPFMV在中國僅有部分序列報道,尚未見關于SPFMV-O株系和SPFMV-RC株系中國分離物基因組全序列的報道。【擬解決的關鍵問題】從我國甘薯樣品中克隆獲得SPFMV-O和SPFMV-RC株系中國分離物全基因組序列,將兩個株系的中國分離物與GenBank中登錄的其他國家分離物進行比較分析,了解SPFMV-O和SPFMV-RC株系中國分離物的基因組結構及遺傳進化特征。
1 材料與方法
試驗于2015—2016年在河南省農業科學院植物保護研究所完成。
1.1 材料
1.1.1 植物材料 2015年4月,在河南省南陽市唐河縣采集植株矮化、葉脈發黃、葉片皺縮等癥狀的甘薯病毒病樣品1份,將甘薯植株種植于25—28℃防蟲溫室中,16 h光照、8 h黑暗培養。同時采集甘薯莖蔓上部幼嫩葉片保存于-70℃冰箱中備用。
1.1.2 主要試劑 Trizol總RNA提取試劑盒購自生工生物工程(上海)股份有限公司;Reverse Transcriptase XL反轉錄試劑盒、2×Premix LADNA聚合酶、dNTP、T4 DNA連接酶、pMD19-T載體和DL5000分子量標準購自寶生物工程(大連)有限公司;DNA膠回收試劑盒和柱式質粒小量抽提試劑盒均購自美國OMEGA公司;大腸桿菌TG1感受態細胞為筆者實驗室制備并保存于-70℃冰箱;其他生化常用試劑為進口或國產分析純。
1.1.3 引物設計與合成 根據GenBank中登錄的SPFMV-O株系和SPFMV-RC株系不同分離物的基因組全序列(表1)設計了2對簡并引物和3對特異性引物,引物由生工生物工程(上海)股份有限公司合成,引物序列見表2。
1.2 方法
1.2.1 總RNA提取 稱取約0.1 g甘薯病樣葉片,在液氮中迅速研磨成粉末,按照Trizol總RNA提取試劑盒說明書提取甘薯病樣葉片總RNA,保存于-70℃冰箱中備用。
1.2.2 RT-PCR擴增基因組序列 第一鏈cDNA合成參照Reverse Transcriptase XL反轉錄試劑盒說明書進行,以OligodT為引物,以總RNA為模板合成cDNA。反轉錄反應體系(10 μL):5×AMV Buffer 2 μL、OligodT引物1 μL、dNTP mixture(10 mmol·L-1)1 μL、AMV反轉錄酶0.5 μL、RNA酶抑制劑0.5 μL、RNA模板5 μL;42℃保溫60 min,16℃保溫5 min。PCR反應體系(50 μL):cDNA 5 μL、2×Premix LADNA聚合酶25 μL、上游和下游引物(10 μmol·L-1)各1 μL、加ddH2O補足至50 μL。反應條件:94℃預變性3 min,94℃變性30 s,53℃退火30 s,根據目的條帶大小不同,72℃延伸2—6 min,循環30次后,72℃延伸10 min。RT-PCR擴增產物經1%瓊脂糖凝膠電泳檢測。將電泳鑒定正確的目的條帶利用OMEGA公司的瓊脂糖凝膠回收試劑盒對目的片段進行切膠純化。
1.2.3 PCR產物的克隆及測序 參照TaKaRa公司pMD19-T載體說明書,將純化的目的片段分別連接至pMD19-T載體,轉化至大腸桿菌TG1感受態細胞。經菌液PCR鑒定篩選到陽性克隆,每個樣品選取3—5個克隆進行測序,測序由生工生物工程(上海)股份有限公司完成,由寶生物工程(大連)有限公司進行5′和3′端RACE試驗。利用BLAST比對測序結果。
1.2.4 全基因組序列拼接及分析 利用DNAMAN軟件對測序成功的基因片段進行序列拼接,利用軟件MEGA 7.0將SPFMV-O-Ch1和SPFMV-RC-Ch1分離物與GenBank上登錄的其他分離物(表1)進行多序列比對分析,利用鄰接法(neighbor-joining,NJ)構建系統進化樹,系統進化樹中各分支置信度(Bootstrap)進行1 000次重復分析。基因重組分析采用Recombination Detection Program(RDP v5.3)軟件,利用RDP5軟件中的RDP、GENECONV、Chimaera、MaxChi、Bootscan、Siscan和3Seq 7種方法對序列進行重組分析,以≤0.05為標準,當3種以上方法同時檢測到重組時,認為其為有意義的重組事件[21-23]。

表1 本研究所用分離物的名稱、株系、來源和登錄號信息
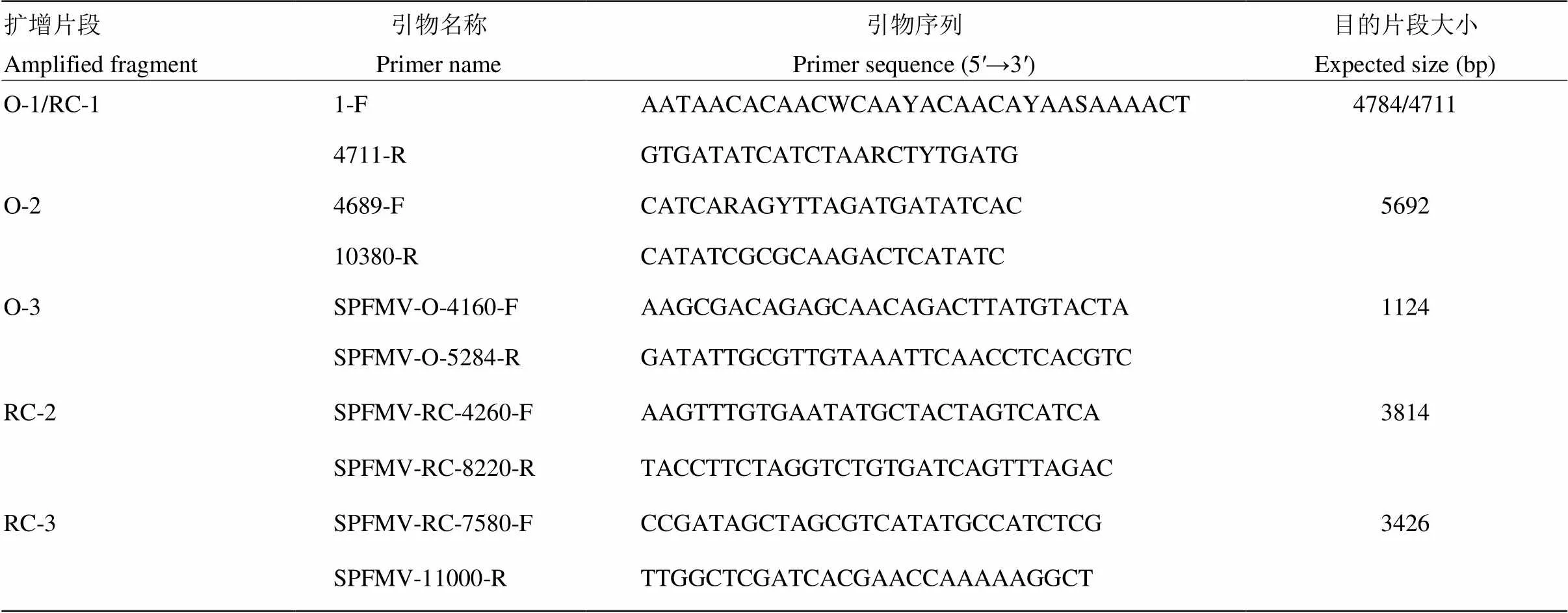
表2 引物序列及目的片段大小
2 結果
2.1 RT-PCR擴增
利用設計的1-F/4711-R、4689-F/10380-R、SPFMV- O-4160-F/SPFMV-O-5284-R、SPFMV-RC-4260-F/ SPFMV- RC-8220-R、SPFMV-RC-7580-F/SPFMV-11000-R等引物擴增SPFMV-O株系中國分離物和SPFMV-RC株系中國分離物的不同基因組片段,擴增產物經1%瓊脂糖凝膠電泳分析,結果表明獲得了預期大小的目的條帶,條帶大小分別約為4 790 bp(O-1)、1 270 bp(O-2)、5 700 bp(O-3)、4 710 bp(RC-1)、3 820 bp(RC-2)、3 420 bp(RC-3)(圖1)。
2.2 SPFMV-O和SPFMV-RC株系中國分離物基因組全序列克隆及結構分析
重組質粒經PCR鑒定為陽性,對陽性克隆進行序列測定,通過RACE試驗獲得了兩個分離物的5′和3′末端序列,確保引物及重疊區域堿基無變異,拼接后獲得了SPFMV-O和SPFMV-RC株系中國分離物的全基因組序列,分別命名為SPFMV-O-Ch1(GenBank登錄號為KY296450)和SPFMV-RC-Ch1(GenBank登錄號為KY296451)。SPFMV-O-Ch1和SPFMV-RC- Ch1與已報道的其他分離物基因組結構特征相一致,3′末端均包含一個poly (A)尾序,編碼一個大的多聚蛋白,加工產生10個成熟蛋白,從N端到C端依次為P1、HC-Pro、P3、6K1、CI、6K2、VPg、NIa-Pro、NIb和CP。除多聚蛋白外,還通過移碼策略分別編碼一個P1N-PISPO和P3N-PIPO蛋白。SPFMV-O-Ch1基因組全長為10 922 nt,5′和3′非翻譯區(UTR)分別含有114和251 nt,多聚蛋白基因位于基因組的115—10 671處,由10 557 nt組成,多聚蛋白大小為3 518 aa。SPFMV-RC-Ch1基因組全長為10 851 nt,5′和3′非翻譯區(UTR)分別含有121和248 nt,多聚蛋白基因位于基因組的122—10 603處,由10 482 nt組成,多聚蛋白含3 493 aa。SPFMV-O-Ch1編碼的P1蛋白含有689 aa,SPFMV-RC-Ch1編碼的P1蛋白含有664 aa,兩者大小不同。兩個分離物編碼的其他蛋白大小相同,依次為HC-Pro(458 aa)、P3(352 aa)、6K1(52 aa)、CI(643 aa)、6K2(53 aa)、VPg(192 aa)、NIa-Pro(243 aa)、NIb(521 aa)和CP(315 aa)。
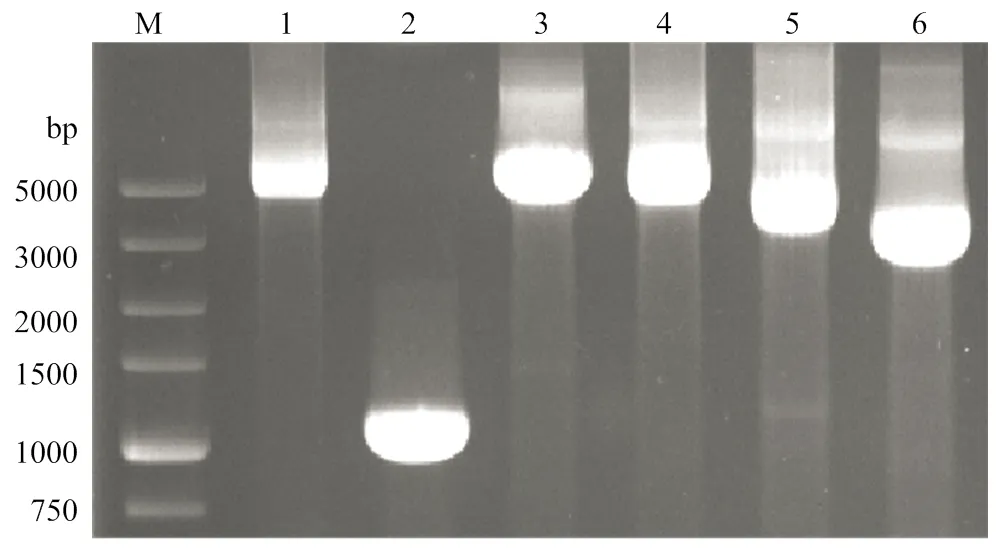
M:DL5000分子量標準 DL5000 marker;1:O-1擴增片段Amplification of O-1;2:O-2擴增片段Amplification of O-2;3:O-3擴增片段Amplification of O-3;4:RC-1擴增片段Amplification of RC-1;5:RC-2擴增片段Amplification of RC-2;6:RC-3擴增片段Amplification of RC-3
2.3 基因組序列一致性分析
利用DNAMAN軟件對SPFMV-O-Ch1、SPFMV-RC-Ch1分離物以及GenBank中登錄的其他48個分離物基因組序列進行比對分析(表3),結果表明SPFMV-O-Ch1與SPFMV-RC-Ch1之間的全序列核苷酸一致性較低,為87.3%,多聚蛋白的核苷酸和氨基酸一致性分別為88.2%和94.0%,屬于不同的株系。SPFMV-O-Ch1與GenBank中登錄的48個分離物基因組全序列核苷酸一致性為86.0%—95.8%,與烏干達的Ruk73分離物一致性最高(95.8%),與美國的11-1分離物一致性最低(86.0%)。多聚蛋白的比對結果表明,SPFMV-O-Ch1與48個分離物核苷酸一致性范圍為87.0%—95.8%,與Ruk73分離物一致性最高,為95.8%,與11-1和澳大利亞Aus3-2BC分離物一致性最低,為87.0%;氨基酸一致性范圍為92.1%—97.6%,與阿根廷O-Arg分離物一致性最高,為97.6%,與肯尼亞SRF109a分離物一致性最低,為92.1%。
SPFMV-RC-Ch1與其他48個分離物全基因組核苷酸一致性為85.9%—98.7%,與韓國的IS90分離物一致性最高(98.7%),與澳大利亞的Aus1-2B分離物一致性最低(85.9%)。多聚蛋白的比對結果表明,SPFMV-RC-Ch1與韓國IS90分離物的核苷酸和氨基酸一致性最高,分別為98.8%和99.3%,與澳大利亞Aus1-2B分離物的核苷酸和氨基酸一致性最低,分別為87.0%和92.8%。
在SPFMV編碼的10個蛋白中,P1蛋白變異最大,O-Ch1、RC-Ch1分離物與48個分離物氨基酸序列一致性分別為81.2%—96.4%和80.6%—99.2%,而且不同株系P1蛋白的大小不同,屬于O株系的O-Ch1分離物P1蛋白包含689 aa,屬于RC株系的RC-Ch1分離物P1蛋白包含664 aa,而屬于EA株系的Piu3分離物P1蛋白包含724 aa。最保守的是CI蛋白,O-Ch1、RC-Ch1分離物與48個分離物氨基酸序列一致性分別為97.0%—99.7%和97.5%—99.8%。
2.4 系統進化關系分析
基于SPFMV多聚蛋白基因核苷酸序列構建的系統進化樹分析表明,O-Ch1分離物與來自烏干達的O株系Ruk73分離物形成一個小分支,親緣關系最近,與其他屬于O株系的分離物在一個大分支上,而與來自秘魯的EA株系Piu3分離物親緣關系也較近。RC-Ch1分離物與來自韓國RC株系的IS90分離物聚類到同一個小分支上,親緣關系最近,與屬于RC株系其他31個分離物形成一個大分支。對變異最大的P1蛋白基因核苷酸序列構建進化樹,結果表明O-Ch1分離物與阿根廷的O-Arg、日本的Ordinary及17-O等屬于O株系的分離物形成一個小分支,親緣關系較近。RC-Ch1分離物與RC株系的IS90分離物(韓國)聚類在同一個小分支上,親緣關系最近(圖2)。

表3 O-Ch1、RC-Ch1與其他分離物核苷酸和氨基酸序列一致性
-:沒有相應的氨基酸No corresponding amino acid

圖2 SPFMV分離物多聚蛋白和P1蛋白核苷酸序列遺傳進化分析
比較保守的CI蛋白核苷酸序列進化樹分析結果顯示,O-Ch1分離物與來自東帝汶的O株系TM66B分離物親緣關系最近,RC-Ch1分離物與IS90分離物親緣關系最近。對CP蛋白核苷酸序列構建進化樹,結果與P1蛋白系統進化樹相似,O-Ch1與O-Arg、10-O、Ordinary及17-O等屬于O株系的分離物形成一個小分支,親緣關系較近,RC-Ch1與IS90分離物在同一個小分支上,親緣關系最近(圖3)。
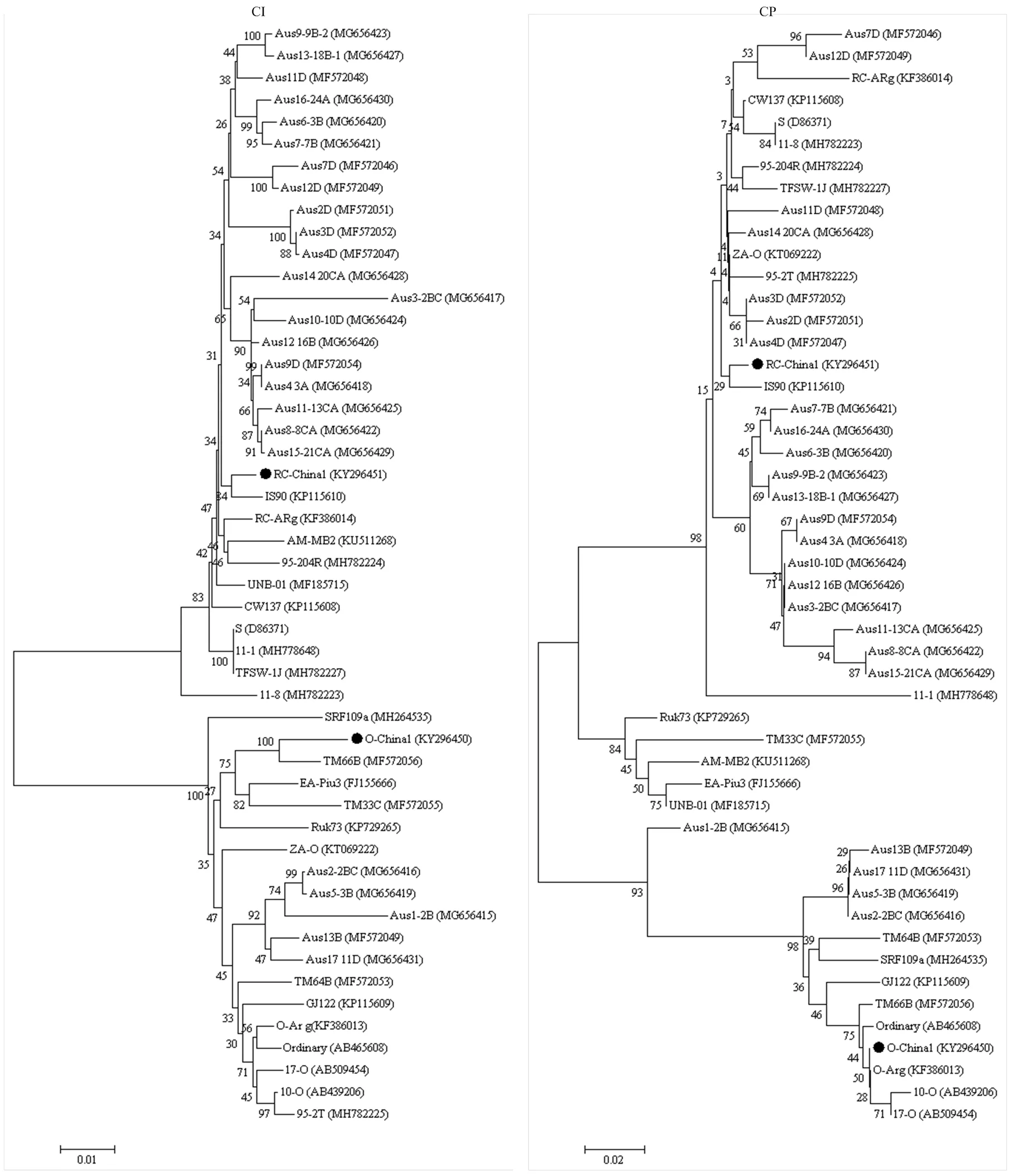
圖3 SPFMV分離物CI蛋白和CP蛋白核苷酸序列遺傳進化分析
綜上所述,基于O-Ch1分離物基因組不同編碼區域構建的系統進化樹,親緣關系最近的分離物不盡相同;而RC-Ch1分離物基因組不同編碼區域構建的系統進化樹分析結果相一致,均與來自韓國RC株系的IS90分離物親緣關系最近。推測O-Ch1分離物可能發生了重組。
2.5 基因組全序列重組分析
為了明確我國的O-Ch1、RC-Ch1分離物是否存在潛在的重組事件,利用RDP5軟件中的7種算法(≤0.05為顯著),將O-Ch1、RC-Ch1與GenBank登錄的其他48個分離物基因組全序列進行重組分析。結果表明(表4),在O-Ch1分離物中檢測到3個潛在的重組事件(其他分離物重組事件未列出)。重組事件1發生在7 731—9 710 nt,獲得7種算法支持,最低值為1.095×10-44(3Seq),最高值為8.317× 10-17(GENECONV),主要親本為日本的O株系Ordinary分離物,次要親本為東帝汶的O株系TM33C分離物。重組事件2發生在135—10 012 nt,獲得4種算法支持,最低值為1.561×10-56(3Seq),最高值為7.437× 10-3(Chimaera),主要親本為秘魯的EA株系Piu3分離物,次要親本為澳大利亞的O株系Aus5-3B分離物。重組事件3發生在4 825—6 948 nt,獲得3種算法支持,最低值為1.104×10-3(MaxChi),最高值為1.256×10-2(3Seq),主要親本為澳大利亞的O株系Aus13B分離物,次要親本為秘魯的EA株系Piu3分離物。RC-Ch1分離物未檢測到重組事件發生。

表4 O-Ch1分離物的重組分析
3 討論
SPFMV是甘薯上分布最廣泛的病毒之一,其可以與SPCSV發生協生作用,形成毀滅性病害SPVD。自我國首次檢測到SPFMV的發生以來,SPFMV在我國逐漸擴散蔓延,成為甘薯上最重要的病毒之一。國內對SPFMV開展了多項研究,主要集中在病毒的分離純化[2,24-25]、生物學性狀[26]、抗體制備[27]、檢測技術[28-38]、分布及遺傳多樣性[9-11,39-40]等方面,而對SPFMV中國分離物的基因組序列測定還未開展相關研究。筆者研究室前期對SPFMV的發生、分布及分子變異等進行了研究,明確了我國SPFMV的主要株系類型為O和RC,在此基礎上,繼續對我國SPFMV O株系和RC株系的基因組開展了相關研究,利用RT-PCR方法結合RACE技術獲得了這兩個主要株系的基因組全序列,獲得的O-Ch1和RC-Ch1分離物是中國SPFMV完整基因組的首次報道。
對O-Ch1和RC-Ch1兩個分離物的基因組結構及編碼的主要蛋白分析發現,O株系和RC株系編碼的P1蛋白大小不同,不同株系(分離物)之間P1蛋白的氨基酸一致性最低,變異最大;而其余蛋白大小均相同,氨基酸一致性高于P1蛋白。Yamasaki等[14]對SPVC和SPFMV的O、RC和EA 3個株系代表分離物的基因組進行了比較分析,表明不同株系之間P1蛋白的核苷酸和氨基酸一致性低于其余9個蛋白;Kwak等[15]對韓國SPFMV等5種侵染甘薯的馬鈴薯Y病毒屬病毒基因組分析結果表明,無論是種間還是株系間,P1蛋白的變異均最大。本研究結果與前人的報道[14-15]相一致。
早期的研究表明,依據CP基因序列分析,SPFMV可分為C、O、RC和EA 4個株系,C株系與其他3個株系遺傳關系較遠,已將其作為新種重新命名為甘薯病毒C(,SPVC),EA株系僅在東非的一些地區存在,而O和RC株系不存在地理局限性[41]。近年來,隨著種質資源的擴散和種薯的調運,EA株系現在已不僅局限于東非地區,我國已報道了EA株系的存在[10]。本研究獲得的O-Ch1和RC-Ch1兩個分離物分別與O株系和RC株系的分離物聚類成一個分支,O-Ch1變異較大,推測可能是由于O株系為我國的優勢株系,地理分布上最廣泛,更易隨著頻繁種質資源交換和調運,而發生變異重組等。
SPFMV經常發生種內重組,不同株系之間重組很常見,比如對10-O分離物的分析結果表明,該分離物是由O、RC和EA 3個株系重組形成的,P1蛋白是重組的熱點區域[20]。韓國的GJ122分離物與10-O分離物類似,也發生了株系間重組,在P1蛋白區域內存在重組位點,是由O株系和RC株系重組產生的[15]。本研究獲得的O-Ch1分離物可能是由O株系和EA株系重組產生的,重組位點發生在NIa-NIb基因、P1基因、CI基因和CP基因處。由于田間常常發生SPFMV不同株系混合侵染現象,不同株系基因組之間容易發生重組變異,從而增強病毒群體的適應能力,種內重組可能是造成SPFMV株系分化較復雜的主要原因,不同株系之間的重組還可能形成新的株系,甚至產生更多的新種等,分析病毒的重組并明確重組熱點區域對了解病毒的進化至關重要。
4 結論
完成了甘薯羽狀斑駁病毒O株系和RC株系中國分離物基因組全序列測定。基因組結構分析結果顯示,我國兩個分離物與其他國家分離物的基因組結構相一致。兩個分離物之間序列相似性較低,變異較大,分別屬于兩個不同的株系。遺傳進化樹分析結果表明,SPFMV-O-Ch1與O株系的分離物形成一個分支,SPFMV-RC-Ch1與RC株系的分離物形成一個分支,基因組重組分析結果顯示,O-Ch1分離物檢測到3個重組事件,RC-Ch1未檢測到重組事件。
[1] R?NN?LI M, CZEKAJ V, JONES R A C, FLETCHER J D, DAVIS R I, MU L, VALKONEN J P T. Molecular characterization of(SPFMV) isolates from Easter Island, French Polynesia, New Zealand and southern Africa., 2009, 93(9): 933-939.
[2] 孟清, 張鶴齡, 張喜印, 楊永嘉, 邢繼英, 宋伯符. 甘薯羽狀斑駁病毒的分離與提純. 植物病理學報, 1994, 24(3): 227-232.
MENG Q, ZHANG H L, ZHANG X Y, YANG Y J, XING J Y, SONG B F. Isolation, purification of., 1994, 24(3): 227-232. (in Chinese)
[3] MUKASA S B, TAIRO F, KREUZE J F, KULLAYA A, RUBAIHAYO P R, VALKONEN J P T. Coat protein sequence analysis reveals occurrence of new strains ofin Uganda and Tanzania., 2003, 27(1): 49-56.
[4] MOYER J W, KENNEDY G G. Purification and properties of., 1978, 68: 998-1004.
[5] KARYEINA R F, KREUZE J F, GIBSON R W, VALKONEN J P T. Synergistic interactions of a potyvirus and a phloem-limited crinivirus in sweet potato plants., 2000, 269(1): 26-36.
[6] UNTIVEROS M, FUENTES S, KREUZE J. Molecular variability ofand other potyviruses infecting sweet potato in Peru., 2008, 153(3): 473-483.
[7] TAIRO F, MUKASA S B, JONES R A C, KULLAYA A, RUBAIHAYO P R, VALKONEN J P T. Unravelling the genetic diversity of the three main viruses involved in sweet potato virus disease (SPVD), and its practical implications., 2005, 6(2): 199-211.
[8] 李汝剛, 蔡少華, Salazar L F. 中國甘薯病毒的血清學檢測. 植物病理學報, 1990, 20(3): 189-194.
LI R G, CAI S H, SALAZAR L F. Serological detection of viruses on sweet potato in China., 1990, 20(3): 189-194. (in Chinese)
[9] 喬奇, 張振臣, 張德勝, 秦艷紅, 田雨婷, 王永江. 中國甘薯病毒種類的血清學和分子檢測. 植物病理學報, 2012, 42(1): 10-16.
QIAO Q, ZHANG Z C, ZHANG D S, QIN Y H, TIAN Y T, WANG Y J. Serological and molecular detection of viruses infecting sweet potato in China., 2012, 42(1): 10-16. (in Chinese)
[10] QIN Y H, ZHANG Z C, QIAO Q, ZHANG D S, TIAN Y T, WANG Y J. Molecular variability of(SPCSV) and five potyviruses infecting sweet potato in China.,2013, 158(2): 491-495.
[11] 王曉華, 張振臣, 喬奇, 秦艷紅, 張德勝, 田雨婷. 甘薯羽狀斑駁病毒外殼蛋白基因的分子變異. 植物保護, 2012, 38(2): 114-116.
WANG X H, ZHANG Z C, QIAO Q, QIN Y H, ZHANG D S, TIAN Y T. Molecular variation ofcoat protein gene., 2012, 38(2): 114-116. (in Chinese)
[12] SAKAI J, MORI M, MORISHITA A, TANAKA M, HANADA K, USUGI T, NISHIGUCHI M. Complete nucleotide sequence and genome organization of(S strain) genomic RNA: the large coding region of the P1gene., 1997, 142(8): 1553-1562.
[13] KREUZE J F, PEREZ A, UNTIVEROS M, QUISPE D, FUENTES S, BARKER I, SIMON R. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: A generic method for diagnosis, discovery and sequencing of viruses., 2009, 388(1): 1-7.
[14] YAMASAKI S, SAKAI J, FUJI S, KAMISOYAMA S, EMOTO K, OHSHIMA K, HANADA K. Comparisons among isolates ofusing complete genomic RNA sequences., 2010, 155(5): 795-800.
[15] KWAK H R, KIM J, KIM M K, SEO J K, JUNG M N, KIM J S, LEE S C, CHOI H S. Molecular characterization of five potyviruses infecting Korean sweet potatoes based on analyses of complete genome sequences., 2015, 31(4): 388-401.
[16] MINGOT A, VALLI A, RODAMILANS B,LEON D S, BAULCOMBE D C, GARCIA J A, LOPEZ-MOYA J J. The P1N-PISPO trans-frame gene of sweet potato feathery mottle potyvirus is produced during virus infection and functions as RNA silencing suppressor., 2016, 90(7): 3543-3557.
[17] MAINA S, BARBETTI M J, EDWARDS O, DE ALMEIDA L, XIMENES A, JONES R A C.andfrom East Timorese and Australian sweetpotato: Biological and molecular properties, and biosecurity implications., 2018, 102(3): 589-599.
[18] MAINA S, BARBETTI M J, MARTIN D P, EDWARDS O R, JONES R A C. New isolates ofand: Biological and molecular properties, and recombination analysis based on complete genomes., 2018, 102(10): 1899-1914.
[19] CLARK C A, DAVIS J A, ABAD J A, CUELLAR W J, FUENTES S, KREUZE J F, GIBSON R W, MUKASA S B, TUGUME A K, TAIRO F D, VALKONEN J P T. Sweetpotato viruses: 15 years of progress on understanding and managing complex diseases., 2012, 96(2): 168-185.
[20] UNTIVEROS M, QUISPE D, KREUZE J. Analysis of complete genomic sequences of isolates of thestrains C and EA: molecular evidence for two distinct potyvirus species and two P1 protein domains., 2010, 155(12): 2059-2063.
[21] LIU Q L, WANG Y J, ZHANG Z C, LV H, QIAO Q, QIN Y H, ZHANG D S, TIAN Y T, WANG S, LI J Q. Diversity of sweepoviruses infecting sweet potato in China., 2017, 101(12): 2098-2103.
[22] 張鳳桐, 程林發, 耿超, 田延平, 原雪峰, 白艷菊, 李向東. 一株PVYNTN-NW黑龍江馬鈴薯分離物的檢測鑒定. 植物病理學報, 2019, 49(4): 512-519.
ZHANG F T, CHENG L F, GENG C, TIAN Y P, YUAN X F, BAI Y J, LI X D. Detection and identification of a(PVY)NTN-NWisolate from potato in Heilongjiang, China., 2019, 49(4): 512-519. (in Chinese)
[23] 李科, 時洪偉, 荊陳沉, 孫現超, 周常勇, 青玲. ACLSV山東蘋果分離物基因組重組及序列多樣性分析. 中國農業科學, 2015, 48(14): 2857-2867.
LI K, SHI H W, JING C C, SUN X C, ZHOU C Y, QING L. Analysis of genome recombination andsequence diversity of ACLSV apple isolate from Shandong., 2015, 48(14): 2857-2867. (in Chinese)
[24] 李汝剛, 朱笑梅, 薛愛紅, 蔡少華, 王小鳳. 甘薯病毒病的研究. I甘薯羽狀斑駁病毒的分離、鑒定. 植物病理學報, 1992, 22(4): 319-322.
LI R G, ZHU X M, XUE A H, CAI S H, WANG X F. Identification of Langfang isolate of., 1992, 22(4): 319-322. (in Chinese)
[25] 朱作為, 薛啟漢, 楊永嘉, 邢繼英. 甘薯羽狀斑駁病毒的分離和提純. 江蘇農業學報, 1994, 10(1): 47-49.
ZHU Z W, XUE Q H, YANG Y J, XING J Y. Isolation and purification of., 1994, 10(1): 47-49. (in Chinese)
[26] 楊崇良, 路興波, 王升吉, 尚佑芬, 趙玖華, 李長松. 甘薯羽狀斑駁病毒(SPFMV)生物學性狀研究. 山東農業科學, 2001, 33(1): 26-29.
YANG C L, LU X B, WANG S J, SHANG Y F, ZHAO J H, LI C S. Biological characteristics research on., 2001, 33(1): 26-29. (in Chinese)
[27] 張振臣, 李大偉, 陳健夫, 于嘉林, 喬奇, 靳秀蘭. 甘薯羽狀斑駁病毒外殼蛋白基因在大腸桿菌中的表達及特異抗血清的制備. 農業生物技術學報, 2000, 8(2): 177-179.
ZHANG Z C, LI D W, CHEN J F, YU J L, QIAO Q, JIN X L. Overexpression ofcoat protein inand preparation of its specific antiserum., 2000, 8(2): 177-179. (in Chinese)
[28] 張盼, 蘭新芝, 喬奇, 張德勝, 秦艷紅, 田雨婷, 王爽, 張振臣. 甘薯病毒病害(SPVD)的多重RT-PCR檢測方法及其應用. 植物保護, 2013, 39(2): 86-90.
ZHANG P, LAN X Z, QIAO Q, ZHANG D S, QIN Y H, TIAN Y T, WANG S, ZHANG Z C. Development and application of a multiplex RT-PCR detection method for sweet potato virus disease (SPVD)., 2013, 39(2): 86-90. (in Chinese)
[29] 許泳清, 李華偉, 劉中華, 邱永祥, 羅文彬, 紀榮昌, 湯浩, 邱思鑫, 余華. 甘薯羽狀斑駁病毒(SPFMV)ELISA鑒定及RT-PCR檢測方法的建立. 福建農業學報, 2013, 28(12): 1267-1272.
XU Y Q, LI H W, LIU Z H, QIU Y X, LUO W B, JI R C, TANG H, QIU S X, YU H. ELISA identification and development of RT-PCR detection of., 2013, 28(12): 1267-1272. (in Chinese)
[30] 王麗, 王振東, 喬奇, 秦艷紅, 張德勝, 田雨婷, 王爽, 張立軍, 張振臣. 甘薯羽狀斑駁病毒實時熒光定量PCR檢測方法的建立. 沈陽農業大學學報, 2013, 44(2): 129-135.
WANG L, WANG Z D, QIAO Q, QIN Y H, ZHANG D S, TIAN Y T, WANG S, ZHANG L J, ZHANG Z C. Development of real-time fluorescent quantitative PCR assay for detection of., 2013, 44(2): 129-135. (in Chinese)
[31] 何海旺, 何虎翼, 譚冠寧, 劉義明, 何新民, 唐洲萍, 李麗淑, 王暉. 反向斑點雜交法快速檢測甘薯羽狀斑駁病毒和甘薯G病毒. 南方農業學報, 2014, 45(1): 43-48.
HE H W, HE H Y, TAN G N, LIU Y M, HE X M, TANG Z P, LI L S, WANG H. Detection of SPFMV and SPVG by using reverse dot blot hybridization system., 2014, 45(1): 43-48. (in Chinese)
[32] 許泳清, 李華偉, 邱思鑫, 劉中華, 邱永祥, 羅文彬, 紀榮昌, 湯浩, 余華. 甘薯羽狀斑駁病毒和褪綠矮化病毒雙重RT-PCR檢測方法的建立. 福建農業學報, 2014, 29(11): 1114-1117.
XU Y Q, LI H W, QIU S X, LIU Z H, QIU Y X, LUO W B, JI R C, TANG H, YU H. Development of dutiplex RT-PCR for the detection of SPFMV and SPCSV in sweet potato., 2014, 29(11): 1114-1117. (in Chinese)
[33] 李華偉, 許泳清, 邱思鑫, 劉中華, 邱永祥, 羅文彬, 湯浩, 余華. 侵染甘薯的SPCSV、SPVG、SPFMV多重RT-PCR檢測方法的建立及應用. 核農學報, 2015, 29(8): 1464-1470.
LI H W, XU Y Q, QIU S X, LIU Z H, QIU Y X, LUO W B, TANG H, YU H. Establishment and application of a multiplex RT-PCR detection method for SPCSV, SPVG and SPFMV Infecting sweetpotato., 2015, 29(8): 1464-1470. (in Chinese)
[34] 盧會翔, 呂長文, 吳正丹, 羅凱, 尹旺, 楊航, 王季春, 張凱. 甘薯羽狀斑駁病毒(SPFMV)和甘薯褪綠矮化病毒(SPCSV)熒光定量RT-PCR檢測方法的建立. 中國農業科學, 2016, 49(1): 90-102.
LU H X, Lü C W, WU Z D, LUO K, YIN W, YANG H, WANG J C, ZHANG K. Development of detection method for(SPFMV) and(SPCSV) through fluorescence quantitative RT-PCR., 2016, 49(1): 90-102. (in Chinese)
[35] 蔣素華, 程喜梅, 宋彩霞, 許申平, 梁芳, 崔波. 三種甘薯病毒多重RT-PCR檢測技術的建立. 植物保護, 2017, 43(1): 126-130.
JIANG S H, CHENG X M, SONG C X, XU S P, LIANG F, CUI B. Establishment of multiplex RT-PCR system for detection of three viruses in sweet potato., 2017, 43(1): 126-130. (in Chinese)
[36] 姜姍姍, 馮佳, 張眉, 王升吉, 辛志梅, 吳斌, 辛相啟. 甘薯羽狀斑駁病毒RT-LAMP快速檢測方法的建立. 中國農業科學, 2018, 51(7): 1294-1302.
JIANG S S, FENG J, ZHANG M, WANG S J, XIN Z M, WU B, XIN X Q. Development of RT-LAMP assay for rapid detection of(SPFMV)., 2018, 51(7): 1294-1302. (in Chinese)
[37] 黃廣學, 孟利前, 朱建晨, 張進, 李龐博, 肖海峻. 甘薯羽狀斑駁病毒(SPFMV)和褪綠矮化病毒(SPCSV)的雙重RT-PCR檢測技術體系構建. 沈陽農業大學學報, 2018, 49(6): 724-729.
HUANG G X, MENG L Q, ZHU J C, ZHANG J, LI P B, XIAO H J. Establishment of duplex RT-PCR detection method for SPFMV and SPCSV infecting sweet potato., 2018, 49(6): 724-729. (in Chinese)
[38] 肖海峻, 孟利前, 朱建晨, 張進, 李龐博, 黃廣學. 甘薯羽狀斑駁病毒的RT-PCR檢測技術構建. 分子植物育種, 2019, 17(13): 4302-4306.
XIAO H J, MENG L Q, ZHU J C, ZHANG J, LI P B, HUANG G X. Construction of RT-PCR detection technique for., 2019, 17(13): 4302-4306. (in Chinese)
[39] 張振臣, 喬奇, 秦艷紅, 張德勝, 田雨婷. 我國發現由甘薯褪綠矮化病毒和甘薯羽狀斑駁病毒協生共侵染引起的甘薯病毒病害. 植物病理學報, 2012, 42(3): 328-333.
ZHANG Z C, QIAO Q, QIN Y H, ZHANG D S, TIAN Y T. First evidence for occurrence of sweet potato virus disease (SPVD) caused by dual infection ofandin China., 2012, 42(3): 328-333. (in Chinese)
[40] 張新新, 王旭芳, 林堅淳, 余竟成, 黃立飛, 董章勇. 甘薯毀滅性病毒病害(SPVD)的研究進展. 中國農學通報, 2019, 35(1): 118-126.
ZHANG X X, WANG X F, LIN J C, YU J C, HUANG L F, DONG Z Y. Sweetpotato virus diseases (SPVD): research progress., 2019, 35(1): 118-126. (in Chinese)
[41] KREUZE J F, KARYEIJA R F, GIBSON R W, VALKONEN J P T. Comparisons of coat protein gene sequences show that Ease African isolates ofform a genetically distinct group., 2000, 145(3): 567-574.
Complete nucleotide sequence analysis and genetic characterization of theO and RC strains isolated from China
QIN YanHong, WANG YongJiang, WANG Shuang, QIAO Qi, TIAN YuTing, ZHANG DeSheng, ZHANG ZhenChen
(Institute of Plant Protection, Henan Academy of Agricultural Sciences/Henan Key Laboratory of Crop Pest Control/ IPM Key Laboratory in Southern part of North China, Ministry of Agriculture and Rural Affairs, Zhengzhou 450002)
【】The objective of this study is to clone the complete nucleotide sequence of Chinese isolate of(SPFMV) O and RC strains, elucidate the genomic structural characterization and variation of SPFMV-O-Ch1 and SPFMV-RC-Ch1, and to lay a foundation for the study of pathogenic mechanism of SPFMV.【】According to the SPFMV genome sequences available in GenBank database, 2 pairs of degenerate primers and 3 pairs of specific primers were designed, the whole genome of SPFMV O and RC strains isolated from China was amplified by RT-PCR from sweet potato leaves infection with SPFMV and subsequently cloned into vector pMD19-T and sequenced. The complete genome sequences of SPFMV-O-Ch1 and SPFMV-RC-Ch1 isolates were assembled by using DNAMAN. Genetic variation analyses of complete genomic sequences, polyproteins, and individual protein sequences were performed using DNAMAN. Neighbor-joining phylogenetic tree of SPFMV-O-Ch1 and SPFMV-RC-Ch1 isolates with other isolates was constructed using MEGA7.0 software. Recombination analyses were carried out using RDP software.【】The amplification and sequencing revealed that the complete nucleotide sequence of SPFMV-O-Ch1 and SPFMV-RC-Ch1 isolates was 10 992 nucleotides (nt) and 10 851 nt in length, respectively. The viral genome of SPFMV-O-Ch1 isolate contained a single open reading frame (ORF) of 10 557 nt encoding a polyprotein of 3 518 aa. SPFMV-RC-Ch1 isolate polyprotein consisted of 10 482 nt and encoded 3 493 aa. Two small ORFs, P1N-PISPO and P3N-PIPO were identified in the P1 and P3 proteins of these two isolates. Pairwise comparisons of the complete genome nucleotide sequence showed that O-Ch1 had 87.3% identity with RC-Ch1 isolate and shared 86.0%-95.8% sequence identity with other SPFMV isolates. It was most closely related to the isolate Ruk73 with 95.8% nt identity and lowest nt identity with 11-1 isolate (86.0%). RC-Ch1 and other SPFMV isolates shared 85.9%-98.7% sequence identity at the complete genome nucleotide sequence level. It had the highest nt identity with IS90 isolate (98.7%), and had lowest nt identity with Aus1-2B isolate (85.9%). Phylogenetic tree analysis based on polyprotein gene indicated that SPFMV-O-Ch1 formed a branch with the isolates of O strain containing Ordinary, 10-O and 17-O, and SPFMV-RC-Ch1 formed a branch with the isolates of RC strain containing S, IS90 and CW137.Recombination analysis showed that there were three potential significant recombination events occurred in 7 731- 9 710, 135-10 012 and 4 825-6 948 nt of O-Ch1 isolate genome, respectively. No recombination event was detected in the complete genome of RC-Ch1 isolate. 【】The genomic organizations of SPFMV-O-Ch1 and SPFMV-RC-Ch1 isolates were same to other isolates. O-Ch1 isolate was closely related to the isolates of O strain and RC-Ch1 isolate was closely related to those isolates of RC strain. Three recombination events were detected in O-Ch1 isolate, but no recombination event was detected in RC-Ch1 isolate.
(SPFMV); strain; complete nucleotide sequence; genetic variation; recombination analysis

10.3864/j.issn.0578-1752.2020.11.007
2019-11-07;
2019-11-28
國家甘薯產業技術體系(CARS-10-B13)、河南省自然科學基金(162300410160)、河南省農業科學院自主創新基金(2019ZC37)
秦艷紅,E-mail:qinyanhong6040@163.com。通信作者張振臣,E-mail:zhangzhenchen@126.com
(責任編輯 岳梅)

