甘薯近緣種Ipomoeacordatotriloba基因組大小測定及高通量調查測序
王珧 鄧逸桐 戴習彬 張安 曹清河 陳艷麗
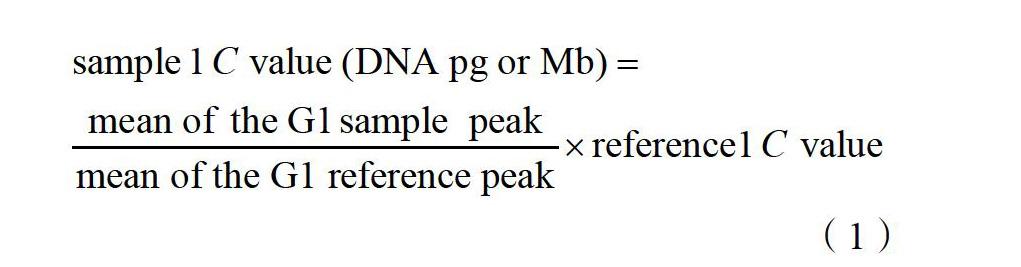
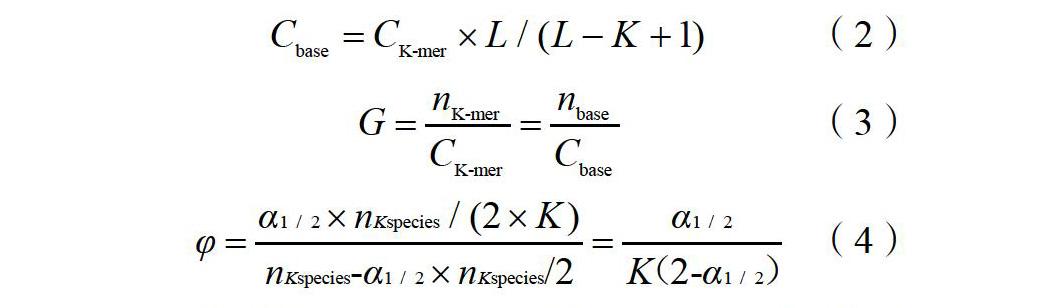
摘? 要:利用流式細胞術,以大豆Williams 82為內參,對甘薯近緣野生種Ipomoea cordatotriloba進行基因組大小測定,結合染色體壓片技術進行倍性鑒定,并利用二代高通量測序技術對測定結果進行驗證。結果表明:利用流式細胞術測得I. cordatotriloba基因組大小為(539.69±13.76)Mb;染色體壓片結果顯示其染色體數目為30條,由于其基數x=15,從而獲知I. cordatotriloba為二倍體;經K-mer計算并修正后得出的C值為560.70 Mb,與流式細胞分析結果相近;同時,測序結果顯示其基因組雜合率為0.40%;重復序列比率為57.93%;GC含量為38.10%。以上結果為此物種全基因組精細圖譜繪制打下基礎,同時為甘薯近緣野生種的利用提供參考。
關鍵詞:甘薯;Ipomoea cordatotriloba;基因組大小;流式細胞法;K-mer計算中圖分類號:S531;Q75 ?????文獻標識碼:A
DNA Amount Estimation and Genome Survey of Sweetpotato Wild RelativesIpomoea cordatotriloba
WANG Yao1, 2, DENG Yitong2, DAI Xibin2, ZHANG An2, CAO Qinghe2, CHEN Yanli1*
1. College of Horticulture, Hainan University, Haikou, Hainan 570228, China; 2. Jiangsu Xuhuai Regional Xuzhou Institute of Agricultural Sciences, Xuzhou, Jiangsu 221131, China
Abstract: DNA content of sweetpotato wild relativesIpomoea cordatotrilobawas determinated by flow cytometry using soybeanWilliams 82 as the internal reference in this study. The ploidy was determined by chromosome counting. In order to verify the reliability of the results, the next-generation sequencing was used to further test and verify. It was a diploid species with base number was 15. The genome size was (539.69±13.76) Mb by the flow cytometry analysis. The C-value determined by sequencing was similar to the results of flow cytometry. Furthermore, the genomic heterozygosity rate, repeat sequence ratio and GC content was 0.40%, 57.93% and 38.10%, respectively. The above results would lay the foundation for the whole genome sequencing and assemble of this species, and provide references for the utilization of sweetpotato relatives.
Keywords: sweetpotato;Ipomoea cordatotriloba; genome size; flow cytometry; sequencing
DOI: 10.3969/j.issn.1000-2561.2020.06.012
甘薯因具有營養豐富、適應性廣等優點大面積種植于熱帶、亞熱帶地區[1],是世界第七大主糧作物(FAO,2018)。甘薯屬植物約有800~900個種,其中有二倍體、四倍體和六倍體等不同倍性的物種資源,且許多野生種具有優良的抗性基因[2-3]。基因組特征研究是植物基因資源開發、利用的前提。基因組大小、重復序列比例、雜合率和GC含量等參數會影響基因組的拼接組裝過程[4]。基因組大小是指物種單倍體所含的DNA量,是表征生物基因組多樣性的基本參數[5]。目前測定基因組大小的方法很多,流式細胞法(Flow cytometry,FCM)因其快速、準確的特點而被廣泛應用[6],超過80%的物種都是采用該方法進行基因組大小測定[7],如棗、月季、草莓、靈芝[8-11]等。近年來,采用低深度測序后進行K-mer分析也是常用方法之一[12]。通過低深度高通量測序(Survey分析)判斷基因組大小及復雜程度,以此選擇合適的后續測序方案,從而增加測序的準確性[13]。
由于倍性復雜,甘薯遺傳基礎薄弱,導致甘薯的物種起源和基因組學研究落后于其他主要農作物[14]。近年來甘薯基因組研究逐漸興起,然而甘薯屬多數物種仍缺乏基本的基因組信息。Ipo?moeacordatotriloba是甘薯組與栽培甘薯親緣關系較近的13個物種之一,對研究甘薯親緣關系及物種起源具有重要意義。本研究利用流式細胞術和染色體計數法,結合高通量測序技術,對I. cordatotriloba的基因組大小、倍性及基因組特征進行評估,為下一步全基因組測序提供參考并為甘薯野生資源利用提供支撐。
1? 材料與方法
1.1材料
Ipomoea cordatotriloba(PI 518495)引自美國國家種質資源庫,來源地為墨西哥Tabasco州。用已知基因組大小的大豆Williams 82(1?C= 1127?Mb[15])作為內參。
1.2方法
1.2.1? 流式細胞術? 將搜集的野生種種子浸種催芽之后置于25?℃的溫室內生根,于1~2?d后取200?mg根尖進行實驗;參照Peggy Ozias-Akins[16]的實驗方法,取樣后用蒸餾水洗凈、晾干,放入預冷的無菌培養皿中,加入預冷4?℃的改良硫酸鎂裂解液600 μL。材料須浸沒在裂解液中,用一次性滅菌刀片在一分鐘內快速切碎該組織;用38?μm篩網過濾到1.5?mL EP管中;以2000 r/min離心6?min,棄上清液;加入200?μL PI染液(含濃度為50?μg/mL的RNase酶)置于4?℃冰箱避光染色15?min,添加染料后震蕩EP管;最后用BD公司C6流式細胞儀收集10 000個事件,重復測定3次。通過公式(1)計算基因組大小[17](單位用pg或Mb來表示,兩者間的換算關系為1?pg=978 Mb[18])。
1.2.2? 染色體壓片? 參照楊慧嫻等[19]的方法,取I. cordatotriloba新鮮1 cm根尖于8-羥基喹啉中處理2~3 h,經蒸餾水洗凈后轉移至卡諾固定液中,24 h后于70%酒精中保存備用。實驗時,取出根尖先用無菌水沖洗2~3次,再用檸檬酸沖洗2次,吸干檸檬酸后加入200 μL 2%纖維素酶和1%果膠酶的混合酶液進行酶解。切取根尖于載玻片上,用濾紙吸走多余水分,取干凈的鑷子輕輕敲打根尖。制片成功后加DAPI染色,于熒光顯微鏡下觀察并拍照。
1.2.3? 基因組survey分析? 取4~6片甘薯野生種I. cordatotriloba葉片,洗凈后置于液氮保存,委托北京諾禾致源科技股份有限公司進行測序。通過Covaris超聲波破碎儀將DNA樣品隨機打斷,構建250 bp小片段測序文庫。將得到的原始序列數據進行質控,去除污染序列,得到有效數據,采用K-mer計算方法[20]分析有效數據。通過公式(2)、(3)可估計基因組大小。通過公式(4)(適用于重復序列影響較小的基因組)計算雜合率,參照霍愷森等[12]的方法(純合峰深度1.8倍后面的K-mer數/K-mer總數?100%)來估計重復序列比例。
1.2.4? 基因組初步組裝? 通過被reads截斷的更小序列片段之間的重疊關系構建de Brujin圖,去掉測序錯誤造成的分支,隨機選擇少量雜合位點并合并。將簡化后的de Brujin圖的每個分叉位點的序列截斷,即為最初的Contigs。再將reads和Contigs進行比對,將Contigs組裝成Scaffolds。
2? 結果與分析
2.1流式分析結果
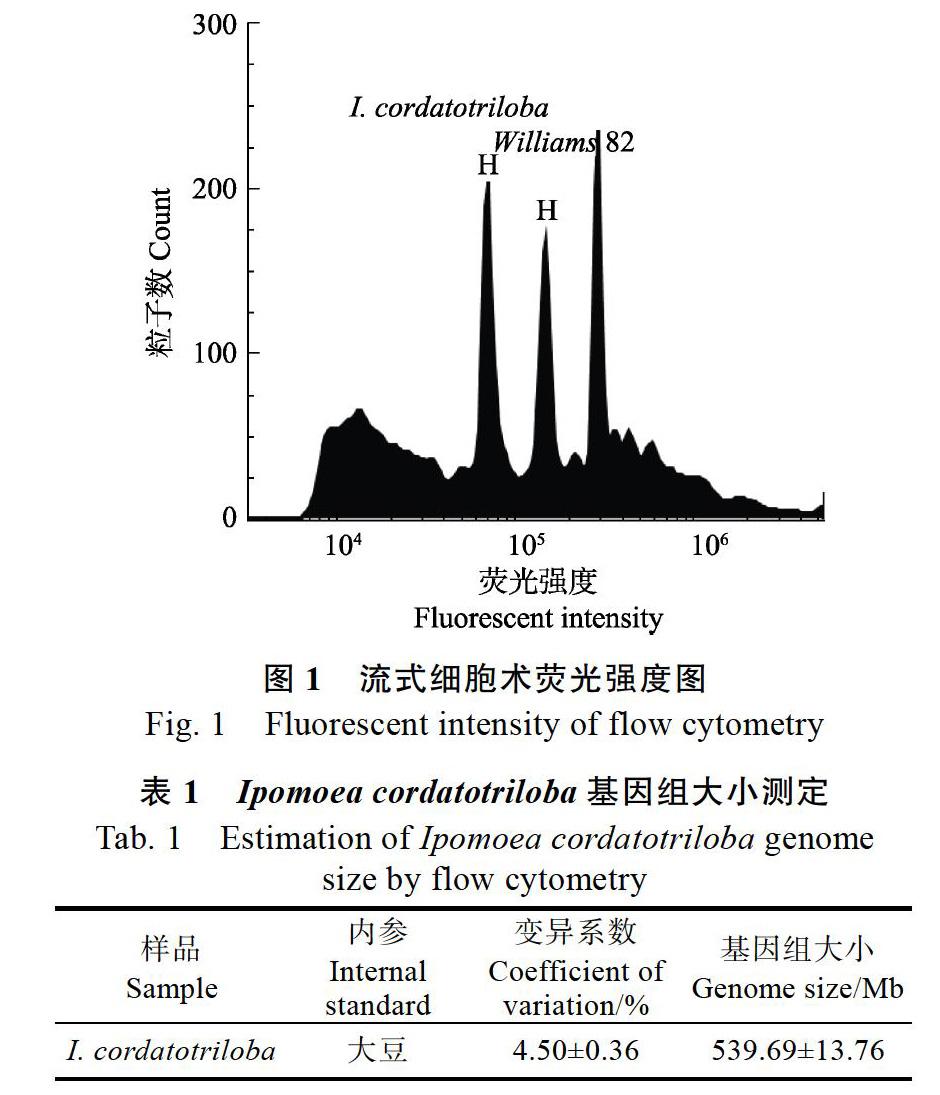
圖1為大豆作為內參時,參照物與待測樣本的熒光強度圖。圖中I. cordatotriloba和大豆主峰清晰可見,區分明顯。圖1中3個峰分別為I. cor?datotriloba的G1峰,Williams 82大豆的G1峰以及G2峰。參照物和樣本峰的CV值均小于5%,表明測定結果穩定可靠。通過對參照物和待測樣品3個生物學重復測定,I. cordatotrilobaG1峰的平均熒光強度為71?291.67,Williams 82大豆G1峰的平均熒光強度為148 601.00,根據公式(1)計算得出(表1),I. cordatotriloba基因組大小為(539.69±13.76)Mb。
2.2染色體壓片結果
通過染色體壓片計數法,確定I. cordatotriloba的倍性。DAPI染液只對染色體進行特異性染色,雜質無法被染色,因此較易判斷染色體的輪廓,背景干凈,計數較方便。I. Corda?totri?lo?ba染色體形態清晰,呈長棒狀,分散良好,粘連現象較少,存在拖尾現象但不影響計數(圖2)。通過觀察10個細胞的分裂相,染色體數目均為30條,由于甘薯屬植物染色體基數x=15,從而獲知I. cordatotriloba為二倍體。
2.3基因組survey分析結果
2.3.1? 測序產出數據統計? 對經過Illumina高通量測序技術得到的數據進行嚴格過濾,得到24.46?Gb的有效數據用于K-mer分析。由表2可知,測序質量≥20的堿基比率為97.01%,測序質量≥30的堿基比率達到92.05%,說明I. cordatotriloba基因組的測序質量較高。
2.3.2? K-mer分析及基因組特征預估? 本研究選用K-mer=17進行分析,其頻率分布圖(圖3)顯示,I. cordatotriloba的期望深度即圖中的主峰,在38附近,且主峰的2倍處有凸起,表明樣品有一定重復率,經計算I. cordatotriloba的重復序列比率為57.93%,并根據公式(4)得到雜合率為0.40%。由表3可知,I. cordatotriloba的K-mer數量為18 867 813 720,通過公式(3)計算得出I. cordatotriloba基因組大小為571.75 Mb,經修正后為560.70 Mb。
2.3.3? 基因組組裝結果分析? K-mer值取41時可得到最優的拼接效果。將24.46?Gb有效數據進行拼接,分別得到978?566條Contigs和961?850條Scaffolds;由表4可知,I. corda?to?triloba經組裝后得到Contigs總長度為466?005?979?bp,占基因組大小的81.3%,其中最長為67?068?bp。Scaffolds總長度為467?229?510?bp,占基因組大小的81.5%,最長為67?068?bp。
圖4顯示I. cordatotriloba只有1個明顯的峰,與K-mer結合分析得知,該樣品為純合峰。如圖5,右方是測序深度分布,上方是GC含量分布,圖中顏色越紅,表示密度越大。GC含量分布在25%~55%,由上方含量分布峰可知40%?左右最密集,預估樣本的GC含量為38.10%。圖中未出現明顯分層現象,且無高GC含量區域,故認為I. cordatotriloba無細菌污染。
3? 討論
基因組大小是分析種群進化、物種分類和生態學等領域研究的重要參照[21]。本研究通過對I. cordatotriloba基因組大小進行測定,為探究甘薯近緣野生種的基因組學信息提供參考。張志珂等[22]用蘿卜、大豆、番茄做外參,通過流式細胞術估算‘解放鐘枇杷基因組大小為654.399 Mb,與測序得到的結果773?Mb有較大的差異,誤差達15.3%。Bennett等[23]以新桿狀線蟲和果蠅作為內標測得擬南芥的基因組大小為157 Mb,與早期測序得到的125?Mb也存在較大差異,誤差為基因組大小的25.6%。本研究以Williams 82大豆為內參,通過流式細胞儀測定I. cordatotriloba的基因組大小為(539.69±13.76)Mb。基于K-mer分析得到的I. cordatotriloba的基因組大小為560.70?Mb,與流式細胞術得到的結果誤差僅為3.7%,說明2種測定方法得到的結果較為接近。前人研究表明I. cordatotriloba是四倍體,基因組大小為3.3?pg/2C[16],與本研究得出的基因組大小差異較大。隨著科學技術的發展,現代的流式細胞儀器更先進,準確性更高,且本研究根據染色體計數得出I. cordatotriloba為二倍體,結合survey預估的數據認為本研究的結果更為準確。
植物基因組雜合受到品系、繁殖方式等因素影響[24]。甘薯具有自交不親和性[25],有性繁殖和無性繁殖共存。雜合率低的植物種群內及種群間基因交流程度低,基因資源穩定,環境適應能力較強[26]。郭水良等[27]認為同一科(或屬)內相關物種間的DNA C-值較小的植物可能具有更強的入侵性。甘薯近緣野生種馬鞍藤(Ipomoea pes-ca?praeL.)雜合率為0.99%,DNA C-值為1041.65 Mb[12],I. cordatotriloba雜合率為0.40%,DNA C-值為560.70 Mb;由此推測,I. cordatotriloba環境適應能力比馬鞍藤更強,是進一步篩選優異基因資源、研究甘薯近緣野生種的重要材料。
從Sanger測序到第2代(Roche 454、SoLiDTM、Illumina)測序平臺以及第3代PacBio測序平臺,基因組測序的蓬勃發展使得測序技術普遍應用于植物研究中[28]。基因組越大、倍性越高對測序技術、機器性能要求越高;而雜合度會導致組裝基因組偏大,數值高于0.5%則認為該品種不易組裝,高于1%則認為該品種很難實現組裝[29-30]。本研究利用二代Illumina測序平臺對I. cord?a?totriloba進行K-mer分析,得到I. cordatotriloba基因組大小為560.70?Mb,雜合率為0.40%,重復序列比率為57.93%,GC含量為38.10%,具有一定重復率和較低的雜合度。結合染色體壓片法進行染色體計數可知I. cordatotriloba為二倍體。綜上所述,研究認為I. cordatotriloba為簡單基因組,可盡快實施全基因組測序計劃,可采用低成本二代測序技術進行全基因組測序。以上結果為I. cordatotriloba全基因組測序策略的選取和甘薯近緣野生種的利用提供參考。
參考文獻
- 劉慶昌. 甘薯在我國糧食和能源安全中的重要作用[J]. 科技導報, 2004(9): 21-22.
- 曹清河, 馬代夫, 張? 安. 甘薯近緣種染色體核型及花粉粒超微結構分析[J]. 西北植物學報, 2008(8): 1610-1613.
- Srivastava G, Mehrotra R C, Dilcher D L. Paleocene Ipomoea (Convolvulaceae) from India with implications for an East Gondwana origin of Convolvulaceae[J]. Proceedings of the National Academy of Sciences, 2018, 115(23): 6028- 6033.
- 鐘永達, 張? 新, 李彥強, 等. 鵝掌楸全基因組調查[J]. 分子植物育種, 2017, 15(2): 507-512.
- 馬鵬舉, 周佳熠, 孫會改, 等. 基于流式細胞術和K-mer分析的好好芭基因組大小估測[J]. 北京師范大學學報(自然科學版), 2018, 54(6): 733-737.
- 鄧果特, 劉清波, 蔣建雄, 等. 五節芒基因組大小測定[J]. 植物遺傳資源學報, 2013, 14(2): 339-341, 346.
- 田新民, 周香艷, 弓? 娜. 流式細胞術在植物學研究中的應用——檢測植物核DNA含量和倍性水平[J]. 中國農學通報, 2011, 27(9): 21-27.
- 王利虎, 呂? 曄, 羅? 智, 等. 流式細胞術估測棗染色體倍性和基因組大小方法的建立及應用[J]. 農業生物技術學報, 2018, 26(3): 511-520.
- 武榮花, 葛蓓蓓, 王茂良, 等. 應用流式細胞術測定18個中國古老月季基因組大小[J]. 北京林業大學學報, 2016, 38(6): 94-100.
- 陳丙義, 李金鳳, 霍恒志, 等. 6種野生草莓基因組大小估算[J]. 果樹學報, 2015, 32(1): 51-56.
- 李秋實, 徐? 江, 朱英杰, 等. 基于流式細胞技術的靈芝基因組大小估測[J]. 菌物學報, 2013, 32(5): 899-906.
- 霍愷森, 趙冬蘭, 陳艷麗, 等. 甘薯屬耐鹽植物馬鞍藤基因組大小及特征分析[J]. 植物遺傳資源學報, 2019, 20(3): 728-735.
- 施季森, 王占軍, 陳金慧. 木本植物全基因組測序研究進展[J]. 遺傳, 2012, 34(2): 145-156.
- 楊? 俊, 范維娟, 王紅霞, 等. 番薯屬植物基因組測序最新進展[J]. 植物生理學報, 2017, 53(5): 768-771.
- Schmutz J, Cannon S B, Schlueter J, et al. Genome sequence of the palaeopolyploid soybean[J]. Nature, 2010, 463(7278): 178-183.
- Ozias-Akins P, Jarret R. Nuclear DNA content and ploidy levels in the genus Ipomoea[J]. Journal of the American Society for Horticultural Science, 1994, 119(1): 110-115.
- Dolezel J, Greilhuber J, Suda J. Estimation of nuclear DNA content in plants using flow cytometry[J]. Nature Protocols, 2007, 2(9): 2233-2244.
- Dolezel J, Bartos J, Voglmayr H, et al. Nuclear DNA content and genome size of trout and human[J]. Cytometry Part A: the Journal of the International Society for Analytical Cytology, 2003, 51(2): 127-128.
- 楊慧嫻, 孫文光, 歐曉昆, 等. 中國西南地區6種植物的核型研究及其系統學意義[J]. 西北植物學報, 2019, 39(4): 630-637.
- 張? 偉, 王? 瑞, 楊曉野, 等. 捕食性真菌Duddingtonia flagrans基因組DNA的提取及基因組survey分析[J]. 中國獸醫學報, 2017, 37(11): 2090-2094.
- 尤祿祥, 謝夢夢, 伊賢貴, 等. 早櫻種系基因組大小的研究[J]. 安徽農業大學學報, 2017, 44(6): 1093-1097.
- 張志珂, 王永清, 林順權, 等. 借助細胞流式儀進行枇杷基因組學測序材料的倍性鑒定[J]. 果樹學報, 2012, 29(3): 498-504.
- Bennett M D, Leitch I J, Price J H, et al. Comparisons with Caenorhabditis (~100 Mb) and Drosophila (~175 Mb) using flow cytometry show genome size in Arabidopsis to be ~157 Mb and thus ~25% larger than the Arabidopsis genome initiative estimate of ~125 Mb[J]. Annals of Botany, 2003, 91(5): 547-557.
- 宋立肖, 李國旗, 靳長青, 等. 大麻狀羅布麻的全基因組分析和SSR標記開發[J]. 植物遺傳資源學報, 2019, 20(5): 1309-1316.
- 張? 安, 戴習彬, 周志林, 等. 甘薯祖先種三淺裂野牽牛自交不親和研究進展[J]. 江西農業學報, 2017, 29(5): 17-21, 27.
- 王? 雪, 周佳熠, 孫會改, 等. 新疆沙冬青基因組調查測序與基因組大小預測[J]. 植物遺傳資源學報, 2018, 19(1): 143-149.
- 郭水良, 陳國奇, 毛俐慧. DNA C-值與被子植物入侵性關系的數據統計分析——以中國境內有分布的539種被子植物為例[J]. 生態學報, 2008(8): 3698-3705.
- Dolezel J, Bartos J. Plant DNA flow cytometry and estimation of nuclear genome size[J]. Annals of Botany, 2005, 95(1): 99-110.
- Feuillet C, Leach J E, Rogers J, et al. Crop genome sequencing: lessons and rationales[J]. Trends in Plant Science, 2011, 16(2): 77-88.
- Edwards M A, Henry R J. DNA sequencing methods contributing to new directions in cereal research[J]. Journal of Cereal Science, 2011, 54(3): 395-400.?

