基于Python的TarBase V8網站爬蟲設計與實現
蔣輝 謝云潔
摘要:在數據爆炸的時代,人們通過搜索引擎從網上來查找自己所需要的數據。但并不是所有的網站都提供所需要的數據下載。網絡爬蟲技術可以將查找到的數據抓取下來,以供研究人員研究使用。Scrapy是使用Python語言開發開源的爬蟲框架。該文將利用Scrapy框架和Python語言設計和實現一個TarBase V8網站爬蟲,對其網站miRNA靶基因數據進行獲取以及存儲。
關鍵詞:Scrapy;網絡爬蟲;搜索引擎;信息檢索;miRNA靶基因
中圖分類號:TP391 文獻標識碼:A
文章編號:1009-3044(2020)20-0020-03
Design and Implementation of Web Crawler on rrarBase V 8 Web Site Based in Python
JIANG Hui, XIE Yun-jie
(School of Computer Science. University of South C.hina, Hengyang 421001, China)
Abstract: In the era of data explosion, we can get the information or data from the internet by using search engines. But not all web-sites provide the data downloads we need. These data can be captured from the internet for the researching purpose by using webcrawler technology. Scrapy is an open source web crawler framework which is developed by Python. In the paper, we design and de-velop a web crawler which is based on Scrapy framework and Python f'or capturing the miRNA targets data from the TarBase V8website.
Key words: scrapy; web crawler; search engine; information retrieval; miRNA targets
1背景
miRNA是一類非編碼的小分子RNA,其在生命活動起著重要的作用。越來越多的研究表明miRNA與疾病的產生與發展有著重要的關系[1]。其機理是miRNA綁定到其靶基因上,使得靶基因降解或者翻譯抑制,致使該基因對應的蛋白表達異常,從而導致疾病。在研究miRNA的過程中,生物學實驗已經證實了非常多的miRNA靶基因信息,并以論文的方式發表。也有生物信息學的研究人員通過文獻挖掘的方法將分布在這些發表在論文中的miRNA靶基因信息挖掘出來,并建立了完善的數據庫供相關人員查詢或下載。TarBase就是其中之一,但由于TarBase V8[2]的數據庫其官網只提供查詢和部分信息下載,因此要獲取這些數據就變得比較困難。網絡爬蟲技術可以從網貞上獲取用戶指定的信息,因此本文使用Python語言設計和實現了網絡爬蟲來獲取這些數據。
2 Scrapy框架簡介
Scrapy[3]是Scrapinghub公司開發的開源的網站爬蟲應用框架,該框架基于Twistid異步網絡框架,使用Python語言開發。Scrapy以其開源性及優秀的系統架構獲得了廣泛的應用。Scrapy組件間的低耦合性,極易擴展性和組件的可定制開發性,使其可以靈活的完成各種需求。除爬蟲外,Scrapy框架還應用在數據挖掘、信息處理、存儲歷史數據等方面。
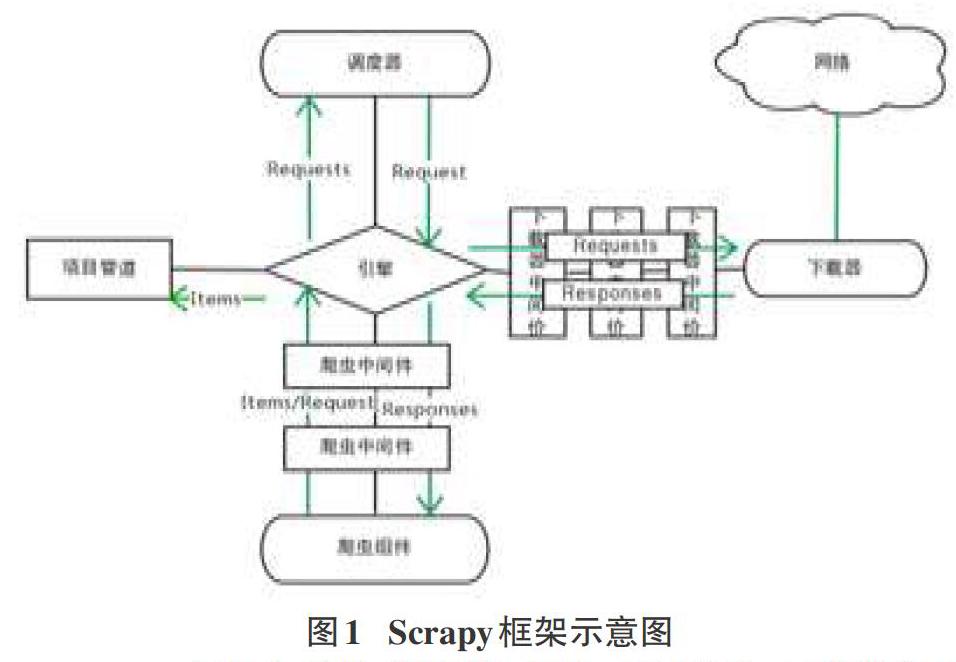
Scrapy框架由引擎、調度器、爬蟲、項目管道、下載器中間件、下載器、爬蟲中間件等組件構成。引擎負責數據和信號在不同組件間的傳遞;調度器是一個隊列,用來儲存請求的URL;爬蟲負責定義爬取的動作及分析網頁;項目管道處理引擎傳過來的數據,如保存,查重和驗證數據;下載器中間件處理從引擎和下載器之間的數據傳遞;下載器負責從網絡上下載引擎傳過來的請求;爬蟲中間件負責處理發送給爬蟲組件的Response及爬蟲組件產生的Item和Request。其框架的結構圖如圖l所示。
3基于python的TarBase V8網站爬蟲設計與實現
大數據時代的到來,使得人們淹沒在數據的海洋,這些數據中蘊含著大量的信息。對這些數據的深入研究可以發現更多有用的知識。網絡爬蟲技術可以從網絡上自動的下載所需要的數據。本文設計了一個基于Scrapy框架的TarBase V8網站爬蟲,獲取其miRNA靶基因數據。
3.1需求分析
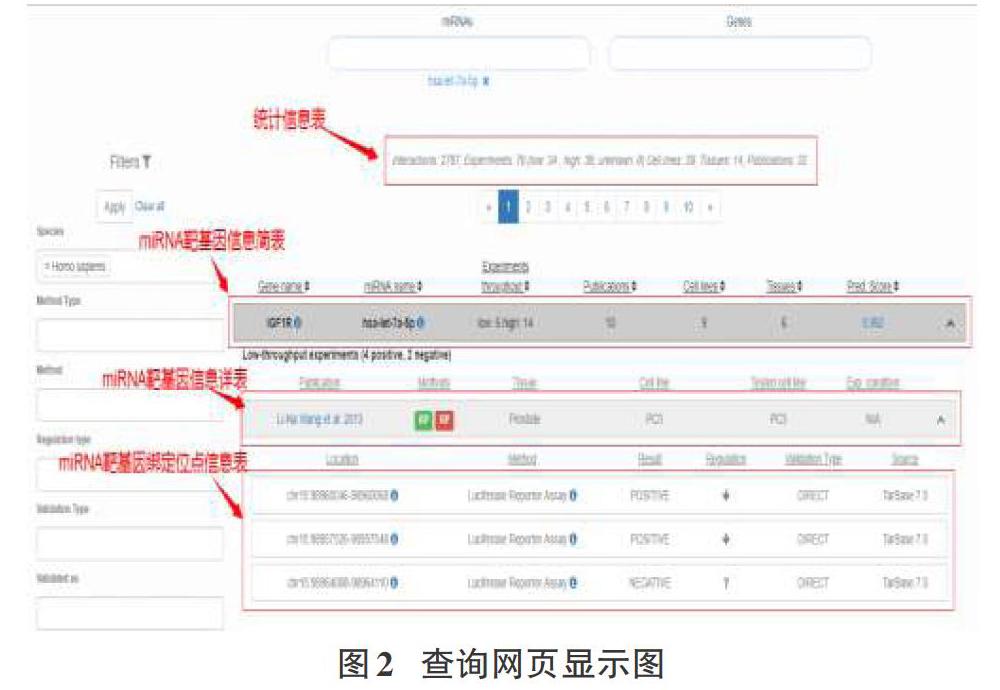
TarBase V8網站上記錄了每一對miRNA靶基因的所屬物種、驗證這miRNA靶基因對的生物實驗種類、支持文獻的PuLMed ID以及靶位點信息。將人類的miRNA名字“hsa-let-7a-5p”提交到網站的查詢頁面,服務器生成了URL用來查詢該miRNA的靶基因,其結果網頁如圖2所示。
我們所需要的數據可以分為四個類型:第一,miRNA靶基因信息簡表,它描述miRNA靶基因對的miRNA名字、基因名、支持文獻篇數以及用來實驗的Cell lines、Tissues數目等數據;第二,miRNA靶基因信息詳表,它描述每對miRNA靶基因的實驗細節,如具體的文獻、用來驗證的生物學實驗方法等;第三,miRNA靶基因綁定位點信息表,它描述miRNA靶基因對之間的綁定細節。如具體的綁定位置、實驗方法等;第四,統計信息表,它描述此miRNA查詢后的統計結果。
通過對服務器返回的html腳本的分析,前三類關系都是在同一個table標簽里面,在
里面的結點包含了miRNA靶基因關系簡表信息,后面的里構建了一個table保存miRNA和與之對應的基因的實驗細節,第三類型數據位于第二個table下面的子節點table()。由以上分析得出,根據標簽的class屬性可以區分所需數據類型。統計表則在另一個diy中,可以用來獲取。
TarBaseV8中的所使用的miRNA名字來源于miRNA信息數據庫miRBase[4](21版)。該數據庫中共有2588條人類的miRNA成熟體信息,可以每次獲取一個miRNA靶基因數據,然后循環2588次,就能將該數據庫中miRNA靶基因信息全部獲取出來,并保存在數據庫中。
3.2系統設計
3.2.1系統架構及其類的設計
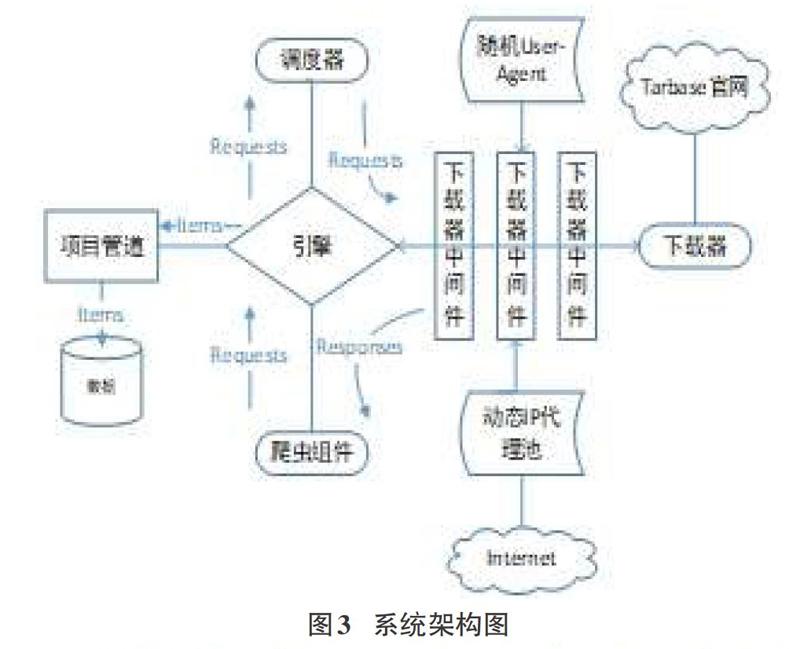
根據需求,并結合SCrapy框架,此爬蟲分為兩個主要的模塊:數據獲取模塊和數據存儲模塊。其整體的結構如圖3所示。
這樣設計使得模塊之間功能獨立相互獨立,降低了模塊間的耦合性。數據庫用來保存從網頁上獲取的數據,隨機User-Agent和動態IP代理池解決了網站反爬蟲的問題。
圖4為系統的類圖,其中getmiRNACount類為獲得統計信息表結果的類,ltem是一個字典類型,用于保存從網頁中解析到的統計信息結果;在search方法中解析網頁,其他的一些方法用于數據庫的操作。TarBase.py.Tarbase類用于爬取miRNA靶基因信息簡表,在start_requests方法中構造請求URL,在parse方法中解析獲取網頁中的數據。Spilder.get_second_third.py.TarhaseSpilder類用于爬取miRNA靶基因對信息詳表以及綁定信息表,在start_requests方法中根據在簡表中已經獲取到的miRNA名和基因名構造請求URL,在parse方法中解析獲取網頁中的數據。Item類型的類用于保存結構性數據,Tmiddler-wares.py.ProxyMiddleware類是一個下載器中間件,用于設置代理IP。
3.2.2數據庫的設計
根據上面對需求和網頁結構的分析,可以創建四個實體類,分別是mirna_info(miRNA靶基因信息簡要信息),through-put_experiments(miRNA靶基因信息詳細信息),binding_site(miRNA靶基因綁定位點信息),mirna_count(統計信息),數據庫的物理模型,如圖5所示。
由于TarBase V8中的miRNA靶基因對數據量比較大,會使得數據表中的數據非常多,從而導致查詢速度變慢。而查詢的主要關鍵詞為MiRNA名稱和基因名稱,因此為所有表的miR-NA name和gene_name屬性創建了索引。由于一個miRNA靶基因對有大于等于一條詳情,以及大于等于一條綁定位點信息,所以其基數比約束都為1:n。
3.2.3遇到的問題及其解決方案
在系統的設計中要遇到如下4個問題。
1)爬蟲獲取網頁數據時無法建立前三個表數據之間關聯
由于TarBase V8網站網頁結構的限制,以及爬蟲的并發操作等原因使得簡表數據與詳細表的數據以及綁定表數據之間沒有關聯。因此爬蟲所獲得的數據就是散亂的、相互之間沒有關系可循的。
為了解決這個問題,本文在設計了一個分步獲取數據的方法,先獲取所有miRNA靶基因信息簡表的數據,為每一對miR-NA靶基因在數據表中生成唯一id號。這樣就可以根據簡表中的每對miRNA名和Gene名來構造相應的請求URL,來獲取miRNA靶基因信息詳表和miRNA靶基因綁定位點信息表的數據,通過每個miRNA靶基因對的唯一id就可以關聯這三張表中的數據。
其偽代碼如下:
BECIN:
//循環從已知miRNA名列表中得到miRNA名稱;
Foreach miRNA_name in miRNA_list(已知數據miRNA名列表)
URL=generate url for miRNA_name;
//根據miRNA名生成的URL請求爬取網頁數據;
Html=spider(“URL”);
//將從網頁中篩選出所需要的信息并保存進數據庫表;
Save(Find the data in Html);
//循環從第一次保存的數據表中得到miRNA名和基因名;
roreach miRNA-gene pair in miRNA_info(簡表)
//根據miRNA名和基因名再次生成請求URL;
URL=generate url for miRNA_name and Gene_name;
Html= spider(“URL”);
Sava(Find the data in Html);
END
2)無法為抓取統計數據頁面構造URL
由于統計數據以ajax異步請求的方式獲得的,因此對獲取miRNA的統計數據帶來了一定的麻煩。對所抓得的統計數據進行分析發現,其URL的請求參數是一條sql語句,該語句中查詢條件是一串無規律的數字,這使得請求的URL構造困難。為了解決這個問題,本文使用selenium和Webdriver[5]來模擬人操作瀏覽器的方式獲取ajax請求返回的數據。
Selenium是ThoughtWorks專門為Web應用程序編寫的運行在瀏覽器上的Web測試工具,它能夠像人操作瀏覽器那樣模擬操作瀏覽器。Wehdriver是基于selenium設計的操作瀏覽器的一套API,它針對不同語言有對應的實現。
3)爬取數據時的翻頁
在查詢結果數據量大的情況下,TarBase V8網站會分頁展示結果。本文將采用遞歸的方式來做分頁請求并解析數據。
4)反反爬蟲技術
TarBase V8網站設置了反爬蟲機制來保證網站的正常運行。,本文采用IP代理和設置隨機User-Agent來解決這個問題.lP代理設置是在爬蟲外部構建一個動態代理lP池,其能不斷的從網上獲取免費的可以使用的代理IP,并定時更新動態池中的數據,然后將這些IP存人Redis數據庫中;隨機User-Agent的設置通過Python的第三方庫fake-useragent來生成。
為了讓此網站不發現是爬蟲程序在訪問,我們降低Scrapy框架中的并發數量并設置為10個。
4爬蟲的實現
本系統采用Python3.7,Selenium 3.141.0,Chromedriver和SCrapyl.6.0,Mysql 8.0來實現。
基于Scrapy框架的可定制性和易擴展性,根據對系統的需求分析,需要創建暫存爬取數據的Item組件,實現spider組件,用于構造請求的URL并調用parse方法解析請求返回的數據,并將其暫存到Item中,經引擎通過項目管道保存到數據庫中。隨后在scrapy框架的下載器中間件組件中,創建ProxyMiddle-ware類和RandomUserAgentMiddleware類,用以設置IP代埋和設置隨機User-Agent,創建getmiRNACount類。用selenium和webdriver來獲取通過ajax返回的統計結果的數據;最后在pipe-line組件中實現數據的持久化。
5結束語
本文在分析TarBase V8網站網頁結構的基礎上,使用py-thon語言和Scrapy框架設計與實現了TarBase V8網站數據爬蟲。針對網頁中各表數據之間無關聯性的問題,設計了分兩步獲取的方法來解決,即先獲取簡表信息,再根據簡表中的數據生成相應的請求URL獲取詳表及綁定表的信息;針對ajax網頁局部渲染更新的問題,使用了selenium加無頭的chromedriver來解決;針對網站反爬蟲機制的問題,使用了隨機User-Agent和IP代理的方式來解決。本爬蟲在網絡狀態良好的情況下,爬取2588條miRNA的數據需要60多小時,共爬取數據約100多萬條。
參考文獻
[1] Huang Y, Shen X J, Zou Q, et al. Biological functions of mi-croRNAs:a review[J]. Journal of Physiology and Biochemistry,2011, 67(1): 129-139.
[2] Karagkouni D. Paraskevopoulou M D, Chatzopoulos S. et al.DIAhrA-TarBase v8:a decade-long collecticm of experimental-ly supported miRNA ~ gene interactions[J]. Nucleic Acids Re-search. 2018, 46(DI): D239-D245.
[3]崔慶才.Python 3網絡爬蟲開發實戰[M].北京:人民郵電出版社,2018.
[4] Kozomara A,Criffiths-Jones S.MiRBase:annotating high confi-dence microRNAs using deep sequencing data[J].Nucleic Ac-ids Research. 2014. 42(D1): D68-D73.
[5]時永坤.基于WehDriver的定向網絡爬蟲設計與實現[J].軟件,2016, 37(9): 94-97.
【通聯編輯:謝媛媛】
收稿日期:2020-05-08
基金項目:湖南省教育廳科研課題(項目編號:17C1377)
作者簡介:蔣輝(1981-),男,湖南衡陽人,講師,碩士,主要研究方向為生物信息學、信息檢索與知識發現、軟件工程;謝云潔(1996一),男,學士。

