蟬擬青霉疏水蛋白PChyd基因表達(dá)模式分析及敲除載體構(gòu)建
王麗芳 彭耀 黨向利 葉良 李雙姣 許慧慧 杜鵑 董育德


摘 ?要:為深入研究昆蟲病原真菌蟬擬青霉疏水蛋白PChyd基因的功能,根據(jù)蟬擬青霉基因組信息克隆疏水蛋白PChyd基因,對(duì)該基因序列進(jìn)行生物信息學(xué)分析,使用qRT-PCR技術(shù)對(duì)其在不同培養(yǎng)條件或階段下的表達(dá)模式進(jìn)行分析,并通過酶切酶連的方法構(gòu)建了該基因的敲除載體。結(jié)果表明:PChyd基因的開放閱讀框序列全長303?bp,編碼100?aa,包含22?aa的信號(hào)肽序列和70?aa疏水蛋白功能區(qū)域。系統(tǒng)發(fā)育分析顯示該基因與粗糙蟲草菌親緣關(guān)系最近。qRT-PCR結(jié)果顯示PChyd基因在PDA培養(yǎng)的菌絲體、誘導(dǎo)的附著胞、誘導(dǎo)的芽生孢子中表達(dá)量顯著高于另外2個(gè)樣品,其中芽生孢子表達(dá)量最高,暗示該基因在蟬擬青霉侵染初期和在昆蟲血腔中定殖階段可能具有重要作用。凝膠電泳結(jié)果表明,成功構(gòu)建了該基因的敲除載體,擴(kuò)增出含有上臂、HPH、下臂的3356?bp左右的片段。本研究為進(jìn)一步探究蟬擬青霉疏水蛋白PChyd基因的致病機(jī)理、生防工程菌的改造奠定了基礎(chǔ)。
關(guān)鍵詞:蟬擬青霉;疏水蛋白;PChyd:表達(dá)模式;敲除載體中圖分類號(hào):Q78;S476.12??????文獻(xiàn)標(biāo)識(shí)碼:A
Analysis of Expression Pattern and Construction of Knockout Vector ofPChydGeneof?Paecilomyces cicadae
WANG Lifang1, PENG Yao2*, DANG Xiangli2, YE Liang2, LI Shuangjiao2, XU Huihui2, DU Juan2,DONG Yude1
1. School of Horticulture, Anhui Agricultural University, Hefei, Anhui 230036, China; 2.?School of Plant Protection, Anhui Agricultural University, Hefei, Anhui 230036, China
Abstract: In order to investigate the function ofPChydofPaecilomyces cicadae, the gene was cloned according to the genomic data ofP. cicadae, and the bioinformatics of the gene were analyzed by the NCBI database and the Expasy online tools. The expression pattern ofPChydwas analyzed under different culture conditions or stages by qRT-PCR. The knockout vector of the gene was constructed by molecular biological methods. The open reading frame ofPChydwas 303 bp encoding 100 aa. Domain analysis revealed that the gene contained a 22 aa signal peptide region and a 70 aa hydrophobin functional region. Phylogenetic analysis showed that the relationship ofP. cicadae PChyd was close toCordyceps confragosa hydrophobin. Expression pattern analysis showed that the expression level ofPChydin mycelium in PDA, appressorium and blastospore were significantly higher than that of the other two samples, and the expression level was the highest in blastospore among five samples, which indicating that this gene had important function in the initial stage of the infection ofP. cicadaeand in the colonization stage of insect blood. The results of gel electrophoresis and sequencing showed that the knockout vector of the gene was successfully constructed. The research would lay a foundation for further exploring of the pathogenic mechanism ofPChydand engineering this entomopathogenic fungusP. cicadae.
Keywords: Paecilomyces cicadae; hydrophobin;PChyd; expression pattern; knockout vector
DOI: 10.3969/j.issn.1000-2561.2020.07.005
隨著害蟲抗藥性的增強(qiáng)、害蟲的再猖獗以及農(nóng)業(yè)投入物對(duì)環(huán)境的污染這一系列問題的出現(xiàn),人們迫切需要尋找以生物為基礎(chǔ)的有害生物控制形式。昆蟲病原真菌作為一種重要的微生物群,廣泛分布于整個(gè)真菌界,已經(jīng)作為害蟲生物防治的主要手段之一并應(yīng)用于農(nóng)業(yè)生態(tài)系統(tǒng)中。當(dāng)前害蟲生物防治中廣泛應(yīng)用的昆蟲病原真菌包括金龜子綠僵菌(Metarhizium anisopliae)、球孢白僵菌(Beauveria bassiana)、玫煙色擬青霉(Paecil omycesfum osoroseus)和蠟蚧輪枝菌(Verticillium lecanii)[1]。蟬擬青霉(Paecilomyces cicadae)是一種在同翅目和鱗翅目昆蟲幼蟲上發(fā)現(xiàn)的昆蟲病原真菌,它能夠侵染竹蟬(Platylomia pieli)、山蟬(Cicada flammata)、小菜蛾(Plutella xylostella)等昆蟲[2-3]。在我國,該菌是研究較早的蟲草菌和生防菌種類之一,目前主要分布于我國華東與西南地區(qū)[4]。早期主要研究了其對(duì)菜粉蝶(Pieris rapae)等鱗翅目農(nóng)業(yè)害蟲的致病性[5]。隨著人們對(duì)蟲草的關(guān)注度逐漸升高,近年來關(guān)于蟲草屬菌種的研究越來越多。研究表明蟬擬青霉對(duì)鱗翅目(Lepidoptera)、半翅目(Hemiptera)、同翅目(Homoptera)等昆蟲都具有較強(qiáng)的致病能力[6-8]。這些研究為后期蟬擬青霉的開發(fā)利用提供了理論基礎(chǔ)。
為提高昆蟲病原真菌的田間穩(wěn)定性、殺蟲效果和殺蟲速度,國內(nèi)外學(xué)者對(duì)病原真菌致病性的分子機(jī)制展開了研究。當(dāng)前白僵菌與綠僵菌致病毒力基因的研究已有大量報(bào)道,但對(duì)蟬擬青霉毒力相關(guān)基因的研究甚少,在一定程度上限制了該菌在害蟲生物防治上的應(yīng)用。在前期的研究中,本課題組對(duì)蟬擬青霉菌株(ZJ1611)基因組進(jìn)行了測(cè)序。根據(jù)已報(bào)道的白僵菌、綠僵菌等昆蟲病原真菌的毒力相關(guān)基因,對(duì)蟬擬青霉基因組中的毒力相關(guān)基因進(jìn)行篩選,從中發(fā)現(xiàn)1個(gè)疏水蛋白(hydrophobin)基因——PChyd。疏水蛋白是一種存在于絲狀真菌中、分子量較小、具有表面活性的分泌蛋白,常分布于分生孢子、菌絲等結(jié)構(gòu)的表面,該蛋白根據(jù)其自身的親水模式、溶解特性、核苷酸序列以及自組裝過程中所形成的結(jié)構(gòu)可分為2種類型(I型和II型)[9-11]。疏水蛋白在真菌中扮演著多種重要的角色。例如,在食用真菌中,Hyd9基因的沉默會(huì)造成金針菇(Flammulina filiformis)菌絲減少,并對(duì)原基形成產(chǎn)生影響[12]。Zhang等[13]研究發(fā)現(xiàn)白靈菇(Pleurotus nebrodensis)疏水蛋白PN1與細(xì)菌抵抗力有關(guān)。在生物防治真菌粉紅螺旋聚孢霉(Clonostachys rosea)中,疏水蛋白對(duì)分生孢子的疏水性、控制分生孢子萌發(fā)以及粉紅螺旋聚孢霉在根內(nèi)定殖都有一定作用[14]。這些研究都表明疏水蛋白在真菌的生長和發(fā)育過程中具有重要的作用。
目前關(guān)于蟬擬青霉疏水蛋白基因的研究尚未見報(bào)道。鑒于此,對(duì)蟬擬青霉基因組中發(fā)現(xiàn)的疏水蛋白PChyd基因進(jìn)行克隆,利用生物信息學(xué)方法對(duì)其序列進(jìn)行分析,對(duì)其在不同培養(yǎng)條件或階段的表達(dá)模式進(jìn)行檢測(cè),并成功構(gòu)建了該基因的敲除載體。為進(jìn)一步探究蟬擬青霉疏水蛋白PChyd基因的致病機(jī)理奠定基礎(chǔ),同時(shí)為該菌生防工程菌的改造以及生防菌的應(yīng)用提供理論依據(jù)。
1 ?材料與方法
1.1 材料
蟬擬青霉(P.cicadae)菌株分離自采集于浙江的野生蟬蟲草子實(shí)體(編號(hào):ZJ1611)。質(zhì)粒pMD19-T和大腸桿菌Escherichia coliDH5α感受態(tài)細(xì)胞購自TaKaRa。Bar-HPH由安徽農(nóng)業(yè)大學(xué)植物保護(hù)學(xué)院郭敏教授饋贈(zèng)。
1.2方法
1.2.1PChyd基因的克隆與鑒定 ?采用Primer 5.0軟件對(duì)預(yù)測(cè)的PChyd基因設(shè)計(jì)克隆引物,引物序列詳見表1,以cDNA為模板對(duì)PChyd基因進(jìn)行PCR擴(kuò)增。以DNA為模板進(jìn)行PCR反應(yīng)來分析基因序列中是否存在非編碼區(qū)域。PCR反應(yīng)根據(jù)2×Taq Plus MasterMix(北京康為)試劑的使用說明在PCR儀(Bio-RAD)中進(jìn)行。產(chǎn)物經(jīng)瓊脂糖凝膠電泳檢測(cè),并使用上海生工膠回收試劑盒對(duì)反應(yīng)產(chǎn)物進(jìn)行膠回收、純化,回收后的產(chǎn)物使用Mighty TA-cloning(TakaRa)試劑盒進(jìn)行TA克隆,克隆好的質(zhì)粒用于大腸桿菌E. coliDH5α的轉(zhuǎn)化,并挑選已成功驗(yàn)證的轉(zhuǎn)化子寄往安徽通用生物公司進(jìn)行測(cè)序,以驗(yàn)證基因組測(cè)序序列的正確性,對(duì)驗(yàn)證正確的基因進(jìn)行后續(xù)生物信息學(xué)及表達(dá)模式分析。
1.2.2PChyd基因的生物信息學(xué)分析 ?將PChyd基因序列于NCBI上利用BLAST進(jìn)行比對(duì)。根據(jù)NCBI提供的InterProScan、dbCAN、CDD
注:下劃線序列為PmeI酶切位點(diǎn)。
Note: The underlined sequences are thePmeI?cleavage site.
databases等在線工具對(duì)該基因結(jié)構(gòu)域進(jìn)行分析。采用ProtParam預(yù)測(cè)蛋白的等電點(diǎn)(pI)、分子量(Mw)和GPI位點(diǎn)。采用SignalP 4.1預(yù)測(cè)該蛋白的理論信號(hào)肽區(qū)域。采用MEGA 6.0軟件構(gòu)建系統(tǒng)發(fā)育樹。
1.2.3??菌株的培養(yǎng) ?根據(jù)PChyd基因可能參與的反應(yīng)階段,選取了2種不同的菌絲培養(yǎng)條件和3個(gè)階段,共5個(gè)樣品(PDA、CM、AP、BS、C)。各種樣品的培養(yǎng)方法如下:將蟬擬青霉接種在PDA固體培養(yǎng)基上于28?℃培養(yǎng)12~15?d,使用含有0.05%吐溫80的無菌水收集蟬擬青霉的分生孢子,配置孢子懸浮液,懸浮液濃度為1×108個(gè)/mL。蟬擬青霉分生孢子接種到CM[15](硝酸鹽溶液50?mL/L,微量元素溶液1?mL/L,維生素溶液1?mL/L,葡萄糖10?g/L,蛋白胨2?g/L,酵母提取物1?g/L,絡(luò)蛋白氨基酸1?g/L,瓊脂粉15?g/L)、PDA液體培養(yǎng)基中,在28?℃條件下180?r/min的搖床中培養(yǎng)3 d后收集菌絲體。使用Junges等[16]報(bào)道的實(shí)驗(yàn)方法對(duì)分生孢子(C)、芽生孢子(BS)(誘導(dǎo)培養(yǎng)基配方:玉米漿固體30?g/L,葡萄糖40?g/L,酵母提取物30?g/L;培養(yǎng)條件:28?℃?180?r/min培養(yǎng)64?h)和附著胞(AP)(配置含有0.004%酵母提取物和0.05%吐溫80的無菌溶液,使用該溶液將1×108個(gè)/mL的孢子懸浮液稀釋至1×106個(gè)/mL。稀釋后的孢子懸浮液均勻涂抹于蓋玻片上,并于28?℃培養(yǎng)16?h后,使用上述配置好的溶液沖洗、離心并收集樣品)進(jìn)行收集。所有試劑均購自生工生物工程(上海)股份有限公司。
1.2.4??基因組DNA、總RNA提取及cDNA合成 ?利用Wizard?Genomic DNA提取試劑盒(Pro mega)提取各樣品的基因組DNA,提取后的基因組DNA用1%的瓊脂糖凝膠進(jìn)行電泳檢測(cè)。根據(jù)Rio等[17]的方法對(duì)所有樣品的總RNA進(jìn)行提取,并使用RNase-free DNase I(TaKaRa)去除總RNA中的基因組DNA污染。通過瓊脂糖凝膠電泳和NanoDrop 2000分光光度計(jì)(Thermo Scientific,DE)對(duì)提取的RNA質(zhì)量和濃度進(jìn)行檢測(cè)。cDNA合成根據(jù)PrimeScript?II 1st Strand cDNA Synthesis試劑盒(TaKaRa)的使用說明進(jìn)行。
1.2.5??qRT-PCR??使用qRT-PCR對(duì)樣品的PChyd基因的相對(duì)表達(dá)水平進(jìn)行檢測(cè)。每個(gè)基因轉(zhuǎn)錄本的相對(duì)熒光定量使用SYBR?Premix Ex TaqTM II(TaKaRa)試劑進(jìn)行,并在Bio-Rad CFX96(Bio-?Rad,Hercules,CA,USA)中進(jìn)行反應(yīng)。熒光定量反應(yīng)使用96孔板,每個(gè)孔包含20?μL反應(yīng)液:10?μL SYBR?Premix Ex TaqTM II、每條引物0.4?μL(0.2?μmol/L)、1?μL(100 ng)cDNA模板、8.2?μL不含核酸酶的無菌水。反應(yīng)條件如下:95?℃?30?s 1個(gè)循環(huán),95?℃?5?s和60?℃?30?s反應(yīng)40個(gè)循環(huán)。蟬擬青霉β-actin基因作為內(nèi)參基因,相對(duì)表達(dá)水平使用2-ΔΔCT方法進(jìn)行分析[18]。所有的實(shí)驗(yàn)進(jìn)行3次重復(fù)。
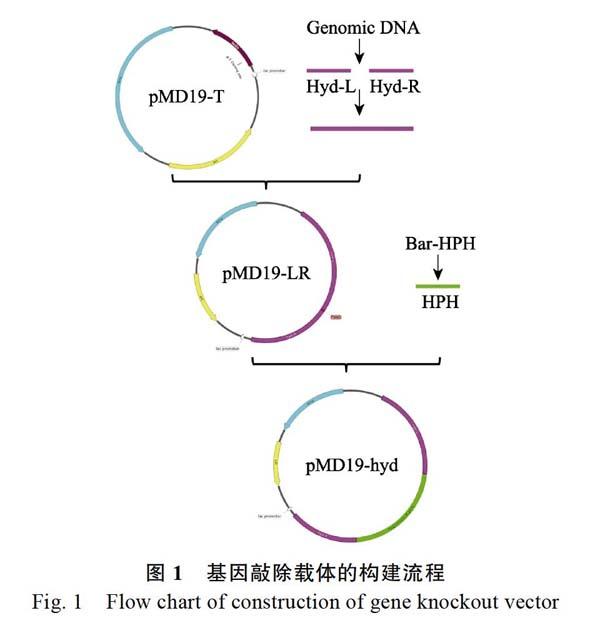
1.2.6??敲除載體構(gòu)建??分別設(shè)計(jì)引物擴(kuò)增敲除基因的上下臂片段及抗性基因(表1),按照?qǐng)D1所示進(jìn)行該基因敲除載體的構(gòu)建,構(gòu)建好的載體送往安徽通用生物公司進(jìn)行測(cè)序驗(yàn)證。具體實(shí)驗(yàn)方法如下:首先進(jìn)行Overlap PCR反應(yīng),構(gòu)建含有PChyd左、右臂的片段(引物提前設(shè)計(jì)同源區(qū)域及PmeI酶切位點(diǎn)),回收PCR產(chǎn)物用于TA克隆,TA克隆后的質(zhì)粒經(jīng)PmeI酶切處理后(酶切后的產(chǎn)物需去磷酸化處理)與HPH進(jìn)行T4連接,挑取連接轉(zhuǎn)化后的大腸桿菌菌落進(jìn)行PCR驗(yàn)證。
1.3數(shù)據(jù)處理
采用GraphPad Prism 7.0和Data Processing System(DPS)軟件進(jìn)行數(shù)據(jù)處理和繪圖。數(shù)據(jù)均為平均值±標(biāo)準(zhǔn)誤,采用SPSS軟件進(jìn)行差異顯著性分析。
2 結(jié)果與分析
2.1 ?PChyd基因克隆與生物信息學(xué)分析
以蟬擬青霉的基因組DNA和cDNA為模板,利用設(shè)計(jì)的特異性引物PC29和PC30進(jìn)行Pchyd基因的擴(kuò)增。凝膠電泳結(jié)果顯示,從DNA和cDNA
模板中分別擴(kuò)增出大約400?bp和300?bp片段(圖2),兩者之間有100 bp左右的差異,暗示該基因的序列中存在非編碼區(qū)域。將2個(gè)PCR產(chǎn)物進(jìn)行測(cè)序,測(cè)序結(jié)果如圖3所示。蟬擬青霉PChyd基因DNA序列長度為417?bp,比mRNA序列大114?bp,該基因中存在2個(gè)非編碼區(qū)域(圖3)。將測(cè)序序列利用NCBI進(jìn)行比對(duì),發(fā)現(xiàn)該基因編碼的蛋白屬于疏水蛋白hydrophobin-2超家族,并且產(chǎn)物測(cè)序結(jié)果與基因組序列相一致,可以確認(rèn)該基因?yàn)橄s擬青霉疏水蛋白基因PChyd。
M:1000 bp DNA Marker;—:為不添加模板的擴(kuò)增,作為陰性對(duì)照;DNA:以蟬擬青霉基因組DNA為模板擴(kuò)增;cDNA:以蟬擬青霉cDNA為模板擴(kuò)增。
M: 1000?bp DNA Marker; —: Negative control (without DNA template); DNA: Genomic DNA as template; cDNA: cDNA as template.
圖2蟬擬青霉PChyd基因擴(kuò)增電泳檢測(cè)
綠色突出部分為起始密碼子,紅色突出部分為終止密碼子。
The green highlight is the start codon and the red highlight is the stop codon.
2.2 ?PChyd基因序列生物信息學(xué)分析
PChyd的開放閱讀框(ORF)序列全長303 bp,編碼蛋白氨基酸序列全長100?aa(圖3、圖4),分子量為10.58 kDa,pI為5.68。結(jié)構(gòu)域分析顯示該蛋白包含信號(hào)肽區(qū)域和疏水蛋白功能區(qū)域。信號(hào)肽區(qū)域包含22?aa,疏水蛋白功能區(qū)域包含70?aa。利用MEGA 6.0軟件對(duì)PChyd基因構(gòu)建系統(tǒng)發(fā)育樹(圖5),結(jié)果顯示真菌疏水蛋白hyd基因可分為小叢殼目(Glomerellales)和肉座菌目(Hypocreales)2個(gè)組,而肉座菌目(Hypocreales)
組疏水蛋白hyd基因又可分為3個(gè)亞組,分別包括肉座菌科(Hypocreaceae)、生赤殼科(Bionectri aceae)和蟲草菌科(Cordycipitaceae)。蟬擬青霉PChyd與其他蟲草菌科的真菌聚在一個(gè)分支上,與粗糙蟲草菌(Cordyceps confragosa)的親緣關(guān)系最近。將hyd基因系統(tǒng)發(fā)育樹中的蟲草菌科序列進(jìn)行氨基酸序列比對(duì)(圖6),結(jié)果發(fā)現(xiàn)蟲草菌科的hyd基因保守度較高,完全保守的氨基酸有56個(gè)。
采用鄰接法并進(jìn)行1000次重復(fù)構(gòu)建系統(tǒng)發(fā)育樹;星號(hào)標(biāo)記的為蟬擬青霉疏水蛋白。
Phylogenetic tree was constructed using neighbor-joining method, and percent bootstrap support from 1000 iterations;P. cicadaehydrophobin is?marked as star.
2.3 ?PChyd表達(dá)模式分析
利用qRT-PCR方法對(duì)PChyd基因在不同培養(yǎng)條件(PDA和CM)或不同發(fā)育階段(AP、BS和C)的表達(dá)模式進(jìn)行檢測(cè)(圖7)。結(jié)果表明,PChyd基因在PDA、AP、BS中相對(duì)表達(dá)量分別為1.029±0.281、0.855±0.221、1.284±0.373。統(tǒng)計(jì)分析顯示,三者之間差異不顯著,但均顯著高于CM(0.056±0.005)和C(0.161±0.045)的相對(duì)表達(dá)量。其中在BS狀態(tài)下,PChyd基因表達(dá)量最高,表明該基因可能在蟬擬青霉附著胞(AP)和芽生孢子(BS)階段具有重要作用。
2.4 ?PChyd敲除載體的構(gòu)建
以蟬擬青霉基因組DNA為模板,使用引物PC31/PC32和PC33/PC34分別對(duì)PChyd基因的左、右臂片段進(jìn)行擴(kuò)增,結(jié)果如圖8所示。通過PCR獲得了長度為1182?bp的左臂和長度為874?bp的右臂。經(jīng)Overlop PCR后將左、右臂融
PDA:在PDA中生長的菌絲體;CM:在CM培養(yǎng)基中生長的菌絲體;AP:誘導(dǎo)的附著胞;BS:誘導(dǎo)的芽生孢子;C:分生孢子。不同小寫字母表示不同樣品之間差異顯著(P<0.05)。
PDA: Mycelium grown in PDA; CM: Mycelium grown in complete medium; AP: Induced appressorium; BS: Induced blastospore;C: Conidia. Different lowercase letters indicatesignificant differences at the 0.05 level.
合(片段大小為2008?bp),融合產(chǎn)物進(jìn)行TA克隆構(gòu)建pMD19-LR,TA克隆后的質(zhì)粒經(jīng)PmeI酶切處理和去磷酸化處理后,與HPH(1348?bp)進(jìn)行T4連接,構(gòu)建pMD19-hyd(載體理論大小為3356?bp),使用引物PC31/PC34進(jìn)行菌落PCR驗(yàn)證,結(jié)果如圖9所示。從左往右的第3、4、9泳道均能擴(kuò)增出3500?bp左右的片段,該片段可能為插入HPH片段的敲除載體pMD19-hyd。經(jīng)測(cè)序驗(yàn)證,該片段大小為3356?bp,與設(shè)計(jì)載體大小一致,包括1158?bp左臂、1348?bpHPH和850?bp右臂。測(cè)序結(jié)果確認(rèn)該片段測(cè)定序列與設(shè)計(jì)序列一致,證明已成功構(gòu)建PChyd的敲除載體,可用于后續(xù)轉(zhuǎn)化實(shí)驗(yàn)。
3 ?討論
在農(nóng)業(yè)上,昆蟲病原真菌作為一種重要的微生物資源,相較于化學(xué)農(nóng)藥更為安全環(huán)保,已廣泛應(yīng)用于農(nóng)林蟲害的生物防治[19]。了解昆蟲病原真菌毒力相關(guān)基因及其致病機(jī)理,對(duì)于構(gòu)建基因工程生防菌、提高防治效果和挖掘應(yīng)用潛力極其重要。當(dāng)前,白僵菌與綠僵菌毒力基因的研究已有大量報(bào)道,但有關(guān)蟬擬青霉致病機(jī)理、毒力相關(guān)基因研究尚未見報(bào)道。在前期研究中,本課題組對(duì)蟬擬青霉菌株(ZJ1611)基因組進(jìn)行了測(cè)序。據(jù)已報(bào)道的白僵菌與綠僵菌的毒力基因特征,對(duì)蟬擬青霉毒力相關(guān)基因進(jìn)行了篩選,共篩選出41類蛋白,共464個(gè)毒力相關(guān)基因。昆蟲病原真菌的侵染過程可以概述為4個(gè)階段:粘附階段、萌發(fā)和穿透階段、血腔中的繁育階段、菌絲穿出蟲體并產(chǎn)孢階段[20]。根據(jù)毒力相關(guān)基因可能參與的昆蟲病原真菌侵染昆蟲的過程,前期篩選出3種可能參與粘附階段的基因,分別是疏水蛋白(hyd rophobin)基因、細(xì)胞壁蛋白(cell wall protein)基因和MAP激酶(mitogen-activated protein kinase)基因。本研究選取1個(gè)可能參與侵染初期的毒力相關(guān)基因——疏水蛋白基因進(jìn)行深入研究。以蟬擬青霉為供試材料,對(duì)PChyd基因進(jìn)行克隆,并對(duì)該基因的核苷酸序列和氨基酸序列進(jìn)行生物信息學(xué)分析。結(jié)果發(fā)現(xiàn),該基因編碼的蛋白屬于hydrophobin-2超家族,并且多重序列比對(duì)發(fā)現(xiàn)該基因與其他幾種蟲草菌科菌株的疏水蛋白氨基酸序列的保守度較高,功能結(jié)構(gòu)域分析也發(fā)現(xiàn)該基因具有疏水蛋白典型的信號(hào)肽和疏水蛋白功能區(qū)域[21-22]。這些結(jié)果都表明該基因?qū)儆谑杷鞍谆颉?/p>
疏水蛋白是一類由高等絲狀真菌在某一特定時(shí)期分泌產(chǎn)生的、具有特殊理化性質(zhì)的小分子量(<20?kDa)蛋白質(zhì)[23]。在昆蟲病原真菌中,疏水蛋白參與了其對(duì)昆蟲的侵染過程,是昆蟲病原真菌重要的毒力相關(guān)基因[24]。早期研究已在多個(gè)昆蟲病原真菌中發(fā)現(xiàn)了疏水蛋白基因。例如,在金龜子綠僵菌(Metarhizium anisopliae)、粉擬青霉(Paecilomyces farinosus)、球孢白僵菌(Beau veria bassiana)中,分別發(fā)現(xiàn)了疏水蛋白基因ssgA、pfah1、mHyd2[25-27]。但這些報(bào)道未對(duì)疏水蛋白基因在昆蟲病原真菌致病機(jī)理方面進(jìn)行闡述。隨著真菌基因敲除技術(shù)的逐步成熟,疏水蛋白在昆蟲侵染過程中的功能有了進(jìn)一步研究。例如,通過對(duì)球孢白僵菌的疏水蛋白基因hyd1和hyd2敲除發(fā)現(xiàn),突變菌株Δhyd1孢子疏水性降低,表面的碳水化合物表位和β-1,3-葡萄糖分布產(chǎn)生變化,毒力降低,但孢子粘附性未受影響。突變菌株Δhyd2孢子表面疏水性和粘附性均降低,但毒力不受影響。菌株Δhyd1、Δhyd2孢子的疏水性、粘附性和毒力均降低[28]。在對(duì)棕色綠僵菌(Metar hizium brunneum)的3個(gè)疏水蛋白基因敲除后發(fā)現(xiàn),3個(gè)突變菌株的產(chǎn)孢能力下降,且分生孢子和芽生孢子侵染甜菜夜蛾(Spodoptera exigua)幼蟲的毒力下降。除突變體Δhyd3外,其余2個(gè)突變體疏水性不變[29]。這些研究都表明,疏水蛋白基因在昆蟲病原真菌侵染昆蟲初期(第1階段)和芽生孢子(第2階段)中扮演著重要的角色。為分析蟬擬青霉疏水蛋白基因PChyd可能的功能,本研究對(duì)其在不同培養(yǎng)條件(PDA、CM)或不同發(fā)育階段(AP、BS、C)的表達(dá)情況進(jìn)行了檢測(cè),結(jié)果發(fā)現(xiàn)PChyd基因在AP(誘導(dǎo)的附著胞,侵染初期的侵染結(jié)構(gòu))、BS(誘導(dǎo)的芽生孢子,參與了昆蟲血腔中定殖過程)中都顯示了較高的表達(dá)量,與早期研究結(jié)果較一致。黃姍等[30]研究發(fā)現(xiàn),蟲生真菌萊氏野村菌hyd基因表達(dá)量隨著分生孢子的產(chǎn)生而升高,到產(chǎn)孢量達(dá)到最大的第8天時(shí),表達(dá)量最高,之后隨著產(chǎn)孢量的下降,基因的表達(dá)量降低,推測(cè)該基因在萊氏野村菌分生孢子的形成過程中起著重要作用。而PChyd基因在C(分生孢子)中相對(duì)表達(dá)量較低,推測(cè)這種低水平的表達(dá)可能與靜息細(xì)胞的代謝活性降低有關(guān)。Junges等[16]在金龜子綠僵菌(M. anisopliae)中也曾發(fā)現(xiàn)這種現(xiàn)象。
綜上所述,在前期研究的基礎(chǔ)上,本研究對(duì)蟬擬青霉疏水蛋白基因PChyd進(jìn)行了克隆,對(duì)其序列進(jìn)行了生物信息學(xué)分析,并分析了其在不同培養(yǎng)條件或不同發(fā)育階段的表達(dá)模式,推測(cè)其可能參與病原真菌的生長、粘附、定值侵染過程,同時(shí)成功構(gòu)建了該基因的敲除載體,為進(jìn)一步研究該基因的致病機(jī)理奠定了基礎(chǔ),也為該菌的生防菌改造和應(yīng)用提供了理論依據(jù)。
參考文獻(xiàn)
- Meng X, Hu J, Ouyang G. The isolation and identification of pathogenic fungi from Tessaratoma papillosa?Drury (Hemi ptera: Tessaratomidae)[J]. Peer J, 2017, 5(10): e3888.
- Zeng W, Yu H, Ge F, et al. Distribution of nucleosides in populations of Cordyceps cicadae[J]. Molecules, 2014, 19(5): 6123-6141.
- Fang M, Chai Y, Chen G, et al. N6-(2-hydroxyethyl)-?adenosine exhibits insecticidal activity against Plutella xylostella?via adenosine receptors[J]. PloS One, 2016, 11(9): e0162 859.
- 萬永紅, 蔡開明, 韋??曙. 蟬花的本草考證與生物學(xué)研究[J]. 江蘇林業(yè)科技, 2013, 40(3): 50-53.
- 陳祝安, 黃基榮, 許益?zhèn)ィ?等. 蟬擬青霉防治菜粉蝶幼蟲試驗(yàn)[J]. 生物防治通報(bào), 1990(3): 131-133.
- 譚艾娟, 歐??翔, 劉愛英. 蟬擬青霉對(duì)蠶豆蚜蟲致病性的初步研究[J]. 天津農(nóng)業(yè)科學(xué), 2009, 15(3): 50-52.
- 李??忠, 金道超, 鄒??曉, 等. 蟬擬青霉對(duì)蚜蟲的致病性研究[J]. 安徽農(nóng)業(yè)科學(xué), 2007(25): 7882-7883.
- 鄒??曉, 劉愛英, 梁宗琦. 蟲生真菌蟬擬青霉誘變株對(duì)溫室白粉虱的致死因素分析[J]. 植物保護(hù), 2007, 33(2): 73-76.
[9]?Kershaw M J, Talbot N J. Hydrophobins and repellents: proteins with fundamental roles in fungal morphogenesis[J]. Fungal Genetics and Biology, 1998, 23(1): 18-33.
[10]?Berger B W, Sallada N D. Hydrophobins: multifunctional biosurfactants for interface engineering[J]. Journal of Biological Engineering, 2019, 13: 10.
[11]?Li W?F, Gong Y?B, Xu H?J,et al. Identification properties of a recombinant class I hydrophobin rHGFI[J]. International Journal of Biological Macromolecules, 2015, 72: 658-663.
[12]?Tao Y?X, Chen R?L, Yan J?J,et al. A hydrophobin gene,Hyd9, plays an important role in the formation of aerial hyphae and primordia inFlammulina filiformis[J]. Gene, 2019, 706: 84-90.
[13]?Zhang R Y, Hu D D, Gu J G,et al. Purification of a novel hydrophobin PN1 involved in antibacterial activity from an edible mushroomPleurotus nebrodensis[J]. European Journal of Plant Pathology, 2015, 143(4): 823-831.
[14]?Dubey M K, Jensen D, Karlsson M. Hydrophobins are required for conidial hydrophobicity and plant root colonization in the fungal biocontrol agentClonostachys rosea[J]. BMC Microbiology, 2014, 14(1): 18.
[15]?Duan Z?B, Chen Y?X, Huang W,et al. Linkage of autophagy to fungal development, lipid storage and virulence inMetar hizium robertsii[J]. Autophagy, 2013, 9(4): 538-549.
[16]?Junges ?, Boldo J T, Souza B K,et al. Genomic analyses and transcriptional profiles of the glycoside hydrolase family 18 genes of the entomopathogenic fungusMetarhizium anisopliae[J]. PloS One, 2014, 9(9): e107864.
[17]?Rio D C, Ares M J, Hannon G J,et al. Purification of RNA using TRIzol (TRI reagent)[J]. Cold Spring Harbor Protocols, 2010(6): 5439.
[18]?Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCTmethod[J]. Methods, 2001, 25(4): 402-408.
[19]?Hajek A E, Delalibera I. Fungal pathogens as classical biological control agents against arthropods[J]. BioControl, 2010, 55(1): 147-158.
[20]?Butt T M, Coates C J, Dubovskiy I M,et al. Entomopathogenic fungi: new insights into host-pathogen interactions[J]. Advances in Genetics, 2016, 94: 307-364.
[21]?He R?L, Li C, Feng J,et al. A class II hydrophobin gene,Trhfb3, participates in fungal asexual development ofTrichoderma reesei[J]. FEMS Microbiology Letters, 2017, 364(8): fnw297.
[22]?Gandier J A, Langelaan D N, Won A,et al. Characterization of a Basidiomycota hydrophobin reveals the structural basis for a high-similarity Class I subdivision[J]. Scientific Reports, 2017, 7: 45863.
[23]?Berger B W, Sallada N D. Hydrophobins: multifunctional biosurfactants for interface engineering[J]. Journal of Biological Engineering, 2019, 13: 10.
[24]?Valero-Jimenez C A, Wiegers H, Zwaan B J,et al. Genes involved in virulence of the entomopathogenic fungusBeau veria bassiana[J]. Journal of Invertebrate Pathology, 2016, 133: 41-49.
[25]?Bidochka M J, Koning J D, Leger R J S. Analysis of a genomic clone of hydrophobin (ssgA) from the entomopathogenic fungusMetarhizium anisopliae[J]. Mycological Research, 2001, 105(3): 360-364.
[26]?Lunkenbein S, Takenberg M, Nimtz M,et al. Characterization of a hydrophobin of the ascomycetePaecilomyces farinosus[J]. Journal of Basic Microbiology, 2011, 51(4): 404-414.
[27]?Kirkland B H, Keyhani N O. Expression and purification of a functionally active class I fungal hydrophobin from the entomopathogenic fungusBeauveria bassianainE. coli[J]. Journal of Industrial Microbiology and Biotechnology, 2011, 38(2): 327-335.
[28]?Zhang S?Z, Xia Y X, Kim B,et al. Two hydrophobins are involved in fungal spore coat rodlet layer assembly and each play distinct roles in surface interactions, development and pathogenesis in the entomopathogenic fungus,Beauveria bassiana[J]. Molecular Microbiology, 2011, 80(3): 811-826.
[29]?Sevim A, Donzelli B G, Wu D,et al. Hydrophobin genes of the entomopathogenic fungus,Metarhizium brunneum, are differentially expressed and corresponding mutants are decreased in virulence[J]. Current Genetics, 2012, 58(2): 79-92.
[30]?黃??姍, 王中康, 陳??環(huán), 等. 萊氏野村菌疏水蛋白基因Nrhyd的克隆及其表達(dá)特征分析[J]. 菌物學(xué)報(bào), 2012, 31(3): 350-358.

