基于SRAP 標記的海雀稗遺傳多樣性及群體結構分析
洪佳琦,孫宗玖, ,楊志民
(1. 新疆農業(yè)大學草業(yè)與環(huán)境科學學院,新疆 烏魯木齊 830052;2. 西部干旱荒漠區(qū)草地資源與生態(tài)重點實驗室,新疆 烏魯木齊 830052;3. 南京農業(yè)大學草業(yè)學院,江蘇 南京 210095)
海雀稗(Paspalum vaginatum),隸屬于禾本科(Gramineae)雀稗屬,廣泛分布于南北半球的沿海地區(qū),在美國南部、非洲、澳大利亞及太平洋沿岸國家最為豐富[1],在中國主要分布于臺灣、云南、廣東和海南島等地[2-3]。海雀稗是一種極具開發(fā)價值的鹽生種質資源,其耐鹽性優(yōu)于其他暖季型草種;并且可在大部分植物生長受到抑制的惡劣環(huán)境中正常生長,具有較強的抗逆性和適應性,對土壤有較強的固著力,可防止水土流失[4-5],適宜于土壤改良、灘涂綠化等環(huán)境修復[6-8],故海雀稗是濱海灘涂發(fā)展草牧業(yè)和改良鹽堿地的優(yōu)良草種。國外對海雀稗的育種非常重視,美國的喬治亞州立大學是全球培育海雀稗新品種最多的單位[9]。我國雖有海雀稗分布,但野生種質資源收集不足40 份[10-11],迫切需要開發(fā)或引進種質,滿足育種需求。
目前,我國對于海雀稗的研究主要集中在國外種質的引種栽培、抗性評價及育種方面[12-13]。在遺傳多樣性方面,解新明等[4]對廣東3 個居群的海雀稗進行RAPD 分析,發(fā)現居群間基因組多態(tài)性比率高達64.77%,認為我國的海雀稗存在豐富的遺傳變異;劉燕等[14]對國內野生與國外引進的42 份海雀稗種質進行SRAP 分析,發(fā)現國內與國外種質間遺傳相似性很低。雖然國內針對海雀稗種質引種及開發(fā)已經開展了部分工作,但總體還較少,特別是對育種而言,缺乏親本選擇可用的遺傳信息基礎。因此本研究以國內外較廣范圍內收集到的49 份海雀稗種質為對象,利用SRAP 標記對其進行遺傳多樣性分析,明確不同種質間的遺傳差異程度,為海雀稗育種提供基礎遺傳信息,也為海雀稗種質核心種質庫的建立提供科學依據。
1 材料與方法
1.1 材料
本研究所用49 份海雀稗材料由南京農業(yè)大學草業(yè)學院提供,其中43 份為2016 年從美國引進(源自12 個國家),5 份為國內收集的海雀稗育成品種,1 份為自主選育新材料。所有供試材料種植于南京農業(yè)大學白馬教學基地育草圃,株距1.5 m,行距2.0 m,并保持完全自然生長的狀態(tài),只定期進行人工除草,不施肥,不修剪,不灌溉,不防治病蟲害,具體材料信息如表1 所列。
1.2 研究方法
1.2.1 樣品采集
2017 年5 月,于南京農業(yè)大學白馬教學基地育草圃分別剪取49 份海雀稗的幼嫩葉片1 g,置于2.0 mL EP 管中,用液氮速凍后置于便攜式車載冰箱帶回實驗室,置于–80 ℃冰箱保存,每份材料4 個樣,均為單株取樣。
1.2.2 DNA 提取、SRAP 反應體系及PCR 擴增產物檢測
(1) DNA 提取:采用改良的CTAB 法[15]對供試材料進行基因組DNA 提取。2017 年5 月采集每份材料4 個重復幼嫩葉片,將幼嫩葉均勻混合提取DNA。0.8%瓊脂糖凝膠電泳檢測DNA 質量,酶標儀檢測DNA 原液濃度和純度,工作液濃度稀釋到20 ng·μL?1,?20 ℃保存?zhèn)溆谩?/p>
(2) SRAP-PCR 反應體系:采用20 μL 體系,包括2 ×PCR mix 10 μL,引物(10 μmol·L?1) 1 μL,DNA(20 ng·μL?1)2 μL,ddH2O 6 μL。
(3) SRAP-PCR 反應程序:94 ℃預變性4 min;94 ℃變性30 s,37 ℃退火45 s,72 ℃延伸45 s (運行5 個循環(huán));94 ℃變性30 s,56 ℃退火45 s,72 ℃延伸2 min(運行30 個循環(huán)) 72 ℃延伸7 min,最后4 ℃保存。
(4) PCR 產物檢測:采用8%非變性聚丙烯酰胺凝膠電泳,上樣量為2.5 μL,電泳上槽緩沖液為1 ×TBE,100 V 電壓下電泳2 h,使用10%硝酸銀快速銀染顯色。
1.3 數據分析與處理
運用Gel-pro 軟件結合人工方法讀帶,進行條帶統計,建立原始“1、0”原始矩陣,“1”表示存在條帶,“0”表示同一位置無條帶或不易分辨的弱帶。根據試驗統計的“1、0”原始矩陣,運用PopGenVer l.3.2 計算多態(tài)性位點的百分率(PP)、有效等位基因數(ne*)、Nei 遺 傳 距 離(h*)、Shannon 信 息 指 數(I*)。運 用NTSYS-pc 軟件計算供試材料的Jaccard 遺傳相似系數,采用UPGMA 進行聚類分析,繪制系統發(fā)育樹狀圖、PCA 散點分布圖。使用Structure 2.3.4 軟件對49 份海雀稗材料進行群體遺傳結構分析,選取群體遺傳結構的最優(yōu)群體數目。
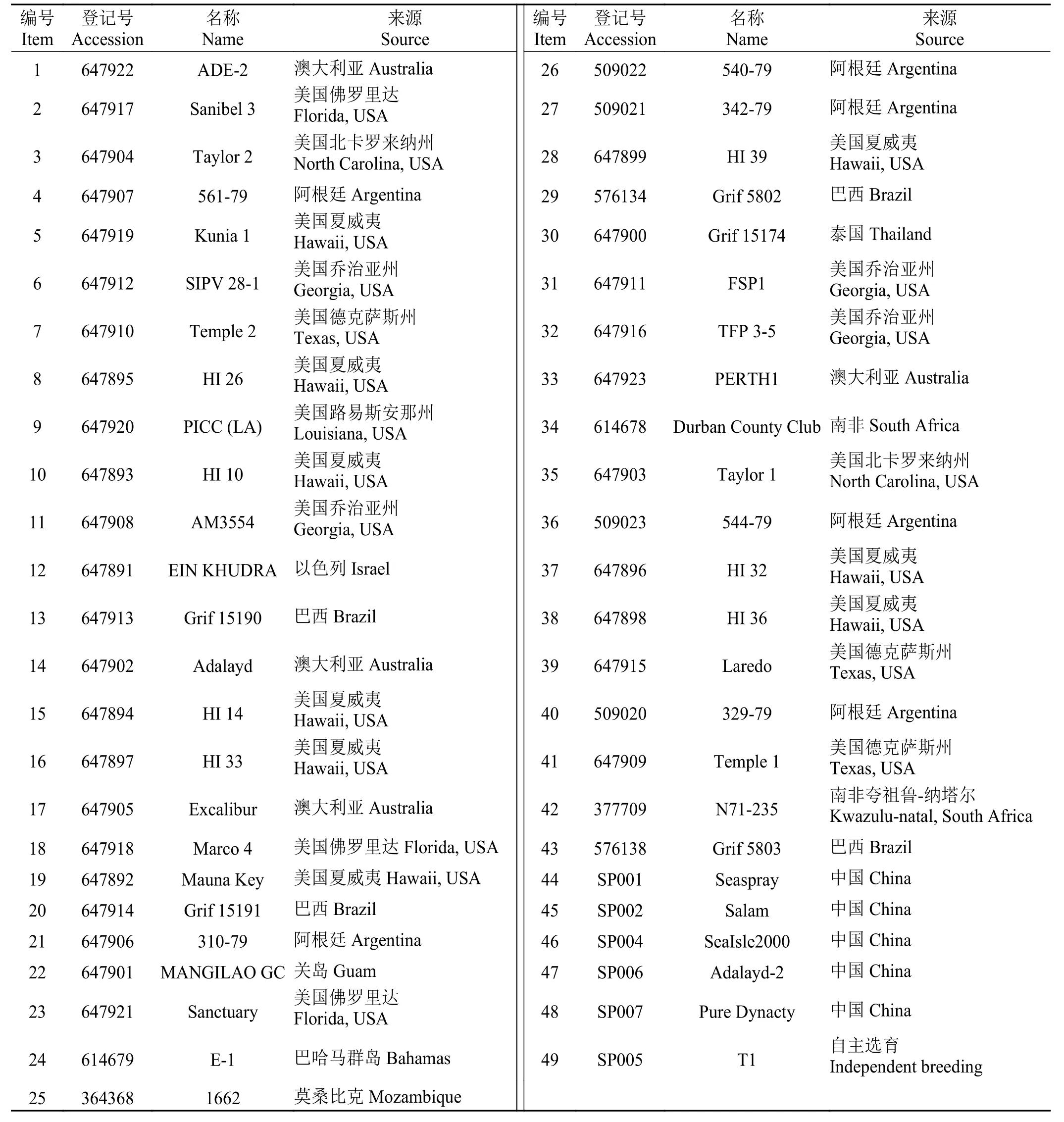
表1 材料編號及來源Table 1 Materials and sources
2 結果與分析
2.1 SRAP 引物多態(tài)分析
運用100 對SRAP 引物(表2)對49 份海雀稗種質資源的基因組DNA 擴增,20 對引物可擴增出條帶清晰且重復性好的條帶,80 對SRAP 不能擴出條帶或擴增出不一致的條帶。利用篩選出的20 對引物組合對49 份海雀稗種質進行PCR 擴增(圖1、表3),共獲得169 個條帶,產物大小介于100~500 bp,各引物擴增條帶為3~12 條,每條引物的平均擴增條帶為8.5 條。在169 條DNA 擴增片段中,有118 條多態(tài)性條帶,每條引物平均擴增多態(tài)性條帶數為5.9,多態(tài)性位點百分率為69.80%。
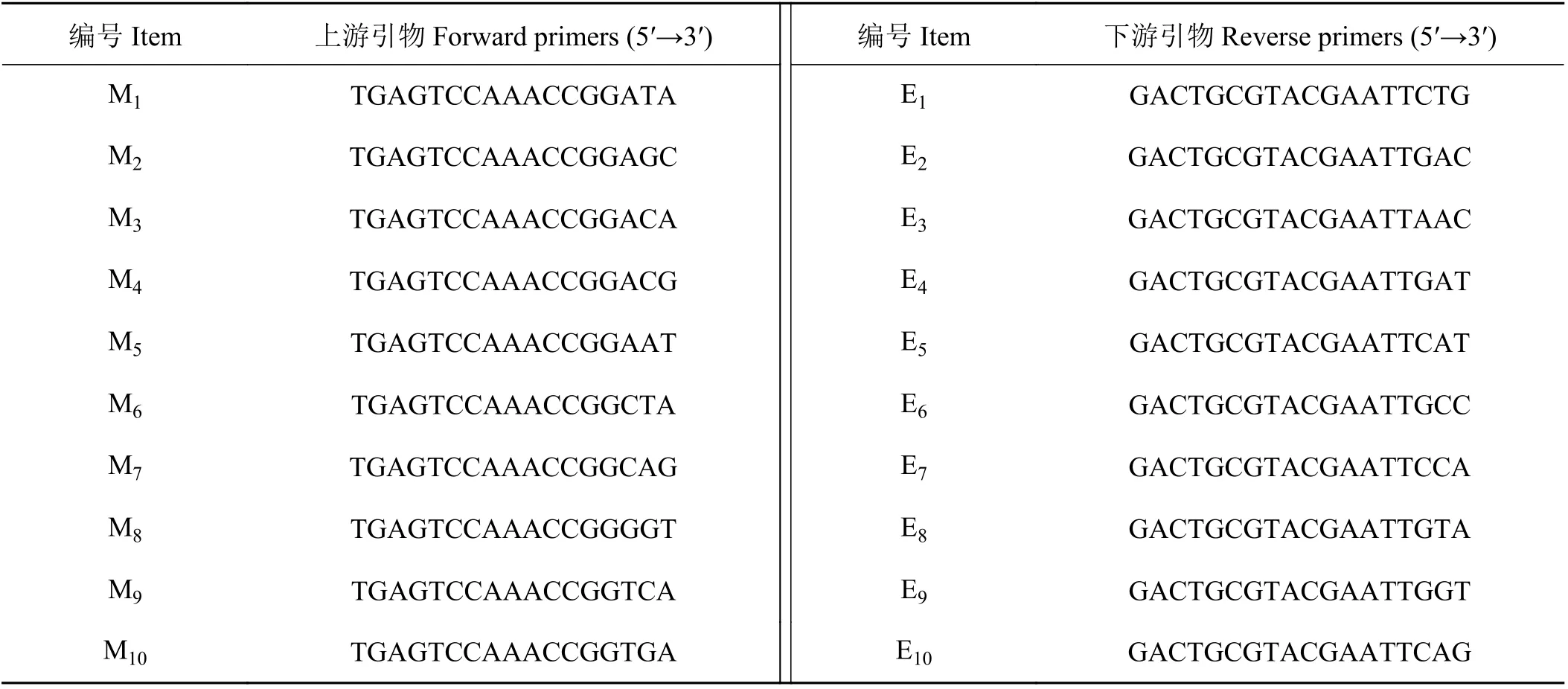
表2 SRAP 引物序列Table 2 Primer sequences used in SRAP analysis

圖1 SRAP 引物篩選聚丙烯酰胺凝膠電泳Figure 1 SRAP primer screening using polyacrylamide gel electrophoresis
供試種質有效等位基因數為1.59~1.97,平均1.83;Nei 遺傳距離為0.36~0.49,平均0.45;Shannon信息指數的范圍在0.55~0.69,平均0.64 (表3),說明供試海雀稗種質資源間存在較豐富的遺傳變異。
2.2 聚類分析
利用NTSYS-pc 計算的Jaccard 遺傳相似系數,49份海雀稗種質資源的遺傳相似系數介于0.52~0.88,平均值為0.67,說明海雀稗種質間存在一定的遺傳差異。其中來自巴西的647913 和來自美國夏威夷的647897 相似系數最大(0.88),而來自美國喬治亞州的647908 和美國夏威夷647896 兩份材料遺傳相似系數最小(0.53),表明它們存在較大遺傳差異。
UPGMA 聚類分析可知(圖2),在遺傳相似系數為0.69 時,可將供試海雀稗材料分為2 大類(Ⅰ、Ⅱ),在遺傳相似系數為0.71 時將材料分為四類(A、B、C、D),其中A 類包含4 份材料,B 類包含12 份材料,C 類包含20 份材料,D 類包含13 份材料。
2.3 主成分分析
材料間的親緣關系可以通過主成分分析(PCA)直觀呈現,種質間位置越近表明它們的相似性越高,越遠則表明種質間的遺傳差異越大。根據統計“1、0”矩陣,用NTsys 軟件進行主成分分析,第1、第2 和第3主成分的貢獻率分別為16.63%、9.06%和5.70%,累計貢獻率31.39%。從圖3 可知,A 類與B 類、C 類海雀稗材料間有交疊,表明他們親緣關系比較近;B 類和D 類有交疊,說明它們存在一定遺傳相似性。
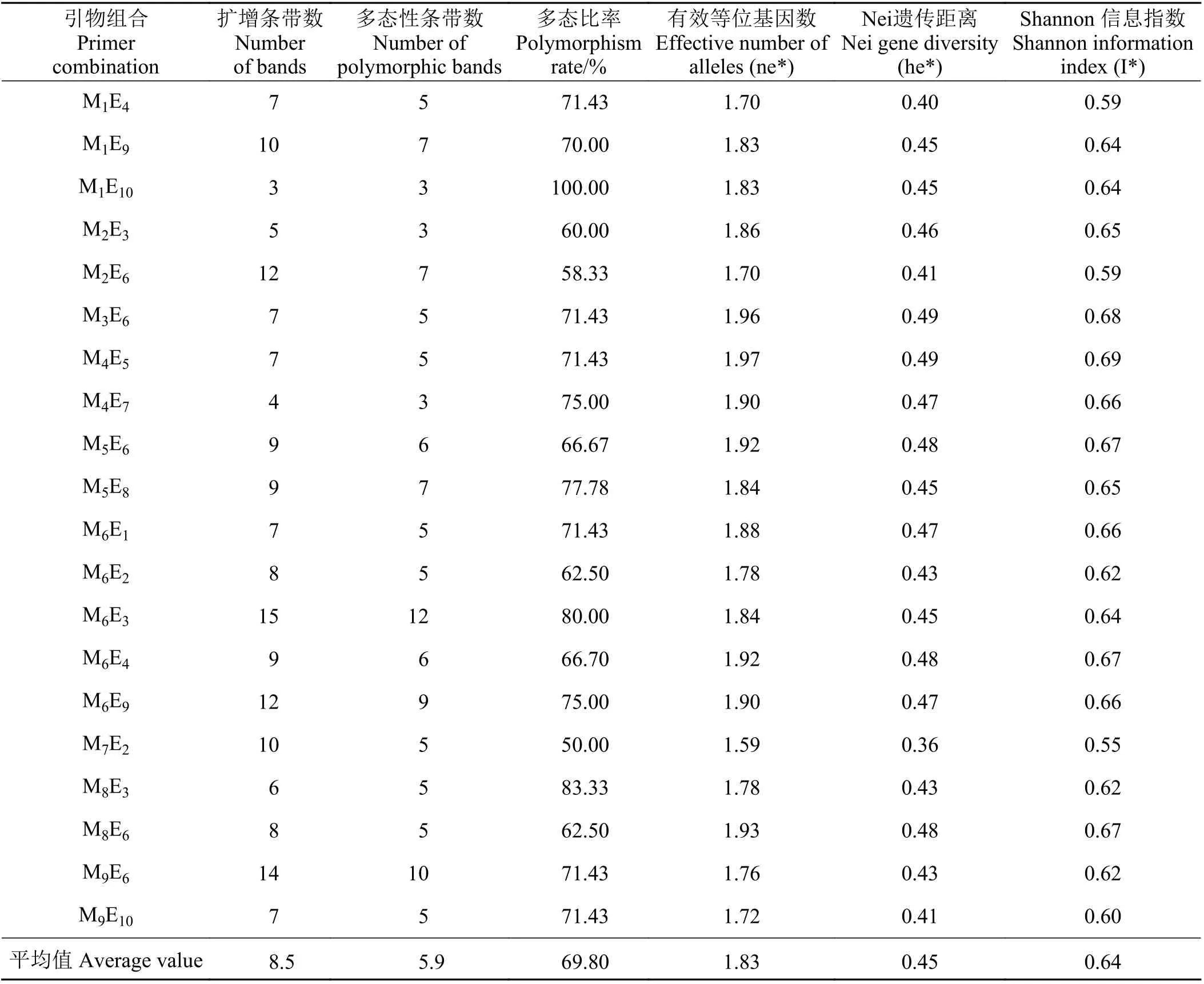
表3 20 對引物組合的多態(tài)性擴增結果Table 3 The results of polymorphic amplification of 20 pairs of primer combinations
2.4 群體結構分析
對49 份海雀稗種質進行群體遺傳結構分析表明,當樣本的等位變異頻率特征類型數K = 2 時,其模型的后驗概率最大。根據K 值將49 份海雀稗種質劃分為PopⅠ、PopⅡ兩個類群,分別以紅色、綠色表示(圖4),且每個亞類群中主要顏色所占的比例情況(Q 值)可以反映種質的來源情況。劉麗華等[16]認為,Q > 0.6 視為血緣單一,Q < 0.6 視為具有混合來源。49 份海雀稗材料的Q 值劃分結果表明,PopⅠ亞群中各份材料只有1 份材料Q = 0.6,說明PopⅠ亞群的種質遺傳背景較單一,與其他亞群間的基因交流不多;PopⅡ亞群部分種質的Q 值 < 0.6,說明種質資源的基因滲透較高,表明其具有混合來源,遺傳背景較復雜。2 個類群中PopⅠ類群包含的海雀稗材料較多(34 份),占供試種質總數71.42%,其典型代表材料是647895 和647892,而其余15 份材料劃分為另一類,典型代表材料是647908 和647899。
3 討論

圖2 海雀稗種質UPGMA 聚類Figure 2 UPGMA cluster map of seashore Paspalum
種質資源是開展育種工作的基礎,種質資源利用的前提是要對其進行多樣性分析和遺傳背景研究[17]。本研究發(fā)現供試海雀稗引物的多態(tài)比率為69.80%,與解新明等[4]多態(tài)比率64.77%的結果相比,本研究海雀稗種質資源遺傳背景相對較豐富。聚類分析與群體結構分析是進行種質資源遺傳多樣性分析的有效手段[18],49 份海雀稗種質資源的遺傳相似系數平均值為0.67,表明49 份海雀稗種質資源親緣關系相對較遠。對供試材料按UPGMA 進行聚類分析,繪制親緣關系樹狀圖(圖2),在遺傳相似系數為0.69 時將材料分為2 大類。第Ⅰ類包含16 份材料,第Ⅱ類包含33 份材料,按UPGMA 進行聚類分析時,其中A 類材料和B 類材料則是按UPGMA 進行聚類分為2 大類中第Ⅰ類材料,C 類材料和D 類材料則是按UPGMA 進行聚類分為2 大類中第Ⅱ類材料,其擬合共相關系數 r = 0.669 表明聚類結果很好的體現了種質之間的遺傳關系。
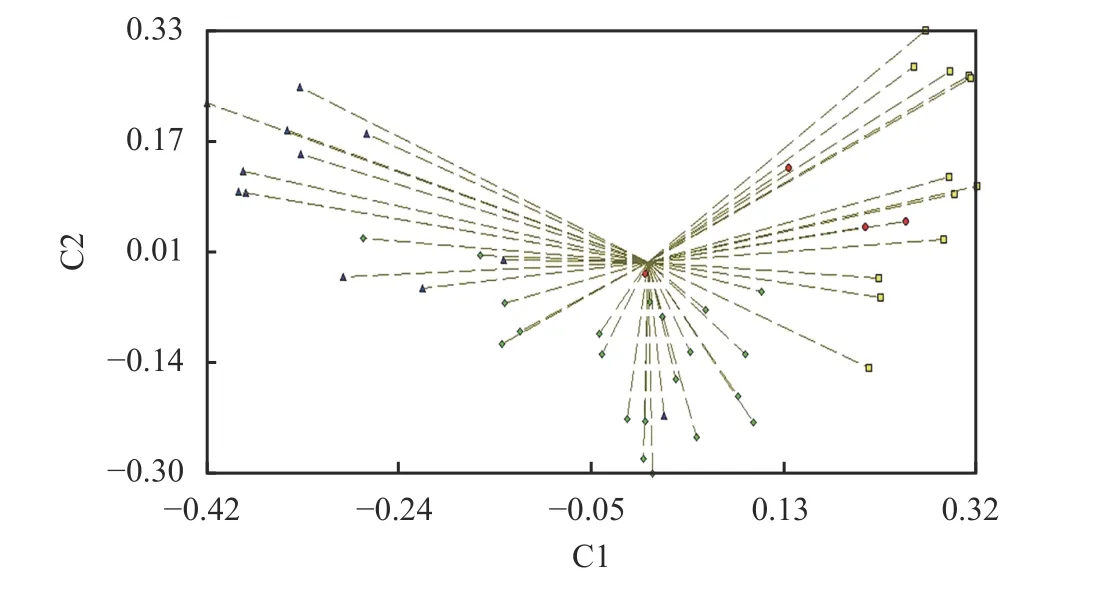
圖3 海雀稗主成分分析Figure 3 Principal coordinate analysis of seashore Paspalum
用Structure2.3.4 軟件對49 份海雀稗材料進行分析,K = 2 時,各群體內海雀稗的相似值最大,群體遺傳結構分析結果將49 份海雀稗份材料劃分為2 個類群。PopⅠ類群包含34 份材料;PopⅡ類群包括15 份材料。當K = 3、4、5 時,海雀稗的邊緣群體出現分化(圖4)。按UPGMA 進行聚類中第Ⅰ類包含的16 份材料都包含在Structure 分析時劃分的PopⅠ類群里。
在Structure 分析時劃分的PopⅡ類群里的15 份材料全部包含于按UPGMA 進行聚類中的第Ⅱ類中。但另有18 份材料的分類在按UPGMA 進行聚類和Structure 分析時出現不一致現象,這18 份材料在按UPGMA 進行聚類全部劃分在第Ⅱ類中,而Structure 分析時則全部都劃分在PopⅠ類群中。這個現象與主成分分析時A 類與B 類材料、C 類材料有交疊的部分,B 類和D 類中的海雀稗材料有交疊部分相呼應。只有部分主成分聚類結果與采用UPGMA進行聚類得出的結果一致,可能是因為主成分分析和系統聚類分析都能對材料進行聚類分析,但它們反映的信息卻不同[19]。
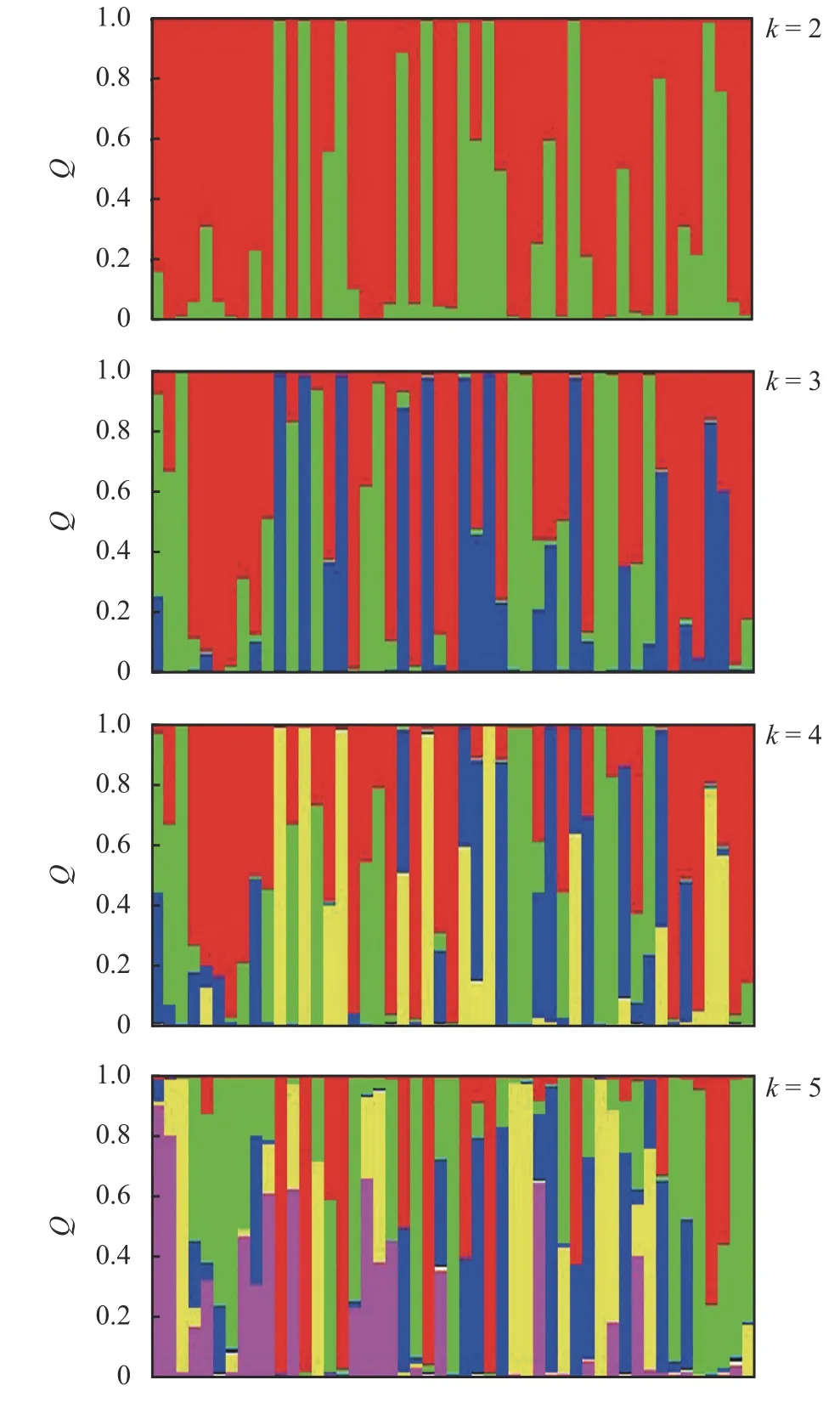
圖4 海雀稗的群體遺傳結構Figure 4 Population genetic structure of seashore Paspalum
綜合分析49 份種質資源在3 種劃分方法中的結果,其中4 份材料(647916、647922、647918、647917)、10 份材料(509022、647903、647906、647921、647899、647897、647913、509021、sp005、sp004)、12 份 材 料(647904、 647895、 647893、 647891、 647902、 647892、647900、 647911、 614678、 647896、 647898、 647909)、18 份材料(647907、647919、647912、647910、647920、647914、 647901、 614679、 364368、 576134、 647923、509023、509020、576138、sp001、sp002、sp006、sp007)在3 種劃分方法時都能各自聚為一類,以上4 種類型的典型代表材料分別是647918、647899、647895、647910,這與前面群體結構分析的結果較為一致。另有5 份材料(647908、647894、647905、647916、377709)在3 種劃分方法聚類時,在大體分類形同的情況下各自分類略有差異,這可能跟劃分的方法與關。比較聚類分析和群體結構分析,群體結構分析能夠反映出育種材料間的基因滲入和交流情況, 能夠更好地揭示材料內在的遺傳結構,對于育種工作者更準確掌握種質間的遺傳關系有很大的幫助。
來自巴西的647913 和來自美國夏威夷的647897相似系數最大(0.88),而來自美國喬治亞州的647908和美國夏威夷647896 兩份材料遺傳相似系數最小(0.53),來自不同國家的兩個材料遺傳相似性最高,而來自同一國家的兩個材料遺傳相似性最小,目前尚缺乏明確的解釋。
4 結論
本研究利用100 對SRAP 標記對49 份種質資源進行遺傳多樣性分析,篩選獲得20 對擴增穩(wěn)定、多態(tài)性好的SRAP 引物;對49 份海雀稗種質資源進行PCR 擴增,共獲得169 個條帶,多態(tài)性位點百分率為69.80%;平均每條引物擴增多態(tài)性條帶數為5.9,有效等位基因數平均值為1.83,Shannon 信息指數平均值為0.64,Nei 遺傳距離平均值為0.45。
49 份海雀稗種質資源的遺傳相似系數介于0.52~0.88,平均值為0.67,進一步采用UPGMA 聚類分析、主成分分析、群體結構分析對其進行分類,44 份材料在3 種方法下均表現為較一致的分類,5 份材料表現出一定程度的變異,表明此研究結果反映的種質親緣關系基本穩(wěn)定,可用于未來育種工作的親本選擇。

