基于16s rRNA的徽派臘肉加工過程中微生物群落結構分析
周瑩 王兆明 涂健 陳興勇 徐寶才
摘要:為探究徽派臘肉加工過程中的微生物群落演替規律,采用高通量測序技術對不同加工階段的徽派臘肉中微生物16S rRNA進行V3~V4區測序,比較不同加工階段(鮮肉、腌制中期、腌制結束、成熟中期、成熟結束)臘肉中細菌群落結構組成及多樣性差異。結果表明:5 個加工階段分別獲得80 155、80 116、80 174、80 114、80 174 條有效序列;在門分類水平上,厚壁菌門(Firmicutes)、變形菌門(Proteobacteria)、放線菌門(Actinomycetes)、擬桿菌門(Bacteroides)相對豐度較高;在屬分類水平上,鮮肉、腌制中期和腌制結束的臘肉中主要優勢菌為乳球菌(Lactococcus)和假單胞菌(Pseudomonas),成熟中期和成熟結束的臘肉中主要優勢菌為葡萄球菌(Staphylococcus)、鹽弧菌(Salinivibrio)和放線菌(Actinomycetes)。微生物群落結構和多樣性在徽派臘肉不同加工階段存在明顯差異。
關鍵詞:徽派臘肉;高通量測序;發酵;微生物群落;細菌多樣性
Analysis of Microbial Community Structure during the Processing of Hui-Style Bacon by 16S rRNA Sequencing
ZHOU Ying1,2,3, WANG Zhaoming2,3, TU Jian1, CHEN Xingyong1, XU Baocai2,3,*
(1.School of Tea and Food Science and Technology, Anhui Agricultural University, Hefei ??230036, China; 2.School of Food and Biological Engineering, Hefei University of Technology, Hefei ??230009, China; 3.Engineering Research Center of Bio-process, Ministry of Education, Hefei University of Technology, Hefei ??230009, China)
Abstract:High throughput sequencing (HTS) was used to investigate the microbial community succession of Hui-style bacon samples at different processing stages (fresh meat, mid-curing, the end of curing, mid-maturation and the end of maturation). The V3-V4 region of the bacterial 16S rRNA gene was sequenced to compare the composition and diversity of bacterial community among the processing stages. The results showed that 80155, 80116, 80174, 80114 and 80174 effective sequences were obtained for the five processing stages, respectively. At ?the phylum level, Firmicutes, Proteobacteria, actinomycetes and Bacteroides were dominant. At the genus level,LactococcusandPseudomonaswere the dominant bacteria in fresh meat, at the mid-cuing stage and at the end of curing, whileStaphylococcus,Salinivibrioandactinomyceteswere the dominant bacteria at the mid-maturation stage and at the end of maturation. These findings showed that different processing stages had significant effects on the microbial community structure and diversity in Hui-style bacon.
Keywords:Hui-style bacon; high throughput sequencing; fermentation; microbial community; bacterial diversity
DOI:10.7506/rlyj1001-8123-20201215-289
中圖分類號:TS251.5?????????????? 文獻標志碼:A???????????? 文章編號:
引文格式:
周瑩, 王兆明, 涂健, 等. 基于16s rRNA的徽派臘肉加工過程中微生物群落結構分析[J]. 肉類研究, 2021, 35(2):? . DOI:10.7506/rlyj1001-8123-20201215-289.??? http://www.rlyj.net.cn
ZHOU Ying, WANG Zhaoming, TU Jian, et al. Analysis of microbial community structure during the processing of hui-style bacon by 16S rRNA sequencing[J]. Meat Research, 2021, 35(2):? . DOI:10.7506/rlyj1001-8123-20201215-289.??? http://www.rlyj.net.cn
腌臘肉制品在我國有上千年的制作歷史[1-2],深受人民群眾的喜愛。生肉經過腌制和成熟后,肉色臘紅、耐貯藏、色味鮮美、食用方便,有著獨特的腌臘風味[3-4]。徽派臘肉俗名“刀板香”,選料精良,制作復雜,原料采用徽州地區土雜豬厚度均勻的五花肉,在適當的環境條件下(溫度、濕度),經預處理、腌制、晾曬(輕發酵)等核心工藝加工而成,是具有典型特征品質的腌臘肉制品。徽派臘肉成品具有高鹽含量和低水分含量的特點,兼具干腌肉制品和發酵肉制品特性。作為徽菜的典型代表,徽派臘肉是徽州文化的一張重要名片,是具有濃郁地方特色的中式傳統肉制品。
微生物的代謝產物與酶會對肌肉蛋白的降解、脂肪的氧化水解等有重要影響,在發酵過程中對臘肉風味輪廓形成具有重要貢獻[5]。有研究表明,腌臘肉制品的質量與微生物的活動密切相關[6-7]。Yu Hai等[8]從如皋火腿中分離出1 株高效的轉氨酶生產菌(肺炎克雷伯菌YZusK-4),并成功將其應用到發酵肉制品中,達到顯著改善產品風味的效果。Zhao Changqing等[9]研究發現,嗜酸乳桿菌作為發酵劑,不僅可以改善肉品風味,還可以有效抑制有害微生物生長,提高食用安全性。Berdague等[10]通過在發酵香腸中接種發酵型乳酸菌和葡萄球菌等,發現可以產生78 種風味物質,如烴、醛、酮等。類似地,Bruna等[11]通過在發酵香腸中接種霉菌,發現接種后香腸的保質期顯著延長,同時香腸的風味物質種類也顯著增加。不難發現,微生物在發酵肉制品風味形成中起重要作用。全面了解臘肉的微生物群落演替有助于解析其風味形成機制,并且可以為天然發酵劑的制備提供一定的理論依據。
高通量測序(high-throughput sequencing,HTS)是食品行業的一項新興技術,可以準確識別不同食物基質中的微生物群落,并且能夠快速、準確地獲取豐富的生物學數據和大量微生物學信息,已成為研究微生物多樣性和群落特性的主要方法之一[12-13]。HTS不僅可以提供有關微生物群落結構的信息,還可以提供實用的基因組成和相關分布[14]。HTS技術在許多發酵食品中得到了廣泛應用,如豆豉[15]、泡菜[16]、酸菜[17]和發酵香腸[18]等。目前,HTS技術也已經被用來研究臘肉中的微生物群落結構[19-20],但對徽派臘肉微生物結構的相關報道還很少。本研究基于HTS技術揭示徽派臘肉發酵過程中微生物的變化,旨在為探明徽派臘肉的品質形成機制和商業化開發提供一定的參考。
1?? 材料與方法
1.1 ??徽派臘肉樣品采集
跟蹤采集徽派臘肉(2019年5月—2019年7月)1 個批次(約1 t)生產的完整過程。根據生產周期確定5 個采樣點(見表1)。取樣完成后,當天將樣品運回實驗室分離,每個樣品不少于1 000 g,每個時間點取3 個平行樣品,混勻。準確稱取10 g樣品,分裝至無菌真空袋中,按不同取樣周期編號。以上過程均在超凈微生物檢測臺上完成。
細菌基因組DNA快速提取試劑盒 ??天根生化科技(北京)有限公司;515F/806R引物 ??武漢天一輝遠生物科技有限公司;DNeasy mericon Food Kit提取試劑盒 ??德國Qiagen公司;5×Trans Start?Fast Pfu緩沖液 ??生工生物工程(上海)股份有限公司;Phusion?High-Fidelity PCR Master Mix with GC Buffer?? 美國New England Biolabs公司;其他試劑均為國產分析純。
1.2?? 儀器與設備
DYY-6c電泳儀 ??北京六一儀器廠;WD-943B凝膠成像系統 ??北京六一生物科技有限公司;ABI Step One Plus實時聚合酶鏈式反應(polymerase chain reaction,PCR)系統 ??美國Life Technologies公司;Illumina Mi Seq PE300高通量測序平臺 ??美國Illumina公司;ND-2000C微量紫外分光光度計 ??美國Nano Drop公司。
1.3?? 細菌測序
對細菌序列16S rDNA V3~V4區域進行相關測序。細菌16S rDNA V3~V4區應用的引物為515F(5′-GTGYCAGCMGCCGCGGTAA-3′)、806R(5′-GGACTACHVGGGTWTCT-3′)。將稀釋后的DNA基因組作為模板,使用帶Barcode的特異引物、Phusion?High-Fidelity PCR Master Mix with GC Buffer和高保真酶進行PCR擴增,確保擴增的效率和準確性。擴增條件:94 ℃、2 min,然后98 ℃、10 s,62 ℃、30 s,68 ℃、30 s進行30 個循環,最后68 ℃、5 min。PCR產物用2%電泳凝膠進行檢測。擴增體系(50 μL):5 μL 10×KOD緩沖液、5 μL 2 mmol/L dNTPs、3 μL 25 mmol/L MgSO4、上下游引物各1.5 μL(10 μmol/L)、1 μL DNA聚合酶、100 ng模板DNA。然后使用1×TAE 2%瓊脂糖凝膠電泳純化PCR產物,切出目標條帶并對其進行回收。文庫建成并測試合格后,上機測序。
1.4?? 數據分析及圖像處理
1.4.1? ?細菌群落α多樣性分析
α多樣性是反映物種豐富度和同質性的綜合指標,可用于分析樣本的復雜性,即樣本中群落的多樣性。利用Spss軟件對實驗數據進行聚類分析。其中包括Observed-otus、Chao1指數、Shannon指數、Simpson指數、ACE指數和覆蓋率。
1.4.2? ?細菌群落組成分析
采用RDP分類算法選擇其中最常見的序列作為代表操作分類單元(operational taxonomic units,OTUs)的序列。OTUs序列的物種注釋,用Mothur與SILVA132[21]的SSUrRNA數據庫[22]進行設定閾值為0.8~1.0的物種注釋剖析,在每個分類層次計算每個樣本的界、門、綱、目、科、屬、種水平群落組成。使用MUSCLE(版本3.8.31)軟件[23]對多個序列進行快速測序,可以得到所有OTUs序列的系統發生關系。最后,每個樣本的數據都是平均值,以數據量最少的樣品為標準進行均一化處理,樣本中的最小數據量為標準。隨后的α多樣性分析和β多樣性分析基于均值數據。按照分析結果,繪制樣品的稀釋曲線、熱圖、菌群分布直方圖和高質量序列分布圖。
2?? 結果與分析
2.1 ??不同加工階段臘肉中細菌測序結果分析
對徽派臘肉中提取的微生物總DNA進行V3~V4區間的高通量測序,在優化軟件數據后,對排序結果進行排序和過濾。共獲得1 202 200 個高質量序列,由表2可知,有效序列平均長度為253 bp。序列長度與所設計出的引物序列長度比較接近,說明臘肉樣本的測序結果比較合理,可以滿足后續的數據分析。
2.2 ??不同加工階段臘肉中細菌多樣性分析
2.2.1? ?不同加工階段臘肉中細菌群落α多樣性分析
α多樣性主要指一定區域或特定生態系統本身的多樣性,是一種能夠反映物種豐富度和同質性的綜合性質指標。α多樣性一方面與菌群豐富度有關,另一方面與菌群多樣性有關。菌群豐富度指數主要包括ACE指數和Chao1指數。菌群多樣性指數主要包括覆蓋率和Shannon指數。Chao1指數可以估計微生物群落中的OTU數量,并計算微生物群落的豐度。通常而言,Chao1指數越大,樣品中的微生物群落物種總數越多。
由表3可知,在加工前期(鮮肉、腌制中期、腌制結束、成熟中期),Chao1指數較大,而成熟結束,Chao1指數明顯減小。ACE指數表現出與Chao1指數類似的變化規律,說明加工后期臘肉樣品中細菌群落豐富度變化較大。Shannon指數和Simpson指數可以表征微生物的豐度和同質性。Shannon指數越高,微生物群落的多樣性就越大。Shannon指數和Simpson指數呈先升高后降低的趨勢,說明臘肉樣品中細菌群落多樣性隨發酵時間的延長發生了顯著變化,在加工后期,微生物多樣性降低。覆蓋率可以反映測序深度,覆蓋率越接近1,測序越合理。本研究中所有樣品覆蓋率均大于0.997,說明徽派臘肉樣品中的序列基本檢測到,測序結果可以反映徽派臘肉樣品中微生物的真實狀況。
2.2.2? ?不同加工階段臘肉中細菌稀釋曲線
稀釋和等級聚類曲線是用來描述樣品中微生物群落多樣性的曲線。稀釋曲線可以直接反映測序量的合理性,也可以間接反映樣品中物種的數量。由圖1可知,所有徽派臘肉樣品的稀釋曲線趨向平坦,表明測序深度合理。隨著測序深度的增加,加工過程中的各個階段臘肉樣本的稀釋曲線呈現先上升后趨于平緩的趨勢。表示樣品選定的序列數據足夠多,可以反映樣品中大多數微生物區域的數據,測序結果合理。
等級聚類曲線是樣本中按相對豐度(或可用序列數)從大到小排序得到的對應排序編號。在等級聚類曲線中,橫坐標方向上,曲線的寬度反映了物種的豐富性。縱坐標方向上,樣品中物種均勻度由曲線的平滑程度來表示。如果曲線顯示平滑,說明物種是均勻分布的[24]。由圖2可知,隨著臘肉加工各個階段的加工時間增加,曲線呈現先下降后趨于平緩的趨勢,說明在測序過程中物種分布較為均勻。
2.2.3? ?不同加工階段臘肉中細菌群落β多樣性分析
由圖3可知,徽派臘肉整個加工過程中,微生物的種間差異逐漸增加,在成熟后期,各物種間的差異達到最大。
2.3 ??不同加工階段臘肉中物種分布情況
2.3.1 ??物種相對豐度
在門分類水平上,共有47 個細菌門被檢出,選取豐度排名前10的細菌繪制相對豐度柱形圖。由圖4可知,厚壁菌門(Firmicutes)、變形菌門(Proteobacteria)、放線菌門(Actinomycetes)、擬桿菌門(Bacteroides)占主導地位。在整個加工過程中,厚壁菌門(Firmicutes)和變形菌門(Proteobacteria)具有主要優勢。其中厚壁菌門(Firmicutes)相對豐度最大,鮮肉、腌制中期、腌制結束、成熟中期和成熟結束的相對豐度分別為51.1%、38.2%、39.4%、38.8%和52.5%。其次,變形菌門(Proteobacteria)是另一優勢微生物,鮮肉、腌制中期、腌制結束、成熟中期和成熟結束的相對豐度分別為33.7%、45.3%、39.0%、52.2%和42.8%。此外,放線菌門(Actinomycetes)在鮮肉、腌制中期、腌制結束、成熟中期和成熟結束的相對豐度分別為3.5%、3.9%、4.3%、1.3%和1.6%,而擬桿菌門(Bacteroides)在鮮肉、腌制中期、腌制結束、成熟中期和成熟結束的相對豐度分別為7.8%、9.3%、14.2%、5.7%和2.3%。
在屬分類水平上,樣品中共檢測到676 種細菌,選取豐度排名前30的細菌繪制相對豐度柱形圖。由圖5可知,在整個加工過程中,葡萄球菌(Staphylococcus)、乳球菌(Lactococcus)、鏈球菌(Streptococcus)、嗜冷桿菌(Psychrobacter)、鹽弧菌(Salinivibrio)和不動桿菌(Acinetobacter)的相對豐度較大,是優勢細菌。李彥虎[25]研究發現,隴西臘肉中主要細菌是環絲菌(Cyclomycetes)、肉桿菌(Botulinum)、乳球菌、假單胞菌(Pseudomonas)和嗜冷桿菌。董蘊等[26]研究發現,湖北恩施臘肉的主要細菌種類為葡萄球菌、嗜冷桿菌、假單胞菌、環孢菌(Cyclospora)、鹽單胞菌(Cobetia)和不動桿菌。Cordero等[27]發現,使用食鹽的西班牙火腿中60%微生物為葡萄球菌。Rodríguez[28]、Lorenzo[29]等分別對意大利和西班牙火腿進行研究,在發酵過程中與發酵后期,葡萄球菌和微球菌(Micrococcus)數量明顯上升,與本研究結果相似。金黃色葡萄球菌(Staphylococcus aureus)有抑制腐敗細菌生長的作用,并在發酵中起良好作用[30]。由于徽派臘肉在發酵階段均處于低溫狀態,導致大量嗜低溫菌與耐鹽菌大量繁殖。而在臘肉的發酵過程中,樣品中的細菌明顯出現演替現象。例如,葡萄球菌相對豐度由鮮肉的0.3%到成熟結束時增加至44.2%,達到最高,可能是由于葡萄球菌對于環境因子的耐受性較高,葡萄球菌同時也是臘肉風味的來源之一。不動桿菌相對豐度呈現先增加后降低的趨勢,在成熟中期時達到最高(16.6%),在成熟結束時降低至7.8%。鹽單胞菌屬(Halomonassp)(屬于圖中Others分類)在腌制過程中也呈現一直增加的趨勢,在成熟結束時達到最高(6.9%)。
2.3.2? ?物種豐度聚類熱圖
為更直觀表現徽派臘肉發酵過程中優勢細菌的豐度變化,挑選相對豐度排名前35的屬繪制熱圖。
由圖6可知,鮮肉優勢菌為乳酸菌(Lactobacillus)、棒狀桿菌(Corynebacterium)、肉桿菌(Carnobacterium)和腸球菌(Enterococcus)等。腌制中期的主要細菌為腸桿菌(Enterobacteriaceae)。腌制結束的優勢菌主要為雙歧桿菌(Bifidobacterium)、鏈球菌(Streptococcus)和嗜冷桿菌。成熟中期樣品的優勢菌主要為嗜冷細菌(Psychrophilic bacteria)、弧菌(Vibrio),成熟結束,弧菌(Vibrio)、耐鹽菌和葡萄球菌(Staphylococcus)最為顯著。
在腌制階段,由于環境溫度較低,滲透壓較高,使得這個階段的優勢菌以耐鹽菌與嗜冷菌為主。到了成熟階段,主要的變化以水分含量為主,并且不同類型的微生物相互抑制,使臘肉樣品中的菌群得到進一步優化。陳競適等[31]在湘西臘肉中發現了微球菌、乳桿菌及葡萄球菌。Wang Xinghong[32]、Wang Xinhui[33]等在宣威火腿中發現了微球菌、棒狀桿菌和乳酸桿菌,在四川臘肉中發現了葡萄球菌、乳球菌和芽孢桿菌。Papamanoli等[34]對廣式臘腸進行研究,發現主要微生物為乳桿菌、片球菌和葡萄球菌,與本研究較為一致。
2.3.3? ?LEfSe分析
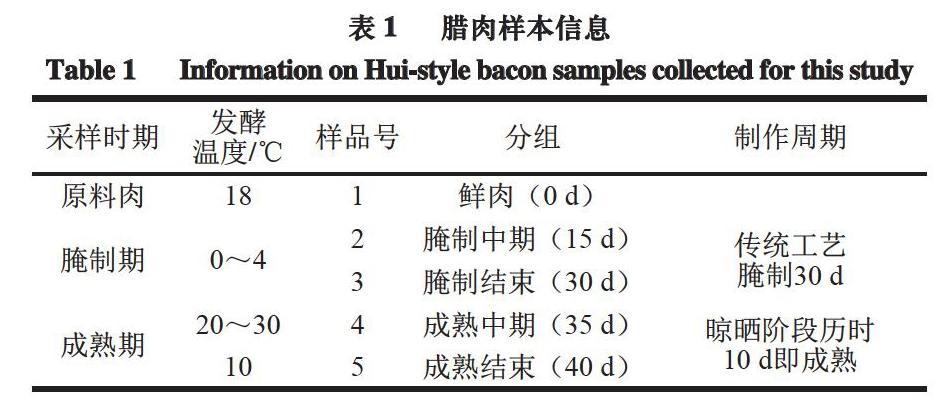
LEfSe分析(線性判別分析(Linear Discriminant Analysis,LDA)對數評分閾值≥4)展現的是有顯著性差異的物種,本研究用于分析明確不同階段處理過程中群落組成的異同。由圖7可知,鮮肉、成熟中期和成熟結束時,樣品中共有24 個明顯不同的類群。其中,成熟結束時的細菌群落明顯比鮮肉和腌制結束時多,并以弧菌、葡萄球菌和耐鹽菌為主。
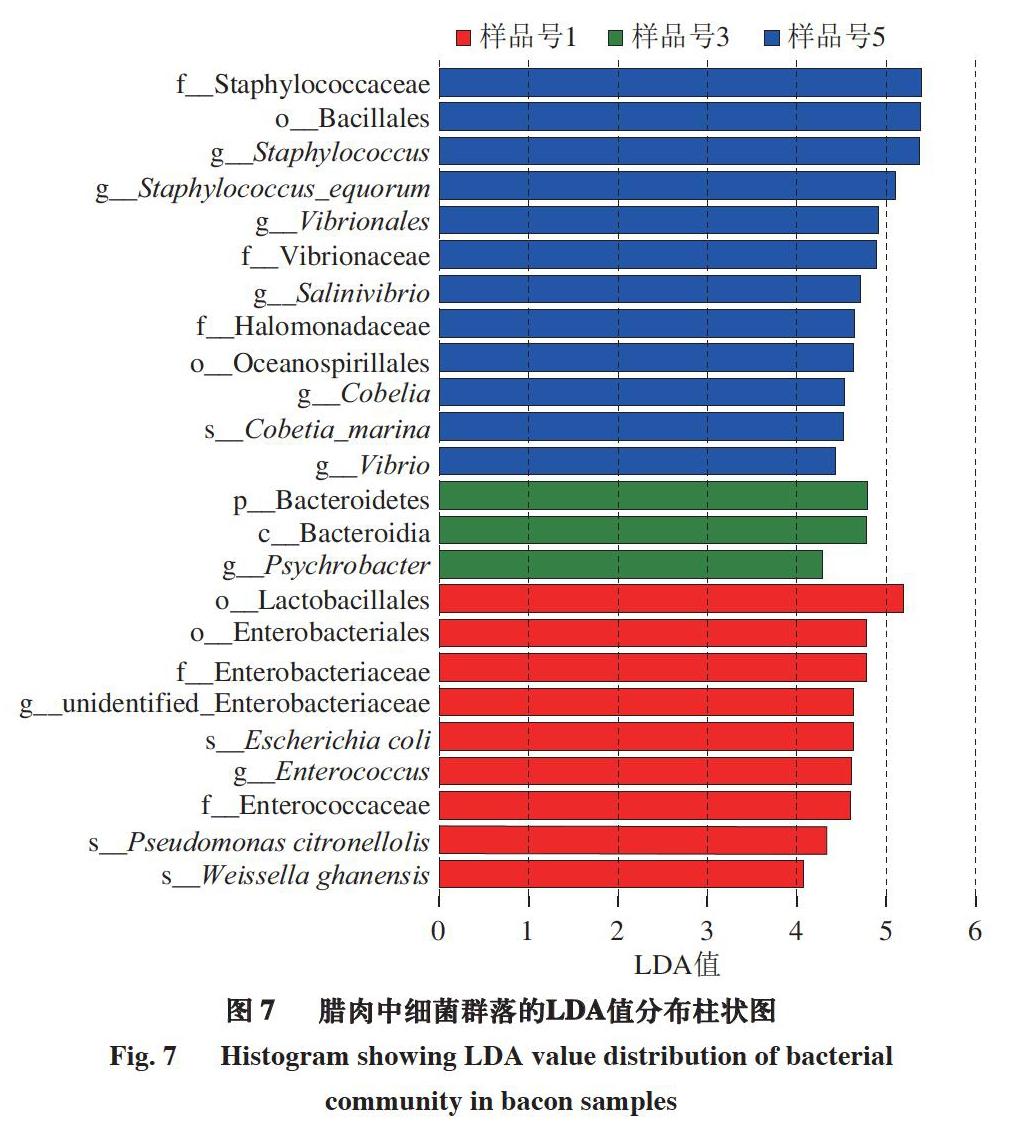
由圖8可知,在鮮肉、成熟中期和成熟結束時,分別觀察到共有9、3、12 個分類群存在顯著差異,表明隨著生產過程的進行,不同加工時間的群落組成相似度先減小后增加,說明在加工后期臘肉中逐漸形成優勢菌。
3?? 結論
本研究采用HTS技術,對徽派臘肉中的微生物群落構成進行研究。結果表明:在臘肉發酵過程中,微生物群落發生較大變化,優勢菌由乳球菌(Lactococcus)、雙歧桿菌(Bifidobacterium)和芽孢桿菌(Bacillus)等逐漸演替為嗜冷桿菌(Psychrobacter)、弧菌(Vibrio)、鹽單胞菌(Halomonas)和葡萄球菌(Staphylococcus);發酵時間越久,菌群分布越穩定,優勢菌越明顯。因此,對徽派臘肉進行微生物測序研究有助于解析其獨特風味的來源,同時為徽派臘肉生產標準的制定等提供理論依據。
參考文獻:
[1] 柴子惠. 低鈉臘肉加工和貯藏期間理化特性和菌相變化的研究[D]. 重慶: 西南大學, 2019.
[2] GUO Xin, HUANG Feng, ZHANG Hong, et al. Classification of traditional Chinese pork bacon based on physicochemical properties and chemometric techniques[J]. Meat Science, 2016, 117: 182-186. DOI:10.1016/j.meatsci.2016.02.008.
[3] 賀雪華, 李林, 白登榮, 等. 改進型城口臘肉貯藏過程中的品質變化及貨架期預測[J]. 食品科學, 2017, 38(11): 249-255. DOI:10.7506/spkx1002-6630-201711040.
[4] 粟桂蓉, 彭鈺媛, 周璐璐, 等. 傳統土家臘肉加工過程中風味物質研究[J]. 食品科技, 2017, 42(3): 118-123.
[5] KO?O?YN-KRAJEWSKA D, DOLATOWSKI Z J. Probiotic meat products and human nutrition[J]. Process Biochemistry, 2012, 47(12): 1761-1772. DOI:10.1016/j.procbio.2012.09.017.
[6] COMI G, ANDYANTO D, MANZANO M, et al. Lactococcus lactis and Lactobacillus sakei as bio-protective culture to eliminate Leuconostoc mesenteroides spoilage and improve the shelf life and sensorial characteristics of commercial cooked bacon[J]. Food Microbiology, 2016, 58: 16-22. DOI:10.1016/j.fm.2016.03.001.
[7] ZHAO C J, SCHIEBER A, GAENZLE M G. Formation of taste-active amino acids, amino acid derivatives and peptides in food fermentations: a review[J]. Food Research International, 2016, 89: 39-47. DOI:10.1016/j.foodres.2016.08.042.
[8] YU Hai, YIN Yongqi, XU Lin, et al. Genome sequence of Klebsiella pneumoniae YZUSK-4, a bacterium proposed as a starter culture for fermented meat products[J]. Genome Announcements, 2015, 3(4): e00774-15. DOI:10.1128/genomea.00774-15.
[9] ZHAO Changqing, LU Ziyang, HUANG Jing, et al. Optimum for pork jerky fermented with Lactobacillus acidophilus[J]. Journal of Biobased Materials and Bioenergy, 2017, 11(1): 21-26. DOI:10.1166/jbmb.2017.1640.
[10] BERDAGU? J L, MONTEIL P, MONTEL M C, et al. Effect of starter cultures on the formation of flavor compounds in dry sausage[J]. Meat Science, 1993, 35(3): 275-287. DOI:10.1016/0309-1740(93)90033-E.
[11] BRUNA J M, HIERRO E, DE LA HOZ L, et al. Changes in selected biochemical and sensory parameters as affected by the superficial inoculation of Penicillium camemberti on dry fermented sausages[J]. International Journal of Food Microbiology, 2003, 85(1/2): 111-125. DOI:10.1016/S0168-1605(02)00505-6.
[12] CHEN K, PACHTER L. Bioinformatics for whole-genome shotgun sequencing of microbial communities[J]. PLoS Computational Biology, 2005, 1(2): 106-112. DOI:10.1371/journal.pcbi.0010024.
[13] TRINGE S G, RUBIN E M. Metagenomics: DNA sequencing of environmental samples[J]. Nature Reviews Genetics, 2005, 6(11): 805. DOI:10.1038/nrg1709.
[14] ZHU Qianglong, LIU Shi, GAO Peng, et al. High-throughput sequencing technology and its application[J]. Journal of Northeast Agricultural University (English Edition), 2014, 21(3): 84-96. DOI:10.1016/S1006-8104(14)60073-8.
[15] YANG Lin, YANG Huilin, TU Zongcai, et al. High-throughput sequencing of microbial community diversity and dynamics during Douchi fermentation[J]. PLoS One, 2016, 11(12): e0168166. DOI:10.1371/journal.pone.0168166.
[16] LIANG Huipeng, YIN Liguo, ZHANG Yahao, et al. Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcai paocai, a traditional Chinese fermented vegetable food, as assessed by Illumina MiSeq sequencing, DGGE and qPCR assay[J]. Annals of Microbiology, 2018, 68(2): 111-122. DOI:10.1007/s13213-017-1321-z.
[17] YANG Hongyan, WU Hao, GAO Lijuan, et al. Effects of Lactobacillus curvatus and Leuconostoc mesenteroides on Suan cai fermentation in Northeast China[J]. Journal of Microbiology and Biotechnology, 2016, 26(12): 2148-2158. DOI:10.4014/jmb.1607.07010.
[18] WANG Xinghui, WANG Songhu, ZHAO Hai. Unraveling microbial community diversity and succession of Chinese Sichuan sausages during spontaneous fermentation by high-throughput sequencing[J]. Journal of Food Science and Technology-Mysore, 2019, 56: 3254-3263. DOI:10.1007/s13197-019-03781-y.
[19] 文開勇, 汪月, 文鵬程, 等. 四川傳統臘肉中微生物群落結構研究[J]. 食品與發酵工業, 2020, 46(3): 36-42. DOI:10.13995/j.cnki.11-1802/ts.022133.
[20] 畢旺來, 胡雷鳳, 明亮, 等. 煙熏臘肉貨架期主要微生物的研究[J]. 武漢輕工大學學報, 2019, 38(2): 16-20. DOI:10.3969/j.issn.2095-7386.2019.02.003.
[21] EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996. DOI:10.1038/NMETH.2604.
[22] WANG Q G, GARRITY M, TIEDJE J M, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07.
[23] QUAST C, PRUESSE E, YILMAZ P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41: D590-6. DOI:10.1093/nar/gks1219.
[24] LUNDBERG D S, YOURSTONE S, MIECZKOWSKI P, et al. Practical innovations for high-throughput amplicon sequencing[J]. Nature Methods, 2013, 10(10): 999. DOI:10.1038/nmeth.2634.
[25] 李彥虎. 傳統隴西臘肉制作過程中微生物群落演替與臘肉風味的相關性分析[D]. 蘭州: 甘肅農業大學, 2020.
[26] 董蘊, 王玉榮, 王堯, 等. 基于變性梯度凝膠電泳和MiSeq高通量測序技術分析恩施地區臘肉的細菌多樣性[J]. 肉類研究, 2018, 32(10): 37-42. DOI:10.7506/rlyj1001-8123-201810007.
[27] CORDERO M R, ZUMALAC?RREGUI J M. Characterization of micrococcaceae isolated from salt used for Spanish dry-cured ham[J]. Letters in Applied Microbiology, 2000, 31(4): 303-306. DOI:10.1046/j.1472-765x.2000.00818.x.
[28] RODR?GUEZ M, N??EZ F, C?RDOBA J J, et al. Characterization of Staphylococcus spp. and Micrococcus spp. isolated from Iberian ham throughout the ripening process[J]. International Journal of Food Microbiology, 1994, 24(1/2): 329-335. DOI:10.1016/0168-1605(94)90131-7.
[29] LORENZO J M, GARC?A FONT?N M C, G?MEZ M, et al. Study of the Micrococcaceae and Staphylococcaceae throughout the manufacture of dry-cured Lacón (a Spanish traditional meat product) made without or with additives[J]. Journal of Food Research, 2012, 1(1): 200-211. DOI:10.5539/jfr.v1n1p200.
[30] 李珊珊, 祝超智, 崔文明, 等. 發酵肉制品中微生物發酵劑分離篩選及應用研究進展[J]. 肉類研究, 2019, 33(7): 61-66. DOI:10.7506/rlyj1001-8123-20190424-089.
[31] 陳競適, 劉靜, 任海姣, 等. 湘西陳年臘肉微生物群落分析及高產脂肪酶細菌的篩選[J]. 肉類研究, 2017, 31(3): 1-6. DOI:10.7506/rlyj1001-8123-201703001.
[32] WANG Xinghong, MA Ping, JIANG Dongfu, et al. The natural microflora of Xuanwei ham and the no-mouldy ham production[J]. Journal of Food Engineering, 2006, 77(1): 103-111. DOI:10.1016/j.jfoodeng.2005.06.047.
[33] WANG Xinhui, ZHANG Yalin, REN Hongyang. Effects of grape seed extract on lipid oxidation, biogenic amine formation and microbiological quality in Chinese traditional smoke‐cured bacon during storage[J]. Journal of Food Safety, 2018, 38(2): e12426. DOI:10.1111/jfs.12426.
[34] PAPAMANOLI E, KOTZEKIDOU P, TZANETAKIS N, et al. Characterization of Micrococcaceae isolated from dry fermented sausage[J]. Food Microbiology, 2002, 19(5): 441-449. DOI:10.1006/fmic.2002.0503.
收稿日期:2020-12-15
基金項目:中央高校基本科研業務費專項資金項目(JZ2020HGQA0150);

