基于圖像分析的玉米抗擬輪枝鐮孢穗腐病的QTL定位
聞競,沈彥岐,王梓鈺,李世界,莫藍月,雷宇豪,張艷,韓四平
基于圖像分析的玉米抗擬輪枝鐮孢穗腐病的QTL定位
聞競,沈彥岐,王梓鈺,李世界,莫藍月,雷宇豪,張艷,韓四平
吉林省農業科學院農業生物技術研究所,長春 130033
【】玉米穗腐病是一種在全世界廣泛發生且危害嚴重的真菌性病害,其中,擬輪枝鐮孢引起的穗腐病(ear rot,FER)在中國發生最為普遍。通過圖像分析方法進行FER抗病QTL定位,并對前期通過病害評級方法定位的FER抗病QTL進行驗證,探索一種新的玉米穗腐病的病害鑒定方法,為玉米穗腐病的遺傳改良提供依據。利用高感FER的自交系(ZW18)分別與3個高抗自交系(承351、丹598和吉V203)構建F2群體(F2-C、F2-D和F2-J)和相應的F2﹕3家系,通過圖像分析的方法獲得每個F2﹕3家系的果穗病斑百分比,進而定位玉米FER抗病QTL。3個群體共定位到18個FER抗病QTL,其中,分別位于2.02—2.03 bins、4.06—4.07 bins和8.06 bin上的3個QTL(、和)可解釋的表型變異率分別高達21.80%、25.80%和27.40%。F2-J群體的與F2-C群體的和F2-D群體的在第1染色體均有重疊區間,可解釋的表型變異率達到16.50%。來自F2-D群體的與F2-J群體的在第8染色體8.05 bin有重疊區間,且抗性基因均來源于抗性親本。F2-C群體的與F2-J群體的在第4染色體4.06—4.07 bins有重疊區間。另外,與之前通過病害評級方法定位的結果相比,、和在1.06—1.07 bins與評級方法定位的抗病位點重合,和5在4.06—4.07 bins與評級方法定位的抗病位點和重疊,與在2.04 bin的定位區間完全重合,與在S2重復中定位到9.03—9.05 bins的重疊區間,且來源于相同的抗源。定位到18個FER抗性位點,其中,位于1.04—1.07 bins、4.06—4.07 bins和8.05 bin上的抗病位點在不同群體中均可以被檢測到,位于2.04 bin和9.03—9.05 bins上的抗病位點用不同的檢測方法可以被檢測到,表明在這些區間可能存在FER的抗性位點。QTL的定位區間在不同群體中的重疊性在一定程度上驗證了定位區間的真實性,不同方法之間定位到重疊區間,說明利用圖像分析方法定位FER抗病QTL具有一定的準確性。
玉米;擬輪枝鐮孢穗腐病;抗性;QTL
0 引言
【研究意義】玉米是糧飼兼用作物,又是重要的工業原料,在中國糧食安全保障體系中占有舉足輕重的地位。病蟲害是中國玉米生產的主要限制因子,每年造成的產量損失占總產的10%以上,嚴重威脅到中國的糧食安全。玉米穗腐病是一種在全世界廣泛發生且危害嚴重的真菌性病害[1-4],也是中國各個玉米產區的主要病害。一般年份玉米穗腐病的發病率為10%—20%,嚴重年份可達30%—40%,感病品種的發病率高達50%左右[5],而且其發病程度呈逐年加重的趨勢。玉米穗腐病不僅會引起果穗腐爛而導致直接減產,而且有些病原菌還會在被侵染的籽粒中產生大量的有害毒素,嚴重影響畜牧業生產,進而威脅人類的身體健康和生命安全[6-7]。因此,定位玉米抗穗腐病主效QTL、發展分子標記,為抗病分子育種服務,無論在抗病理論探索還是育種實踐應用上都具有重要的意義。【前人研究進展】玉米穗腐病的致病菌種類繁多,國內外報道有40多種,這些病原菌可以單獨或者復合侵染引起發病[8-9]。目前已報道的玉米穗腐病的主要致病菌包括鐮孢菌(spp.)、木霉菌(spp.)、青霉菌(spp.)、曲霉菌(spp.)、平臍蠕孢菌(spp.)和鏈格孢菌(spp.)等[10-12]。不同國家及地區玉米穗腐病病原菌種類及優勢種類差異明顯,國內外報道不盡一致,而且不同區域環境下,穗腐病的主要致病菌存在差異[13-15]。已有研究結果表明,國內玉米穗腐病的病原菌主要以擬輪枝鐮孢()和禾谷鐮孢()為優勢種,分別引起擬輪枝鐮孢穗腐病(ear rot,FER)和禾谷鐮孢穗腐病(ear rot,GER),其中,FER在中國發生最為普遍[16-21]。目前,國內外已利用多種方法開展對FER抗病QTL的定位研究[22-30]。有研究利用重組自交系進行QTL定位,定位到的抗性QTL位于第3、4、5、6染色體[31]。也有研究結果顯示在3.04 bin可能含有多個抗病基因[32]。通過全基因組關聯分析,在第4染色體上定位到了FER的抗病QTL,表型變異率達18%[33]。也有研究通過全基因組關聯分析與基因組預測相結合的方法,將FER的抗病QTL定位在第3、7、9、10染色體上[34]。【本研究切入點】現有的針對玉米穗腐病的防治措施(如噴施殺菌劑、翻耕、間混作等化學防治和耕作措施)效果均不理想[35]。另外,玉米籽粒直收已成為玉米育種的方向,而籽粒直收過程中,無法對發生穗腐病的果穗進行剔除。機械化收獲的不斷推進,將會對品種穗腐病抗病方面的要求變得更為嚴苛。因此,培育和推廣抗病品種成為目前亟需解決的問題。然而,目前還沒有可以直接用于分子育種的抗穗腐病遺傳穩定的QTL或者基因被報道。【擬解決的關鍵問題】本研究通過借助圖像處理軟件計算果穗病斑面積的百分率獲得群體的病害數據進行玉米FER抗病QTL的定位,探索一種新的玉米穗腐病抗性鑒定方法,同時對前期通過病害評級方法定位的FER抗病QTL進行驗證。
1 材料與方法
1.1 試驗材料
植物材料:高感FER的自交系ZW18分別與高抗FER的玉米自交系承351、丹598和吉V203構建3個F2群體(F2-C、F2-D和F2-J)和F2﹕3家系,F2群體大小分別為124、200和176(圖1)。前期已分別構建3個F2群體的遺傳連鎖圖譜,總遺傳距離分別為2 754.2、1 862和2 086.1 cM,平均每兩個相鄰標記之間的遺傳距離分別為5.91、4.22和3.75 cM。在構建遺傳連鎖圖譜時將感病親本的基因型設為“1”,抗病親本的基因型設為“2”。F2群體的F2﹕3家系用于玉米FER的病害鑒定。
病原菌:擬輪枝鐮孢()由中國農業大學徐明良教授提供。
1.2 試驗方法
1.2.1 田間設計 將親本自交系和3個F2群體的F2﹕3家系分別種植于吉林省公主嶺地區和松原地區2個試驗點,每個點設置2個重復。每份材料的每個重復種植1行,行長5 m,行距0.6 m,株距0.25 m,每行種植21株。
1.2.2 病原菌的培養 將擬輪枝鐮孢菌接種到PDA培養基平板上,在培養箱中28℃培養約1周,待菌絲長滿平板,4℃保存待用。取40 g綠豆加入適量水煮沸20 min,過濾后加入20 g蔗糖,1 g KH2PO4,定容至1 L,121℃高壓蒸汽滅菌30 min,冷卻后4℃保存備用。將1/2板已培養好的帶菌平板中的培養基切成0.25 cm2的小塊放入500 mL 5%綠豆煎汁液體培養基中,28℃ 150 r/min震蕩培養2 d,過濾菌絲,收集孢子置于4℃保存備用。在接種前一天,用無菌蒸餾水配制成濃度為5×106個/mL的孢子懸浮液。在孢子懸浮液中加入表面活性劑吐溫-80,吐溫-80在孢子懸浮液中濃度為2 μl·mL-1,混勻。
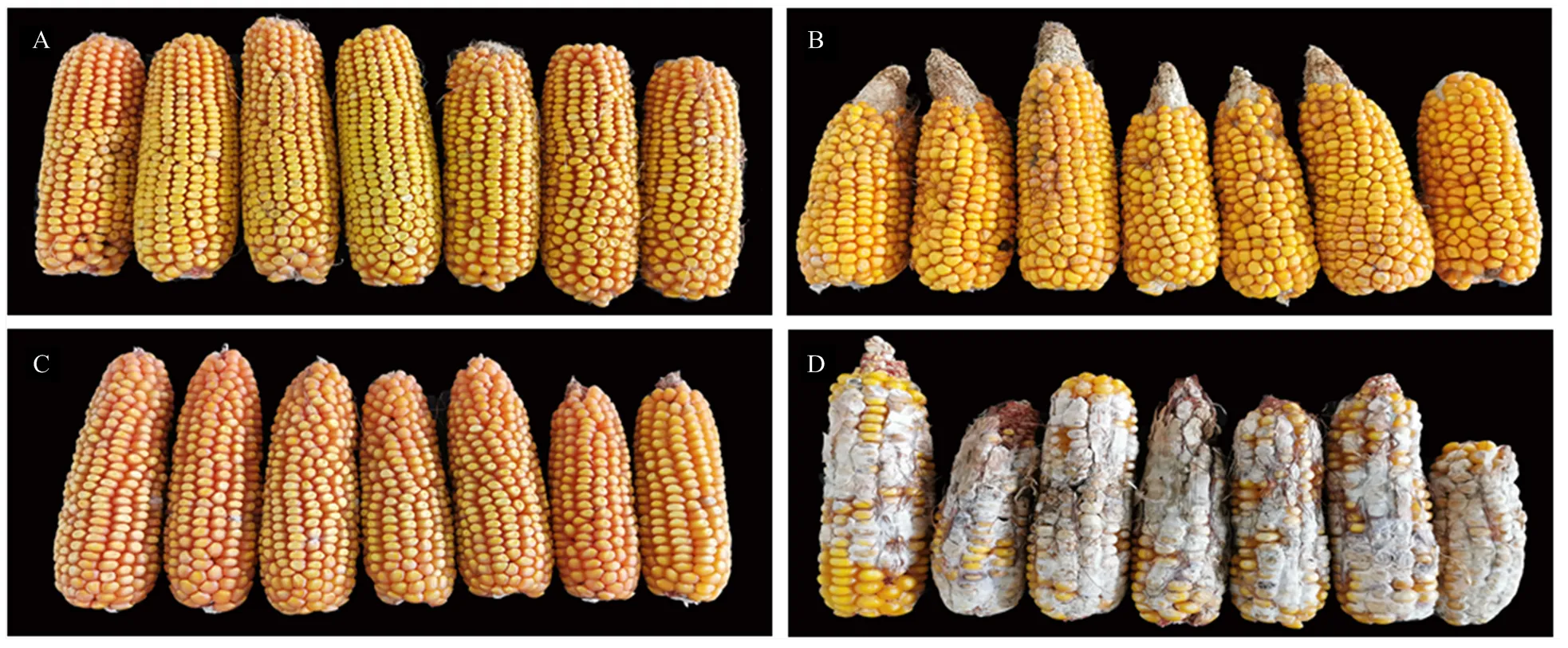
A:承351;B:丹598;C:吉V203;D:ZW18 A: Cheng351; B: Dan598; C: JiV203; D: ZW18
1.2.3 人工接種 根據往年經驗,接種的最佳時期為玉米吐絲后12 d,用注射器在果穗中部注射2 mL孢子懸浮液,接菌后用手指輕捏傷口處,防止菌液外流。
1.2.4 病害鑒定 在玉米成熟期(吉林省公主嶺和松原9月下旬)進行田間發病調查。將各接種處理的玉米果穗剝去苞葉,并對每個重復中的每個F2﹕3家系的所有果穗發病最嚴重的部位拍照,拍照設置比例尺,保證每張圖片中玉米籽粒的大小一致,然后利用Photoshop軟件處理圖片。首先利用磁性套索工具將玉米果穗從圖片中分離,利用魔棒工具選中病斑位置,并用綠色填充;隨后選擇反向,選中果穗的未發病部位,將其填充為紅色。分別選中果穗發病和未發病的部位,點擊“分析”,選擇“數據測量”,記錄測量結果中各部分的面積大小,計算每個重復中每個F2﹕3家系全部玉米果穗的發病面積占總面積的百分比(圖2)。以每個F2﹕3家系中所有個體的病害鑒定值的均值作為對應的F2個體的病害鑒定值。
1.2.5 方差分析方法 利用R軟件進行表型的方差分析,采用=μ+α+β+()+εGLR的模型。其中:α表示基因型的效應,β表示環境的效應,()表示基因型和環境的互作效應,表示殘差效應。
1.2.6 QTL初定位方法 采用F2群體進行玉米抗穗腐病QTL的初定位。表型數據采用4個重復(G1、G2、S1、S2)、每個地點2個重復的平均值(G、S)和最佳線性無偏估計值(best linear unbiased estimation,BLUE)的數據進行計算。結合F2群體的遺傳連鎖數據和表型數據利用WinQTLCart軟件[36]采用復合區間作圖法(composite interval mapping,CIM)[37]進行玉米抗穗腐病QTL的掃描,LOD值的閾值采用排列測驗的方法由軟件自動產生,其中排列測驗的次數為300,顯著水平為0.05。
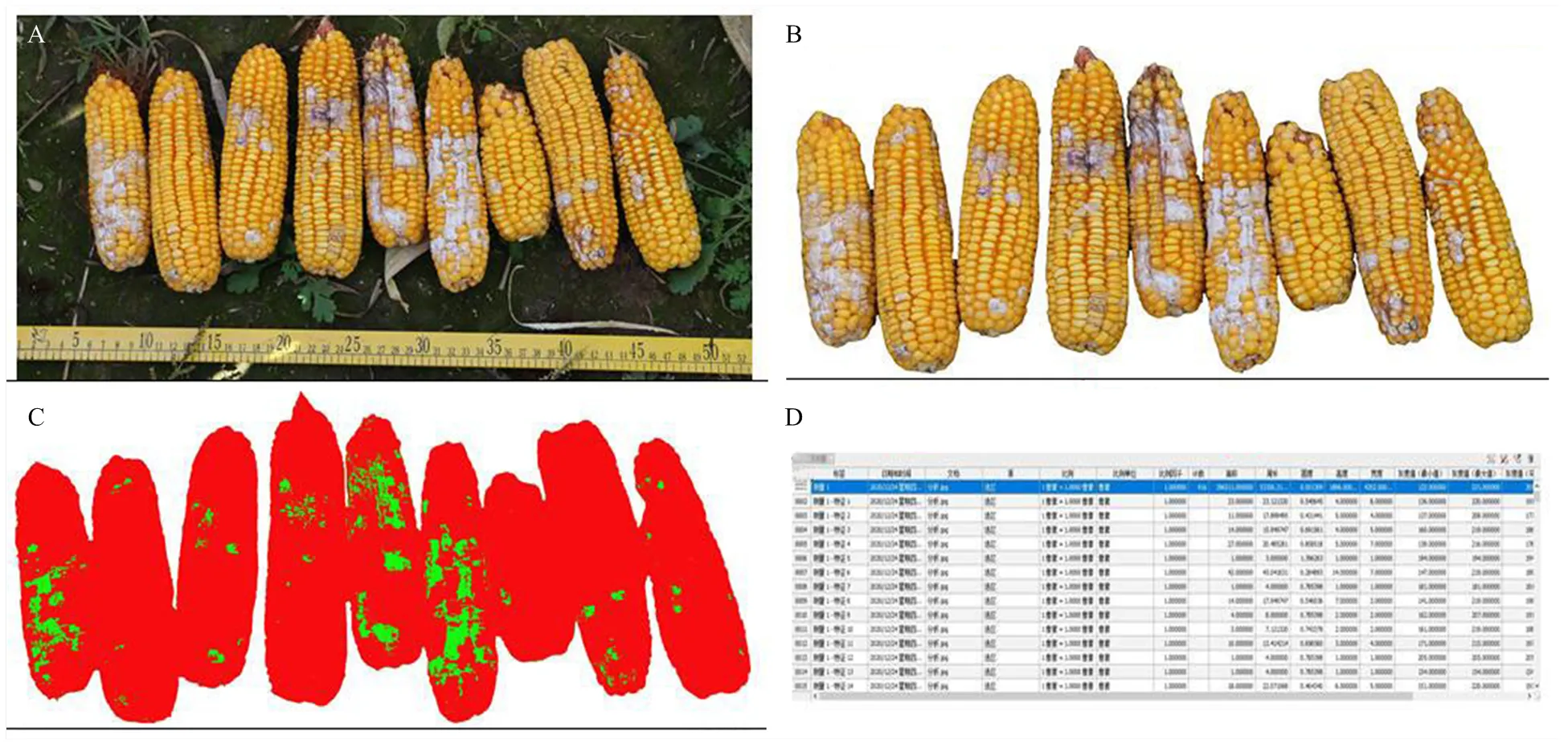
A:原圖;B:去除背景的玉米果穗圖片;C:用不同顏色標注病斑和未發病部位的圖片;D:數據分析
2 結果
2.1 表型分析
對定位群體的材料進行玉米穗腐病抗性的鑒定。人工接種擬輪枝鐮孢菌后,在公主嶺的第1個重復(G1)中親本自交系承351、丹598、吉V203和ZW18的發病面積百分比分別為0、0、0和49.22%;在公主嶺的第2個重復(G2)中親本自交系承351、丹598、吉V203和ZW18的發病面積百分比分別為0、0、0和82.26%;在松原的第1個重復(S1)中親本自交系承351、丹598、吉V203和ZW18的發病面積百分比分別為0、0、1.30%和63.85%,在松原的第2個重復(S2)中親本自交系承351、丹598、吉V203和ZW18的發病面積百分比分別為0、2.40%、0和55.38%。在F2群體中,對玉米抗穗腐病的抗性沒有呈現雙峰的分布(圖3),說明玉米對穗腐病的抗性不是由單基因控制的,而是由數量抗性位點控制的。
2.2 玉米穗腐病抗性的方差分析
對由抗病親本承351、丹598和吉V203分別與感病親本ZW18構建的F2群體(F2-C、F2-D和F2-J)的不同重復進行方差分析,結果表明,每個群體中不同重復間的發病水平基本相當。抗病親本(承351、丹598和吉V203)在不同的地點不同重復中均表現一定的抗性,感病親本ZW18均表現明顯感病。根據F2-C、F2-D和F2-J群體在不同重復的表型數據估算的FER抗性遺傳力分別為0.63、0.29和0.56(表1)。方差分析結果表明,在F2-C群體中,不同基因型之間和不同地點之間的FER抗性存在顯著性差異,另外,基因型與環境之間的互作效應顯著。在F2-D群體和F2-J群體中,不同基因型之間和不同地點之間的FER抗性存在顯著性差異(表2)。另外,3個F2群體的殘差的偏度分析結果表明殘差符合正態分布(表2和圖4)。
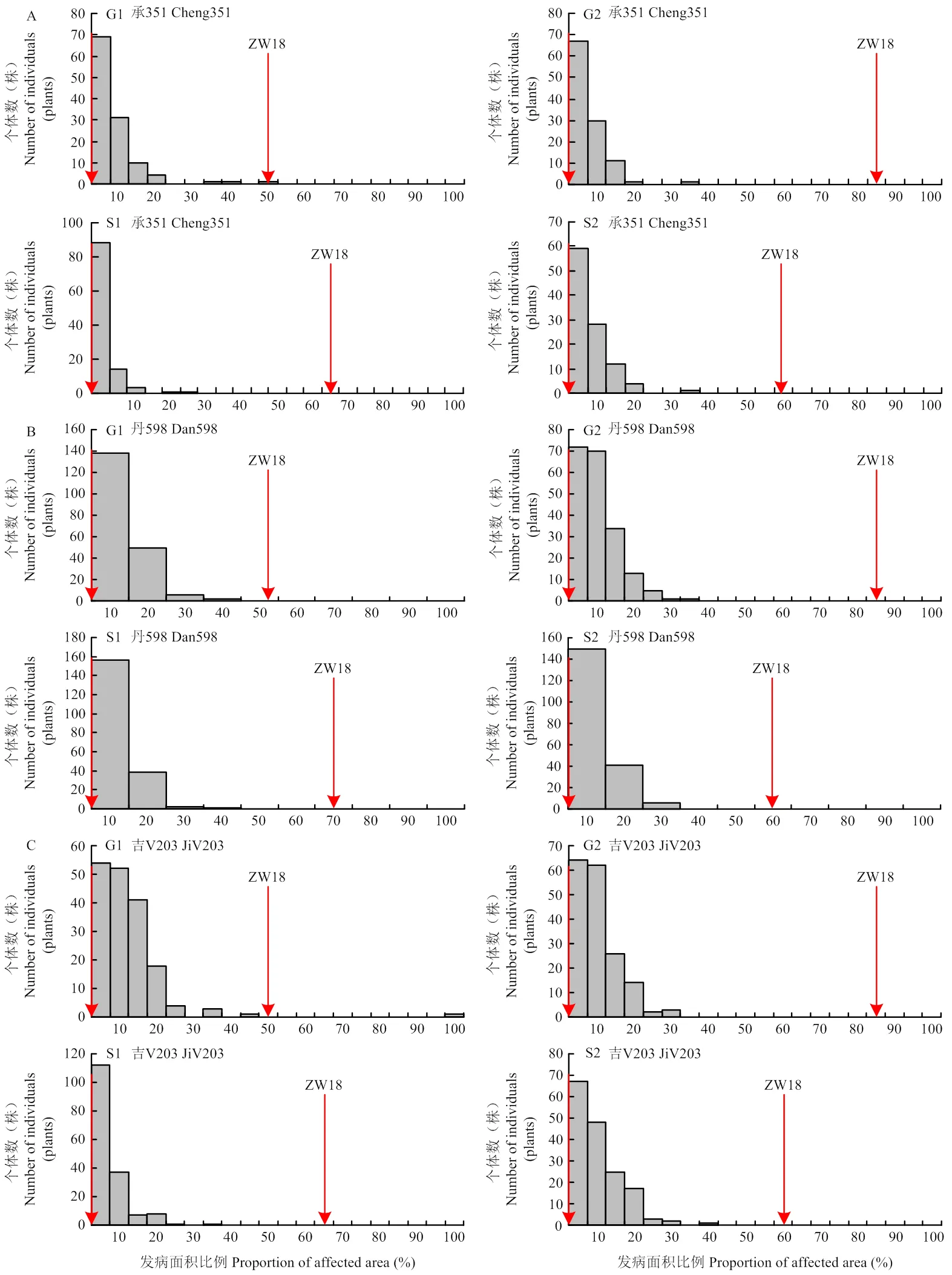
A:F2-C;B:F2-D;C:F2-J。G1:公主嶺第1個重復;G2:公主嶺第2個重復;S1:松原第1個重復;S2:松原第2個重復。下同
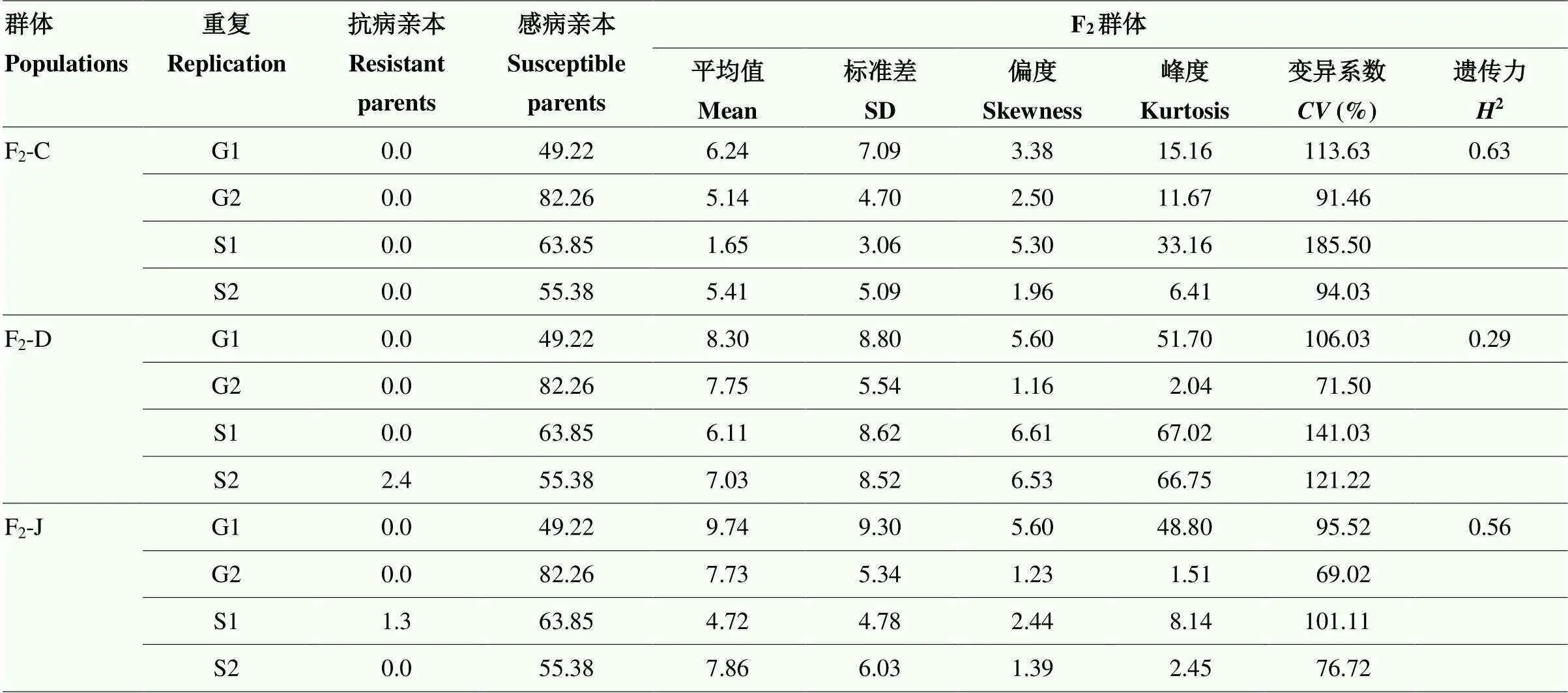
表1 親本及F2群體的發病面積百分比分析結果
G1:公主嶺第1個重復;G2:公主嶺第2個重復;S1:松原第1個重復;S2:松原第2個重復。下同
G1: 1streplicate in Gongzhuling; G2: 2ndreplicate in Gongzhuling; S1: 1streplicate in Songyuan; S2: 2ndreplicate in Songyuan. The same as below
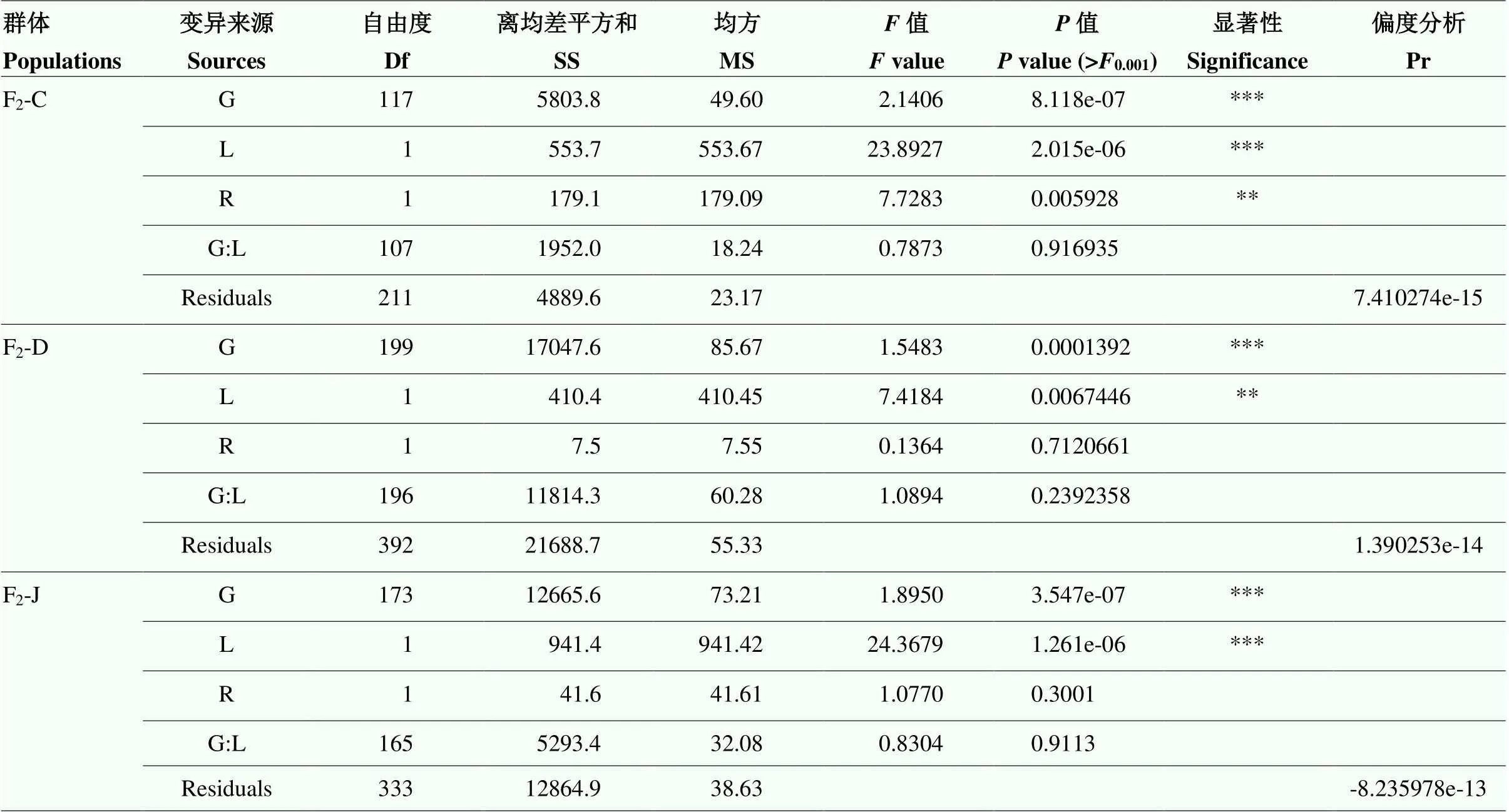
表2 人工接種擬輪枝鐮孢菌后玉米穗腐病抗性的方差分析
G:基因型;L:地點;R:重復;G:L:基因型與環境間的互作;Residuals:殘差效應
G: Genotype; L: Location; R: Replicate; G:L: Interaction between genotype and environment; Residuals: Residual effect
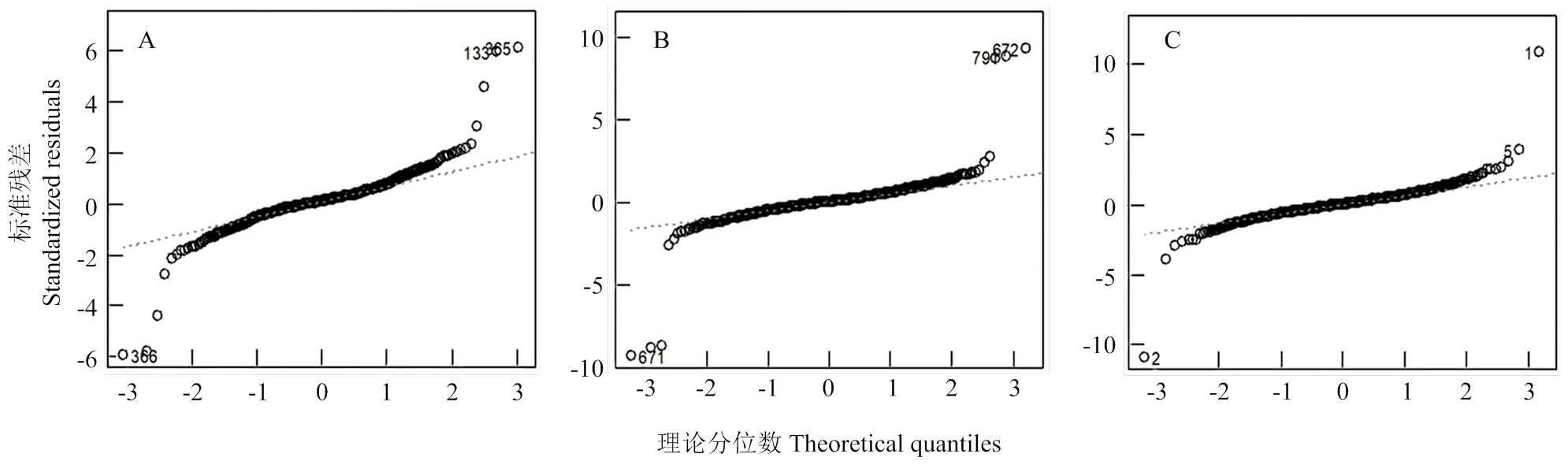
A:F2-C;B:F2-D;C:F2-J
2.3 玉米FER抗病QTL的定位
利用3個F2群體(F2-C、F2-D和F2-J)對玉米FER抗病QTL進行了定位,WinQTLCart經過300次排列測驗之后生成相應的LOD閾值,共定位到18個抗性QTL。利用F2-C群體共定位到5個抗性QTL,分別位于1.04—1.06 bins、2.02—2.03 bins,4.06—4.07 bins、8.06 bin和8.07—8.08 bins。其中、、和的抗性等位基因來源于抗病親本承351,的表型變異率最大,為26.00%—27.40%;的抗性等位基因來源于感病親本ZW18,解釋了7.1%的表型變異率(圖5-A和表3)。利用F2-D群體定位到5個抗性QTL,分別位于1.07 bin、2.04 bin、3.03—3.04 bins、8.05—8.06 bins和10.06 bin。其中、、和的抗性等位基因來源于抗病親本丹598,單個QTL的表型變異率為7.94%—12.32%;的抗性等位基因來源于感病親本ZW18,表型變異率為10.82%(圖5-B和表3)。利用F2-J群體定位到8個抗性QTL,分別位于1.06—1.07 bins、3.04—3.05 bins、3.06—3.07 bins、4.01—4.03 bins、4.05—4.07 bins、8.03—8.05 bins、9.03—9.05 bins和9.06—9.07 bins。其中、、和的抗性等位基因來源于抗病親本吉V203,的表型變異率最大,達到17.96%;、、和的抗性等位基因來源于感病親本ZW18,表型變異率最大的為,達到13.46%(圖5-C和表3)。另外,已定位到的18個QTL由加性效應和顯性效應共同遺傳(表3)。
3個F2群體在第1和第8染色體均定位到抗性QTL。其中,F2-C群體的與F2-J群體的在1.06 bin(e1178476—e1188929)有重疊區間,F2-D群體的與F2-J群體的在1.07 bin(e1197737—e1200620)區間重疊。這3個QTL除的抗性等位基因來源于感病親本ZW18外,與的抗性等位基因均來源于抗性親本。F2-C群體的在BLUE中被定位,F2-J群體的在S2重復中被定位。與和均有重疊區間,可解釋的表型變異率達到16.50%,說明可能是FER的抗病“熱點”。另外,3個F2群體在第8染色體上均有定位區間。其中,來自F2-D群體的與F2-J群體的在8.05 bin(e8130926—e8144487)有重疊區間。在BLUE中被定位,表型變異率為10.85%;在S重復中被定位,表型變異率為8.15%。二者的抗性等位基因均來源于抗性親本。此外,F2-C群體的與F2-J群體的在第4染色體4.06—4.07 bins(e4168742—e4171651)有重疊區間。在G1和BLUE中被定位,可解釋的表型變異率分別為25.80%和19.60%;在G2和S2中被定位,可解釋的表型變異率分別為17.96%和8.16%。
前期利用病害評級的方法進行抗病QTL定位,共挖掘到20個抗病位點,其中,位于1.03 bin、1.04—1.07 bins、2.04—2.08 bins、4.05—4.08 bins、6.03—6.06 bins和7.01—7.02 bins上的6個位點在不同的群體中都能檢測到,說明這6個位點可能是擬輪枝鐮孢穗腐病的抗病“熱點”[38]。利用圖像分析的方法共定位到18個抗病QTL,其中,、和在1.06—1.07 bins與評級方法定位的抗病位點重合(表4)。在S1和S2同時被定位到,在S2中的表型變異率為16.73%,同樣在S2群體中定位到,表型變異率為16.50%,二者的表型變異率極為接近,且抗性等位基因均來源于抗性親本吉V203,均受加性效應的影響。此外,利用圖像分析的方法定位到的和5在4.06—4.07 bins與評級方法定位的抗病位點和重疊[38](表4)。和5在G1、G2、S2和BLUE重復中被定位,可解釋的表型變異率為8.16%—25.8%;和在G1、G2、G和BLUE重復中被定位,可解釋的表型變異率為12.02%—21.81%。2種方法定位到的位點表型變異率差異不大,抗性等位基因均來源于抗性親本承351和吉V203,均受加性效應的影響。與在2.04 bin的定位區間完全重合,的表型變異率為7.94%,的表型變異率為7.02%,二者的表型變異率差異不大,且均在BLUE重復中定位到,抗性等位基因均來源于抗性親本丹598,受加性效應的影響[38](表4)。另外,與也同時在S2重復中定位到9.03—9.05 bins的重疊區間,二者的表型變異率分別為3.61%和4.10%,抗性等位基因均來源于感病親本ZW18[38](表4)。利用圖像分析的方法定位到的抗性位點與病害評級法定位的QTL有重疊區間,說明該方法較為可靠,定位結果的準確性較高。、、、和定位到的抗性QTL用不同的方法均可定位到,說明這5個位點可能是擬輪枝鐮孢穗腐病的抗病“熱點”。
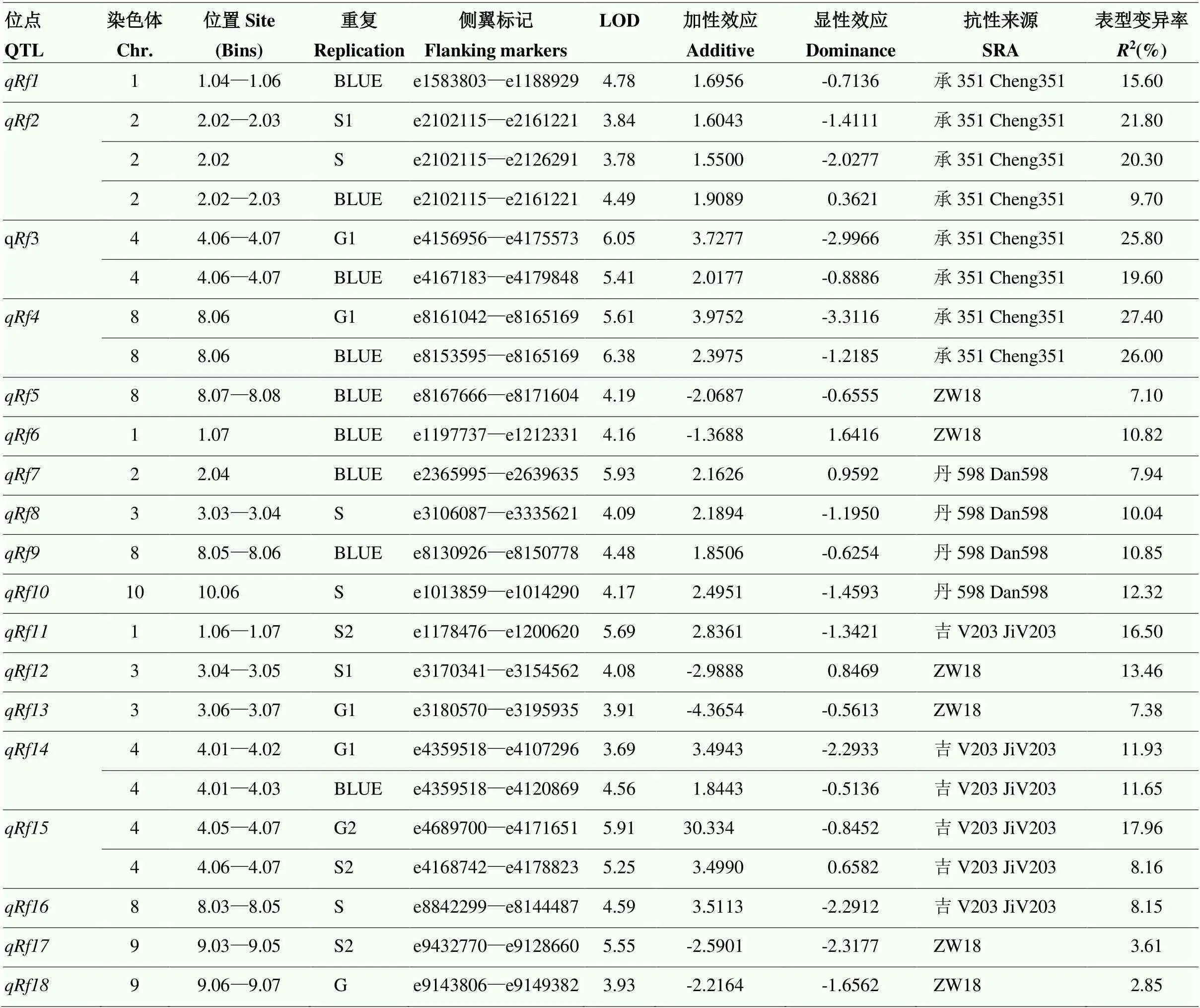
表3 玉米FER抗病QTLs及其參數
G:G1和G2的平均值;S:S1和S2的平均值;BLUE:最佳線性無偏估計值。下同
G: Average of G1 and G2; S: Average of S1 and S2; BLUE: Best linear unbiased estimation. The same as below
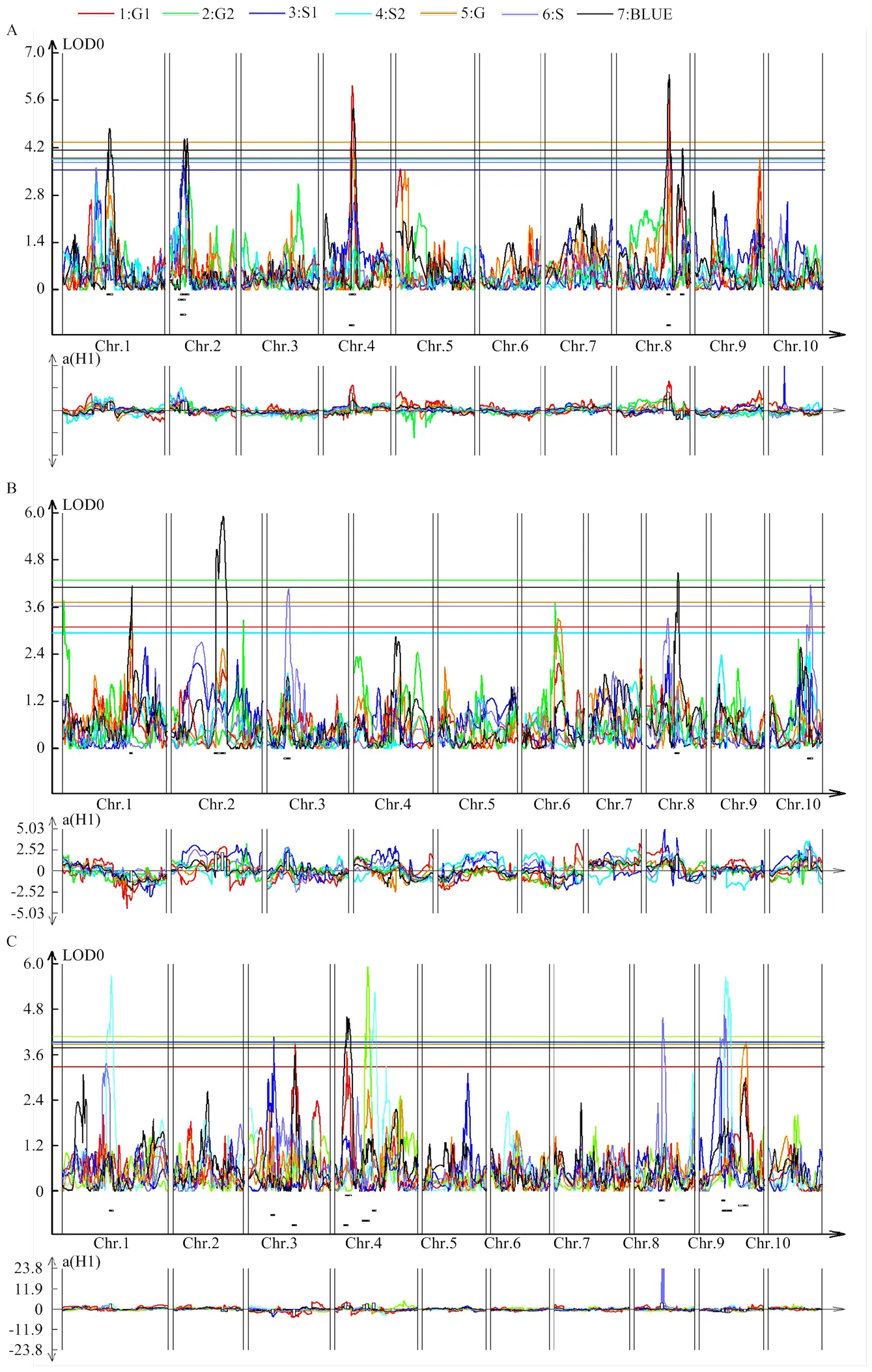
A:F2-C;B:F2-D;C:F2-J。G:G1和G2的平均值;S:S1和S2的平均值;BLUE:最佳線性無偏估計值
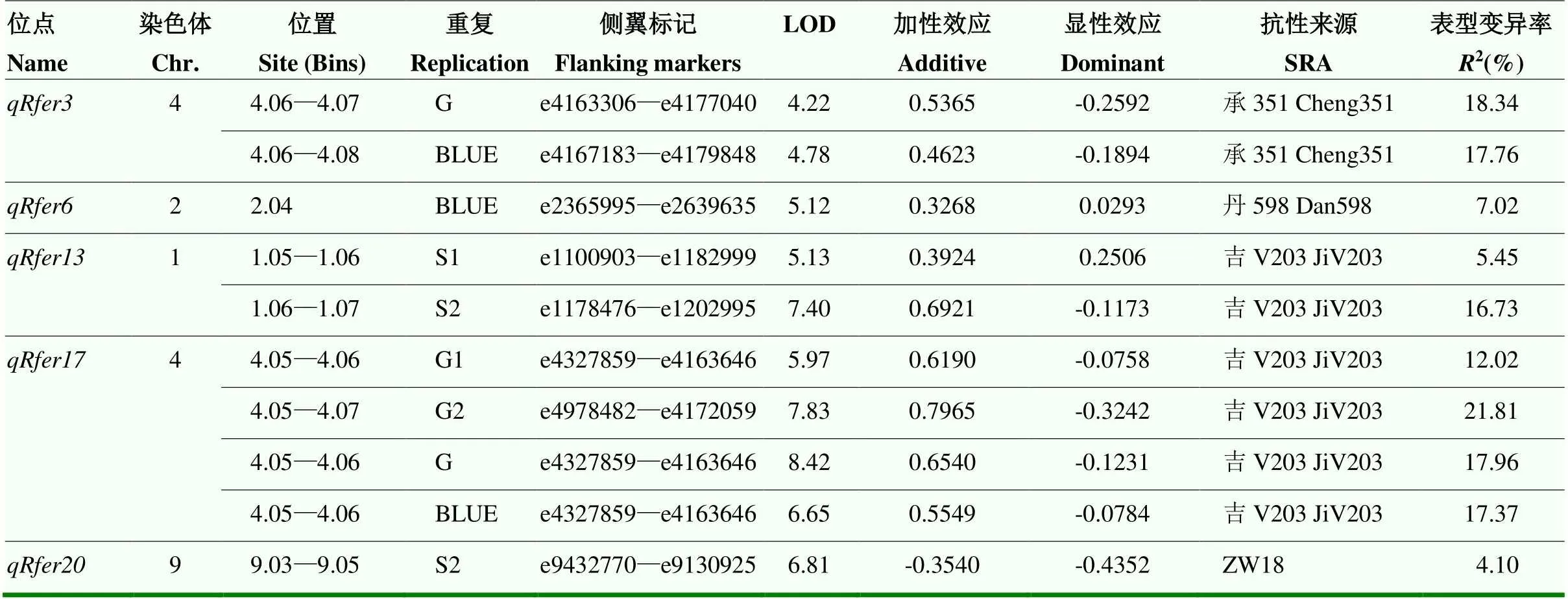
表4 利用評級法分析的玉米FER抗病QTL及其參數
3 討論
在以往的玉米穗腐病病害調查中,大多采用的是病情分級的方式(《玉米抗病蟲性鑒定技術規范,NY/T 1248.8—2016》)。該種鑒定方法是根據病斑所占的百分比進行人為評級,存在一定的主觀因素,而且同一評級的發病面積占果穗總面積的比例的范圍較大[39-40]。以9級為例,發病面積占果穗總面積的51%—100%均定為9級,同一等級的果穗發病面積百分比可能會有較大差異。因此,單從評級水平無法精確評價果穗的真實發病情況。為了探索一種新的玉米FER的病害鑒定方法,本研究采用對發病果穗的圖片通過軟件分析并計算出發病面積所占的百分比的方法獲取病害數據。該種方法雖然工作量相對較大,耗費時間較長,操作較為繁瑣,但可以精確計算不同果穗的發病面積所占的百分比。
本研究共定位到18個FER抗性位點,其中位于1.04—1.07 bins、4.06—4.07 bins和8.05 bin上的抗病位點在不同的群體中均可以檢測到,位于2.04 bin和9.03—9.05 bins上的抗病位點用不同的檢測方法可以檢測到,說明在這些區間可能會存在FER的抗性位點。目前,國內外已有一些關于玉米穗腐病有效QTL位點的研究報道。有研究以BT-1和N6為親本構建了250個重組自交家系材料,定位到的抗性QTL位于4.06 bin,本研究也得到了相同的結果[31]。此外,有研究結果表明在3.04—3.05 bins、9.04 bin、9.05 bin、9.06—9.07 bins均有QTL與FER抗性顯著相關,這些區域與本研究中、和的定位區間重疊[22-28,33]。但也有研究結果顯示定位到的FER抗性QTL在1.01 bin、5.05 bin、5.08 bin、5.09 bin、7.02—7.04 bins、9.00—9.01 bins和10.03—10.04 bins,本研究并未找到與之重疊的QTL[23-25,29-30,32-34,41]。與前期利用病害評級的方法進行的抗病QTL的定位結果相比,有多個QTL的定位區間重疊,、、、和分別與之前通過病害評級的方法定位到的、、、和20的定位區間重疊,而且來源于相同的抗源[38]。以上QTL的定位區間的重疊性在一定程度上驗證了其真實性,但是還需要通過精細定位和遺傳效應分析進一步驗證以上QTL對玉米FER的抗病功能。在2種定位方法中有未能同時定位到的QTL,可能是由于2種方法在發病情況的表型鑒定方面存在差異,從而造成定位偏差。在本研究中定位到的18個QTL中,來源于抗病親本承351的、和可解釋的表型變異率分別可高達21.80%、25.80%和27.40%,而且可在不同處理的表型數據的定位中檢測到。后期對、和的精細定位、遺傳效應分析以及功能基因的克隆將對玉米FER的抗病遺傳改良提供重要的遺傳資源和理論參考。然而,在本研究中并未定位到可以在所有不同處理的表型數據(G1、G2、S1、S2、G、S和BLUE)中均能定位到的QTL。根據前期報道,玉米穗腐病的遺傳機制非常復雜[42],而且發病對環境條件非常敏感[43-44],所以很難在不同的環境條件下獲得較為一致的群體病害數據,從而影響對抗病QTL的定位結果。因此,對于玉米穗腐病這樣的復雜的數量性狀,可通過與不同的方法的結合進行功能位點挖掘,比如全基因組關聯分析,轉錄組分析、代謝組分析等。
4 結論
共定位到18個FER抗病QTL。其中,不同的群體均存在相同的定位區間,與、有重疊區間,與在8.05bin有重疊區間,與在4.06—4.07bins重疊。另外,、、、和分別與之前通過病害評級的方法定位到的、、、和20的定位區間重疊,而且來源于相同的抗源。QTL的定位區間在不同的群體中可以檢測到,利用不同的定位方法也可以檢測到,說明這些重疊區間可能存在FER的抗性位點。不同群體中和不同方法中的重疊性在一定程度上驗證了定位區間的真實性,也說明利用圖像分析的方法進行FER抗病QTL定位的可行性。
[1] ULLSTRUP A J. An undescribed ear rot of corn caused by. Phytopathology, 1946, 36: 201-212.
[2] BEZUIDENHOUT H, MAXASAS W F O. Botryosphaeria zeae: the cause of grey ear rot of maize () in South Africa. Phytophylaetica, 1978, 10(1): 21-24.
[3] KUMAR V, SHETTY H S. A new ear and kernel rot of maize caused bypers. ex Fries. Current Science, 1982, 5(12): 620-621.
[4] 張艷, 譚靜. 玉米穗粒腐病的研究進展. 現代農業科技, 2014, 21(1): 121-125.
ZHANG Y, TAN J. Research progress on ear rot in maize. Modern Agricultural Science and Technology, 2014, 21(1): 121-122. (in Chinese)
[5] 任金平. 玉米穗腐病研究進展. 吉林農業科學, 1993(3): 39-43.
REN J P. Progress in researching of ear rot of maize. Jilin Agricultural Sciences, 1993(3): 39-43. (in Chinese)
[6] 胡南, 章紅. 吉林省玉米穗腐病病原真菌中鐮刀菌毒素的研究. 玉米科學, 1997, 5(2): 66-68.
HU N, ZHANG H. A study on production of threeof corn ear rot pathogenic fungi in Jilin province. Journal of Maize Sciences, 1997, 5(2): 66-68. (in Chinese)
[7] WANG J H, NDOYE M, ZHANG J B, LI H P, LIAO Y C. Population structure and genetic diversity of thespecies complex. Toxins, 2011, 3(8): 1020-1037.
[8] 王曉鳴, 石潔, 晉齊鳴, 李曉, 孫世賢. 玉米病蟲害田間手冊. 北京: 中國農業科學技術出版社, 2010: 55-61.
WANG X M, SHI J, JIN Q M, LI X, SUN S X.. Beijing:China Agricultural Science and Technology Press, 2010: 55-61. (in Chinese)
[9] 徐書法, 陳捷, 高增貴, 鄒慶道, 紀明山, 劉海南. 中國玉米莖基腐病和穗腐病研究進展. 植物病理學報, 2006, 36(3): 193-203.
XU S F, CHEN J, GAO Z G, ZOU Q D, JI M S, LIU H N. Maize stalk rot and ear rot in China. Acta Phytopathologica Sinica,2006, 36(3): 193-203. (in Chinese)
[10] 秦子惠, 任旭, 江凱, 武小菲, 楊知還, 王曉鳴. 我國玉米穗腐病致病鐮孢種群及禾谷鐮孢復合種的鑒定. 植物保護學報, 2014, 41(5): 589-596.
QIN Z H, REN X, JIANG K, WU X F, YANG Z H, WANG X M. Identification ofspecies andspecies complex causing maize ear rot in China. Journal of Plant Protection, 2014, 41(5): 589-596. (in Chinese)
[11] 孫華, 張海劍, 郭寧, 石潔, 陳丹, 馬紅霞. 黃淮海夏玉米主產區穗腐病病原菌的分離鑒定. 植物保護學報, 2017, 44(5): 796-802.
SUN H, ZHANG H J, GUO N, SHI J, CHEN D, MA H X. Isolation and identification of pathogens causing maize ear rot in Huang-Huai- Hai summer corn region. Journal of Plant Protection, 2017, 44(5): 796-802. (in Chinese)
[12] HAAFSMA A W, LIMAY-RIOS V, TAMBURIC-ILINCIC L. Mycotoxins andspecies associated with maize ear rot in Ontario, Canada. Cereal Research Communications, 2008, 36: 525-527.
[13] 孫華, 張海劍, 馬紅霞, 石潔, 郭寧, 陳丹, 李坡. 春玉米區穗腐病病原菌組成、分布及禾谷鐮孢復合種的鑒定. 物病理學報, 2018, 48(1): 8-15.
SUN H, ZHANG H J, MA H X, SHI J, GUO N, CHEN D, LI P. Composition and distribution of pathogens causing ear rot in spring maize region and identification ofgraminearum species complex. Acta Phytopathologica Sinica, 2018, 48(1): 8-15. (in Chinese)
[14] ATANASOVA V, PONS S, PINSON L, PICOT A, RICHARD F. Chlorogenic acid and maize ear rot resistance: a dynamic study investigatingdevelopment, deoxynivalenol production and phenolic acid accumulation. Molecular plant-microbe interactions, 2012, 25(12): 1605-1616.
[15] 張婷, 孫曉東, 呂國忠. 我國東北地區玉米穗腐鐮孢菌的種類及其分離頻率. 菌物研究, 2011, 9(1): 9-14.
ZHANG T, SUN X D, Lü G Z.species and its isolation frequency from rot ears of maize in northeast China. Journal of Fungal Research, 2011, 9(1): 9-14. (in Chinese)
[16] 潘惠康, 張蘭新. 玉米對穗粒腐病菌的抗病性. 華北農學報, 1987, 2(3): 86-89.
PAN H K, ZHANG L X. Disease resistance of corn to ear and kernel rot. Acta Agriculturae Boreali-Sinica, 1987, 2(3): 86-89. (in Chinese)
[17] 劉春元, 李洪連, 吳建宇, 劉建華. 穗粒腐病菌對玉米幼苗的致病性研究. 河南農業科學, 2005, 11: 58-62.
LIU C Y, LI H L, WU J Y, LIU J H. Studies on pathogenicity of pathogen of ear and seed rot in maize hybrids to maize seedling blight. Journal of Henan Agricultural Sciences, 2005, 11: 58-62. (in Chinese)
[18] 陳廣泉. 河西走廊玉米穗粒腐病侵染規律及發病因子研究. 玉米科學, 2006, 14(1): 158-160.
CHEN G Q. Study on infection law and disease factor of corn spike kernel rotten in Hexi corridor. Journal of Maize Sciences, 2006, 14(1): 158-160. (in Chinese)
[19] 劉振庫, 賈嬌, 蘇前富, 孟玲敏, 晉齊鳴. 齊齊哈爾玉米穗腐病病原菌的鑒定和致病性測定. 吉林農業科學, 2014, 39(6): 28-30.
LIU Z K, JIA J, SU Q F, MENG L M, JIN Q M. Identification of pathogen and pathogenicity of maize ear rot in Qiqihaer. Journal of Northeast Agricultural Sciences, 2014, 39(6): 28-30. (in Chinese)
[20] ZHOU D N, WANG X M, CHEN G K, SUN S L, YANG Y, ZHU Z D, DUAN C X. The majorspecies causing maize ear and kernel rot and their toxigenicity in chongqing, China. Toxins, 2018, 10(2): 90-103.
[21] 席靖豪, 趙清爽, 林煥潔, 袁虹霞, 丁勝利, 李洪連. 河南省及周邊地區玉米穗腐病病原菌的分離及鑒定. 河南科學, 2018, 36(5): 688-692.
XI J H, ZHAO Q S, LIN H J, YUAN H X, DING S L, LI H L. Isolation and characterization of fungal pathogenic species causing maize ear rot in Henan and beyond provinces. Henan Science, 2018, 36(5): 688-692. (in Chinese)
[22] CHEN J F, SHRESTHA R, DING J Q, ZHENG H J, MU C H, WU J Y, GEORGE M. Genome-wide association study and QTL mapping reveal genomic loci associated withear rot resistance in tropical maize germplasm. Genes Genomes Genetics, 2016, 6(12): 3803-3815.
[23] COAN M M D, SENHORINHO H J C, PINTO R J B, SCAPIM C A, TESSMANN D J, WILLIAMS W P, WARBURTON M L. Genome-wide association study of resistance to ear rot byverticillioides in a tropical field maize and popcorn core collection. Crop Science, 2018, 58(2): 564-578.
[24] YAO L S, LI Y M, MA C Y, TONG L X, DU F L, XU M L. Combined genome-wide association study and transcriptome analysis reveal candidate genes for resistance toear rot in maize. Journal of Integrative Plant Biology, 2020, 62(10): 1535-1551.
[25] GUO Z F, ZOU C, LIU X G, WANG S H, LI W X, JEFFERS D, FAN X M, XU M L, XU Y B. Complex genetic system involved inear rot resistance in maize as revealed by GWAS, bulked sample analysis, and genomic prediction. Plant Disease, 2020, 104(6): 1725-1735.
[26] ZILA C T, SAMAYOA L F, SANTIAGO R, BUTRON A, HOLLAND J B. A genome-wide association study reveals genes associated withear rot resistance in a maize core diversity panel. G3:Genes Genomes Genetics, 2013, 3: 2095-2104.
[27] JU M, ZHOU Z J, MU C, ZHANG X C, GAO J Y, LIANG Y K, CHEN J F, WU Y B, LI X P, WANG S W, WEN J J, YANG L M, WU J Y. Dissecting the genetic architecture ofseed rot resistance in maize by combining QTL mapping and genome-wide association analysis. Scientific Reports, 2017, 7(1): 1109-1115.
[28] PEREA B D, HEFFERS D, GONZALEZ D, KHAIRALLAH M, CORTES M, VELAZQUEZ G, AZPIROZ S, SRINIVASAN G, QTL mapping ofear rot resistance in highland maize, Mexico.Agrociencia, 2001, 35: 181-196.
[29] BUTRON A, SANTIAGO R, CAO A, SAMAYOA L F, MALVAR R A, QTLs for resistance toear rot in a multiparent advanced generation intercross (MAGIC) maize population. Plant Disease, 2019, 103: 897-904.
[30] ROBERTSON L A, JINES M, BALINT P J, KLEINSCHMIDT C E, WHITE D G, PAYNE G A, MARAGOS C M, MOLNAR T L, HOLLAND J B, QTL mapping forear rot and fumonisin contamination resistance in two maize populations. Crop Science, 2006, 46(4): 17434-1743.
[31] LI Z M, DING J Q, WANG R X, CHEN J F, SUN X D, CHEN W, SONG W B, DONG H F, DAI X D, XIA Z L, WU J Y, 2011. A new QTL for resistance toear rot in maize. Journal of Applied Genetics, 2011, 52(4): 403-406.
[32] DING J Q, WANG X M, CHANDER S, YAN J B, LI J S, QTL mapping of resistance toear rot using a RIL population in maize. Molecular Breeding, 2008, 22(3): 395-403.
[33] CHEN J F, DING J Q, LI H M, LI H, LI Z M, SUN X D, LI J J, WANG R X, DAI X D, DONG H F, SONG W B, CHEN W, XIA Z L, WU J Y. Detection and verification of quantitative trait loci for resistance toear rot in maize. Molecular Breeding, 2012, 30(4): 1649-1656.
[34] LIU Y B, HU G H, ZHANG A, LOLADZE A, HU Y X, WANG H, QU J T, ZHANG X C, OLSEN M, VICENTE F S, CROSSA J, LIN F, PRASANNA B M. Genome-wide association study and genomic prediction ofear rot resistance in tropical maize germplasm. The Crop Journal, 2020, doi:10.1016/j.cj.2020.08.008.
[35] 賈玉芳. 玉米穗腐病的發病原因及防治措施. 中國農業信息, 2015(7): 51.
JIA Y F. Causes of corn ear rot and its control measures. China Agricultural Informatics, 2015(7): 51. (in Chinese)
[36] ZENG Z B. Precision mapping of quantitative trait loci. Genetics, 1994, 136(4): 1457-1468.
[37] GARCIA S A, THORNSBERRY J M, TH B E. Structure of linkage disequilibrium in plants. Annual Review of Plant Biology, 2003, 54(1): 357-374.
[38] WEN J, SHEN Y Q, XING Y X, WANG Z Y, HAN S P, LI S J, YANG C M, HAO D Y, ZHANG Y. QTL mapping ofear rot resistance in maize. Plant Disease, 2020, doi: 10.1094/PDIS-02-20- 0411-RE.
[39] 張艷, 張葉, 王梓鈺, 聞競, 韓四平, 郭嘉, 邢躍先. 44份玉米自交系對鐮孢穗腐病的抗性鑒定. 植物遺傳資源學報, 2019, 20(2): 276-283.
ZHANG Y, ZHANG Y, WANG Z Y, WEN J, HAN S P, GUO J, XING Y X. Evaluation of resistance toear rot in 44 maize inbred lines. Journal of Plant Genetic Resources, 2019, 20(2): 276-283. (in Chinese)
[40] 鄒成佳, 崔麗娜, 章振羽, 張小飛, 李榮進, 陳耕, 李曉. 玉米自交系對輪枝鐮孢菌穗腐病的抗性評價, 西南農學報, 2017, 30: 1346-1349.
ZOU C J, CUI L N, ZHANG Z Y, ZHANG X F, LI R J, CHEN G, LI X. Evaluation of maize inbred lines for resistance toear rot. Southwest China Journal of Agricultural Sciences, 2017, 30: 1346-1349. (in Chinese)
[41] MASCHIETTO V, COLOMBI C, PIRONA R, PEA G, STROZZI F, MAROCCO A, ROSSINI L, LANUBILE A. QTL mapping and candidate genes for resistance toear rot and fumonisin contamination in maize. BMC Plant Biology, 2017, 17(20): 20.
[42] ALESSANDRA L, VALENTINA M, BORRELLI V M, LORENZO S, LOGRIECO A F, ADRIANO M. Molecular basis of resistance toear rot in maize. Frontiers in Plant Science, 2017, 8: 1774.
[43] JONG G D, PAMPLONA A K A, PINHO R G, BALESTRE M. Genome-wide association analysis of ear rot resistance caused byin maize. Genomics, 2018, 110(5): 291-303.
[44] KING S B, SCOTT G E. Genotypic differences in maize to kernel infection bymoniliforme. Phytopathology, 1981, 71: 1245-1247.
QTL Mapping of Resistance toEar Rot in Maize Based on Image Analysis
WEN Jing, SHEN YanQi, WANG ZiYu, LI ShiJie, MO LanYue, LEI YuHao, ZHANG Yan, HAN SiPing
Institute of Agricultural Biotechnology, Jilin Academy of Agricultural Sciences, Changchun 130033
【】Ear rot in maize is a kind of fungal disease that is prevalent and giving serious threat to maize production worldwide, among which,ear rot (FER) is the most commonly reported in China. The location of FER disease resistance QTL is very important for the genetic improvement of maize ear rot. Locating FER disease-resistant QTL by an image analysis method not only explores a new disease evaluation method for FER in maize, but also validated the FER resistance QTLs located in the early stage through the disease rating method. 【】In this study, three F2populations (namely F2-C, F2-D and F2-J) and their corresponding F2:3families were used for QTL mapping of resistance to FER, which were constructed from a highly FER-susceptible inbred line ZW18 and three highly FER-resistant inbred lines (Cheng 351, Dan 598 and JiV203) respectively. The percentage of diseased spots was obtained by image analysis for the ears of F2:3families, then the FER resistance QTLs were mapped.【】18 FER resistance QTLs were mapped. Among them,,andlocated in 2.02-2.03 bins, 4.06-4.07 bins and 8.06 bin explained the phenotypic variation as high as 21.80%, 25.80% and 27.40%, respectively. Theof the F2-J population and theof the F2-C population and theof the F2-D population all have an overlapping interval, and the explainable phenotypic variation rate reached 16.50%. Thefrom the F2-D population and thefrom the F2-J population have an overlapping interval in 8.05 bin, and thefrom the F2-C population and thefrom the F2-J population have an overlapping interval in the 4.06-4.07 bins of 4thchromosome.In addition, compared with the results of the previous positioning by the disease rating method,,andoverlap with the disease resistance sitelocated by the rating method at 1.06-1.07 bins, andandat 4.06-4.07 bins and the resistance location located by the rating method. The disease siteandoverlap,andcompletely overlap in the 2.04 bin location interval,andare located in the overlap interval of 9.03-9.05 bins in the S2 duplication, and are derived from the same source of resistance.【】The 18 FER resistance sites located in 3 populations, among which the resistance sites located in 1.04-1.07 bins, 4.06-4.07 bins and 8.05 bin can be detected in different populations, located in 2.04 bin and 9.03-9.05 bins can be detected by different detection methods, indicating that FER resistance sites may exist in these intervals. The overlap of QTL positioning intervals in different populations verifies the authenticity of the positioning intervals to a certain extent. The overlapping intervals are located between different methods, indicating that the image analysis method is used to locate FER disease-resistant QTLs with certain accuracy.
maize;; resistance; QTL

10.3864/j.issn.0578-1752.2021.13.003
2020-12-09;
2021-01-18
中央引導地方科技發展資金吉林省重點實驗室基礎研究專項(202002072JC)、2020年吉林省預算內基本建設資金(2020C019-4)
聞競,E-mail:jlruby1988@126.com。通信作者韓四平,E-mail:hansp@cjaas.com
(責任編輯 李莉)

