谷子RNA干擾相關酶類基因家族的鑒定與分析
張司雯,鄧 欣,王 龍,羅皓天,王禹茜,王 月,李清竹,王紅艷
(遼寧大學生命科學院/ 植物表觀遺傳與進化實驗室,遼寧沈陽 110036)
RNA 干擾(RNA interference,RNAi)現象普遍存在于動物和植物中,是一種在進化上高度保守的基因表達調節機制。其是在3種核心酶類的作用下,利用雙鏈RNA(double-stranded RNA,dsRNA)誘導形成的小RNA(small RNA, sRNA),對目標序列的mRNA進行降解從而特異性地阻斷或抑制相應基因表達的過程[1-2]。與RNA 干擾相關的酶類主要包含三大類:Dicer 酶、Argonautes(AGO)蛋白家族、RNA 依賴的RNA 聚合酶(RNA-dependent RNA polymerases,RDRs),分別參與RNA 干擾過程中的啟動階段、效應階段和擴增階段[3-4]。其中,RNA 干擾的啟動階段需要通過多種途徑產生dsRNA,例如DNA的雙向轉錄途徑、自身互補的RNA 折疊途徑或異常合成的mRNA 的RNA 依賴性轉錄途徑等[5]。然后,互補的dsRNA 被具有RNaseⅢ型活性Dicers蛋白加工成長度為21~26個核苷酸的小干擾RNA(small interfering RNA, siRNA)或微小RNA (microRNA,miRNA)。其中一條RNA 通過AGO蛋白與沉默效應復合物(RNA-induced silencing complex,RISC)結合,使這些復合物序列具有特異性的導向功能,從而找到與sRNA 互補的靶RNA,進而通過阻斷翻譯或裂解靶mRNA 而導致靶基因的沉默或阻遏。這些sRNA 還可以通過將組蛋白和/或DNA 甲基轉移酶募集到靶基因的調控序列中介導轉錄基因沉默[5-6]。
目前已知的所有動植物中均存在Dicer、AGO和RDR 基因的多個拷貝,并且部分基因的功能已被闡明[4]。以禾本科為例,水稻(Oryza sativa)中含有8個DCL類基因,分別是OsDCL1a、OsDCL1b、OsDCL1c、OsDCL2a、OsDCL2b、OsDCL3a、OsDCL3b、OsDCL4[4,7]。其中,OsDCL1在水稻miRNA 的加工過程中起到了十分重要的作用[8]。OsDCL3a和OsDCL3b的dsRB(double-stranded RNA‐binding)結構域差異很大,因此有學者建議將其歸類為完全不同的第五類Dicer 蛋白[7]。OsDCL4負責正常植物發育所需的21個核苷酸的反式作用干擾小RNA(trans-acting siRNA, ta-siRNA)的加工[8-9],且與水稻穗部的sRNA 生成有關[10]。
AGO蛋白家族是RNA 誘導的沉默復合物中的重要組成部分,該沉默復合物基于序列互補性使目標轉錄本沉默。水稻中鑒定出了19 種AGO基因[4],在RNAi 和相關途徑中起著至關重要的作用,并調節植物的生長和發育[11]。如水稻中的AGO2已經有研究表明在冷脅迫、鹽脅迫和干旱脅迫的響應過程中特異上調表達[12]。AGO1和AGO18具有協同抗病毒的能力[13]。OsAGO7控制水稻葉片向上卷曲[14]。此外,OsMEL1是一種獨特的AGO蛋白,可能通過由H3K9甲基化介導的染色質修飾來調節生殖細胞的減數分裂以及雌雄配子的正常發育。OsMEL1是否直接與調節性sRNA 相互作用以在染色質上產生所需的改變尚不清楚[4]。水稻SHL2、SHL4/SHO2和SHO1分別編碼擬南芥(Arabidopsis thaliana)RNA 依賴性RNA 聚合酶6、AGO7和DICER-like 4的直系同源物,其突變通過ta-siRNA 影響葉片發育以及莖尖分生組織(shoot apical meristem,SAM)的完全缺失或異常形成[15]。OsPNH1作為AGO蛋白家族中的一員,與SAM和葉片的發育有關[16]。
RNA 依賴性RNA 聚合酶(RDRs)由RDR 基因編碼,在發育調控、維持基因組完整性和防御外源核酸等方面發揮著重要作用[17]。高等植物已經進化出多個依賴于RNA 的RDRs,其與Dicers(DCL)蛋白共同作用,產生具有特殊分子功能的不同種類的sRNA[10]。水稻中已經報道5個RDRs相關基因[4]。擬南芥中RDR1、RDR2或RDR6功能的喪失可以增強植物對RNA 和DNA 病毒的敏感性[18],水稻中OsRDR6介導的RNAi 途徑也與植物對病毒的防御反應以及植物對細菌和真菌病原體的防御有關[19]。
谷子(Setaria italica)是適合于干旱地區種植的重要糧食作物和飼料作物。因其基因組較小(約515 Mb),是二倍體C4類禾本科植物,自花授粉,易栽培,生育周期及繁殖期短,且與水稻基因組具有良好的共線性,因此經常被視作分子遺傳學研究的模式作物[20-21]。近年來,隨著組學技術的發展,越來越多的谷子品種的基因組測序工作已經完成,如xiaomi、Zhanggu、Yugu1、TT8等[22-24],并開發出了多種谷子功能組學綜合數據庫,如SIFGD和MDSi等[22,25],但是對于谷子表觀遺傳學的研究仍然較少。因此,本研究以谷子RNA 干擾相關酶類基因為研究對象,通過對兩個代表性的谷子品種(xiaomi和Yugu1)進行全基因組篩查與比較基因組學的研究,獲得谷子DCL 酶、AGO蛋白家族和RDR 類基因家族所有成員的序列信息,構建系統進化樹,并對其進行保守結構域分析和不同組織器官的特異性表達分析,為更深入地開展谷子表觀遺傳學研究提供理論依據。
1 材料與方法
1.1 供試材料
本試驗選取谷子測序品種xiaomi和Yugu1(豫谷1)為研究對象,其基因組信息可在MDSi(http://foxtail-millet.biocloud.net/home)和Phytozome (https://phytozome.jgi.doe.gov/pz/portal.html)中得到。
1.2 試驗方法
1.2.1谷子RNA 干擾相關酶類基因的探尋
水稻基因組中RNA 干擾相關酶類基因的氨基酸序列通過文獻報道[4]和RGAP(http://rice.plantbio logy.msu.edu/)網站搜索得到,將這些基因序列本身或序列名稱在NCBI上校正,再與Phytozome[26]和MDSi[22]網站谷子數據庫進行比對,選取相似度最高且E值最低的序列作為谷子中RNA 干擾相關酶類基因的候選序列。
1.2.2蛋白質序列理化性質分析
谷子RNA 干擾相關酶類基因的蛋白質理化性質[氨基酸數、分子量、理論等電點、分子式、脂肪指數、親水性的平均值(Grand average of hydropathicity,GRAVY)等]的分析使用Protparam (https://web.expasy.org/protparam/)在線工具完成。
1.2.3亞細胞定位預測及分析
利用BUSCA 亞細胞定位網站(http://busca.bioc omp.unibo.it/)對谷子RNA 干擾相關酶類基因的蛋白質序列進行亞細胞定位分析。
1.2.4系統進化樹的構建
運 用MEGA X (Molecular Evolutionary Genetics Analysis)軟 件 鄰 接 法(neighbor-joining method, NJ)對谷子和水稻的DCL 類、AGO類、RDR 類蛋白質序列進行系統進化樹分析,boot strap設置為1000,Model/Method 設置為p-distance,其余采用默認參數進行,得到的進化樹輸出結果在EvolView(https://www.evolgenius.info/evolview/#login)中進行美化。
1.2.5保守結構域分析
使用HMMER (https://www.ebi.ac.uk/Tools/hmmer/)對谷子和水稻RNA 干擾酶類基因的氨基酸序列進行分析,使用AI CS6 (Adobe illustrator CS6)進行繪圖。
1.2.6谷子RNA 干擾酶類基因的蛋白質保守基序鑒定及分析
利用MEME 在線網站(http://meme-suite.org/)對谷子RNA 干擾酶類基因的蛋白質序列進行保守基序(Motif)分析。
1.2.7組織特異性表達分析
用于探索xiaomi 谷子中RNA 干擾相關酶類基因的不同組織的表達模式的轉錄組表達數據在MDSi中獲得。Yugu1谷子中RNA 干擾相關酶類基因的不同組織的表達模式的轉錄組數據在SIFGD上獲得(原始序列號為SRR442161、SRR442162、SRR442163、SRR442164)[24-25]。 所 得 的FPKM(Fragments per Kilobase of exon model per million mapped fragments)值在TBtools[27]中的Heatmap中進行可視化,Log Scale 中的Base設置為2.0,LogWith設置為1.0。
2 結果與分析
2.1 谷子RNA 干擾相關酶類基因家族的探尋及基本特征分析
通過同源性探索,共獲得24個谷子RNA 干擾相關酶類基因家族序列(表1),其中DCL 類7個(包括SiDCL1a、SiDCL1b、SiDCL1c、SiDCL2、SiDCL3a、SiDCL3b、SiSHO1),AGO類13個(包 括SiAGO1b、SiAGO1c、SiAGO1d、SiAGO2、SiAGO4a、SiAGO4b、SiAGO12、SiMEL1、SiAGO14、SiAGO16、SiSHL4、SiAGO18、SiPNH1),RDR 類4個(包 括SiRDR1、SiRDR2、SiRDR3、SiSHL2)。水稻中3個AGO基因(OsAGO3、OsAGO15、OsAGO17)在谷子中未找到同源序列,在這里不做研究。
通過對24個谷子RNA 干擾相關酶類蛋白序列的分析發現(表1),谷子與水稻的這些同源序列在長度上相近,并且相似性較高,在xiaomi 基因組中,同源序列相似性略高,從53% (SiRDR3/OsRDR3)~91%(SiPNH1/OsPNH1),在Yugu1基因組中,同源序列相似性從50% (SiAGO18/OsAGO18)~91% (SiPNH1/OsPNH1),并且谷子品種間同源基因的整體相似性差異不大,說明谷子間以及谷子與水稻間的這些基因在進化上相對保守,行使的功能較為重要。此外,水稻中的多個蛋白序列,如OsDCL2a和OsDCL2b、OsAGO1a和OsAGO1b、OsAGO13和OsMEL1、OsAGO11和OsAGO12、OsRDR3和OsRDR4與谷子比對后,只能對應到谷子的同一個序列。

表1 谷子與水稻中RNA 干擾相關酶類基因比較分析Table 1 Comparativeanalysisof RNA interference-related enzyme genesin Setaria italica and Oryza sativa
通過對谷子RNA 干擾相關酶類蛋白序列的理化性質的分析發現(表2),氨基酸數從313(SiDCL1b)~1 933 (SiDCL1a),分 子 量在35 044.76(SiDCL1b)~216 227.3 Da(SiDCL1a),理論等電點在5.01(SiDCL1b)~9.55 (SiAGO12),說明這其中既有酸性蛋白質又有堿性蛋白質,帶負電荷的殘基總數(Asp +Glu)在43 (SiDCL1c)~257 (SiDCL1a),帶正電荷的殘基總數(Arg+Lys)在36(SiDCL1b)~241(SiDCL1a),原子總數在4 913(SiDCL1b)~30 290(SiDCL1a),脂肪指 數 在73.1 (SiAGO1b)~94.66 (SiDCL3b),GRAVY在?0.536(SiAGO1b)~?0.172(SiDCL2),除SiAGO1b是親水性蛋白質外,其余均為兩性蛋白質。
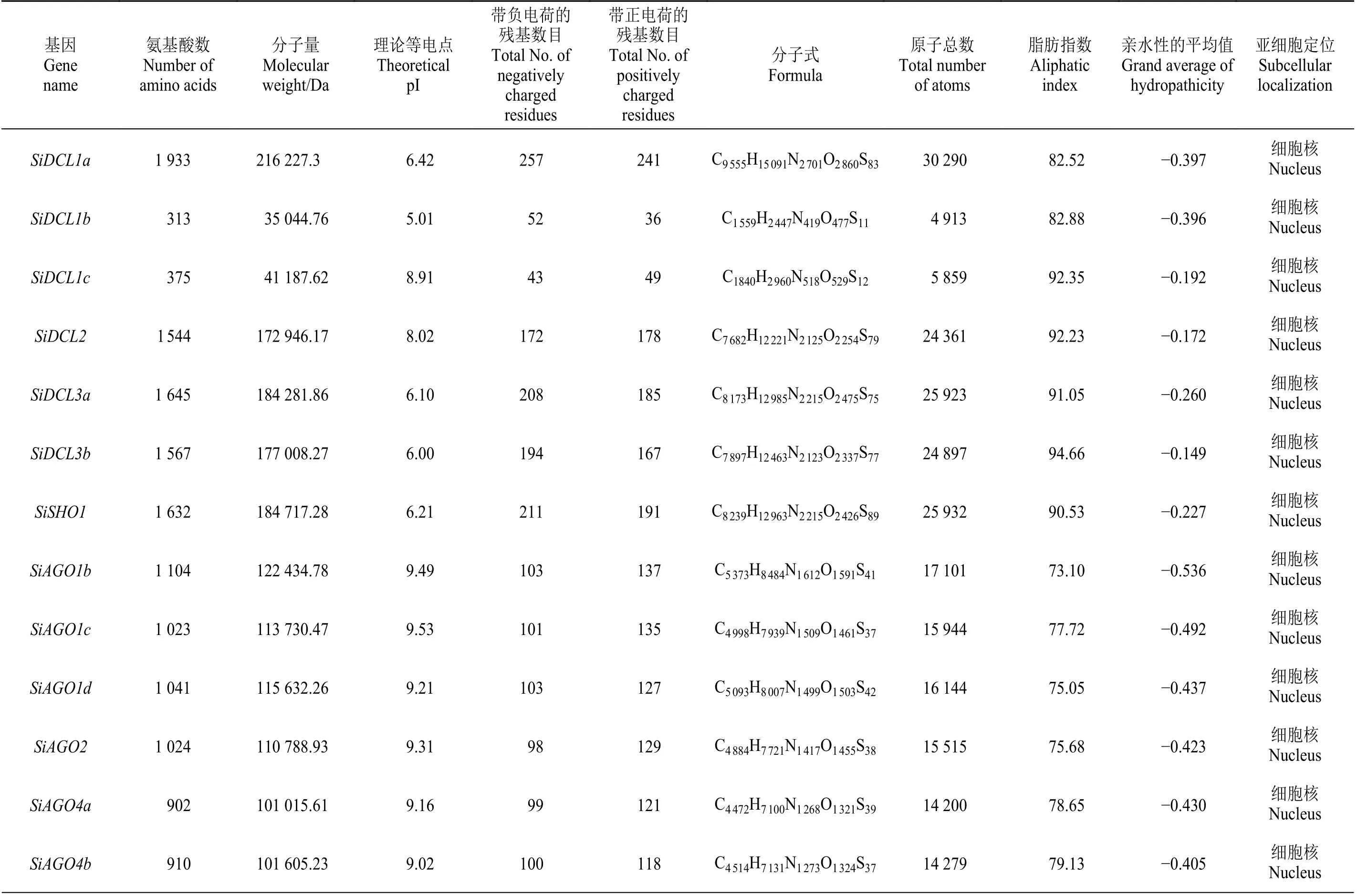
表2 谷子RNA 干擾相關酶類基因的蛋白質理化性質分析Table 2 Protein physical and chemical properties analysis of Setaria italica RNA interference-related enzyme genes
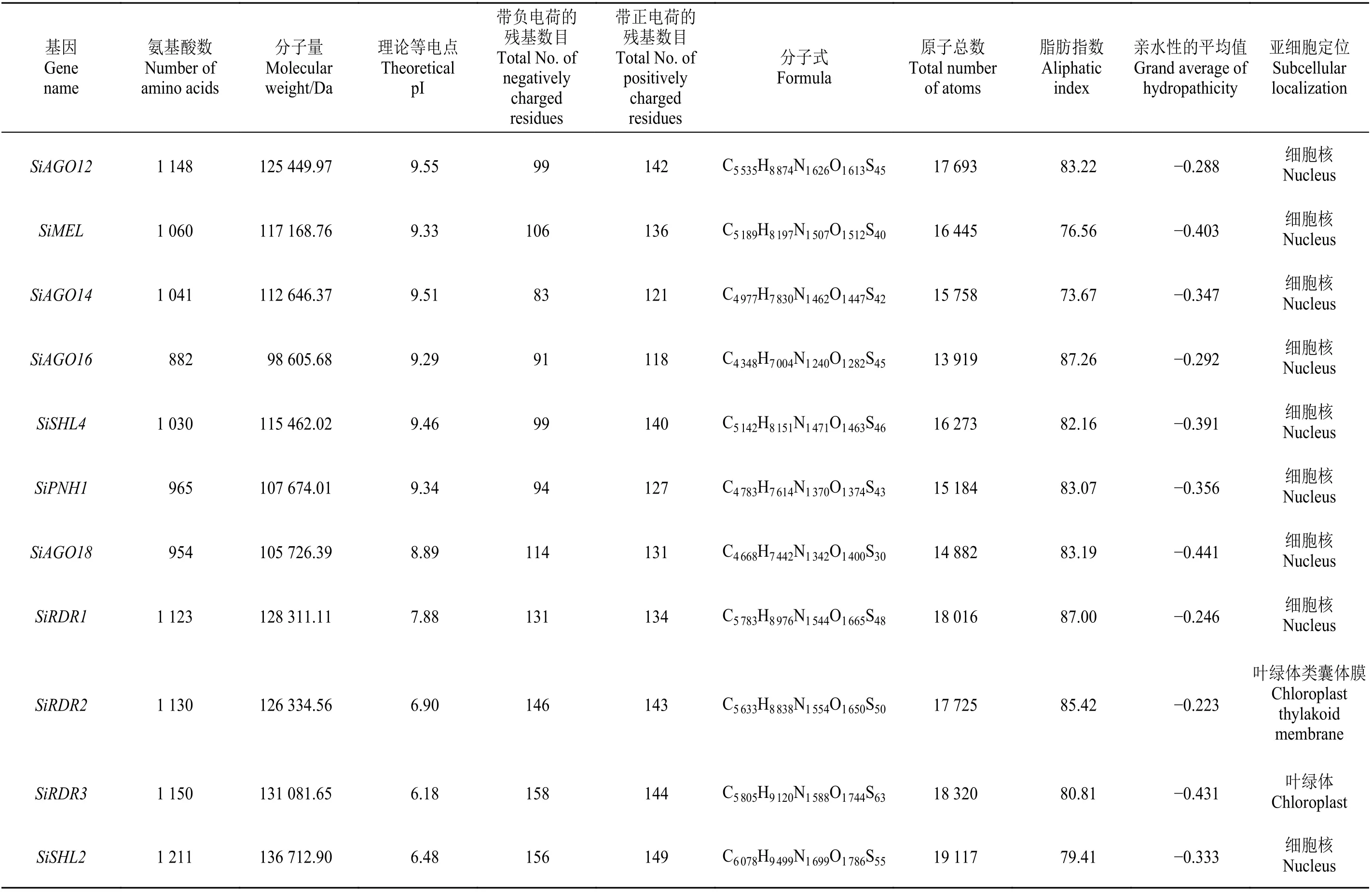
續表 2Table 2 (Continued)
通過對谷子RNA 干擾相關酶類基因的蛋白質序列進行亞細胞定位分析發現(表2),除SiRDR2位于葉綠體類囊體膜、SiRDR3位于葉綠體外,其余均位于細胞核,這說明這些酶類基因會在不同的亞細胞結構中發揮不同的功能。
2.2 谷子和水稻RNA 干擾相關酶類基因系統進化樹分析
為探究谷子和水稻RNA 干擾相關酶類基因的系統進化關系,對篩選出的谷子和水稻RNA 干擾相關酶類蛋白進行系統進化樹構建,結果發現(圖1),RNA 干擾相關酶類基因共分為3個進化支。第一個進化支包括所有的Dicer 家族和SHO1:Si/OsDCL1a、Si/OsDCL1b、Si/OsDCL1c、SiDCL2、Si/OsDCL3a、Si/OsDCL3b、Si/OsSHO1、OsDCL2a、OsDCL2b。第二個進化支包括所有的RDR 家族和SHL2:Si/OsRDR1、Si/OsRDR2、Si/OsRDR3、OsRDR4、Si/OsSHL2。第三個進化支包括所有的AGO蛋白家族以及MEL1、SHL4、PNH1:Si/OsAGO1b、OsAGO1a、Si/OsAGO1c、Si/OsAGO1d、Si/OsAGO2、OsAGO3、Si/OsAGO4a、Si/OsAGO4b、Si/OsAGO12、OsAGO11、OsAGO13、Si/OsAGO14、OsAGO15、Si/OsMEL1、Si/OsAGO16、Si/OsSHL4、Si/OsPNH1、OsAGO17、Si/OsAGO18。
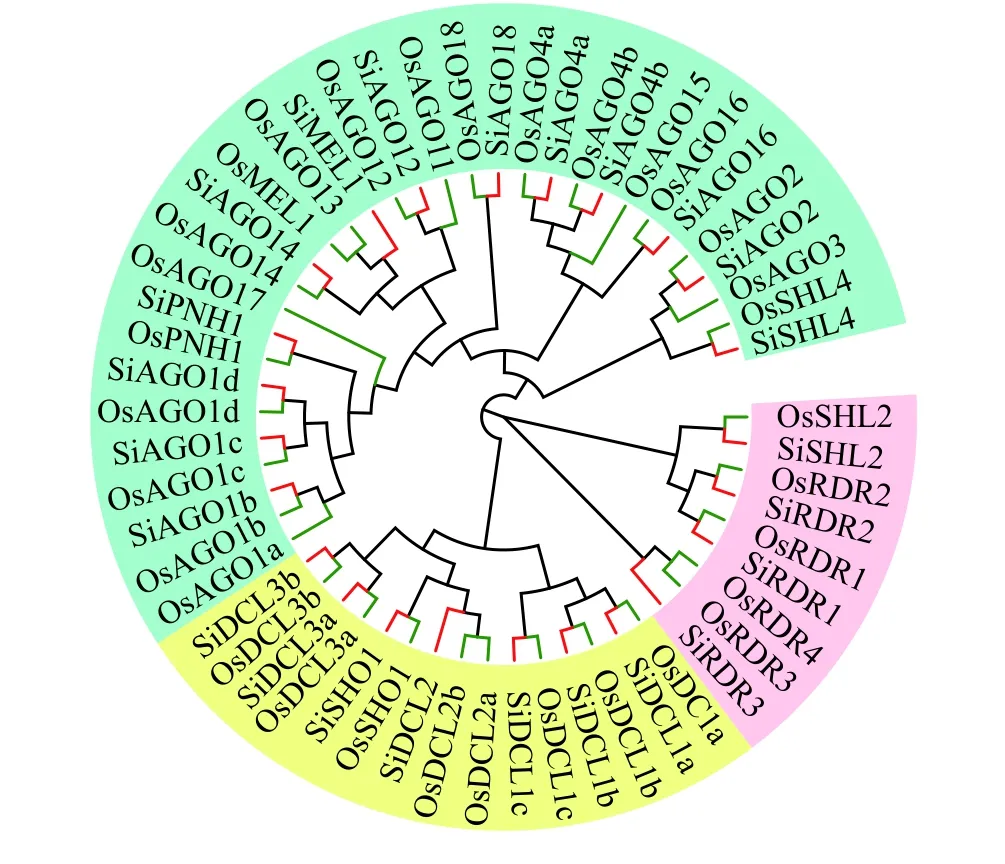
圖1 谷子和水稻RNA 干擾相關酶類基因的系統進化樹Figure 1 Phylogenetic tree for the RNA interference-related enzyme genes in Setaria italica and Oryza sativa
2.3 谷子和水稻RNA 干擾相關酶類基因的保守結構域分析
DCL酶類是RNAⅢ家族成員,通過對谷子和水稻DCL酶的保守結構域分析發現(圖2),Si/OsDCL1a、SiDCL2、OsDCL2a、OsDCL2b、SiDCL3a、Si/OsDCL3b、OsSHO1中含有與RNAⅢ家族相同的結構域,即DEAD、Helicase_C、Dicer_dimer、PAZ和Ribonuclease_3結構域。此外,SiSHO1和OsDCL3a 還有一個特有的ResⅢ結構域,是Res亞基中的Ⅲ型限制酶,參與DNA 合成和ATP合成,具有水解酶活性。Dsrm 結構域僅存在于SiDCL1a、Si/OsDCL1b、Si/OsDCL1c、OsDCL2b、Si/OsSHO1中;Si/OsDCL1b、Si/OsDCL1c與其他DCL酶相比只含有Dsrm 和Ribonuclease_3結構域。
谷子和水稻的AGO蛋白家族除OsAGO13和OsAGO17外至少具有ArgoN、ArgoL1、PAZ和Piwi 4 個結構域(圖2),除SiSHL4和OsAGO3之外,還均具有ArgoL2結構域。在Si/OsAGO1、OsAGO4b、OsAGO11、Si/OsAGO12、OsAGO13、Si/OsAGO14、OsAGO18、Si/OsMEL1和Si/OsPNH1中還均具有共同的Argomid 結構域,而Gly-rich_Ago1結構域只存在于OsAGO1a、Si/OsAGO1b和SiAGO1d 中。SiMEL1在進化樹和結構域的結果中均與其他AGO基因聚為一類并與其他AGO酶類基因具有相同的結構域。
在RDR 家族蛋白質結構域的探索中(圖2),發現谷子和水稻的RDR 酶類均包含RdRP結構域。

圖2 谷子和水稻RNA 干擾相關酶類的氨基酸結構域分析Figure 2 Analysis of the amino acid domains of enzymesrelated to RNA interference in Setaria italica and Oryza sativa
2.4 谷子RNA 干擾相關酶類基因蛋白質保守基序分析
谷子RNA 干擾相關酶類基因的蛋白保守結構域主要包含5個保守基序:Motif1、Motif2、Motif3、Motif4、Motif5,并且在大多數的谷子RNA 干擾酶類基因中均存在,說明其基序是高度保守的(圖3)。基序1的序列為RJIFYRDGVSEGQFT;基序2的序列為CHPTEFDFYLC;基序3 的序列為SRPAHYHVL YDENGF;基序4的序列為VPPAYYAHLAA;基序5的序列為GQWNMMNKKMVNGGKIRKWAC。從圖中分析得出,谷子RNA 干擾酶類基因的蛋白序列均含有這5個保守基序,說明這5個基序在谷子RNA 干擾酶類基因蛋白的進化中是高度保守的。

圖3 谷子RNA 干擾相關酶類基因蛋白質保守基序分析Figure 3 Analysis of protein conservative motifs in Setaria italica RNA interference-related enzyme genes
2.5 谷子RNA 干擾相關酶類基因組織特異性表達分析
為了探索RNA 干擾相關酶類基因對谷子生長發育的影響,本研究對xiaomi 的6個發育時期(包括3周齡葉片、孕穗期的倒二葉、抽穗2 d 的穗、授粉時期的穗、灌漿期的穗和莖)以及Yugu1的穗、葉、莖、根4種組織器官的轉錄組數據進行分析,結果表明(圖4)兩者轉錄組顯示的結果有一定的一致性也有各自的特點。xiaomi 中1個基因(SiDCL3b)在所有組織中均未檢測到表達,同時該基因在Yugu1中也僅在穗中檢測到低量表達,在Yugu1中未檢測到表達的SiRDR3在xiaomi 中檢測到的表達量也較低。在xiaomi和Yugu1中分別有5 個基因(SiDCL3a、SiAGO16、SiSHL4、SiRDR3、SiSHL2)和11個 基 因(SiDCL1a、SiDCL1b、SiDCL3b、SiSHO1、SiAGO12、SiAGO14、SiAGO16、SiSHL4、SiRDR2、SiRDR3、SiSHL2)在所有組織中檢測到較低表達[FPKM <7,即log2(FPKM +1)<3]。在xiaomi 和Yugu1中 分 別 有7 個 基 因(SiAGO1b、SiAGO1c、SiAGO1d、SiAGO2、SiAGO4a、SiAGO18、SiPNH1)和6個 基 因(SiAGO1b、SiAGO1c、SiAGO4a、SiPNH1、SiAGO18、SiRDR1)至少在一種組織中出現高表達[FPKM>20,即log2(FPKM+1)>4.39],且xiaomi中SiAGO18僅在灌漿期的穗中特異性高表達,在Yugu1中SiAGO18相對其他時期在穗中表達量最高,SiAGO1b在4種組織中的表達量均最高,暗示了這些基因在組織發育中的關鍵調控作用。同時,在所分析的xiaomi 的6個組織中,所有基因在生長3周的葉片中的表達量最低,在所有時期的穗以及灌漿期的莖中表達量較高,且分別有11、4、5、2個基因在抽穗2 d 的穗、授粉時期的穗、灌漿期的穗和灌漿期的莖中的表達達到了最高值。在所分析的Yugu1中AGO蛋白家族基因在4種組織中均有較高水平的表達,說明在RNA 干擾過程中,AGO酶類基因處于核心位置[4],谷子RNA 干擾相關酶類基因在穗和莖中相比其他組織中的表達量更高,這與其在穗和莖的生長發育中發揮的重要調節作用有關[15]。

圖4 谷子RNA 干擾相關酶類基因組織特異性表達分析Figure 4 Transcriptome analysisof RNA interference-related enzyme genesin different Setaria italica tissues
3 討論與結論
由DCL、AGO和RDRs三類家族成員介導的RNAi 是一個重要的生理調控過程,在植物營養和生殖生長過程,特別是在SAM 的發育以及從SAM 到花分生組織的轉變過程中具有重要作用[4]。但是,谷子中的這些基因尚未見報道。xiaomi作為一種超早熟材料,因其具有生長周期短、基因組數據精準、轉錄組數據全面,并已成功建立出高效穩定的再生體系和遺傳轉化體系等特點,對谷子的功能基因組研究和功能基因利用起到重要的指導作用[22],而Yugu1是第一個谷子基因組測序的品種,在谷子分子生物學和基因組學研究中具有重要的參考價值。因此,本研究選取這兩個代表性的谷子品種,對其RNA 干擾相關酶類基因家族進行了全基因組鑒定。本研究共挖掘出24個谷子RNA 干擾相關酶類基因,包括DCL 類7個,AGO類13個,RDR 類4個。分析并注釋了這些基因的長度、相似度及氨基酸序列號,水稻中的多個蛋白序列與谷子比對后,只能對應到谷子的同一個序列。說明谷子在進化過程中既與水稻保持了共線性又有其自身進化的特點[21,28]。并通過對24個蛋白質序列的理化性質分析以及亞細胞定位預測,以確定不同基因間的相互關系。通過系統發育樹分析了各個基因家族之間的進化關系,Dicer 酶可催化dsRNA 產生小RNA,是重要的RNA 酶Ⅲ家族成員之一[29]。在植物中,RDR 參與了RNA 介導的DNA 甲基化途徑(RNA-directed DNA methylation,RdDM),RdDM途徑不僅在突變、印記、基因調控和植物發育中起到重要作用,同時也影響轉座子沉默、基因組的穩定性以及植物的脅迫反應[30]。AGO蛋白作為效應復合物的核心,在RNA 干擾中發揮重要作用[29]。進化樹結果揭示了谷子各基因家族成員的進化過程及這些基因的多樣性。值得注意的是,谷子RNA 干擾相關酶類基因在Yugu1基因組中只是注釋成Dicer、AGO、RDR 基因,并沒有對其進行細化,而本研究結果則是對Phytozome數據庫的豐富和補充。同時,根據進化樹結果可以看出,水稻和谷子的直系同源(orthologous)基因(如OsDCL3a 和SiDCL3a)在進化樹上先聚為一支,再與旁系同源(paralogous)基因(OsDCL3b和SiDCL3b)聚為一大支,驗證了谷子和水稻在進化上的關系和地位。
在對谷子RNA 干擾相關酶類基因家族的保守基序分析中發現這24個基因均含有5個共同的保守基序,說明這些基序在進化過程中相對保守。
在對谷子和水稻RNA 干擾相關酶類基因家族的保守結構域分析中發現,所有的DCL 酶、AGO蛋白家族、RDR 類家族各自具有共同的保守結構域。具有相同結構域的基因在功能上相似,行使相同的功能。DCL酶類家族中除氨基酸長度較短的Si/OsDCL1b、Si/OsDCL1c外,均 具 有Helicase_C、Dicer_dimer、PAZ和Ribonuclease_3 4種結構域;AGO酶類基因所包含的4種結構域中,PAZ和Piwi結構域是核心AGO蛋白家族最主要的功能域,PAZ 有助于siRNA 3′末端的結合,而Piwi 則將siRNA 的5′末端與靶RNA 結合[4]。水稻MEL1主要位于生殖細胞的細胞質中,在減數分裂前生殖細胞的發育和減數分裂的進程中具有特定的功能[31]。RDR 酶類家族中主要包含RdRP結構域。因此,在對SiSHL2和SiSHL4進行歸類時,由于SiSHL2與SiRDR1、SiRDR2具有相同的結構域,其被定義為RDR 酶類相關基因,而SiSHL4是AGO酶類基因,可以看出,雖然它們在命名上相近,卻分別屬于不同的基因家族,在分類時需要特別注意。谷子與水稻同源基因間保守結構域有細微差別,說明谷子和水稻在進化的過程中即有相同點又分別保持了自身的特點[28]。
雖然在組織特異性表達分析時選用xiaomi 對其他谷子品種來說具有局限性,但是也具有一定的啟示作用。在水稻中已報道RNA 干擾途徑在生長發育、花期轉變中的調節作用[32-34],本研究也發現了這一點。高表達[FPKM> 20,即log2(FPKM+1)>4.39]的RNA 干擾相關酶類基因在亞細胞定位預測中均定位在了細胞核中,而已有報道顯示細胞核中存在的生長素信號途徑有助于解釋植物生長發育的調控機制[35]。谷子RNA 干擾相關酶類基因在不同組織器官中的轉錄組分析顯示,xiaomi中3種酶類基因家族在谷子3周齡葉片中表達水平較低,在孕穗期的倒二葉中表達量次之,而在所有發育時期的穗和灌漿期的莖中表達量較高,分別有45.8%、16.7%、20.8%和8.3%的基因在抽穗2 d 的穗、授粉時期的穗、灌漿期的穗和灌漿期的莖中的表達達到了最高值,說明在谷子的穗和莖中生成小RNA 途徑更活躍,暗示其在谷子SAM 的發育以及從SAM到花分生組織的轉變過程中的重要作用,有待于后續深入研究。在Yugu1中不同組織器官的轉錄組分析結果顯示,3種酶類基因家族在谷子葉片和根中的表達水平相對較低,而在穗和莖中的表達量較高,說明在谷子的穗和莖中生成小RNA 途徑更活躍。除此之外,SiDCL1c、SiDCL2、SiRDR1和SiAGO1b在各組織器官中的表達量均為同家族成員中的最高值,說明這些基因在RNA 干擾過程中可能發揮著重要作用。
本研究對谷子的RNA 干擾相關酶類基因家族進行了挖掘和鑒定,共挖掘出24個谷子RNA 干擾相關酶類基因,其中DCL 類7個,AGO類13個,RDR 類4個,并對其進行了蛋白質理化性質分析、亞細胞定位預測、系統發育分析、蛋白質保守基序分析、保守結構域分析表明谷子RNA 干擾相關酶類基因在谷子營養和生長發育的過程中發揮著重要作用,對谷子兩個品種的不同組織器官中的表達分析表明RNA 干擾酶類基因中AGO酶類基因的作用較為關鍵,在穗和莖的生長發育中RNA 干擾酶類基因發揮著關鍵作用,研究結果將對谷子功能基因組研究提供新的參考,對開展谷子分子表觀遺傳學研究提供重要理論基礎。

