基于eDNA技術的白洋淀微型生物群落監測
陳家琪,董麗,麻曉梅,田凱,白潔,趙彥偉
(北京師范大學環境學院,水環境模擬國家重點實驗室,北京 100875)
白洋淀是華北地區最大的內陸淺水湖泊濕地,被譽為“華北之腎”,其對維持區域生態平衡和保護生物多樣性發揮著生態安全屏障作用[1]。長期以來,受人為和自然雙重脅迫的影響,白洋淀面臨水質下降與水體富營養化[2]、濕地萎縮[3]、生物多樣性下降[4]等生態環境問題,因此亟待加強生態管理與綜合修復。生物與生態監測可以用來評估生態現狀,查明存在的生態問題,識別退化的驅動因子,為管理與修復提供重要的基礎信息與依據,開展白洋淀生物監測具有重要意義。
微型生物群落是生物監測的重要類群之一,其主要由細菌、真菌、藻類、原生動物以及小型后生動物等在顯微鏡下才能觀測到的微小生物組成。微型生物群落是濕地生態系統中生物要素的重要組成,對外界環境條件的變化十分敏感,在物質循環和能量流動中發揮著重要作用,其個體或群落變化可以客觀地反映水體變化[5-8],在生態監測實踐中應用廣泛。傳統的基于形態學與生理生化特征的生物分類鑒定雖然能基本滿足自然生態系統中常規物種的監測,但該方法在檢測數量和種類上有先天不足,加之受鑒定人員的主觀影響,很難對體積微小的微型生物實現精準識別。為實現對微型生物的全面評估,需要突破傳統生物監測方法的瓶頸,探索高靈敏度的生物監測新技術。
環境DNA(Environmental DNA,eDNA)技術為微型生物群落監測提供了新手段。該技術是DNA 條形碼與高通量測序方法的結合,又稱eDNA 宏條形碼(Metabarcoding)技術,其主要通過對目標DNA 片段進行高通量測序,將所得序列與標準數據庫進行比對,獲取環境中的物種組成及豐度等信息[9-10]。eDNA技術是近年生態學領域最重要的技術之一,其快速發展,推動了物種識別方法從傳統形態學鑒定向分子生物學領域轉變,極大提高了人類從生態系統中獲取數據的能力。因具有通量高、易發現新物種,在探究生物群落組成和檢測物種多樣性方面更準確快捷等優勢[11],eDNA 技術已被廣泛應用在魚類[12]、哺乳動物[13]、藻類[14-15]等多種生物群落的監測中,并逐漸被應用到入侵物種[16]和瀕危物種[17]的研究中。eDNA 宏條形碼技術在白洋淀的浮游動物監測[18]、沉積物細菌多樣性研究[19]等方面有過初步應用,但尚未對白洋淀的微型生物群落開展過全面的監測和研究。
本研究采用eDNA 宏條形碼技術,對白洋淀微型生物群落進行監測和分析,明確了白洋淀微型生物的分布和組成,比較論證了eDNA 方法在白洋淀生物監測中的靈敏度和高效性,揭示了白洋淀微型生物群落的多樣性變化,探究了季節變化和空間變化對白洋淀微型生物群落的影響。
1 材料與方法
1.1 樣品采集及形態學鑒定
選取白洋淀9 個點位采集水樣(圖1),采集時間分別為2020年4月(春季)、6月(夏季)、9月(秋季)和11 月(冬季)。每個采樣點用水質采樣器(JC-8000B)采集2 L 地表水,其中1 L 用于eDNA 宏條形碼分析,將水樣立即轉移到冰中,并在6 h內進行抽濾,濾膜孔徑為0.45 μm,直徑為50 mm,抽濾體積為250~300 mL。抽濾后立即將濾膜取下,放置在2 mL 無菌離心管內,并在-20~-80 ℃范圍內保存直至DNA提取。
為便于分析對比,采用形態學鑒定方法進行藻類鑒定。用于形態學鑒定的1 L水樣立即加入10 mL 魯哥試劑固定,靜置48 h 后,抽取上層清液并將下層沉淀濃縮至30 mL 左右,用光學顯微鏡對濃縮后的樣品進行鑒定。
1.2 DNA提取、PCR擴增和高通量測序
采用Magen Hipure 土壤DNA 試劑盒從樣本中提取DNA,使用Qubit?dsDNA HS 分析試劑盒檢測提取到DNA的濃度。
16S rRNA 測序被廣泛應用在原核生物的分類鑒定中,真核生物則主要應用18S rRNA 測序,因微型生物既有原核生物又有真核生物,本研究同時進行了16S rRNA 和18S rRNA 測序。以20~30 ng DNA 為模板,采用包含序列“CCTACGGRRBGCASCAGKVRVGAAT”的正向引物和包含序列“GGACTACNVGGGTWTCTAATCC”的反向引物擴增原核生物16S rRNA 的V3 和V4 區。對于真核生物,擴增的是18S rRNA 的V7 和V8 區,其正反向引物分別包含序列“CGWTAACGAACGAG”和“AICCATTCAATCGG”。PCR 擴增體系為25 μL,其中包含TransStart 緩沖液2.5μL,dNTPs 2μL,正向引物和反向引物各1μL,模板DNA 20 ng。原核生物和真核生物的擴增方案不同,原核生物:94 ℃初始變性3 min,94 ℃變性5 s,57 ℃退火90 s,72 ℃延伸10 s,72 ℃最終延伸5 min,共循環24 次。真核生物:94 ℃初始變性5 min,94 ℃變性30 s,57 ℃退火30 s,72 ℃延伸30 s,72 ℃最終延伸5 min,共循環25次。擴增后的產物經1.5%瓊脂糖凝膠電泳檢測。
通過Qubit3.0熒光儀(Invitrogen,Carlsbad,CA)檢測DNA文庫濃度。將文庫定量到10 nmol·L-1,按Illumina MiSeq(Illumina,San Diego,CA,USA)儀器使用說明書進行PE250/FE300 雙端測序,由MiSeq 自帶的MiSeq控制軟件(MCS)讀取序列信息。
1.3 生物信息學分析
雙端測序得到的正反向Reads 首先進行兩兩組裝連接,過濾拼接結果中含有N 的序列,保留序列長度大于200 bp的序列。經過質量過濾、去除嵌合體序列后,最終得到的序列用于可操作分類單元(Operational Taxonomic Units,OTU,等同于傳統分類學中的“種”)分析,使用VSEARCH(1.9.6)進行序列聚類,序列的相似性設為97%。之后用RDP Classifier(Ribosomal Database Program)貝葉斯算法對OTU 的代表性序列進行物種分類學分析,并在不同物種分類水平下統計每個樣本的群落組成,最后綜合多篇文獻中的微型生物名錄,對檢測到的結果進行篩選分類[20-22]。其中16S rRNA 的參考數據庫為Silva 132[23],18S rRNA的參考數據庫為Protist Ribosomal Reference(PR2)[24]。
本文所有的數據分析均是基于高通量測序獲得的OTUs序列。為了直觀地展示所檢測到的微型生物群落的序列和分類信息,本研究將門水平的序列數和OTU 數進行了對數(lg)轉換。需特別說明的是,真核生物的注釋數據庫存在分類信息和正常分類級別無法對應的情況(真菌類群和無脊椎后生動物類群),為進行統一,本研究在統計各分類級別數目時遵循了數據庫的規則,但在后續的分析中仍然按照正常的分類處理。
基于上述結果,主要對藻類優勢度、季節間差異及微型生物類群的多樣性指數進行了計算,同時也比較分析了檢出的白洋淀微型生物群落的組成和豐度以及序列數的四季變化。
1.3.1 藻類優勢度
物種優勢度(Y)表示原核藻類或真核藻類類群某一物種所占的優勢程度,計算公式為:
式中:ni為第i種藻類的個數;N為所有藻類總個數;fi為第i種藻類在各采樣點出現的頻率[25]。為便于比較,本文對原核藻類和真核藻類優勢度分別進行計算,其中傳統形態學計算的是種水平的優勢度,而eDNA 宏條形碼計算的是OTU 的優勢度。本文將優勢度Y>0.02的藻類確定為優勢種。
1.3.2 ANOSIM分析
ANOSIM 用于計算季節間差異,它是一種非參數檢驗方法,用于評估兩組實驗數據整體的相似性及相似的顯著性。該方法主要有兩個數值結果:R用于評估不同組間是否存在差異,P用于說明組間差異是否顯著。
R值計算公式為:
式中:rb表示組間距離排名的平均值;rw表示組內距離排名的平均值;n表示樣品總數[26]。R的范圍為[-1,1],R>0說明組間差異大于組內差異,R<0說明組間差異小于組內差異。
P值通過置換檢驗(Permutation test)獲得。當P<0.01時,差異顯著。
本研究在R 語言中用vegan 包的anosim()函數分別對檢測到的原核生物和真核生物進行了ANOSIM分析。
1.3.3 α多樣性指數
選用香農-威納指數(H)反映樣本中不同微型生物群落的生物多樣性,在R 語言中用vegan 包的diversity()函數進行計算,計算公式為:
式中:S為群落物種豐富度指數,即OTU 數;pi為物種i的相對豐度;x通常為2或e[27],本研究中選取2 為底數。
2 結果與分析
對36 個水樣進行高通量測序,獲得2 730 899 條原核生物序列和2 801 272 條真核生物序列。為保證數據可比性,按照每個樣本36 897條原核生物序列和33 484條真核生物序列的標準對測序結果進行隨機抽平處理,36個樣本最終共得到1 328 292條原核生物序列(占抽平前原核生物序列的48.64%)和1 205 424 條真核生物序列(占抽平前真核生物序列的43.03%)。對抽平后的序列按照97%的相似性進行OTU 聚類,獲得4 629個OTU(原核生物1 517個、真核生物3 112個),其中4 580 個OTU 能被數據庫注釋到門水平以上(占總OTU的98.94%)。
2.1 白洋淀微型生物群落多樣性
2.1.1 微型生物群落組成
依據eDNA 宏條形碼測序中的OTU 注釋結果進行分類,檢測到的白洋淀微型生物群落分為7 個類群,包括原核與真核生物,原核生物包括古細菌、細菌(除藍藻門)、原核藻類,真核生物包括真核藻類、真菌、原生動物、無脊椎后生動物。其中,細菌(除藍藻門)的序列數和OTU 數在原核生物中最多,原核藻類次之;真菌的序列數和OTU 數在真核生物中最多,原生動物次之,無脊椎后生動物最少,這些微型生物類群的序列數和OTU 數詳見表1。按照這些OTU 所能注釋到的最高分類水平進行統計,它們分屬于50 個門、119個綱、228個目、386個科、614個屬、567個種。
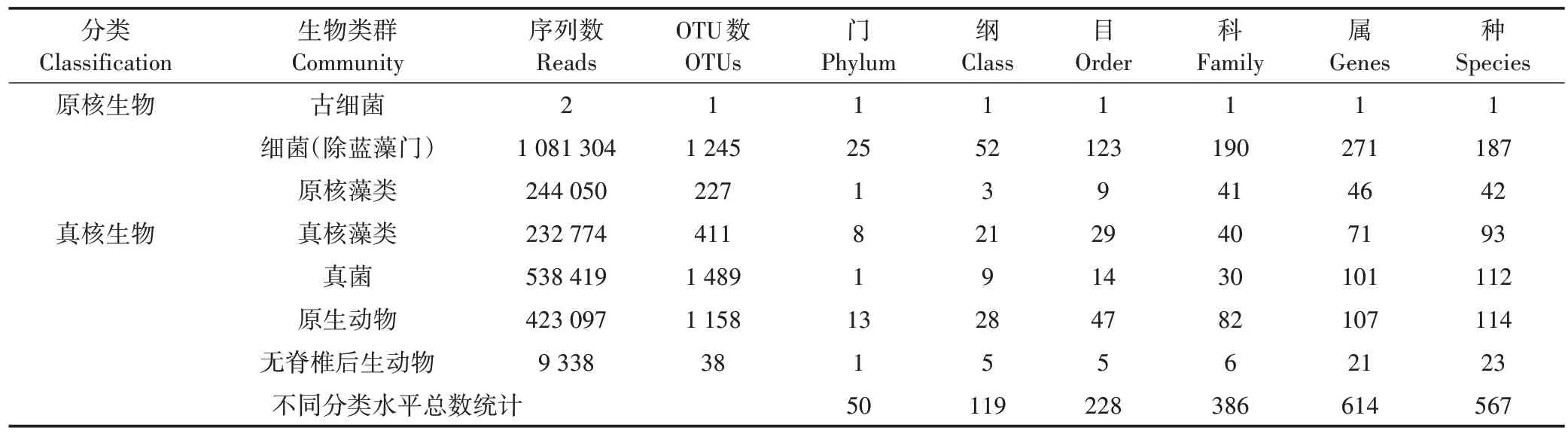
表1 微型生物群落注釋結果Table1 The annotation results of microbial community
7個微型生物群落的組成見圖2。由圖2可見,變形菌門(Proteobacteria)、擬桿菌門(Bacteroidetes)和放線菌門(Actinobacteria)三者序列數和OTU 數在細菌(除藍藻門)中占主要優勢;隱藻門(Cryptophyta)在真核藻類中最豐富,其序列數占真核藻類總序列數的82.65%,OTU 數占總OTU 數的54.74%;真菌中最豐富的門類是壺菌門(Chytridiomycota)和隱真菌門(Cryptomycota),二者序列數分別占真菌總序列數的67.16%和22.15%,OTU 數占總OTU 數的56.15%和20.89%;絲足蟲類(Cercozoa)占原生動物總序列數的53.15%,領鞭毛蟲門(Choanoflagellida)占序列數的25.81%,二者在原生動物中占主要優勢,它們的OTU數分別占原生動物總OTU 數的39.90%和27.98%。腔腸動物門(Cnidaria)和輪蟲類(Rotifera)在無脊椎后生動物中最豐富。原核藻類中僅檢測到藍藻門(Cyanobacteria),其序列數和OTU數也較高。
2.1.2 藻類監測結果
基于eDNA 宏條形碼技術共注釋到196個原核藻類OTU 和287 個真核藻類OTU,分屬于9 門21 綱34目76 科100 屬112 種,其中藍藻門(196 個OTU)和隱藻門(163 個OTU)的種類最豐富,Ochrophyta(55 個OTU)和Katablepharidophyta(31 個OTU)次 之(圖3A)。共有14 個OTU 被確定為優勢種(表2),藍藻門9 個,隱藻門5 個。OTU3 的優勢度最大(0.290 4),OTU134 的優勢度最小(0.021 7),這兩個物種均屬于隱藻門。
為驗證eDNA 宏條形碼技術用于生物監測的可靠性,同步對秋季樣品進行藻類的形態學鑒定。共鑒定出73種藻類,基本被eDNA宏條形碼技術的監測結果所覆蓋,分屬于7 門9 綱18 目31 科46 屬,其中綠藻門(Chlorophyta,35 種)和硅藻門(Bacillariophyta,17種)種類最豐富,分別占鑒定出所有藻類的47.95%和23.29%(圖3B)。鑒定出的73 種藻類中,15 種被確定為優勢種(表2),其中綠藻門種類最多,為6 種,藍藻門(Cyanophyta)4 種,硅藻門3 種,隱藻門1 種,甲藻門(Dinophyta)1 種。偽魚腥藻(Pseudanabaenasp.)為絕對優勢種,優勢度為0.750 2,四角十字藻(Crucigenia lauterbornii)為次優勢種,優勢度為0.195 3。
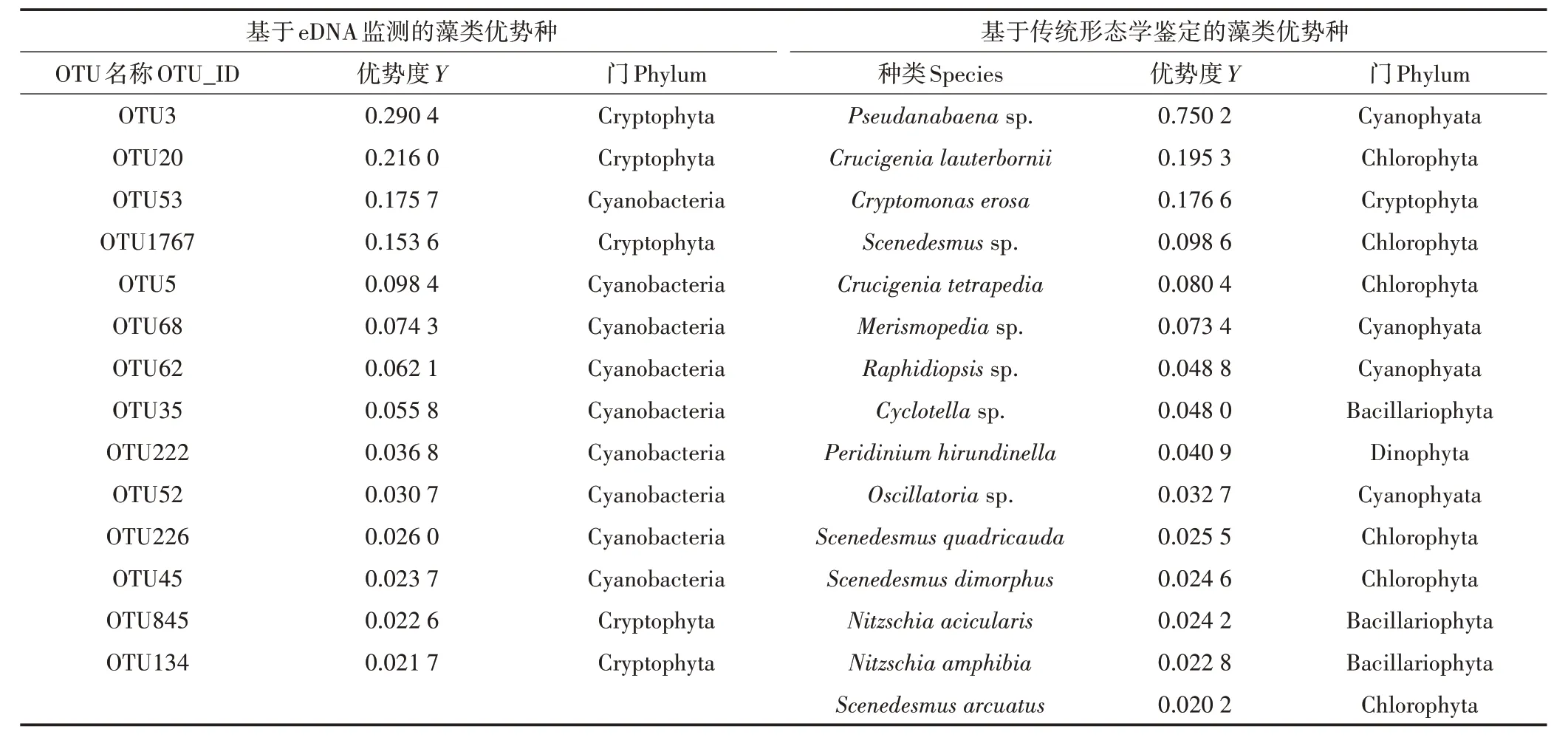
表2 基于eDNA和傳統形態學方法確定的藻類優勢種Table2 Dominant species of algae determined by eDNA and traditional morphological methods
2.2 白洋淀微型生物組成的時空分布
2.2.1 微型生物群落的季節分布
對檢出的原核生物和真核生物分別進行ANOSIM 分析,結果顯示:原核生物和真核生物的R值分別為0.648 和0.621,且P值均小于0.01,季節間差異顯著(圖4)。
如圖5 所示,在白洋淀檢出的6 個主要微型生物類群中,秋季的香農-威納指數均明顯高于其他3 個季節,說明秋季微型生物的種類最豐富,生物多樣性最高,除無脊椎后生動物外,其他微型生物類群夏季的香農-威納指數僅次于秋季。春季和冬季的生物多樣性在各微型生物類群之間的分布不一致,其中原生動物和無脊椎后生動物春季的生物多樣性要高于冬季,而其他4 個微型生物類群正好相反。細菌(除藍藻門)的香農-威納指數在6 個微型生物類群中最大,介于5.17~7.56 之間,說明該類群的生物多樣性最高;無脊椎后生動物最小,范圍在0.74~3.10。
序列數對比分析結果表明,白洋淀各微型生物類群分布有明顯不同的季節特點(圖6)。細菌(除藍藻門)序列數在四季中均占絕對優勢;真菌次之,且四季變化明顯,表現為夏季>秋季>春季>冬季。序列占比同樣較高的原生動物序列秋季分布要明顯高于其他3 個季節,無脊椎后生動物的序列數相對較少,且在冬季分布最多。原核藻類和真核藻類的季節變化特征較為相似,但和其他類群差異較大,它們的序列分布在冬季最多,夏季最少。OTU數量的總體季節變化趨勢為秋季>夏季>春季>冬季,與多樣性季節變化特點相吻合。
各微型生物類群代表物種在門和屬水平的相對豐度變化,也各自呈現出明顯的季節特點。放線菌門是細菌(除藍藻門)的優勢群體,其秋季相對豐度要明顯低于其他季節,而藍藻門冬季和秋季的相對豐度顯著高于春季和夏季(圖7A)。壺菌門是真菌的優勢群體,也是真核生物中最豐富的門類,其夏季相對豐度最高,平均值達到49.96%,冬季相對豐度最低,平均值僅有15.92%,季節差異明顯。隱藻門在真核藻類中所占比例具有絕對優勢,該門類在冬季相對豐度最高,這與真核藻類序列分布在冬季最多的結果相一致(圖8A)。和門水平相比,各微型生物類群在屬水平上的分布更為復雜,季節差異更為顯著,詳細見圖7B和圖8B。
2.2.2 微型生物群落的空間分布
某些生物類群門水平和屬水平的相對豐度變化在采樣點之間也有明顯差異(圖7和圖8)。隸屬于細菌(除藍藻門)的Epsilobacteracota 菌群,在南劉莊、棗林莊和光淀張莊3 個點位秋季相對豐度較高,尤其是在棗林莊地區,該菌群相對豐度占比要遠高于其他采樣點。與之相似,屬水平上隸屬于原核藻類藍藻門的Planktothrix-NIVA-CYA-15,在采蒲臺和王家寨兩個點位秋季相對豐度明顯高于其他點位。隸屬于原生動物領鞭毛蟲門的Monosigidae_Group_O_X,在秋季棗林莊點位相對豐度顯著高于其他點位。絲足蟲類作為真核生物的優勢種群,在四季各點位相對豐度的變化呈現出相似趨勢,即在采蒲臺和圈頭這兩個地區相對豐度較高,而在其他地區相對豐度較少。
3 討論
3.1 白洋淀微型生物群落組成主要受時間變化影響
主成分分析結果表明(圖9),白洋淀微型生物群落受時間變化的影響要大于受空間變化的影響,尤其是原核生物,在同一季節內不同采樣點之間的相似度很高,而季節間的差異較為明顯。真核生物同一季節內不同采樣點之間的差異要大于原核生物,但相較而言,空間差異沒有季節差異顯著。因此,白洋淀微型生物群落組成主要受時間變化的影響。
白洋淀地處亞歐大陸東部季風區,暖溫帶半干旱地區,全年四季分明。在春季和夏季,適宜的水溫有利于微型生物群落的生長繁殖;秋季,隨著水溫的逐漸下降,微型生物的種類逐漸減少;冬季水溫最低,微型生物的種類最少。為進一步分析本研究中出現的秋季微型生物群落多樣性高于夏季的情況,對采樣時間段內白洋淀水溫的歷史監測值進行了統計,結果表明:白洋淀4 月末(春季)的水溫在18 ℃左右,6 月初(夏季)的水溫在24 ℃左右,9月中下旬(秋季)的水溫在23 ℃左右,11 月中下旬(冬季)的水溫在11 ℃左右。本研究中,夏季的采樣時間處于春末夏初,水溫未達到最高值,是微型生物群落大量繁殖的前期;而秋季采樣時間處于夏末秋初,水溫和夏季接近,處于微型生物群落大量繁殖的后期。已有研究表明,水溫是對微型生物物種數量和豐度影響最大的環境因子[28],因此夏、秋兩季采樣時間早于典型季節性環境條件發生時間可能是造成本研究秋季多樣性指數偏高的主要原因。
陽小蘭等[29]的研究發現,白洋淀整體處于輕度富營養化狀態,部分區域呈中度富營養化,富營養程度在夏秋兩季較高。藻類是水體富營養化的重要指示生物[30],本研究中,藻類(原核藻類和真核藻類)相對豐度在夏季和秋季較低,在冬季最高。推測造成這種現象的原因可能是夏秋兩季水溫適合包括藻類在內的多種微型生物的繁殖,尤其是真菌類群中的壺菌門,在夏季的平均相對豐度占比達到49.96%,這在一定程度上削弱了部分微型生物,包括藻類的相對豐度,盡管這些生物(如部分藻類)的絕對豐度可能顯著高于其他季節,也可能是受eDNA 宏條形碼技術無法判定檢測到的是否為活體生物[31]的影響,檢測到較多死亡個體殘留的DNA所致。
空間上,各采樣點的微型生物組成、多樣性、序列數等信息分布規律性不夠突出,變形菌門、擬桿菌門、放線菌門、藍藻門等原核生物的優勢門類及壺菌門、絲足蟲類、領鞭毛蟲門、隱藻門等真核生物的優勢門類的相對豐度在不同采樣點均較高,且差異較小。相反,相對豐度較低的某些門類,如原核生物的Epsilonbacteraeota、真核生物的Basidiomycota 等的相對豐度在不同采樣點之間差異明顯。原因可能有兩方面:一是白洋淀生態補水隨機性較強,補水不穩定,會頻繁擾動水體,使得水動力條件不穩定,底泥與水體的交換頻繁,部分死體生物也進入了樣本,對不同采樣點的微型生物群落檢測造成干擾;二是采樣點密度較小,未覆蓋整個淀區,可能會使監測到的結果具有片面性,今后應根據具體情況合理設計優化布點與采樣方案,高密度設置采樣點,開展重復校驗性檢測,進行長期跟蹤監測,使之更能反映實際,更有利于分析時空變化的影響。
3.2 eDNA 技術用于白洋淀微型生物群落監測的優勢與不足
在針對白洋淀微型生物群落進行的16S rRNA 和18S rRNA 高通量測序研究中,大量細菌(除藍藻門)、原核藻類、真核藻類、真菌、原生動物類群,少量的無脊椎后生動物類群以及微量的古細菌類群被檢出,填補了白洋淀微型生物群落多樣性調查的空缺,也印證了eDNA 宏條形碼技術用于生物多樣性監測的潛力。相較于傳統形態學方法,eDNA 宏條形碼技術可識別出更多的物種,包括定鞭藻門(Haptophyta)、紅藻門(Rhodophyta)、鏈型植物(Streptophyta)、Katablepharidophyta 等顯微鏡未識別到的物種,以及以往白洋淀未報道過的藻類[32-34],豐富了白洋淀藻類名錄和微型生物群落數據庫。王靖淇等[35]、張麗娟等[36]和楊海樂等[37]的研究已表明,eDNA 宏條形碼對傳統形態學監測結果有較高的還原性,并可檢測到一些傳統方法無法識別的稀有物種和隱匿物種,說明eDNA 技術在生物多樣性監測中有重要應用潛力。
本研究也揭示了eDNA 宏條形碼技術存在的一些問題,如無法將所有的OTU 注釋到種水平、eDNA檢測對傳統形態學監測到的物種覆蓋度低等。原因在于自然界中有大量未被發現、培養和測序的物種,其基因序列在數據庫中不存在,導致數據庫信息與自然界實際存在的物種不對等,無法注釋[35,38]。測序平臺[39]、特異基因、引物的選擇[40-41]等實驗條件以及季節、水溫、流速等自然條件變化也會影響產物的有無和相對豐度。
可見,eDNA 宏條形碼技術發展初期,仍需要與傳統形態學鑒定方法緊密結合[42],才能構建可靠的多維生物監測系統。但隨著eDNA 技術的不斷完善和發展,該方法不僅會用于大規模的生物監測,更有望融合到生物多樣性監測的標準程序中。
4 結論
(1)白洋淀水樣eDNA 宏條形碼分析共檢測到7個微型生物類群,分別為古細菌、細菌(除藍藻門)、原核藻類、真核藻類、真菌、原生動物和無脊椎后生動物,注釋得到4 569 個OTU,按這些OTU 所能注釋到的最高分類水平統計,分屬于567 個種。其中,細菌(除藍藻門)和真菌分別是原核生物和真核生物的優勢類群。
(2)白洋淀微型生物群落分布與組成受時間影響更大。原核生物和真核生物均有顯著的季節間差異,且各主要微型生物類群的生物多樣性、序列數和物種相對豐度都呈現出季節分布特征。優勢門類在各采樣點的微型生物組成、多樣性、序列數等信息分布規律性不夠突出,在不同采樣點的空間分布沒有明顯差異,Epsilonbacteraeota、Planktothrix-NIVA-CYA-15等豐度較低的種群呈現出區域分布特征。
(3)eDNA 技術和傳統形態學的藻類監測結果具有相似性,且eDNA 技術可檢測到更多傳統形態學不能識別的藻類,兩種監測手段結合可獲得更全面的生物組成信息。

