霸王葉綠體基因組測序及結構分析
李曼芝 朱強 曾繼娟 牛錦鳳


摘? ? 要:為探索霸王植物葉綠體基因的系統進化,以銀川植物園沙生植物種質資源庫提供的霸王為試驗材料,采用Illumina HiSeq X Ten測序系統對其葉綠體基因組DNA建庫測序,借助近緣參考序列,得到霸王的葉綠體全基因組序列,并采用MEGA7軟件通過鄰接法基于葉綠體全基因組序列對牻牛兒苗科、蒺藜科共20個物種構建系統發育樹。結果表明,霸王葉綠體基因組序列全長105 349 bp,包括長單拷貝區79 818 bp、短單拷貝區16 955 bp和2個反向重復序列4 288 bp,共編碼97種基因,包括64種蛋白編碼基因、29種tRNA基因和4種rRNA,檢測到7種重復基因,包括4種蛋白質編碼基因和3種tRNA;系統發育樹中霸王與蒺藜科四合木的親緣關系最近,與牻牛兒苗科老鸛草屬、老鸛葵屬親緣關系較遠。研究結果為進一步開展霸王植物資源的引種馴化提供參考依據。
關鍵詞:霸王;葉綠體基因組;測序與分析
中圖分類號:Q941+.2? ? ? ? ? 文獻標識碼:A? ? ? ? ?DOI 編碼:10.3969/j.issn.1006-6500.2021.10.004
Sequencing and Stuctural Analysis of Chloroplast Genome in Zygophyllum xanthoxylum
LI Manzhi1, ZHU Qiang2,3, ZENG Jijuan2,3, NIU Jinfeng4
(Ningxia Ningmiao Ecological Garden Co., Ltd., Yinchuan, Ningxia 750004, China; Ningxia Forestry Institute Co., Ltd.,Yinchuan, Ningxia 750004, China; State Key Laboratory of Seeding Bioengineering, Yinchuan, Ningxia 750004, China; Ningxia State-owned Forest Farm and Forest Seedling Station, Yinchuan, Ningxia 750004, China)
Abstract: To explore the phylogenetic evolution of chloroplast genes in Zygophyllum xanthoxylum plant, Zygophyllum xanthoxylum provided by the psammophyte germplasm bank of Yinchuan Botanical Garden were used as the material, and Illumina high-throughput sequencing technology was employed to sequence its chloroplast genome. By relative reference sequence, the complete chloroplast genome sequence of Zygophyllum xanthoxylum was obtained. Phylogenetic tree of 20 species belonging to Geraniacea and Zygophyllaceae based on chloroplast genome were constructed using neighbor-joining method through MEGA7 software. The result showed that the chloroplast genome sequence of Zygophyllum xanthoxylum was 105 349 bp in length, including a long single copy region of 79 818 bp, a short single copy region of 16 955 bp and two inverted repeats of 4 288 bp. A total of 97 genes were encoded, including 64 protein-coding genes, 29 tRNA genes and 4 rRNAs. A total of 7 repetitive genes were detected, including 4 protein-coding genes and 3 tRNAs. According to the phylogenetic tree, Zygophyllum xanthoxylum had the closest relationship with Tetraena mongolica, but had the relative farther relationship with Geranium and California which belonged to Geraniacea. The results could provide a reference for the further introduction and domestication of Zygophyllum xanthoxylum plant resources.
Key words: Zygophyllum xanthoxylum; chloroplast genome; sequencing and analysis
霸王(Zygophyllum xanthoxylum),蒺藜科(Zygophy-
llaceae)多漿旱生灌木,是分布于亞洲中部荒漠區的特有種[1],在維持干旱區脆弱的生態與環境方面具有重要作用[2]。作為我國西北荒漠區植被組成的主要優勢種,霸王在漫長的進化過程中形成了獨特的適應機制,蘊含著豐富的抗逆基因資源[3],備受關注。目前,國內外學者已從不同角度對霸王在干旱環境下的適應機理[4-6]、藥用成分及生態修復[7]等方面進行了研究。隨著研究的不斷深入,Khalik等[8]從61種形態特征方面討論了霸王屬植物在蒺藜科的系統位置;Beier等[9]分析了霸王屬(Sarcozygium Bunge)與Augea Thunb屬、四合木屬(Tetraena Maxim)和Fagonia L.之間的關系;我國學者也從分子角度推測了國內一些蒺藜科植物的親緣關系[10-11],這些研究主要集中在霸王屬的系統位置和屬內發育關系,較少涉及霸王物種的系統發育分析。
葉綠體全基因組在種以及種以上較高階元的系統發育方面有顯著的優勢,可以作為物種分類的依據。目前,有關葉綠體基因組在霸王種質資源的相關報道較少。本研究以銀川植物園沙生植物種質資源庫的霸王植物為材料,首次進行葉綠體全基因組測序以及結構和功能基因分析,以期為霸王的系統進化發育研究奠定基礎,并對寧夏地區霸王植物資源的育種及引種馴化提供理論依據。
1 材料和方法
1.1 試驗材料
以寧夏銀川植物園采摘的霸王葉片為供試材料,葉片采摘后立即放置于保鮮盒,低溫避光保存。植物葉綠體基因組DNA提取試劑盒(BTN120308)購自北京天根生化科技有限公司。
1.2 試驗方法
1.2.1 霸王葉綠體基因組DNA的提取與組裝? 采用Illumina HiSeq X Ten測序共得到一定數量讀長為150 bp的Paired-End Reads,經預估最終組裝覆蓋率,隨機取部分數據用于葉綠體基因組組裝。采用參考文獻[12]的MITObim v1.9程序,借助近緣參考序列,最終得到本項目霸王樣品的葉綠體全基因組序列,通過GenBank現有近緣種葉綠體基因組比對的方式進行注釋,利用OGDraw在線工具[13](https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)進行基因組圖譜制作。
1.2.2 葉綠體基因組的系統發育分析 選取GenBank現有16種牻牛兒苗科(Geraniacea)和3種蒺藜科植物葉綠體基因組與霸王進行比對,共20個物種的葉綠體基因組進行系統發育分析。借助Geneious R11軟件提取單個蛋白質編碼基因編碼序列并進行比對,選取若干比對效果良好的基因,連接成單個超級基因(supergene),導出至MEGA7軟件 [14]構建系統發育樹。
2 結果與分析
2.1 基因組組分特征與分析
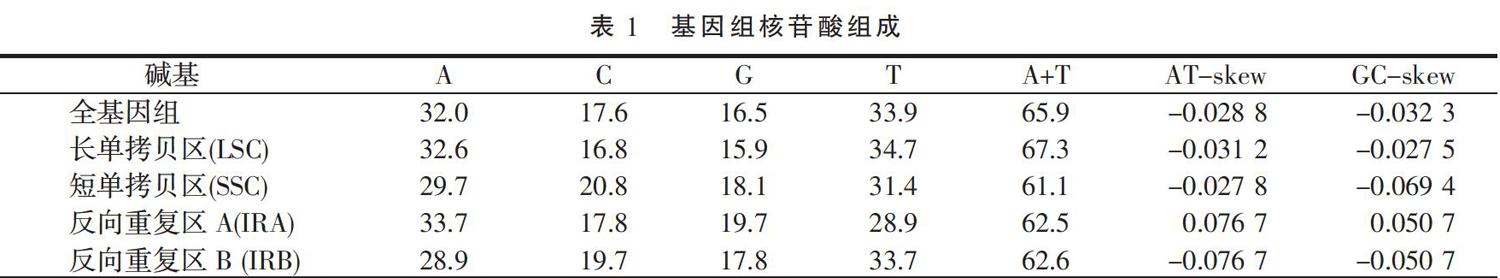
構建霸王葉綠體基因組物理圖譜(圖1),分析其葉綠體基因組特征及數量。結果表明,霸王葉綠體基因組呈環狀雙鏈,與大多數高等植物葉綠體基因組一樣,存在2個反向重復序列(Inverted repeat, IR),即IRA和IRB;反向重復序列之間有一個長單拷貝區(Large single-copy region, LSC)和一個短單拷貝區(Small single-copy region, SSC)。霸王葉綠體基因組全長105? 349 bp,其中長單拷貝區(LSC)為79 818 bp,短單拷貝區(SSC)為16 955 bp,反向重復區(IRs)為4 288 bp。
由表1可知,在全基因組中,A+T含量為65.9%,G+C含量為34.1%;長單拷貝區中A+T含量為67.3%,G+C含量為32.7%;短單拷貝區中A+T含量為61.1%,G+C含量為38.9%;反向重復區A中A+T含量為62.5%,G+C含量為 37.5%;反向重復區B中A+T含量為62.6%,G+C含量為37.5%。
通過分析霸王基因組成及內含子情況,由表2可知,霸王共包含97種基因,包括64種蛋白質編碼基因,29種tRNA和4種rRNA。其中重復的基因有7種,包括4種蛋白質編碼基因,分別是rpl2、 rpl23、rps19、ycf15;3種轉運RNA,分別是trnH-GUG、trnI-CAU和trnL-CAA。有11種基因含有內含子,包括6種蛋白質編碼基因,其中5種含有1個內含子,分別是atpF*、 clpP*、rpl2*、rpoC1*、rps12* ,另外1種基因含有2個內含子,即ycf3**;5種轉運RNA,均含有1個內含子,分別是trnA-UGC*、trnG-UCC*、trnI-GAU*、trnL-UAA*和 trnV-UAC*。此外,86種基因不含有內含子。
2.2 系統發育樹的構建
葉綠體基因組對于了解植物進化等具有十分重要的價值[15]。為確定霸王的系統發育關系,本研究采用鄰接法(Neighbor-Joining,NJ),選取牻牛兒苗科、 蒺藜科共20個物種構建霸王的系統進化樹(圖2),分析確定霸王在植物中的系統進化位置。選擇的序列比對總長度為16 677 bp,包含atpB、matK、petA、psaA、psaB、psbC、psbD、rbcL、rpoB和rpoC1這10個基因。從構建的系統進化樹中看到,有16個節點的檢驗分值達到了100%,聚類結果的可靠性比較高,如Geranium、California、Erodium、Monsonia、Pelargonium
等屬的物種聚在了一起,表明這些屬植物之間的葉綠體相似性較高。同時發現,霸王與四合木(Tetraena mongolica)的親緣關系最近,其次和團香木屬(Larrea)、愈瘡木屬(Guaiacum)較近,同屬于蒺藜科;與牻牛兒苗科老鸛草屬(Geranium)、老鸛葵屬(California)親緣關系最遠。
3 結論與討論
植物葉綠體基因組總長度一般約為107~218 kb,其中小單拷貝區長約18~20 kb,大單拷貝區長約81~90 kb,2個反向重復區長約20~30 kb[16]。葉綠體基因組的差異主要由反向重復區的收縮與擴張或者缺失引起[17],反向重復區在葉綠體基因組架構的穩定和大小方面具有關鍵作用[18]。本研究通過對霸王葉綠體基因組進行測序與分析,發現霸王葉綠體基因組全長105 349 bp,為環狀DNA分子,其中長單拷貝區域(LSC)79 818 bp,短單拷貝區域(SSC)16 955 bp,反向重復區域(IRs)4 288 bp;霸王共編碼97種基因,包括64種蛋白編碼基因、29種tRNA基因和4種rRNA;IR區中有7種基因,包括4種蛋白質編碼基因(rpl2、rpl23、rps19、ycf15)和3種tRNA(trnH-GUG、trnI-CAU、trnL-CAA)。
近年來,植物葉綠體基因組的研究得到國內外許多學者的關注,現已成為葉綠體轉化技術和植物親緣關系鑒定的重要手段之一[19]。目前,有關蒺藜科在分類學上的歸屬問題存在一定的爭議,如《中國植物志》蒺藜科的霸王、四合木均被分類到了蒺藜目[20],然而在國外的植物學科學研究與教學中被廣泛應用的是APG植物分類系統[21],其將蒺藜科的霸王分類到牻牛兒苗目,而蒺藜科的四合木被分類到了蒺藜目。本研究為確定霸王的進化地位和親緣關系,選取了牻牛兒苗科(Geraniales) 、蒺藜科(Zygophyllales) 共20個物種葉綠體基因組進行系統發育分析,發現霸王與四合木(Tetraena mongolica)為最近的姐妹種;其次和團香木屬(Larrea)、愈瘡木屬(Guaiacum)較近,同屬于蒺藜科;與牻牛兒苗科老鸛草屬(Geranium)、老鸛葵屬(California)親緣關系最遠。牻牛兒苗屬(Erodium)分支和老鸛葵屬(California)分支、鳳嘴葵屬(Monsonia)分支、天竺葵屬(Pelargonium)分支聚攏,依次成為姐妹關系,參與牻牛兒苗科植物的組成。
在整個系統樹中,親緣關系較近的同屬植物、同屬不同種的植物更容易聚在一起,這與傳統分類學的結果一致。霸王與四合木的親緣關系最近,具有相似的遺傳背景,研究結果為進一步探索蒺藜科植物的起源、演化以及親緣關系提供參考,同時為后期引種馴化及種質資源開發利用奠定基礎。
參考文獻:
[1] 趙一之, 朱宗元. 亞洲中部荒漠區的植物特有屬[J]. 云南植物研究, 2003, 25(2): 113-121, 122.
[2] 宛濤, 史雪松, 伊衛東, 等. 阿拉善荒漠駝蹄瓣屬7種植物的花粉形態研究[J]. 西北植物學報, 2006, 26(8): 1704-1708.
[3] 王文穎. 多漿旱生植物霸王HKT轉運蛋白的功能研究[D]. 蘭州: 蘭州大學, 2019.
[4] 馮燕, 王彥榮, 胡小文. 水分脅迫對幼苗期霸王葉片生理特性的影響[J]. 草業科學, 2011, 28(4): 577-581.
[5] 耿生蓮, 王占林. 霸王、沙木蓼和銀水牛果在不同水分條件下的生理研究[J]. 山西林業科技, 2007, 12(4): 24-26, 30.
[6] 吳斌. 霸王抗旱性的分子機理及相關基因克隆研究[D]. 北京: 中國林業科學研究院, 2006.
[7] 孫安安. 西鄂爾多斯三種強旱生植物在礦區治理中的生理生態適應性[D]. 呼和浩特: 內蒙古大學, 2019.
[8] ABDEL KHALIK K N. A numerical taxonomic study of the family Zygophyllaceae from Egypt[J]. Acta Botanica Brasilica, 2012, 26(1): 165-180.
[9] BEIER B A, CHASE M W, THULIN M. Phylogenetic relationships and taxonomy of subfamily Zygophylloideae (Zygophyllaceae) based on molecular and morphological data[J]. Plant Systematics and Evolution, 2003, 240(1/4): 11-39.
[10] 韓燕燕. 基于核糖體ITS和葉綠體trnL-F基因片段的蒺藜科植物分子系統學研究[D]. 烏魯木齊: 新疆大學, 2007.
[11] 張辰波, 史雪松, 宛濤, 等. 內蒙古駝蹄瓣屬植物親緣關系的RAPD分析[J]. 中國草地學報, 2006, 28(5): 86-90.
[12] HAHN C, BACHMANN L, CHEVREUX B. Reconstructing mitochondrial genomes directly from genomic next-generation sequencing reads—a baiting and iterative mapping approach[J]. Nucleic Acids Research, 2013,41(13): e129.
[13] LOHSE M, DRECHSEL O, KAHLAU S, et al. Organellar Genome DRAW—a suite of tools for generating physical maps of plastid and mitochondrial genomes and visualizing expression data sets[J]. Nucleic Acids Research, 2013, 41(Web Server issue): W575-W581.
[14] KUMAR S, STECHER G, TAMURA K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[Z]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[15] WU M, LAN S, CAI B, et al. The complete chloroplast genome of guadua angustifolia and comparative analyses of Neotropical-Paleotropical bamboos[J]. PLOS One, 2015, 10(12): e0143792.
[16] DANIELL H, LIN C S, YU M, et al. Chloroplast genomes: diversity, evolution, and applications in genetic engineering[J]. Genome Biology, 2016, 17(1): 134.
[17] 路東曄, 張磊, 郝蕾, 等. 臭柏葉綠體基因組結構與系統進化分析[J]. 西北植物學報, 2018, 38(8): 1464-1475.
[18] WANG R J, CHENG C L, CHANG C C, et al. Dynamics and evolution of the inverted repeat-large single copy junctions in the chloroplast genomes of monocots[J]. BMC Evolutionary Biology, 2008, 8: 36.
[19] 趙志常, 高愛平, 黃建峰, 等. 黃皮葉綠體基因組測序與分析[J]. 安徽農業科學, 2019, 47(11): 115-118.
[20] 中國科學院中國植物志編輯委員會. 中國植物志: 第43卷[M]. 北京: 科學出版社, 1997.
[21] 劉文哲, 趙鵬. APGⅣ系統在植物學教學中的應用初探[J]. 高等理科教育, 2017(4): 104-109.

