同源建模和分子對接揭示甜菊糖苷糖基化關鍵酶SrUGT76G1的構效關系
張紹山,楊云舒,呂承承,陳靳松,莊 藝,劉 圓,吳 衛*
? 藥材與資源 ?
同源建模和分子對接揭示甜菊糖苷糖基化關鍵酶SrUGT76G1的構效關系
張紹山1, 2, 3,楊云舒2,呂承承2,陳靳松2,莊 藝2,劉 圓1, 3,吳 衛2*
1. 西南民族大學青藏高原研究院,四川 成都 610041 2. 四川農業大學農學院,四川 成都 611130 3. 四川省羌彝藥用資源保護與利用技術工程實驗室,四川 成都 610225
揭示甜菊糖苷糖基化關鍵酶SrUGT76G1的構效關系。采用RT-PCR法克隆各甜葉菊材料中的變異序列;采用原核表達系統—大腸桿菌和體外催化反應驗證各變異序列功能;采用Modeller 9.17軟件對SrUGT76G1各變異序列蛋白進行同源建模,利用AutoDock Vina工具進行對接分析。除變異序列序列和序列外,還分別從110和B1188材料中獲取了2個變異序列序列和序列。各變異序列蛋白體外催化反應和催化效率比較分析結果表明,N02蛋白相較于UGT76G1(N01-1,AY345974.1)活性顯著下降,特別是以甜茶苷、甜菊糖苷(stevioside,ST)和萊鮑迪苷D(rebaudioside D,RD)為糖基受體底物時,B1188、N03和110-3蛋白對糖苷的糖基化功能已喪失;同源建模和分子對接分析發現由于各變異序列蛋白中氨基酸殘基的改變,導致翻譯形成的蛋白三維空間結構改變,導致無法完全包裹催化底物。SrUGT76G1形成的活性口袋能否較好地包裹糖基受體底物是其活性強弱和有無的關鍵。
甜葉菊;甜菊糖苷;SrUGT76G1;同源建模;分子對接
甜葉菊(Bertoni) Hemsl.為菊科(Asteraceae)甜葉菊屬L.多年生草本植物,為藥食兩用植物,主要藥食部位為干燥葉片,是繼甘蔗、甜菜之后的第3種天然甜味劑[1-2]。我國目前已成為世界第一大甜葉菊種植國、生產國和出口國,約占世界市場的90%[3]。甜葉菊葉片的甜味主要來源于甜菊糖苷(stevia glycosides,SGs),其混合物甜度相當于蔗糖的250~300倍,但所含熱量僅為蔗糖的1/300,故具有高甜度、低熱量等優點[4-7]。目前,甜菊糖苷的安全性已得到聯合國糧食及農業組織(Food and Agriculture Organization of the United Nations,FAO)和世界衛生組織(World Health Organization,WHO)等國際組織的認可,是飲料、食品、醫藥及日用化工等領域的理想甜味替代劑[8-11]。我國衛生部于1985年和1990年分別批準甜菊糖苷作為不限量使用的天然甜味劑和醫藥用甜味劑輔料[12]。
甜菊糖苷代謝的糖基化途徑中,糖基轉移酶SrUGT76G1被鑒定能催化甜菊糖苷形成1,3-β--葡萄糖苷鍵,參與包括甜茶苷、甜葉菊苷B(rebaudioside B,RB)、甜葉菊苷A(rebaudioside A,RA)和甜葉菊苷M(rebaudioside M,RM)的形成[13-14]。通過RNA干擾、堿基突變、體外酶活性分析、同源建模和分子對接等方法研究的功能和分子機制,充分證明了在甜菊糖苷糖基化步驟中的重要作用。農桿菌介導的瞬時基因沉默降低了基因的表達,導致總糖苷含量顯著下降,表明該酶的功能對糖苷含量有很大的作用[15]。根據氨基酸列相似性對目的蛋白進行同源建模,并將蛋白和底物進行分子對接,是一種被證明可以有效識別位于蛋白活性口袋(底物結合位點)中目標氨基酸殘基的方法[16-17]。
課題組利用逆轉錄-聚合酶鏈反應(reverse transcription-polymerase chain reaction,RT-PCR)的方法從糖苷含量有差異的不同基因型甜葉菊材料中克隆分析了基因,最終獲的了該基因的5個變異序列~序列,其中~序列可形成完整的蛋白結構,且其功能存在顯著差異[18]。在一定程度上揭示了該基因序列變異導致其蛋白空間構象的改變,進而導致其催化功能的差異,但受限于模板蛋白同源性較低(ID=30%),無法獲取各SrUGT76G1變異序列蛋白的高質量三維構象[18]。幸運的是,2019年底Yang等[19]解析了SrUGT76G1(AY345974.1)蛋白的三維結構,這為通過同源建模構建各SrUGT76G1變異序列的高質量蛋白三維結構提供了保障。因此,本研究利用已發表的的2個變異序列序列和序列[18],以及利用RT-PCR方法從其他甜菊糖苷含量不同的甜葉菊材料中克隆基因,分析其序列差異并篩選變異序列;通過在大腸桿菌系統中表達重組蛋白,鑒定不同變異序列的功能。在此基礎上,以SrUGT76G1(AY345974.1)為模板蛋白,采用同源建模和分子對接的方法探究SrUGT76G1的構效關系,以期進一步揭示基因催化糖苷糖基化的機制并為獲取更加高效的變異序列提供支撐。
1 材料與試劑
1.1 材料
課題組前期研究基因的2個變異序列序列和序列[18]。甜葉菊材料經四川農業大學吳衛教授鑒定為甜葉菊(Bertoni) Hemsl.,命名為“110”和“B1188”,材料種植于四川農業大學成都校區實驗圃。
1.2 試劑
大腸桿菌菌株DH5α和BL21(DE3)(TsingKe公司,北京);克隆載體Pclone007 blunt vecter(TsingKe公司,北京);原核表達載體pET-28a(+)由本實驗室保存;總RNA提取試劑盒-E.Z.N.A.?Plant RNA Kit(OMEGA Bio-Tek,美國);反轉錄試劑盒-HiScript?II Q RT SuperMix for qPCR(+gDNA wiper)Kit(Vazyme公司,南京);基因克隆試劑盒-Phanta Max Super-Fidelity DNA Polymerase(Vazyme公司,南京);酶切所用酶-H I和d III(Takara公司,日本);DNA膠回收試劑盒-Gel Extraction Kit(100)(OMEGA公司,美國);同源重組試劑盒-ClonExpressTM II One Step Cloning Kit(Vazyme公司,南京);聚丙烯酰氨凝膠電泳試劑盒-10%聚丙烯酰氨凝膠電泳預混液試劑盒(新賽美公司);50 mg/mL異丙基-β--硫代半乳糖苷(IPTG)溶液(Solarbio公司,北京)。
2 方法
2.1 RT-PCR克隆甜葉菊材料中SrUGT76G1變異序列
RT-PCR克隆及其變異序列篩選參照Zhang等[18]報道相關方法。
2.2 各變異序列功能驗證及其催化效率比較
由于SrUGT76G1參與糖苷1,3-β--葡萄糖苷鍵的形成,因此選擇雙糖苷甜菊雙糖苷(steviolbioside)和甜茶苷、3糖苷甜菊糖苷(stevioside,ST)以及5糖苷萊鮑迪苷D(rebaudioside D,RD)分別作為底物,其中甜菊雙糖苷理論上可在其C13位的第1個和第2個葡萄糖基上形成1,3-β--葡萄糖苷鍵,甜茶苷和ST理論上可在其C13和C19位的第1個葡萄糖基上形成1,3-β--葡萄糖苷鍵,RD理論上可在其C19位的第1個葡萄糖基上形成1,3-β--葡萄糖苷鍵(圖1)。這4種糖苷底物可實現SrUGT76G1的各變異序列對糖苷C13和C19催化偏好以及糖基數量對其催化效率影響的考察。
參照Wang等[20]在比較其克隆到的3個變異序列催化效率時所用的方法,即將收集的各變異序列的誘導菌體經PBS緩沖液重懸,并經PBS稀釋至菌體質量濃度為50 mg/mL,然后準確吸取一定體積的重懸菌液進行破碎,破碎方法一致,以保證獲取的重組目的蛋白量一致,破碎液離心后,取350 μL上清液,6.25 μL、40 mmol/L的尿苷二磷酸葡萄糖(uridine 5′-diphosphoglucose-[glucose-1-3H],UDPG)、75 μL2.5 mmol/L的SGs于1.5 mL離心管中,30 ℃、200 r/min下誘導1 h,然后立即加入等體積水飽和的正丁醇終止反應并萃取產物,萃取液減壓旋干后加入等體積的色譜甲醇溶解,經0.22 μm濾膜過濾后按以下高效液相色譜(HPLC)法檢測:色譜柱TSKgel Amide-80 column(250 mm×4.6 mm,5 μm,TOSOH,日本);柱溫40 ℃;檢測波長210 nm;流動相體積流量1.0 mL/min;進樣量10 μL;流動相為二元洗脫體系乙腈(A)-0.1%磷酸水(B),洗脫梯度:0~5.0 min,10%~15% B;5.0~16.0 min,15%~25% B;16.0~22.0 min,25%~30% B;22.0~40.0 min,30% B[18]。產物與糖苷標準品進行比較確認,以產物的相對峰面積代表產物的產量,以SrUGT76G1(AY345974.1)催化的產物峰面積為分母,其余變異序列對應的催化產物峰面積為分子,計算各變異序列較SrUGT76G1(AY345974.1)的催化效率。
2.3 變異序列的同源建模及其與甜菊糖苷對接
利用Yang等[19]解析的SrUGT76G1(AY345974.1)蛋白三維結構,在PDB蛋白數據庫(https://www.rcsb.org/)中下載PDB號為“6inh”的SrUGT76G1,該蛋白復合體包括了底物(甜茶苷)和二磷酸尿苷(uridine diphosphate,UDP),以該蛋白的結構為模型,采用Modeller 9.17軟件對SrUGT76G1各變異序列進行同源建模。根據molpdf、DOPE和GA341 potential評分選擇最好的模型。通過在線(http://servicesn.mbi.ucla. edu/ SAVES/)檢查模型的幾何形狀和質量。甜菊雙糖苷、甜茶苷、ST和RD均是SrUGT76G1的糖基化底物,其中催化ST生成RA是最重要的反應,因此分子對接中采用ST為糖基受體,利用Chem3D軟件生成ST,并優化其三維結構。利用AutoDock Vina工具進行對接分析,對接時根據“6inh”中配體的坐標位置來確定配體坐標[19]。使用PyMOL軟件對結果進行分析。
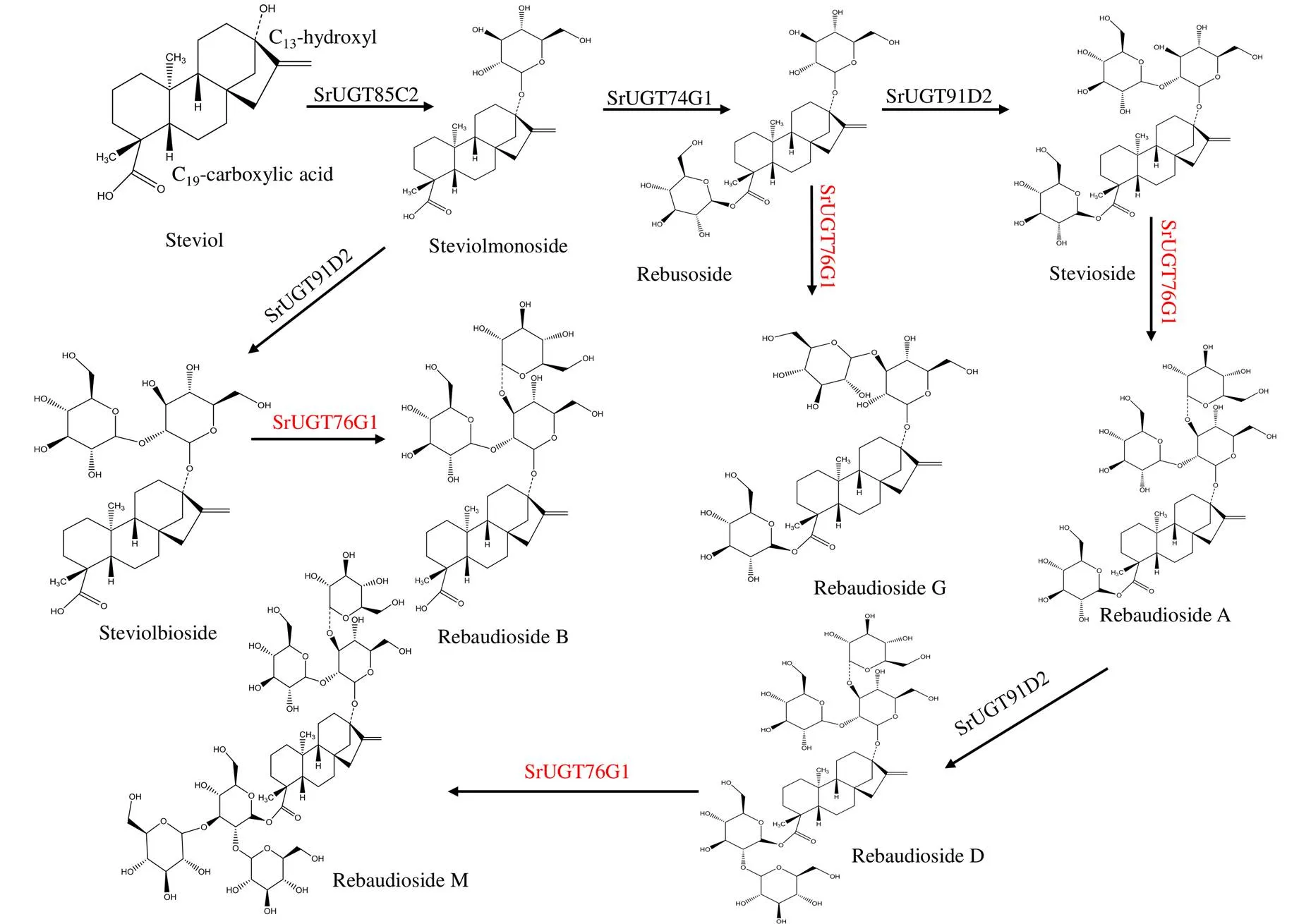
圖1 SrUGT76G1在甜菊醇糖基化代謝網絡中的合成路線
3 結果與分析
3.1 SrUGT76G1基因在110和B1188材料中序列比較分析
利用特異性引物,通過RT-PCR從110和B1188材料中克隆了基因的開放閱讀框(open reading frame,ORF)。通過構建到pclone007 blunt vector后測序,成功獲得了2份不同基因型材料葉片組織中的,擴增產物大小約為1.4 kb,與預期一致。
將2份材料中獲得的堿基序列與已研究報道的(AY345974.1)的比對分析發現:送測序的B1188材料中獲得的單克隆,分析發現其中3個單克隆序列一致,但較AY345974.1序列發生了109個堿基的替換,以及在346位缺失了“AAGAGG”6個堿基,在這115個單核苷酸多態性(single nucleotide polymorphisms,SNP)中,沒有引起氨基酸翻譯的提前終止,用序列代表該差異序列;從110材料中獲得的單克隆測序后發現1個單克隆序列較AY345974.1序列觀察到了121個SNPs,包括在346位缺失了“AAGAGG”6個堿基,但沒有導致蛋白翻譯提前終止,用序列代表該差異序列。
包括課題組前期發表文章中的2個變異序列序列和序列在內,共獲得了4個能形成完整蛋白的變異序列,分別是、、和序列,堿基序列經NCBI的ORFfinder(www.ncbi.nlm.nih.gov/orffinder/)翻譯,進一步確定各變異序列均能形成包括C端和N端在內的完整蛋白(圖2)。利用DNAMAN軟件進行序列分析,確定以上各變異序列與(AY345974.1)的核苷酸相似度在92%~98%(圖3)。

彩色背景的為各變異序列間的差異殘基

彩色背景的為各變異序列間差異堿基
3.2 SrUGT76G1變異序列的功能及催化效率比較分析
各SrUGT76G1變異序列菌體破碎離心后上清液的十二烷基硫酸鈉聚丙烯酰胺凝膠(Sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)檢測重組目的蛋白表達情況結果見圖4,由圖可得各變異序列均成功誘導表達出重組目的蛋白。具有催化活性的變異序列破碎液催化雙糖苷steviobioside和甜茶苷、3糖苷ST和5糖苷RD的HPLC色譜圖見圖5,由于無法購得瑞鮑迪苷(rebaudioside G,RG),參照其同分異構體RB苷的保留時間對其進行確認。由圖可知,N01-1蛋白和N02蛋白均可催化steviobioside形成RB苷和ST形成RA,N01-1蛋白可催化甜茶苷形成RD和催化RD形成少量的RM,而N02蛋白催化甜茶苷和RD的產物中沒有檢測到對應產物;N01-1蛋白催化甜茶苷苷的產物中,僅檢測到了C13位糖基化的RD,而C19位糖基化的產物沒有檢測到,在催化ST的產物中也發現了同樣的現象即沒有檢測到C19位進一步糖基化的產物;而N03、110-3和B1188蛋白均沒有檢測到對所用糖苷底物的催化活性。具有催化功能的N01-1和N02蛋白催化4種底物的催化效率分析結果見表1,可以看出,N02和N01-1蛋白催化steviobioside的效率無顯著差異,而N02蛋白催化ST的效率顯著低于N01-1蛋白。
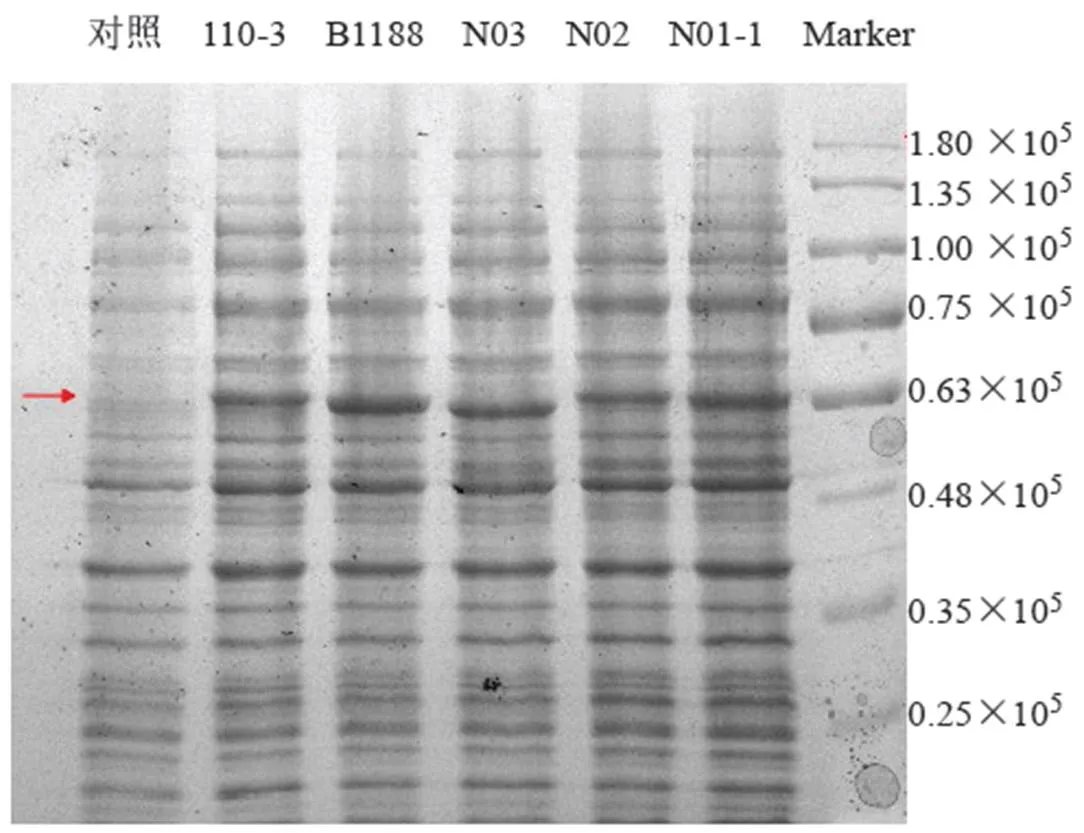
箭頭指示重組目的蛋白
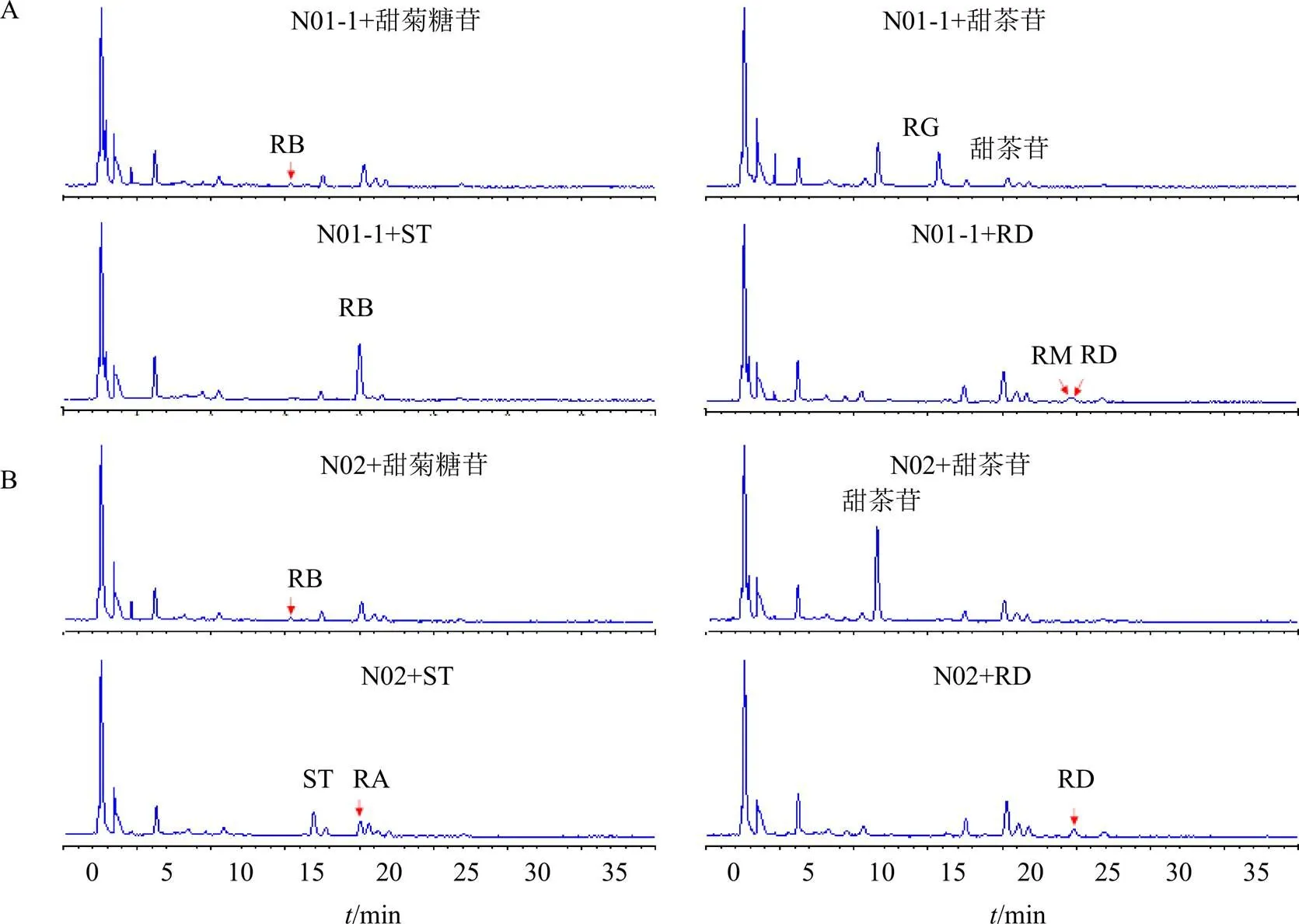
圖5 N01-1 (A)和N02(B)重組蛋白分別與各糖苷底物催化反應的HPLC檢測結果
3.3 同源建模及分子對接揭示SrUGT76G1變異序列功能差異
為了解這些氨基酸缺失、替換如何影響糖基化過程,導致酶活性的差異,對SrUGT76G1同拷貝進行了同源建模,并將這些模型與糖基受體底物ST進一步對接分析。結果顯示SrUGT76G1變異序列蛋白的三級結構包括典型的由β折疊和α螺旋構成的面對面的2個Rossmann結構域,這是GT-B折疊型糖基轉移酶的特征。將這4個SrUGT76G1變異序列的模型結構與建模模板進行疊加,表明C-末端的三級結構高度保守;N-端區域變化更大,說明它負責結合多變的底物受體。此外,4個變異序列的模型疊加后發現:N02蛋白與模板蛋白SrUGT76G1(AY345974.1)即N01蛋白疊加后二者除部分β折疊和loop區外,其余部分幾乎能完全疊加,特別是在C端,這與二者均有催化活性一致;無催化活性的N03蛋白和SrUGT76G1(AY345974.1)疊加后除少部分非活性口袋處有loop區外,其余部位亦能良好的重疊;110-3蛋白和SrUGT76G1(AY345974.1)疊加后可看出它們間重疊性較差,特別是在N端,且不能正確折疊的loop區更多,說明較SrUGT76G1(AY345974.1)其蛋白構型發生了顯著的變化,這與該變異序列的蛋白無糖苷催化活性一致;B1188蛋白和SrUGT76G1(AY345974.1)疊加后可看出它們間的重疊性也較差,同樣的在N端的重疊性最差,以及不能正確折疊的loop區更多,說明較SrUGT76G1(AY345974.1)其蛋白構型也發生了顯著的變化,這與該變異序列的蛋白無糖苷催化活性一致(圖6)。

表1 SrUGT76G1各變異序列重組蛋白催化各糖苷底物的相對催化效率
“?”表示沒有檢測到
“?” means no detection
在Yang等[20]的研究中,由于去質子化的甜菊糖苷對UDPG的親核反應太快,只能結晶到包含糖苷底物和UDP的SrUGT76G1晶體,但UDP的三維坐標仍然能反應出UDPG在該蛋白中的結合情況。因此,進一步將構建好的各變異序列的蛋白模型與ST和UDP對接分析,結果顯示,各變異序列的活性口袋均有明顯差異,且差異的大小與氨基酸序列相似性趨勢一致,特別是結合ST的活性口袋差異巨大,具體差異和分析如下:N01-1蛋白與ST和UDP對接后可以看到N01-1蛋白形成的活性口袋較好地包裹了配體和底物,且底物反應基團的位置在同一個方向(圖7-A),對識別ST底物的活性口袋分析,可以看到F22、P84、L85、G87、M88、I90、L126、W197、I199、L200、I203和L204構成的疏水口袋包裹著ST的甜菊醇母核和C19羧基上的葡萄糖基,而S283、T284、H356和H25與C13上的葡萄糖基形成了直接的相互作用,將STC13位上的反應基團錨定在于UDPG糖基供體中葡萄糖基相同的方向,從而讓糖基化反應順利進行(圖7-B);N02蛋白與ST和UDP對接后可以看到N02蛋白形成的活性口袋亦較好的包裹了配體和底物,且底物反應基團的位置也在同一個方向(圖7-C),對識別ST底物的活性口袋分析,可以看到F22、P84、L85、G87、M88、I90、L126、W197、I199、L200、I203、L204和G379構成的疏水口袋包裹著ST的甜菊醇母核和C19羧基上的葡萄糖基,這與N01-1蛋白的情況一致,而H357和Q382與C13上第1個葡萄糖基C4上的羥基形成了直接的相互作用,將STC13位上的反應基團(第1個葡萄糖基C3上的羥基)錨定在于UDPG糖基供體中葡萄糖基相同的方向,從而讓糖基化反應順利進行(圖7-D);N03蛋白與ST和UDP對接后,可以看到N03蛋白形成的活性口袋仍然能較好的包裹配體和底物,且底物反應基團的位置也在同一個方向(圖8-A),對識別ST底物的活性口袋分析,可以看到相比具有催化活性的N01-1和N02蛋白,N03蛋白由于氨基酸替換后不再構成疏水性的口袋,其中K199側鏈上的氨基以及L85和M88中羧基上的羰基與ST C19位羧基上的葡萄糖基形成了氫鍵,這可能導致了ST不能轉動,從而無法與UDPG完成糖基化反應(圖8-B);110-3蛋白與ST和UDP對接后,可以看到雖然ST和UDPG底物反應基團的位置仍然在同一個方向,但110-3蛋白形成的活性口袋已經不能完整的包裹底物,ST中C19位上的葡萄糖已經裸露在蛋白外面(圖8-C),對識別ST底物的活性口袋分析,可以看到110-3蛋白由于疏水性的口袋不能完整包裹ST,導致了ST與UDPG的距離較具有催化活性的N01-1和N02蛋白的更遠,這可能導致了C13位上的葡萄糖基無法與UDPG完成糖基化反應(圖8-D);B1188蛋白與ST和UDP對接后,可以看到ST的位置發生了顯著的變化,ST中C13位上的參與糖基化的葡萄基已經遠離了UDPG(圖9-A),對識別ST底物的活性口袋分析,可以看到構成疏水口袋的殘基較之前的已經發生了顯著變化,導致口袋的形狀和大小也發生改變,最終導致ST僅能在S87和E284極性相互作用下固定在活性口袋口,這可能導致了該變異序列的蛋白活性喪失(圖9-B)。
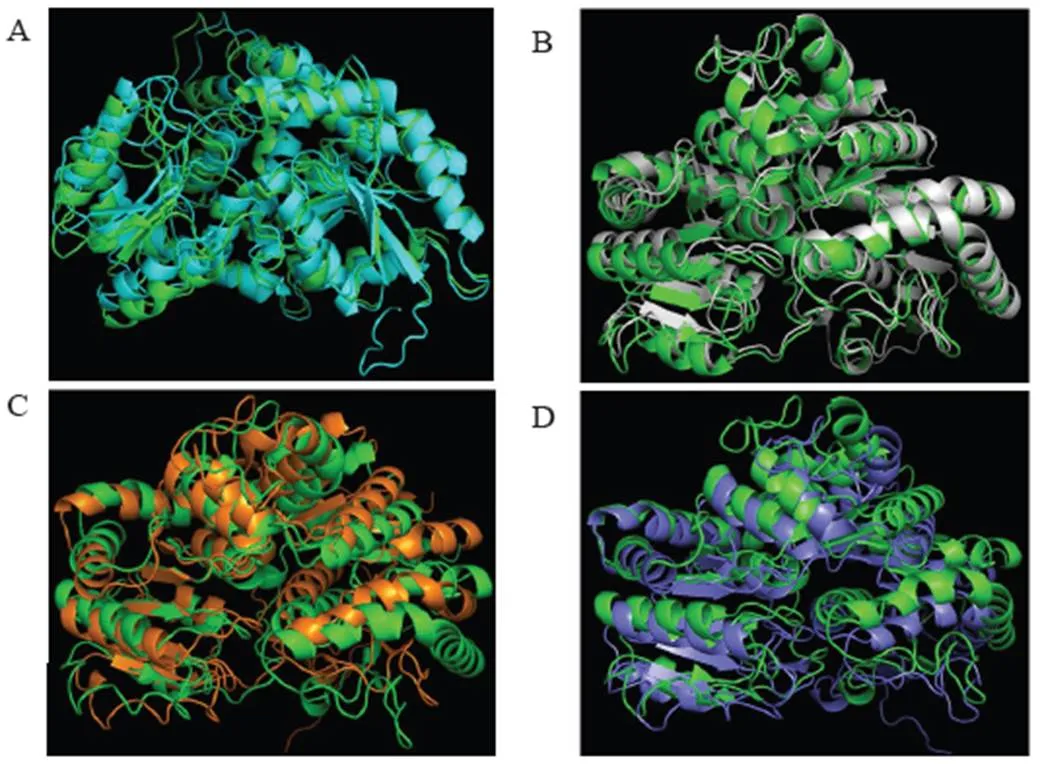
綠色為SrUGT76G1(6INH)、青色為N02、灰色為N03、菊紅色為110-3、天藍色為B1188
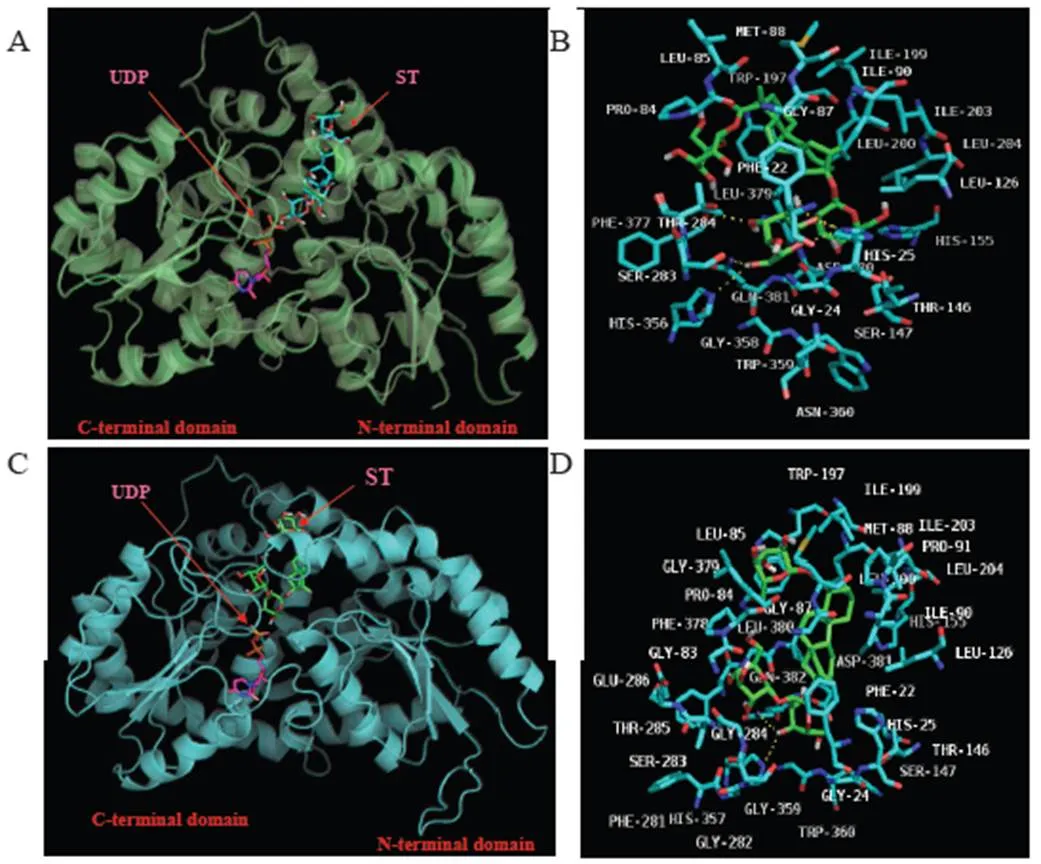
A-N01-1蛋白與ST和UDP對接后整體結構圖 B-A圖中識別ST底物的活性口袋 C-N02蛋白與ST和UDP對接后整體結構圖 D-C圖中識別ST底物的活性口袋
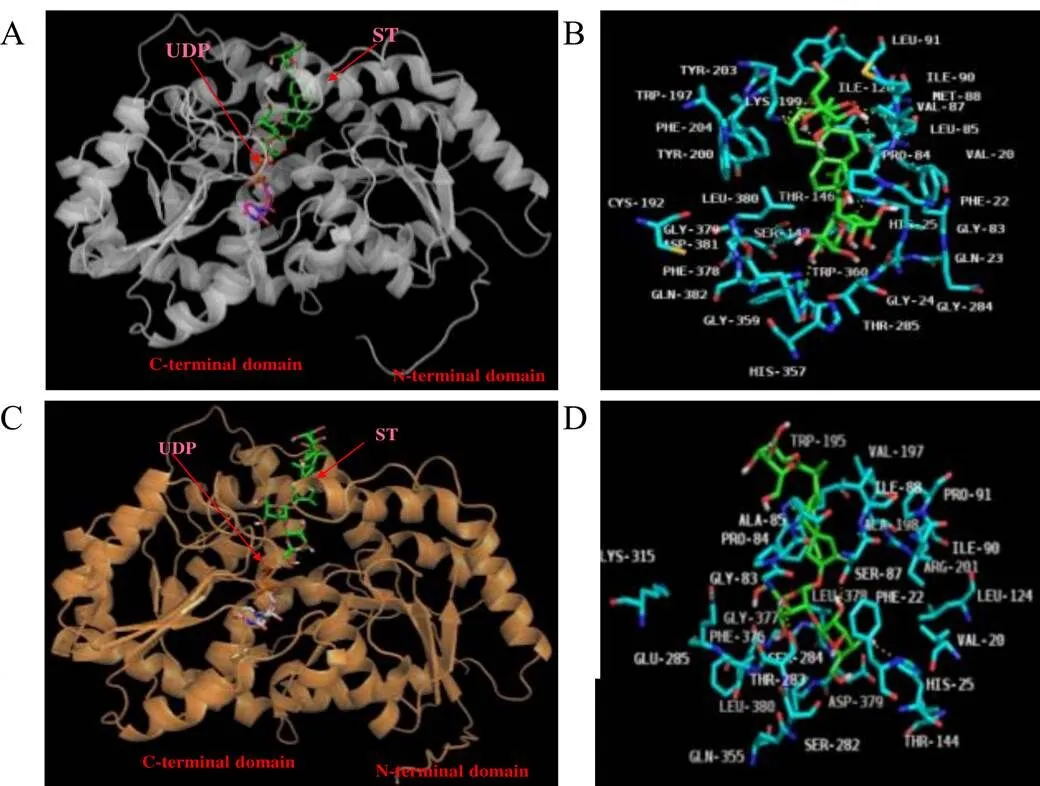
A-N03蛋白與ST和UDP對接后整體結構圖 B-A圖中識別ST底物的活性口袋 C-110-3蛋白與ST和UDP對接后整體結構圖 D-C圖中識別ST底物的活性口袋
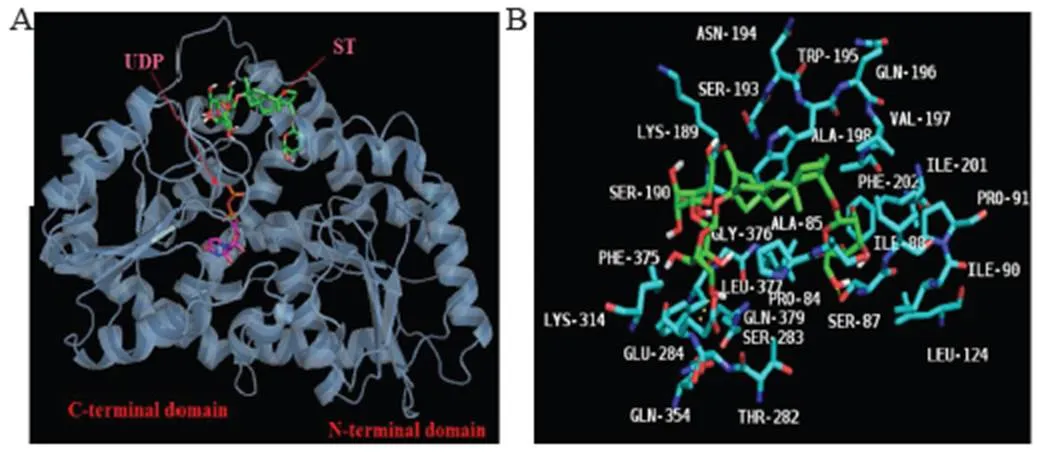
A-與ST和UDP對接后整體結構圖 B-A圖中識別ST底物的活性口袋
4 討論
各SrUGT76G1變異序列的體外糖苷催化結果說明,SrUGT76G1是一個可識別多種糖苷底物的酶,氨基酸序列的改變對其功能有重要影響,其對各底物的催化偏好依賴于糖苷中糖基化基團的位置和糖苷上糖基的數量,具體表現為更偏好糖基化C13位羥基上的第1個葡萄糖,更偏好糖基化糖基數目相對較少的糖苷。N02蛋白對已連有1,2-β--葡萄糖基的糖苷(甜菊雙糖苷和ST)仍具有催化活性,而對沒有1,2-β--葡萄糖基的糖苷(甜茶苷和RD)則喪失了活性,說明SrUGT76G1更偏好糖基化已連有1,2-β--葡萄糖基的糖苷,這與Kishore等[14]和Houghton-Larsen等[21]的研究結果一致。此外,也說明甜葉菊中(AY345974.1)是經長期進化和自然選擇的具有高效催化效率的序列。
據前人對甜葉菊中基因的序列分析研究結果,該基因在甜葉菊存在著廣泛的序列變異,且這些變異對該基因的功能有顯著影響[22],這與本研究中由于序列的差異導致N02、N03、110-3和B1188等蛋白活性喪失一致。本研究中變異序列N01-1、N02和N03蛋白氨基酸序列相似度較高,但功能差異較大,其中N01-1(AY345974.1)蛋白和N02蛋白對于steviobioside和ST具有相同的功能,說明N03、110-3和B1188酶活性喪失的主要原因可能不是和序列中的SNPs引起。在分子對接分析中,構成SrUGT76G1疏水口袋的殘基是否有極性的變化,殘基的差異是否導致活性口袋的形狀和大小改變,活性口袋是否能較好的包裹糖基受體底物是該酶能否催化糖苷糖基化的關鍵;SrUGT76G1酶活的有無與強弱受蛋白的整體結構影響更大,可能單個殘基的改變不會顯著提高其催化效率,但卻可能導致其活性的喪失。2016年Olsson等[13]利用其他物種中已解析三維結構的糖基轉移酶和SrUGT76G1的氨基酸序列,構建了SrUGT76G1的蛋白模型,與糖苷底物和UDPG對接分析后識別到了49個活性口袋的氨基酸殘基,進一步構建了一個包含1748個SrUGT76G1變異序列庫,酶活驗證后鑒定出了一些關鍵殘基。充分證明利用同源建模和分子對接技術能較好的預測和揭示甜葉菊SrUGT76G1蛋白的三維空間結構和構效關系。
綜上所述,本研究基于功能有差異的各SrUGT76G1變異序列,采用同源建模和分子對接的方法揭示了其結構和功能之間的關系,這為今后通過分子設計來改變其空間結構,從而獲取高效變異序列積累了一定的科學數據。
利益沖突 所有作者均聲明不存在利益沖突
[1] Yadav A K, Singh S, Dhyani D,. A review on the improvement of[(Bertoni)] [J]., 2011, 91(1): 1-27.
[2] 萬會達, 李丹, 夏詠梅. 甜菊糖苷類物質的功能性研究進展 [J]. 食品科學, 2015, 36(17): 264-269.
[3] 王亞平, 李世光, 趙金慶, 等. 中國甜菊糖產業現狀及其應對 [J]. 對外經貿, 2013(10): 4-6.
[4] Bondarev N I, Sukhanova M A, Reshetnyak O V,. Steviol glycoside content in different organs ofand its dynamics during ontogeny [J]., 2003, 47(2): 261-264.
[5] 趙興華, 張全香, 苑曉威, 等. 大孔吸附樹脂純化甜菊糖苷的工藝研究 [J]. 中草藥, 2012, 43(10): 1971-1974.
[6] Brahmachari G, Mandal L C, Roy R,. Stevioside and related compounds-molecules of pharmaceutical promise: A critical overview [J]., 2011, 344(1): 5-19.
[7] Jeppesen P B, Gregersen S, Alstrup K K,. Stevioside induces antihyperglycaemic, insulinotropic and glucagonostatic effects: Studies in the diabetic Goto- Kakizaki (GK) rats [J]., 2002, 9(1): 9-14.
[8] Takahashi K, Matsuda M, Ohashi K,. Analysis of anti-activity of extract from[J]., 2001, 49(1): 15-24.
[9] Hong J, Chen L, Jeppesen P B,. Stevioside counteracts the alpha-cell hypersecretion caused by long-term palmitate exposure [J]., 2006, 290(3): E416-E422.
[10] Shukla S, Mehta A, Mehta P,. Antioxidant ability and total phenolic content of aqueous leaf extract ofBert [J]., 2012, 64(7/8): 807-811.
[11] Adebiyi O E, Olayemi F O, Tan N H,.antioxidant activity, total phenolic and flavonoid contents of ethanol extract of stem and leaf of[J]., 2017, 6(1): 10-14.
[12] Jeppesen P B, Gregersen S, Alstrup K K,. Stevioside induces antihyperglycaemic, insulinotropic and glucagonostatic effects: Studies in the diabetic Goto-Kakizaki (GK) rats [J]., 2002, 9(1): 9-14.
[13] Olsson K, Carlsen S, Semmler A,. Microbial production of next-generationsweeteners [J]., 2016, 15(1): 207.
[14] Mao G H, Yu X D. Recombinant production of steviol glycosides: US, 10081826 [P]. 2018-09-25.
[15] Guleria P, Yadav S K.mediated transient gene silencing (AMTS) in: Insights into steviol glycoside biosynthesis pathway [J]., 2013, 8(9): e74731.
[16] Osmani S A, Bak S, Imberty A,. Catalytic key amino acids and UDP-sugar donor specificity of a plant glucuronosyltransferase, UGT94B1: Molecular modeling substantiated by site-specific mutagenesis and biochemical analyses [J]., 2008, 148(3): 1295-1308.
[17] Hans J, Brandt W, Vogt T. Site-directed mutagenesis and protein 3D-homology modelling suggest a catalytic mechanism for UDP-glucose-dependent betanidin 5-O-glucosyltransferase from[J]., 2004, 39(3): 319-333.
[18] Zhang S S, Chen H, Xiao J Y,. Mutations in the uridine diphosphate glucosyltransferase 76G1 gene result in different contents of the major steviol glycosides in[J]., 2019, 162: 141-147.
[19] Yang T, Zhang J Z, Ke D,. Hydrophobic recognition allows the glycosyltransferase UGT76G1 to catalyze its substrate in two orientations [J]., 2019, 10(1): 3214.
[20] Wang J F, Li S Y, Xiong Z Q,. Pathway mining-based integration of critical enzyme parts for de novo biosynthesis of steviolglycosides sweetener in[J]., 2016, 26(2): 258-261.
[21] Houghton-Larsen J, Hicks P M, Naesby M, et al. Recombinant production of steviol glycosides, US Patent WO 2013/022989 A2.
[22] Yang Y H, Huang S Z, Han Y L,. Base substitution mutations in uridinediphosphate-dependent glycosy ltransferase 76G1 gene ofcauses the low levels of rebaudioside A: Mutations in UGT76G1, a key gene of steviol glycosides synthesis [J]., 2014, 80: 220-225.
Characterizing structure-activity relationship of key glycosyltransferase SrUGT76G1 by homologous modeling and molecular docking
ZHANG Shao-shan1, 2, 3, YANG Yun-shu2, LYU Cheng-cheng2, CHEN Jin-song2, ZHUANG Yi2, LIU Yuan1, 3,WU Wei2
1. Institute of Qinghai-Tibetan Plateau, Southwest Minzu University, Chengdu 610041, China 2. Agronomy College, Sichuan Agricultural University, Chengdu 611130, China 3. Sichuan Provincial Qiang-Yi Medicinal Resources Protection and Utilization Technology Engineering Laboratory, Chengdu 610225, China
To reveal the structure-activity relationship of SrUGT76G1, which was a key glycosyltransferase involving the glucosylation of steviol glycosides.RT-PCR was used to clone thefrom differentmaterials. The prokaryotic expression systemandcatalytic reaction were used to verify the function of the SrUGT76G1 variants. Modeller 9.17 software was used to model the homology of SrUGT76G1 variants, and the docking analysis was performed with AutoDock Vina tool.In addition toand, two variantsandwere obtained from 110 and B1188 materials respectively. Compared with UGT76G1 (N01-1, AY345974.1), the activity of N02 protein was significantly lower than that of SrUGT76G1, especially using rubusoside, stevioside (ST) and rebaudioside D (RD) as receptor substrates. And the enzyme activities of B1188, N03 and 110-3 variants were lost. Homology modeling and molecular docking analysis showed that the change of amino acid residues in each variant resulted in the change of the three-dimensional structure of the SrUGT76G1 protein, resulting in the failure to completely wrap the substrates.Whether the active pocket formed by SrUGT76G1 can wrap the receptor substrate well is the key to its activity.
(Bertoni) Hemsl.; steviol glycoside; SrUGT76G1; homologous modeling; molecular docking
R286.12
A
0253 - 2670(2021)24 - 7606 - 10
10.7501/j.issn.0253-2670.2021.24.023
2021-05-09
國家自然科學基金資助項目(31671757);西南民族大學人才類(自科)-引進人才科研啟動金資助項目(16011211073)
張紹山(1990—),博士,助理研究員,主要從事藥用植物栽培、質量評價和次生代謝產物調控研究。E-mail: 1007354550@qq.com
吳 衛,博士,教授,博士生導師,從事藥用植物次生代謝產物調控研究。E-mail: ewuwei@sicau.edu.cn
[責任編輯 時圣明]

