小麥籽粒硬度QTL的分析
周淼平 楊學明 張鵬 張平平 姚金保 何漪


摘要:小麥籽粒硬度是小麥分類和定級的重要標準,會影響小麥面粉的最終用途。為了對小麥籽粒硬度的分子設計育種提供支撐,采用CI12633和揚麥158配制的重組自交系群體,進行連續3年種植和籽粒硬度分析,結合該群體遺傳連鎖圖,對影響小麥籽粒硬度的數量性狀座位(QTL)進行分子定位。結果表明,小麥3D、5B、6A、6D和7A染色體上均存在影響小麥籽粒硬度的QTL,單個QTL可以解釋11.3%~25.1%的表型變異;其中6A和6D染色體上的QTL由親本CI12633貢獻,3D、5B和7A染色體上的QTL則來自親本揚麥158;3D、6A、6D和7A染色體為新發現的QTL。這些QTLs可為長江中下游麥區的軟質弱筋小麥和硬質中強筋小麥的培育提供參考。
關鍵詞:小麥;籽粒硬度;數量性狀座位(QTL);分子設計育種
中圖分類號: S512.103? 文獻標志碼: A
文章編號:1002-1302(2022)10-0072-03
小麥籽粒硬度是小麥商品化分類、定級和定價的重要標準,籽粒硬度不僅影響小麥籽粒的出粉率及面粉加工的能耗,還會通過影響面粉的流變學特性進而影響面粉的加工品質。因此,開展小麥籽粒硬度遺傳機制的研究對于指導小麥品種改良具有極其重要的意義。
小麥籽粒的硬度受種植品種、種植環境及栽培措施的影響,其中品種基因型是主要影響因素[1]。研究發現,小麥籽粒硬度是由多基因控制的數量性狀,主要由位于5D染色體短臂Ha位點的Pina和Pinb基因決定,軟質對硬質為顯性,當2個基因均為野生型時,籽粒為軟質,當Pina缺失或Pinb發生突變時,籽粒表現為硬質[2]。除Ha位點外,在1A、1B、4B、5A、5B、5D和7A等染色體上也發現了影響小麥籽粒硬度的數量性狀座位(QTL)[3-7]。由于以往的QTL定位研究多采用多態性相對較低的簡單重復序列(SSR)標記,遺傳連鎖圖的基因組覆蓋率偏低,部分QTL不能被檢出,導致單個研究定位的QTL數目少、總體表型解釋率低。隨著小麥參考基因組組裝的完成及基因組測序技術的發展,高通量標記的開發及高密度遺傳圖譜的構建成為可能,小麥QTL的定位也愈來愈高效和準確。
本研究擬采用籽粒硬度差異明顯的小麥品種CI12633和揚麥158構建的重組自交系群體為材料,結合該重組自交系群體的高密度遺傳連鎖圖,定位影響小麥籽粒硬度的QTLs,以期為今后小麥品質設計育種提供參考。
1 材料與方法
1.1 重組自交系群體和遺傳連鎖圖的構建
1.1.1 親本及重組自交系群體的構建 CI12633品種是提莫菲維小麥的六倍體衍生系,該品系籽粒小,但籽粒硬度較高;揚麥158為江蘇里下河地區農業科學研究所培育的品種,該品種適應性廣,綜合抗病性好,曾在長江中下游地區大面積種植,籽粒硬度中等。
重組自交系群體以揚麥158為母本、CI12633為父本,采用單粒傳的方法培育,該群體含有198個家系,2017年已達第7代,本研究對其中94個家系進行基因型分析和籽粒硬度的測定。
1.1.2 遺傳連鎖圖的構建 采用二代測序方法開發該群體的單核苷酸多態性(SNP)標記,選取含有95%以上家系基因型的SNP標記,去除共分離分子標記,結合群體多態性SSR標記,用JoinMap 4.0軟件構建遺傳連鎖圖,詳見周淼平等的研究[8],該遺傳圖譜覆蓋2 510.7 cM,含有3 355個分子標記,包含31個連鎖群,根據SSR和小麥基因組信息,可以將所有連鎖群分配至相應的染色體上。
1.2 親本和重組自交系群體的籽粒硬度測定
親本和群體各家系于2017、2018、2019年種植于江蘇省農業科學院六合動物科學基地,行長 1.6 m,行寬0.25 m,每行均勻播種20粒種子,每個親本和家系間間隔1行,按常規管理,小麥成熟后收獲種子,曬干。每個親本和家系隨機取150粒籽粒,用Perten SKCS4100單籽粒硬度測定儀測定籽粒硬度指數,以硬度指數指示籽粒硬度,硬度指數數值越高,籽粒硬度越大。
1.3 數據分析和QTL定位
各個試驗重組自交系群體的籽粒硬度指數最小值、最大值和平均值及試驗間相關系數用R軟件計算。籽粒硬度QTL分析用MapQTL 5.0軟件[9],先用QTL區間作圖法(interval mapping)初步定位,然后用復合QTL作圖法(MQM Mapping)進一步進行詳細分析,根據置換檢測(permutation test)推薦的極大似然函數比的常用對數(likelihood of odd,LOD值)判定是否存在QTL。
2 結果與分析
2.1 親本和重組自交系群體籽粒硬度的測定結果
于2017、2018、2019年連續3年在江蘇省農業科學院六合動物科學基地對親本和重組自交系群體各家系進行試驗。由表1可以看出,親本CI12633的籽粒硬度與揚麥158差異明顯,3年的種植結果也進一步證實這一結果;CI12633、揚麥158的3年平均籽粒硬度指數分別為68.5、47.9;群體的籽粒硬度指數呈連續分布,且有超親現象,表明小麥籽粒硬度性狀是由多基因控制的數量性狀。
在本試驗中,小麥籽粒硬度的遺傳力較高,受環境的影響小,可以從群體各家系連續3年試驗的相關系數看出:2017年群體籽粒硬度指數與2018、2019年群體籽粒硬度指數間的相關系數均為0.8,2018年群體籽粒硬度指數與2019年群體籽粒硬度指數間的相關系數為0.7。
2.2 小麥籽粒硬度性狀的QTL定位結果
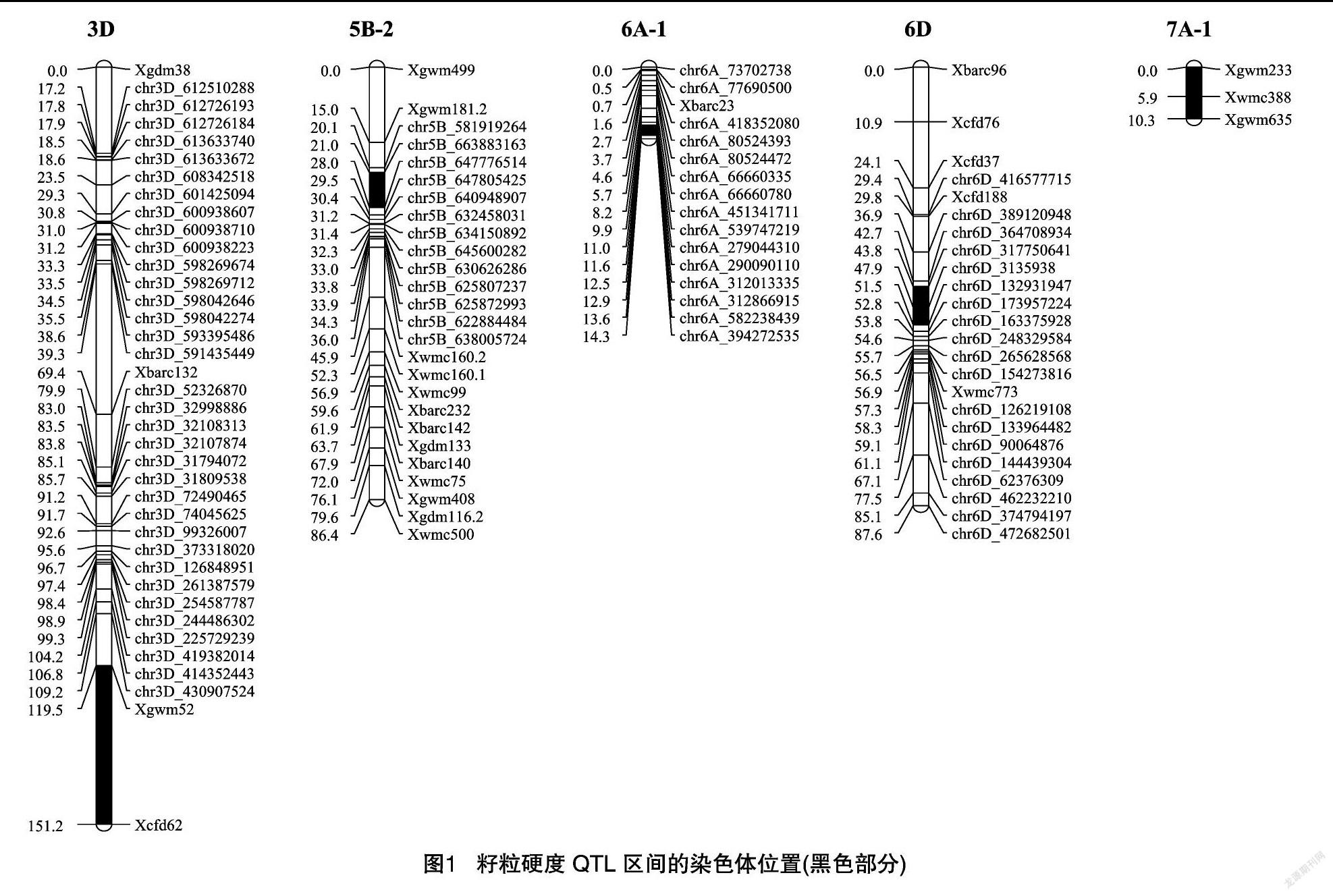
根據3年重組自交系群體籽粒硬度測定資料,結合已經構建的該群體高密度遺傳連鎖圖,采用復合區間作圖,共檢測到5個QTL位點,分別位于3D、5B、6A、6D和7A染色體上(圖1),單個QTL可以解釋11.3%~25.1%的表型變異,其中6A、6D染色體QTL由親本CI12633貢獻,3D、5B、7A染色體的QTL則來自親本揚麥158(表2),7B、7D染色體雖然在區間作圖中檢測到QTL,但未能通過復合區間作圖的檢測。
在本研究發現的QTL中,位于3D、5B和6D染色體的QTL均只在1年表型資料中檢測到,屬于不穩定QTL,可能與特定的環境有關。位于7A、6A染色體的QTL分別在2、3年表型資料中檢測到,屬于較為穩定的QTL,可以在今后的標記輔助育種或設計育種中發揮重要作用,特別是位于6A染色體的QTL,不僅穩定,而且表型解釋率和加性效應均較高,與之緊密連鎖的分子標記是SNP標記,可進行高通量篩選。
3 討論與結論
采用重組自交系群體3年表型資料及遺傳連鎖圖,分別在3D、5B、6A、6D和7A染色體上定位了影響小麥籽粒硬度的QTL。由于親本CI12633和揚麥158籽粒硬度均超過軟質小麥,且對小麥籽粒硬度的影響最大,位于5D染色體的Ha位點在群體中未出現分離,因此推測2個親本在該位點均為硬質突變型。與已有研究相比,位于3D、6A和6D染色體上的QTL尚未見報道,為新發現的QTL,特別是6A染色體的QTL比較穩定,表型解釋率高,加性效應明顯,可在今后小麥籽粒硬度的遺傳育種中發揮重要作用。
Groos等借助2個硬質小麥構建的重組自交系群體,曾在5B染色體上發現1個影響小麥籽粒硬度的QTL,可解釋7.9%~9.4%的表型變異[4],根據SSR標記信息,該QTL與本研究中檢測到的位于5B染色體的QTL位置接近,可能為相同的QTL。Kumar等采用重組自交系群體在2地連續2年進行表型資料分析發現,在7A染色體的364.8~373.1 cM位置存在影響籽粒硬度的QTL,可解釋7.4%的表型變異[7],但是根據小麥基因組信息,本研究檢測到的位于7A染色體上的QTL與之相距較遠,為不同的QTL。
根據籽粒硬度,小麥可以分為軟質麥與硬質麥,用軟質麥磨制的面粉顆粒度小、破損淀粉含量低、面粉吸水率低,適合制作餅干和糕點等;用硬質麥磨制的面粉顆粒度大、破損淀粉含量高、面粉吸水率較高,適合制作面包和優質面條等[1]。在本研究中,親本CI12633為引進的白粉病抗源,后來的研究發現,其對小麥紋枯病和莖基腐病均有較好的抗性[8,10];揚麥158的綜合抗病性好,曾在長江中下游地區大面積種植。因此,可根據最終加工目的及親本特性,結合本研究發現的影響籽粒硬度的QTL,指導針對長江中下游麥區的軟質弱筋及中強筋等品質小麥的品種改良。
參考文獻:
[1]鄭雅月,趙振杰,趙萬春,等. 小麥籽粒硬度研究進展[J]. 麥類作物學報,2017,37(7):915-922.
[2]Bhave M,Morris C F. Molecular genetics of puroindolines and related genes:allelic diversity in wheat and other grasses[J]. Plant Molecular Biology,2008,66(3):205-219.
[3]Igrejas G,Leroy P,Charmet G,et al. Mapping QTLs for grain hardness and puroindoline content in wheat (Triticum aestivum L.)[J]. Theoretical and Applied Genetics,2002,106(1):19-27.
[4]Groos C,Bervasb E,Charmet G. Genetic analysis of grain protein content,grain hardness and dough rheology in a hard×hard bread wheat progeny[J]. Journal of Cereal Science,2004,40(2):93-100.
[5]Heo H,Sherman J. Identification of QTL for grain protein content and grain hardness from winter wheat for genetic improvement of spring wheat[J]. Plant Breeding and Biotechnology,2013,1(4):347-353.
[6]Ibba M I,Kiszonas A M,See D R,et al. Mapping kernel texture in a soft durum (Triticum turgidum subsp. durum) wheat population[J]. Journal of Cereal Science,2019,85:20-26.
[7]Kumar A,Mantovani E E,Simsek S,et al. Genome wide genetic dissection of wheat quality and yield related traits and their relationship with grain shape and size traits in an elite × non-adapted bread wheat cross[J]. PLoS One,2019,14(9):e0221826.
[8]周淼平,姚金保,楊學明,等. 小麥紋枯病抗性QTL的分析[J]. 麥類作物學報,2020,40(5):554-559.
[9]van Ooijen J W. MapQTL5,Software for the mapping of quantitative trait loci in experimental population[M]. Netherlands:Kyazma B V,Wageningen,2004.
[10]周淼平,張 鵬,楊學明,等. 小麥莖基腐病抗性QTL 的分析[J]. 麥類作物學報,2021,41(5):538-543.

