基于免疫信息學的SARS-CoV-2表位篩選及疫苗分子設計分析①
鄧輝雄 王革非 李雁嫘 宋鑫利 谷李銘 李 蕊
(汕頭大學醫學院微生物學與免疫學教研室,病原與免疫學研究中心,汕頭 515041)
新型冠狀病毒(SARS-CoV-2)感染引起的新冠肺炎(Corona Virus Disease 2019,COVID-19)疫情在全球200多個國家和地區流行,對全球公共衛生安全造成巨大威脅,給社會生活及經濟發展造成巨大損失。我國與其他國家的科研人員針對SARS-CoV-2 的表征、候選抗原和表位的鑒定、動物模型的建立、免疫應答的表征以及疫苗設計研究取得了實質性進展[1-2]。截至2020年11月,至少已有5種SARS-CoV-2疫苗進入Ⅲ期臨床試驗。但SARS-CoV-2 的抗原特性、抗原變異對宿主的保護性免疫反應的影響有待進一步驗證[2]。
利用免疫信息學篩選、分析鑒定具有免疫保護性的表位,可有效提高保護性抗體親和力和細胞免疫平衡。病毒抗原,包括Spike 蛋白及RBD 區域存在多種表位。其中部分表位具有免疫保護作用,能夠誘導中和抗體和抑制病毒復制的抗體,以及抑制或殺傷感染細胞及輔助免疫平衡應答的效應T 細胞。大多數表位所誘導的免疫應答不具有中和或抑制病毒復制的作用,反而可能會誘導細胞免疫應答失衡并加重炎癥反應,存在免疫病理損傷、抗體依賴的感染增強(ADE)等負面風險,甚至可能減弱具有免疫保護力表位的免疫保護效應[3-4]。
Spike 蛋白是SARS-COV-2 的關鍵抗體靶點,其同源三聚體在受體結合和病毒入侵的過程中發揮重要作用。全長三聚體Spike 蛋白通常具有很高的免疫原性(約600 kD),其不僅包含RBD 及強效中和抗體的主要靶點,還包含能誘導中和抗體或保護性抗體的非RDB 區域,如N 端結構域。目前已報道了一種基于結構設計的疫苗,確保MERS-CoV Spike蛋白穩定維持在具有最佳抗病毒效果的融合前構象,進而提高基于全長Spike蛋白的疫苗保護效果,該策略能維持宿主體液免疫和細胞免疫的有效平衡[5-6]。
為確保合成肽疫苗效果,本研究基于免疫信息學進行SARS-CoV-2 優勢保護性抗原表位的篩選、鑒定及對接分析,計算機克隆及免疫模擬預測分析抗原表位,構建候選多價表位疫苗,現將結果報道如下。
1 資料與方法
1.1 SARS-CoV-2候選疫苗毒株的確定 選擇NCBI數據庫發表的surface glycoprotein[Severe acute respiratory syndrome coronavirus 2](NC_045512.2)為細胞表位預測模板。通過NCBI(https://www.ncbi.nlm.nih.gov/protein)數據庫收集了579 條序列不同的SARS-COV-2 Spike 蛋白氨基酸序列作為保守性分析模板,所有蛋白數據以FASTA文件格式收集。
1.2 細胞表位預測 通過ABCpred(http://crdd.osdd. net/raghava/abcpred/)和BCPreds(http://ailab.ist.psu.edu/bcpred/)預測候選新冠病毒毒株的線性B 細胞表位。ABCpred 是基于遞歸神經網絡的學習算法,默認閾值為0.51,表位長度為16,其準確率為65.93%[7]。而基于內核方法實現的BCPreds 準確率為75.8%[8]。IEDB 服務器(http://www.iedb.org/)和RANKPEP 服務器(http://imed. med. ucm. es/Tools/rankpep. html)用來預測MHCⅡ型表位[9-10]。選擇IEDB 默認參數設置,并選擇一個MHC HLA 等位基因中人群覆蓋度可達最小等位基因99%覆蓋率的等位基因參考集[11]。RANKPEP 使用定位特異性評分矩陣(PSSMs)或已知與給定MHC 分子結合的一組對齊肽作為MHC肽結合的預測因子,可重點預測保守表位,避免突變引起的免疫逃逸[10],表位與MHCⅡ的結合潛力通過EpiTOP(http://www.pharm‐fac.net/EpiTOP/index.php)評估[12]。IFNepitope(http://crdd. osdd. net/raghava/ifnepitope/)和IL4pred(http://crdd.osdd.net/raghava/il4pred/)預測IFN-γ 誘導性抗原表位和非IL-4誘導性抗原表位[13-14]。MHCⅠ結合CTL表位預測分別用NetMHCpan 4.1服務器(http://www. cbs. dtu. dk/services/NetMHCpan/)、IEDB 服務器(http://tools. iedb. org/mhci/)、Rankpep 服務器(http://imed.med.ucm.es/Tools/rankpep.html)、CTL‐pred(http://crdd. osdd. net/raghava/ctlpred/)、NetCTL server 1.2(http://www.cbs.dtu.dk/services/NetCTL/)進行分析。選擇的所有服務器HLA 等位基因的參數均可達到97%的群體覆蓋率[15]。NetMHCpan4.1 server 利用人工神經網絡預測表位與任何已知序列的MHC分子的結合[16],本研究只選擇了強結合能力的抗原表位進行進一步分析。IEDB 服務器使用ANN、SMM 和CombLib 組成的共識推薦方法[17]。Rankpep 使用位置特異性評分矩陣(PSSM)[10]。CTLpred 利用T細胞表位模式代替MHC 結合物預測CTL 表位[18]。NetCTL 預測方法綜合預測MHCⅠ類結合肽、蛋白酶體C端裂解、TAP 轉運效率。所有預測的表位均由IEDB(http://tools.iedb.org/immunoge‐nicity/)進行免疫原性檢測,并通過ToxinPred(https://webs. iiitd. edu. in/raghava/toxinpred/design.php)確定其無毒性[19-20]。在本研究的表位預測策略中,使用多種工具可比單獨使用一種工具獲得更可靠的結果。
1.3 保護性表位的鑒定 為了選擇合適的表位進行進一步分析,通過比較服務器生成的B細胞表位、Th 表位和CTL 表位,找出重疊度高的最佳片段,使表位同時具有誘導體液免疫反應和細胞免疫反應的能力。選擇MHCⅠ結合物,CTL 和B 細胞表位重疊區域作為潛在的表位,使用IEDB 服務器(http://tools. immuneepitope. org/mhcii/)確定最佳結合物的IEDB評分[9]。
1.4 候選抗原表位保守性評估及T 細胞(HTL&CTL)表位等位基因人群覆蓋分析 通過IEDB(http://tools.iedb.org/conservancy/)上的表位保守性分析工具評估病毒血清型的保守水平[21]。根據已確定的候選表位與579條不同的SARS-COV-2 Spike蛋白質序列比對,計算抗原表位的保守性情況。分析類型和序列識別閾值分別設置為“線性”和“100%”,選擇去除相同模板序列。IEDB 人群覆蓋度工具(http://tools. iedb. org/population/)用于評估預測的T細胞表位(MHCⅠ和MHCⅡ)的人群覆蓋率[22]。
1.5 疫苗構建及抗原性、過敏原性、溶解度的預測 基于上述免疫信息學分析得到的表位,使用KK、AAY 和GPGPG linker 將B 細胞表位、HTL 和CTL 表位按順序連接在一起,50S 核糖體蛋白L7/L1分子佐劑通過EAAAK 在N 端連接[23]。采用Vexigen(http://www. ddg-pharmfac. net/vaxijen/VaxiJen/Vaxi‐Jen.html)和ANTIGENpro(http://scratch.proteomics.ics.uci.edu/)預測構建的多表位疫苗的抗原性[24-25]。AllerTop server(https://www. ddg-pharmfac. net/Aller‐TOP/)用于預測最終疫苗構建的過敏原性,與其他服務器進行的過敏原預測相比,AllerTOPV2.0 的敏感度高達94%[26]。SOLpro(http://scratch.proteomics.ics.uci.edu/)用于預測構建的疫苗蛋白的溶解度[27]。利用ExPASy 工具(https://web.expasy.org/protparam/)對疫苗的分子量、等電點(PI)、半衰期、穩定性等理化性質進行預測[28]。
1.6 疫苗二級、三級結構的預測及二硫鍵設計 以Raptor X(http://raptorx. uchicago. edu/StructPredV2/predict/)和PSIPRED 4.0(http://bioinf.cs.ucl.ac.uk/psipredtest)預測蛋白的二級結構[29-30]。以trRosetta(https://yanglab. nankai. edu. cn/trRosetta/)完成對最終蛋白疫苗的三級結構的預測[31],trRosetta 是一種快速、準確地從頭預測蛋白質結構的算法,其基于一個限制性的Rosetta 模塊直接能量最小化構建蛋白質穩定構象,約束了包括殘基間距離和方向分布,由深度殘基神經網絡進行預測。在CASP1 和CAMEO 派生集的基準測試中,trRosetta 的性能優于所有之前描述的方法。基于Omega、Phi、Distance、Theta、Contact 參數以及TM-score。PyMOL 程序用于三維模型的可視化。在進行下一步之前,有必要提高蛋白模型的穩定性。利用Disulfide by Design2.0(http://cptweb. cpt. wayne. edu/DbD2/index. php)對候選蛋白疫苗進行二硫鍵設計[32]。
1.7 三級結構的重修與驗證 GalaxyRefine(http://galaxy. seoklab. org/cgi-bin/submit. cgi?type=REFINE)用于重修三級結構[33]。為了驗證三級結構模型的質量,使用RAMPAGE 在線服務器(http://mordred.bioc. cam. ac. uk/~rapper/rampage. php)和ProSA-web(https://prosa. services. came. sbg. ac. at/prosa. php)對模型進行檢測[34-35]。
1.8 B 細胞構象表位的預測及蛋白分子對接分析 利用DiscoTope2.0(http://www. cbs. dtu. dk/ser‐vices/DiscoTope/)預測疫苗蛋白三級結構的構象B細胞表位[36]。蛋白質間的相互作用對于理解細胞功能和組織結構非常重要。分子對接是預測受體與配體在穩定構象下相互作用的一種方法。配體和受體間的相互作用取決于他們的親和力,親和力越高,配體和受體分子間的相互作用越穩定。使用ClusPro 2.0(https://cluspro. bu. edu/login. php)預測作為受體的TLR-4(PDB ID:4G8A)和作為配體的蛋白疫苗間的相互作用[37]。TLR-4(PDB ID:4G8A)受體使用pyMol 去掉水分子及其他雜質。該服務器產生的對接配合物具有良好的脫溶自由能和靜電相互作用。LigPLot+用來確定疫苗配體和TLR-4 受體是否具有氫鍵和疏水相互作用的氨基酸殘基。
1.9 計算機克隆及免疫模擬分析 計算機模擬克隆通過SnapGene 軟件完成,而多表位疫苗的免疫原性和免疫應答模式通過C-ImmSim 服務器(http://150.146.2.1/C-IMMSIM/index.php)完成,該服務器基于位置特異性評分矩陣(PSSM)原理,預測候選疫苗模型抗原表位,并基于經典免疫學原理使用機器學習模型預測免疫應答相互作用[38]。在本實驗中,基于群體覆蓋率使用了以下MHC 等位基因:HLA A*11:01、HLA A*31:01、HLA B*35:01、HLA B*53:01、HLA DRB5*01:01、HLA DRB1*09:01。按典型免疫過程間隔1 周注射,共免疫3 次。所有模擬參數均設置為默認值,時間步長設置為1、20 和40(每個時間步長為8 h,時間步長1 為第1 次免疫時間點),抗原分子的氨基酸序列作為疫苗。
2 結果
2.1 SARS-CoV-2 表位的初步篩選 表1 為經鑒定的5 個B 細胞表位,表2 為6 個Th 表位,表3 為5 個CTL 表位。所有候選抗原表位在Spike 蛋白的構象位置如圖1A,獲得能夠中和Spike蛋白與ACE2結合域的中和抗體具有重要意義,本研究選擇的B-1 和B-2 候選抗原表位如圖1B,在Spike 蛋白RBD 和ACE2 結合域上,具有能夠誘導主要中和抗體、破壞其與ACE2結合的潛力。候選Th抗原表位和CTL抗原表位結合的HLA 等位基因對應的不同地區人群覆蓋度分析結果如表4 所示,候選表位可覆蓋全世界95.4%的人群,盡管對應不同地區人群有差異,但依然具有較高的人群覆蓋度,如目前新冠疫情的高發地區南亞有98.39%的人群覆蓋度,歐洲有99.67%的人群覆蓋度,南美有99.95%的人群覆蓋度。對于全球性暴發流行的新冠病毒,開發能夠適應高人群覆蓋度的疫苗具有重要意義。
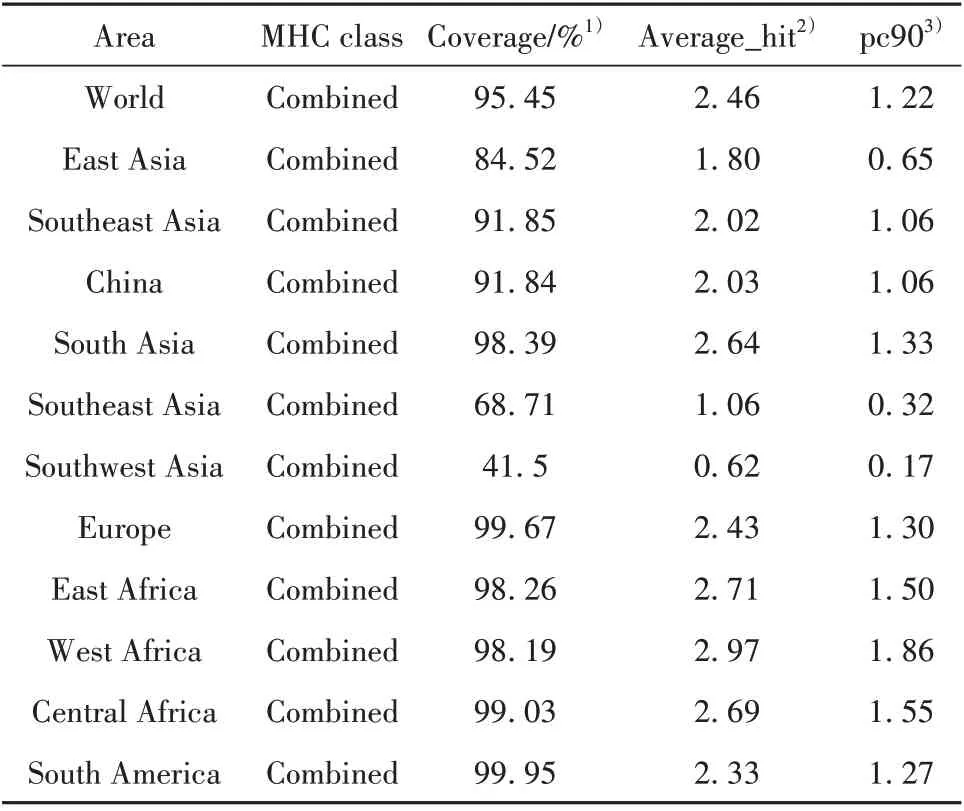
表4 候選Th和CTL表位等位基因中不同地區人群覆蓋率Tab.4 Population coverage calculation of Th and CTL epitopes specific alleles
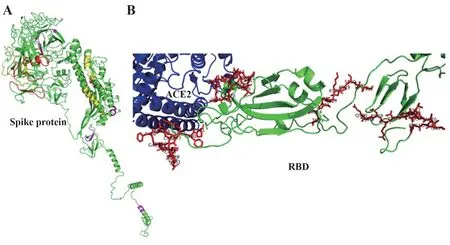
圖1 SARS-CoV-2 Spike蛋白候選抗原表位空間展示Fig.1 Epitopes of SARS-CoV-2 Spike protein candidate antigen
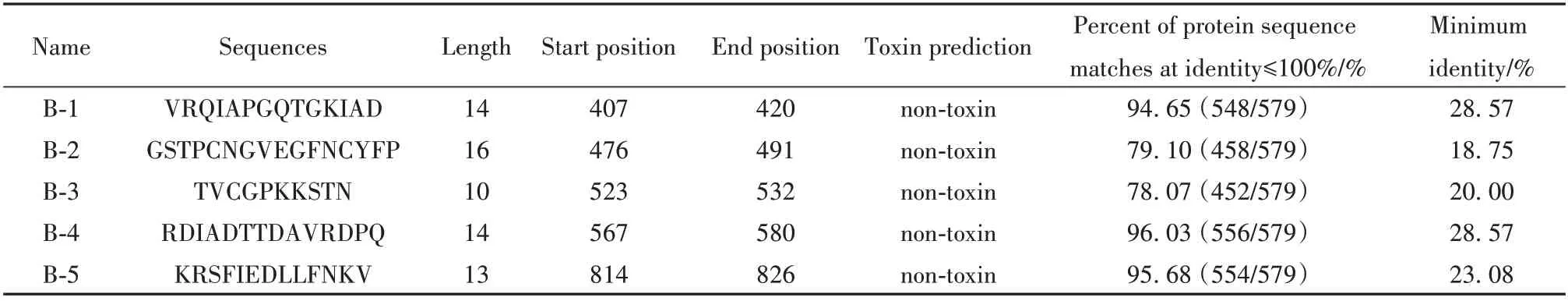
表1 候選B細胞表位Tab.1 Candidate B cell epitopes
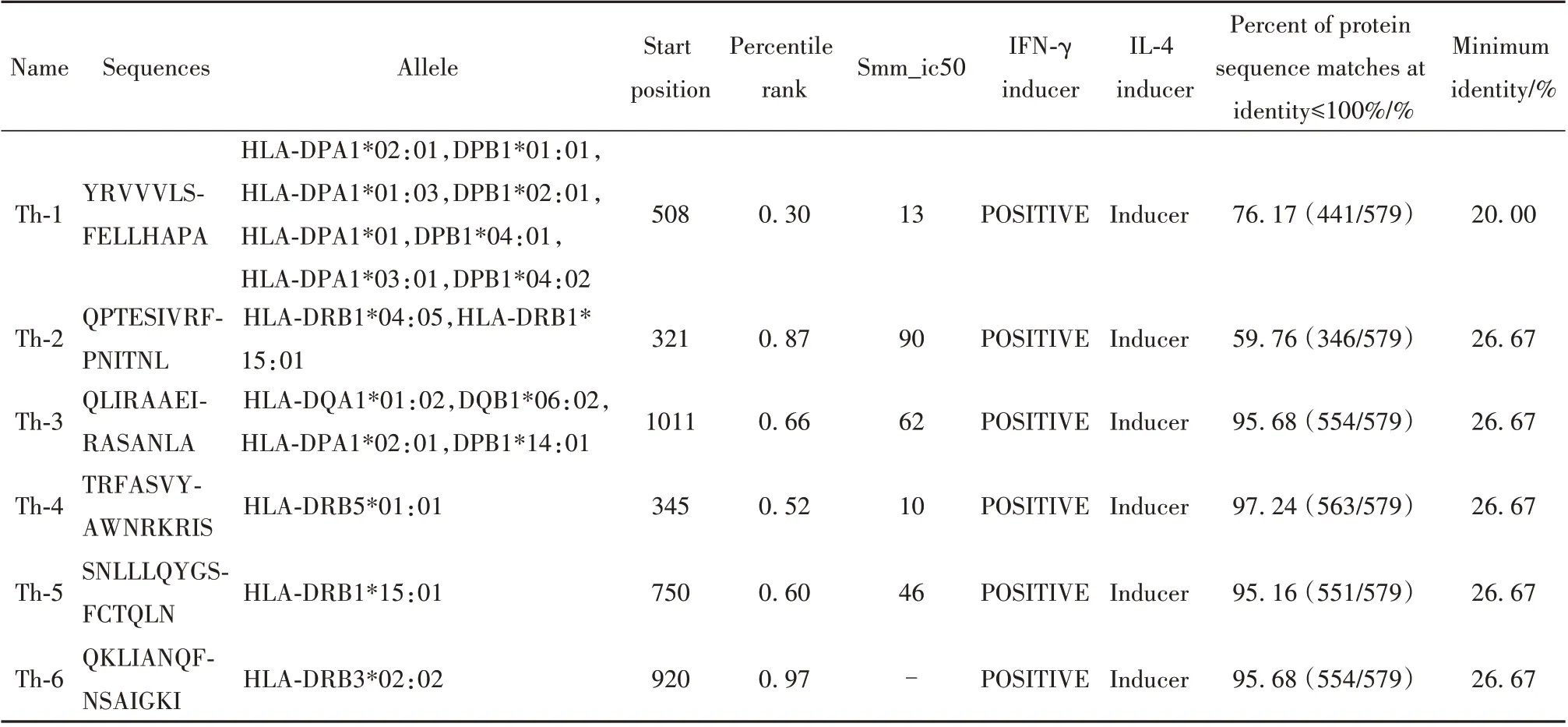
表2 候選Th細胞表位Tab.2 Candidate Th cell epitopes

表3 候選CTL細胞表位Tab.3 Candidate CTL cell epitopes
2.2 SARS-CoV-2表位疫苗構建 基于上述免疫信息學分析得到的表位,使用KK、AAY 和GPGPG linker將B 細胞表位、HTL 和CTL 表位按順序連接在一起,B 細胞表位之間使用雙賴氨酸(KK)linker 保持其獨立的免疫原性活性,MHC 分子在MHCⅡ分子介導的抗原提呈到Th0之前,作用于抗原的KK序列,使用AAY 和GPGPG linker 可增強疫苗亞基的識別能力。Linker 可確保良好的穩定性、結構域取向和折疊率,使用EAAAK linker 連接表位和50S L7/L12分子佐劑氨基酸序列,示意圖如圖2所示。

圖2 SARS-CoV-2表位疫苗氨基酸序列Fig.2 Amino acid residues sequence of SARS-CoV-2 epitopes-based vaccine
2.3 SARS-CoV-2表位疫苗理化性質分析 疫苗全長392 個氨基酸,分子量大小為41.649 92 kD,pI:9.35,在體外哺乳動物網織紅細胞中半衰期為30 h,體內酵母細胞中大于20 h,大腸桿菌中大于10 h,抗原穩定指數為21.82,歸類為穩定蛋白質。Vaxijen v2.0 預測保護性抗原在設定閾值為0.4 時分數為0.416 2,可作為抗原;ANTIGENpro 預測抗原免疫原性為0.964 894;預測過表達溶解度為0.942 995,提示可溶。脂肪族指數為84.52,Grand average of hydropathicity(GRAVY):?0.117,球狀蛋白結構中的脂肪族鏈代表了該蛋白的熱穩定性。蛋白質脂肪族指數的升高表明該蛋白質在性質上是熱穩定的,GRAVY代表蛋白質序列疏水性的平均值。
2.4 SARS-CoV-2表位疫苗二級結構預測 PSIPRED和Raptox 對二級結構的預測結果表明,蛋白疫苗由11.1%的螺旋、35.3%的折疊和53.6%的卷曲組成。溶劑可及性(ACC)由26%的埋藏、25%的居中和48%的外側組成。二級結構預測如圖3所示。
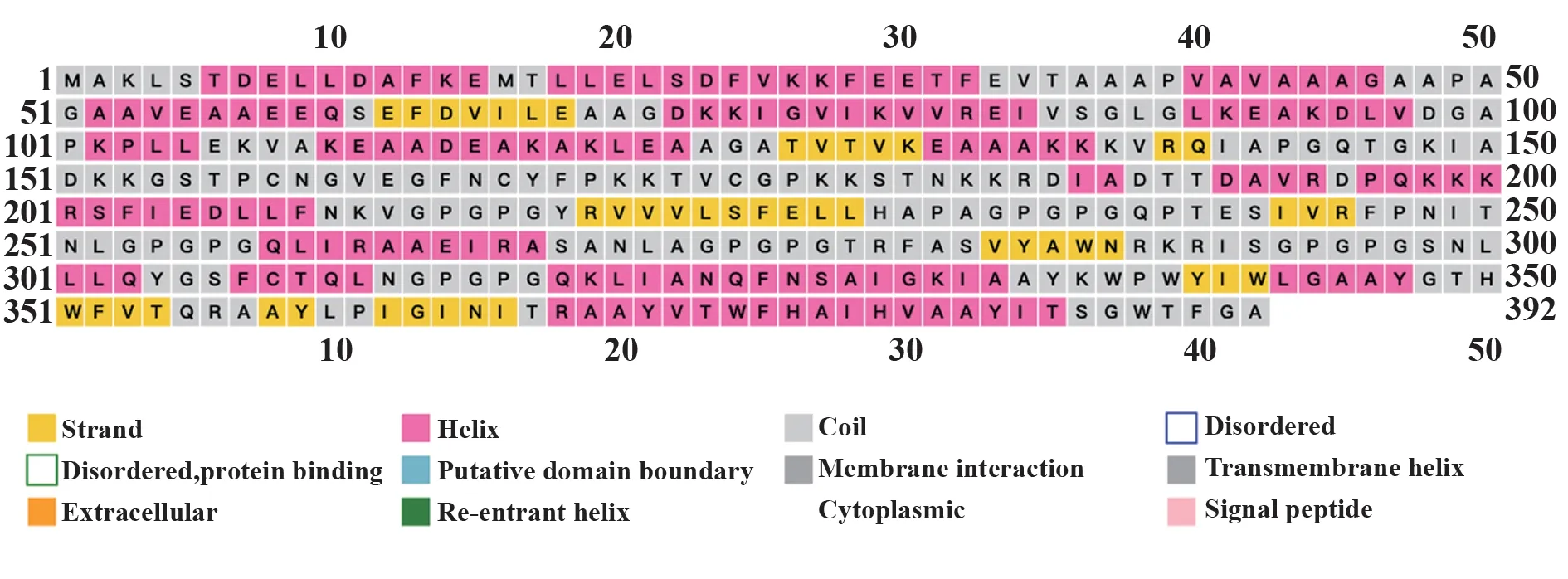
圖3 SARS-CoV-2表位疫苗二級結構預測分析Fig.3 Secondary structure prediction of SARS-CoV-2 epitopes-based vaccine
2.5 疫苗三級結構建模 最佳疫苗三級結構建模結果如圖4F 所示。進一步使用Disulfide by De‐sign2.0進行二硫鍵設計,在默認參數下,分析得到的B-factor 均為0,認為通過trRosetta 能量最小化的蛋白質建模已足夠穩定,不需要進一步的二硫鍵設計。預測模型的Ramachandran 質量分析表明,預測模型中分別有379 個(97.2%)、8 個(2.1%)和3 個氨基酸(0.8%)的氨基酸殘基分別位于有利、允許和異常區域(圖5)。
圖4 SARS-CoV-2疫苗三級結構建模Fig.4 SARS-CoV-2 epitopes-based vaccine tertiary struc?ture modeling
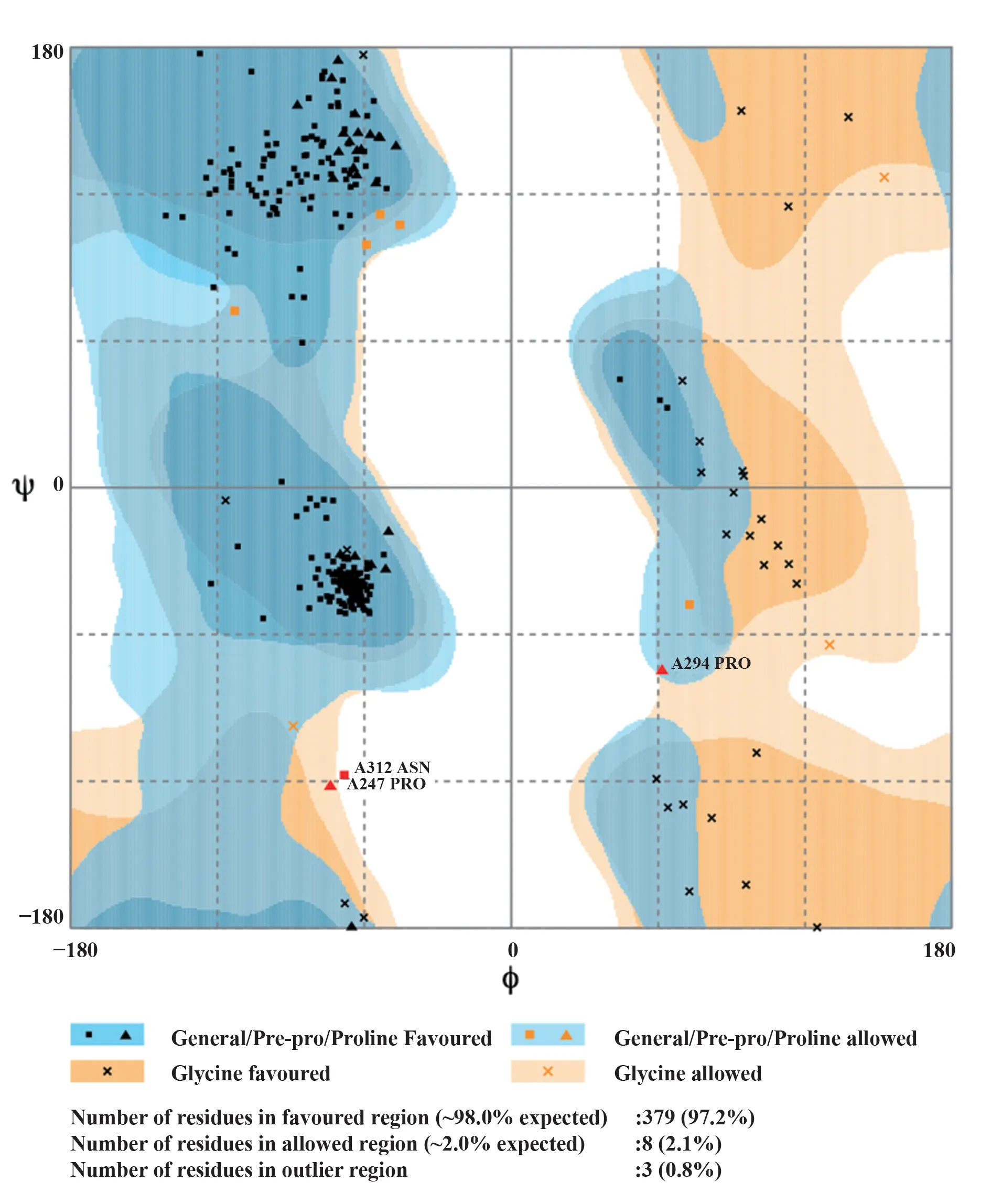
圖5 疫苗三級結構模型refine 前Ramachandran 分析模型質量Fig.5 Validation of vaccine tertiary structure model before refine using Ramachandran plot
2.6 疫苗三級結構的refine 和驗證 應用Galaxy‐Refine 程序對同源性模型結構進行了改進重修。GalaxyRefine 為模型構建了5 個重修模型。重修前蛋白質模型各種參數為GDT-HA(1.000 0)、RMSD(0.000)、Molprobity(2.509)、Clash score(48.7)、poor rotamers(2.0)以及Ramachandran plot(97.2),重修后確認模型1 為最佳候選模型,其具體參數為GDT-HA(0.9458)、RMSD(0.440)、Molprobity(1.995)、Clash score(19.4)、poor rotamers(0.7)以及Ramachandran plot(97.9)。使用Pymol 軟件繪制的卡通圖比對結果如圖6A 所示。Ramachandran 對重修三級結構模型的驗證表明,重修后的模型質量獲得提升(圖6B)。Ramachandran 圖表明,在優化后,有利區域含382個氨基酸(97.9%),允許區域含6個氨基酸(1.5%),不允許區域含2 個氨基酸(0.5%)。而ProSA 網絡(蛋白質結構分析)顯示,蛋白質疫苗結構準確度分析結果在類似大小的天然蛋白質常見的分數范圍內,z值為?7.07(圖6C)。該圖通過繪制氨基酸序列位置函數的能量分布來顯示局部模型質量(圖6D),一般來說,正值對應輸入結構存在問題部分。結果提示,基于同源性建模的多價抗原表位腸道病毒蛋白質結構模型精度可滿足進一步分析。
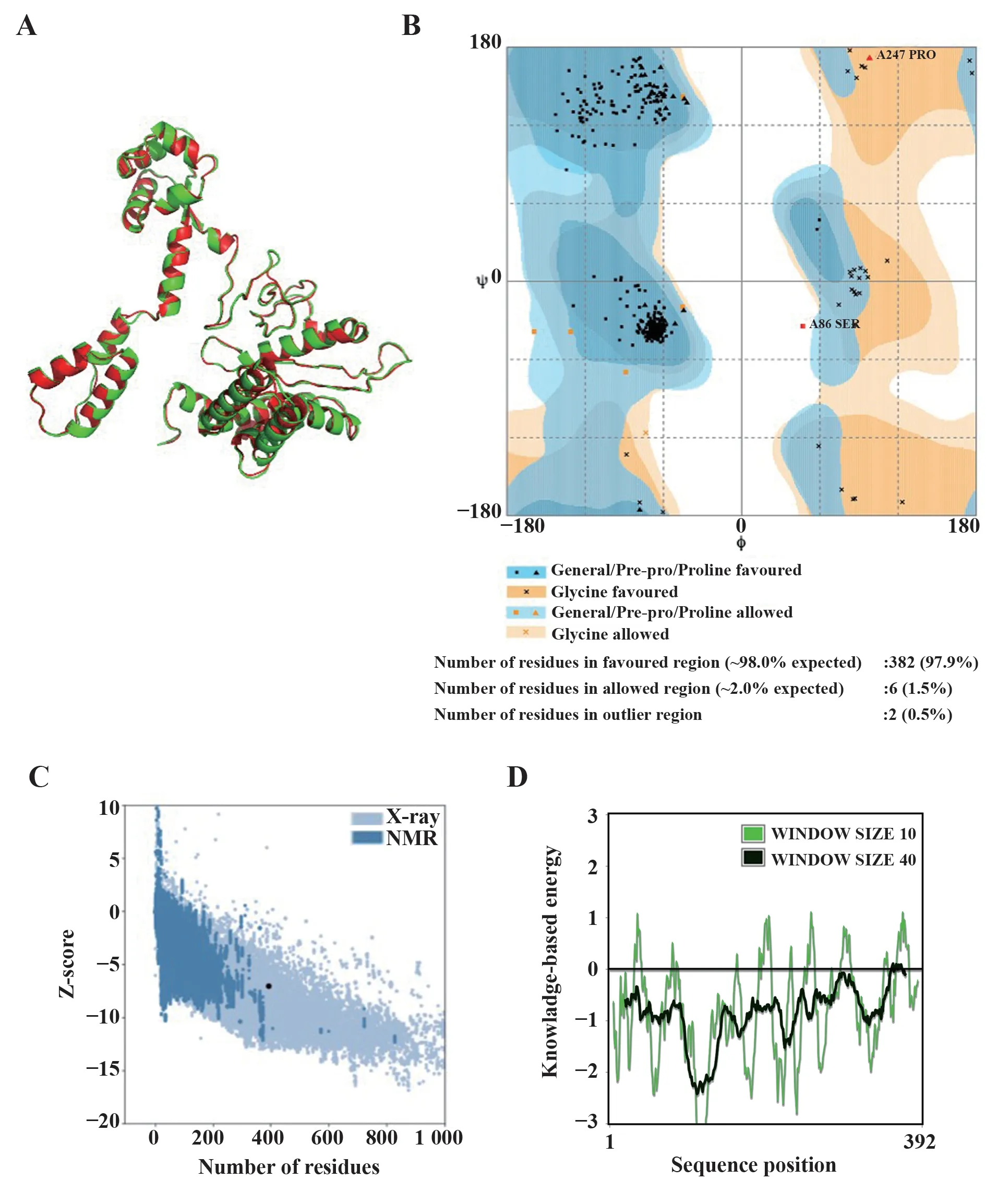
圖6 候選疫苗三級結構的重修和驗證Fig.6 Refinement and validation of vaccine tertiary struc?ture model
2.7 計算機模擬克隆候選多價表位疫苗及構象B細胞表位分析 計算機模擬克隆選擇pGEX-KG 原核表達載體,使用Snapgene 軟件對疫苗的氨基酸序列進行反向翻譯得到疫苗表達基因序列,通過密碼子的優化獲得更好的表達。通過選擇BamHⅠ和HindⅢ限制性內切酶酶切位點構建pGEX-S 新冠疫苗原核表達載體,示意圖如圖7A所示。候選疫苗構象B 細胞表位分析surface 結構如圖7B 所示,共有44個構象B細胞表位。
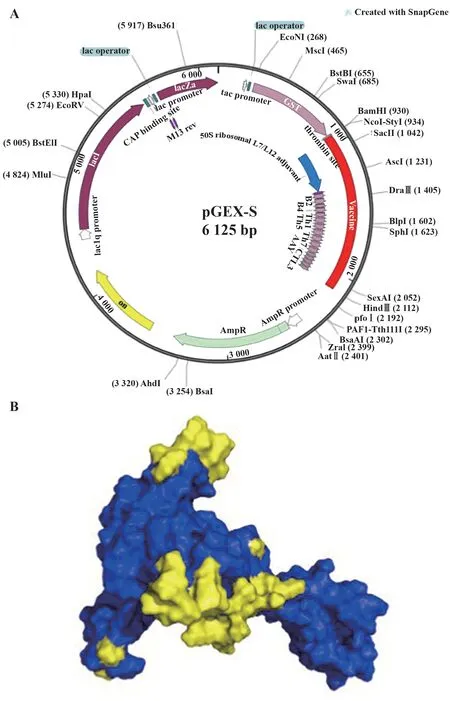
圖7 原核表達載體構建和構象B細胞表位分析Fig.7 Diagram of silico cloning and conformational B-cell epitope prediction
2.8 疫苗與TLR4-MD2 復合體對接 在平衡模式下選擇model.000.05 作為最佳對接模型,對接結果如圖8A 所示,在該對接模式下可以獲得穩定的結合。LigPLot+確定疫苗配體和TLR-4/MD2 受體具有氫鍵的氨基酸殘基為Asn365、Asn181、Gly363、Arg184、Asn210、Lys170、Tyr371、His350、Gly391、Tyr383、Thr389、Glu33、Arg368。具有疏水相互作用的氨基酸殘基為Ile366、Ile362、Ile364、Gly213、Trp374、Val353、Ala357、Phe375、Ala392、Phe390、Trp388、His379、Tyr371、Thr35、Phe375、His376。
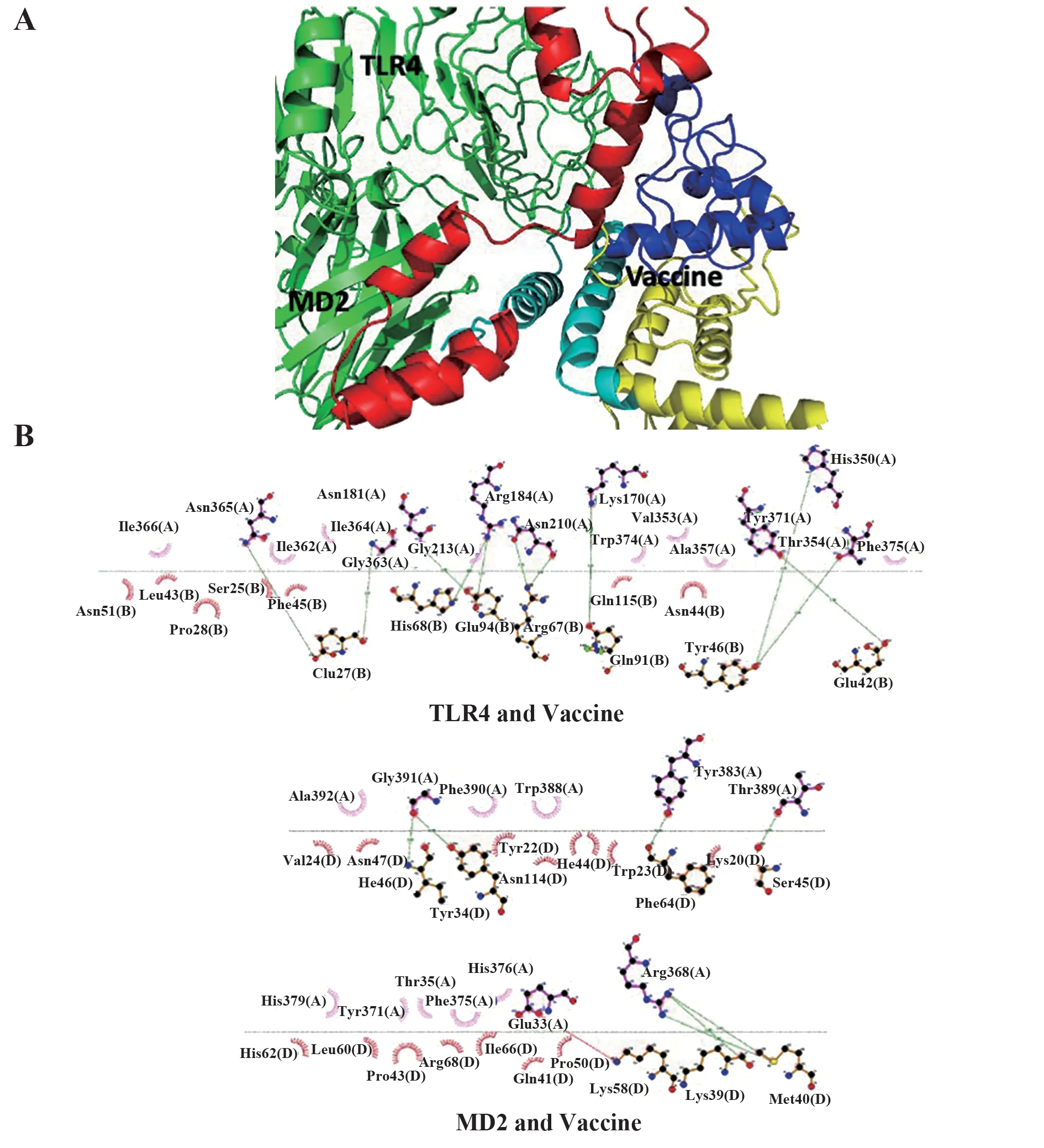
圖8 候選疫苗蛋白與人TLR4/MD2受體對接復合體結果Fig.8 Complexes of candidated vaccine protein docking with human TLR4/MD2 receptor
2.9 計算機模擬免疫應答刺激 多表位疫苗的免疫原性和免疫應答由C-ImmSim 服務器進行,按典型免疫過程間隔1 周注射,共免疫3 次。所有模擬參數均設置為默認值,時間步長設置為1、20 和40(每個時間步長為8 h,時間步長1為第1次免疫時間點),抗原分子的氨基酸序列作為疫苗。通過計算機模擬免疫應答刺激可快速評估疫苗的免疫原性,快速篩選候選疫苗,節約時間和成本。計算機模擬免疫應答刺激可以確定疫苗符合誘導免疫應答規律,具有良好的免疫原性,可有效激活體液免疫和細胞免疫,結果如圖9~12所示。
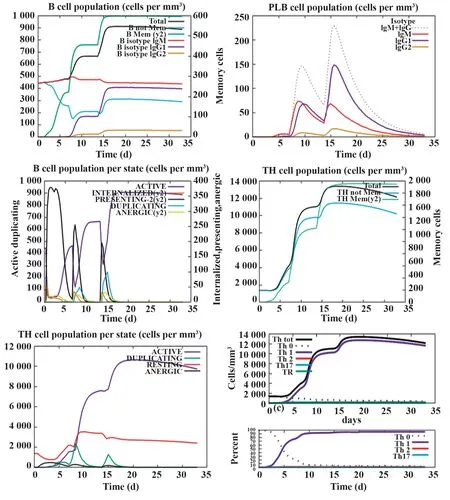
圖9 模擬免疫刺激免疫B細胞和Th數量變化展示Fig.9 B cell and Th counts shown in silico immune simu?lation
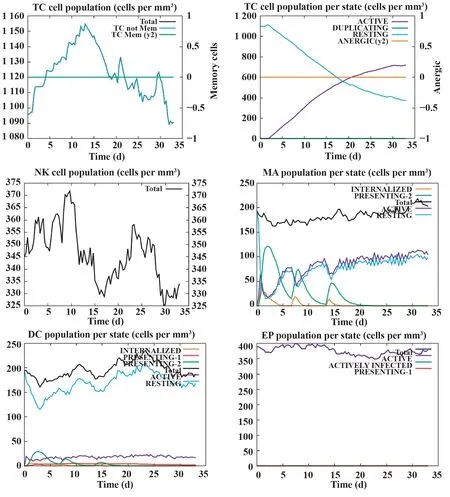
圖10 模擬免疫刺激免疫CTL、NK、MA、DC、EP 細胞數量變化展示Fig.10 CTL,NK,MA,DC,EP cell counts shown in silico immune simulation
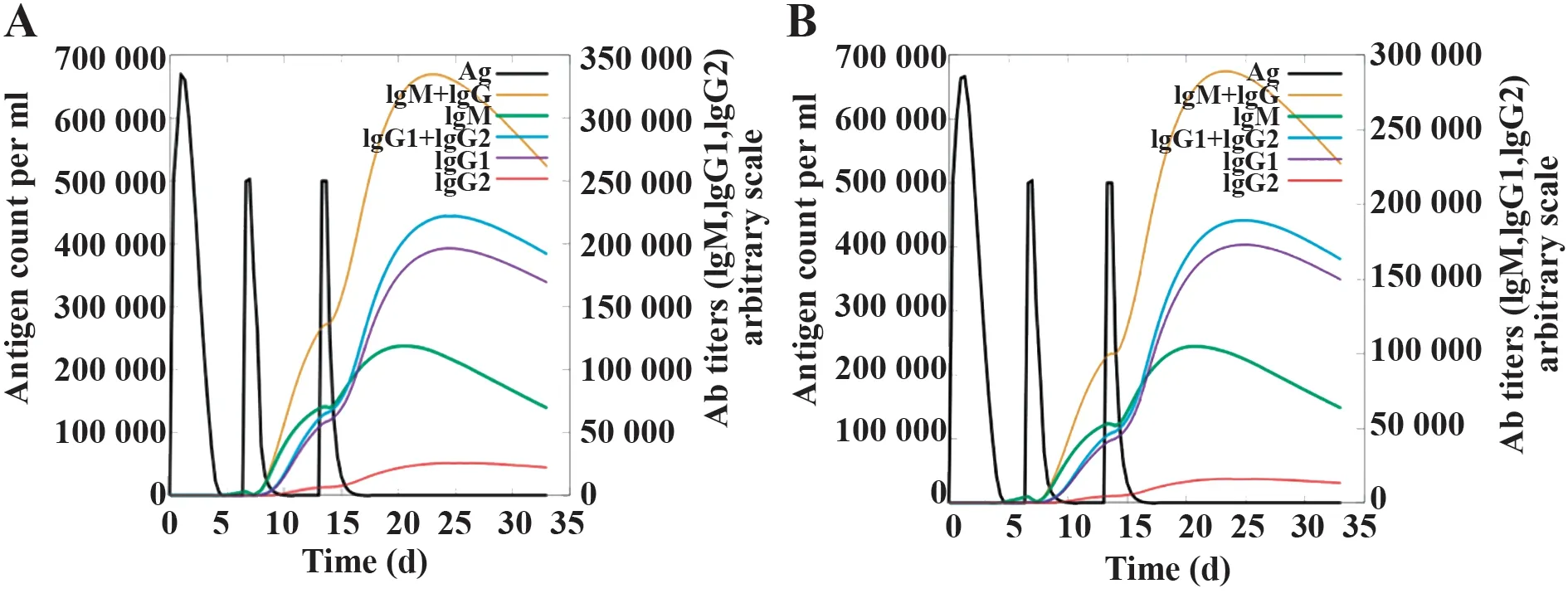
圖11 免疫球蛋白和免疫復合物變化趨勢Fig.11 Trends of immunoglobulins and immunocomplexes
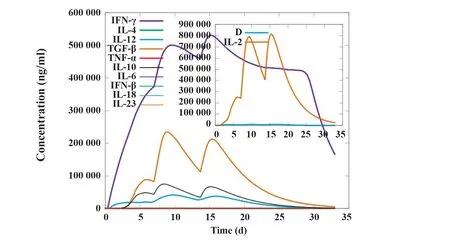
圖12 細胞因子和白細胞介素的濃度變化趨勢Fig.12 Concentration trends of cytokines and interleukins
3 討論
借助免疫信息學和結構生物學開發新冠病毒疫苗是一種有效的策略,目前已有一些借助免疫信息學和結構生物學設計的新冠疫苗研究和本文類似[39-40],不同的是,本研究策略側重于體液免疫和細胞免疫的平衡。疫苗的保護作用除了能夠誘導中和抗體外,還需依賴于細胞毒性CD8 T 細胞和輔助性CD4 T細胞來完全清除病毒。免疫應答的重要特征之一就是輔助性T細胞亞型的激活及相對應的特異性類型細胞因子的釋放。加載在MHCⅡ上的抗原表位可激活Th1 和Th2 型輔助細胞。Th2 主要引發針對細胞外病原體的體液反應,而Th1 激活針對細胞內病原體(病毒、癌癥)的細胞免疫。然而,Th1和Th2 分別與介導細胞免疫和體液免疫不嚴格相等[41],例如Th1 途徑也可刺激適度水平的基于抗體的反應。Th1細胞因子傾向于產生促炎反應,而Th2與抗炎反應相關。不平衡的Th1/Th2 反應可能引起免疫病理學并發癥,例如廣泛的組織損傷炎癥或強烈的過敏反應。因此,在疫苗開發過程中應考慮適當平衡Th1和Th2反應。Th與APC相互作用可依靠IL-4 這樣的細胞因子形成局部正反饋,特異性的激活遞呈了有效表位的APC,同時也擴增了能夠識別有效表位的Th,避免泥沙俱下的胡亂遞呈[14]。針對每個不同表位的Th,可以確立反應方式,是奮力殺死敵人,還是選擇免疫耐受,與之和平相處。理想抗原應由一般人群中的多個MHCⅠ和MHCⅡ等位基因呈遞,并包含與中和抗體相關的線性B 細胞表位。很多抗原并沒有足夠有效的抗原表位,無法有效激活細胞毒性T細胞,所以效率低,準確找到有效的抗原表位具有重要意義。本研究通過免疫信息學篩選出具有平衡體液免疫和細胞免疫特征的新冠病毒候選B 細胞、Th 和CTL 細胞表位,設計具有多表位的高免疫原性候選疫苗。通過計算機免疫模擬刺激實驗分析發現候選疫苗可誘導CD8+T 細胞,同時產生CD4+T 輔助性1型細胞免疫應答,其具有良好的抗病毒特性。佐劑不僅可用于提高蛋白質疫苗引起的抗體反應的強度和持久性,還可影響T 細胞來源的細胞因子模式,從而調節免疫反應,50S L7/L12 分子佐劑已被證明可通過激活TLR4 信號通路增強免疫應答,實驗通過分子對接確定候選疫苗與TLR4 的結合模式和相互作用的氨基酸殘基。在設計疫苗時需要在激發最大的免疫原性的同時盡可能地減少潛在的副作用。冠狀病毒株之間的多樣性為我們提出了另一個挑戰,因為在冠狀病毒屬中至少有6 個不同的亞組。盡管SARS-CoV和SARS-CoV-2 處在相同的亞組,但二者的基因組只有77%相同[42]。了解SARS-CoV-2 的共有和獨特表位有助于為患者設計更好的疫苗或開展診斷性臨床試驗。另一方面,新冠病毒疫苗可能存在的抗體依賴增強(antibody-dependent enhancement,ADE)效應也是一個巨大的挑戰,傳統的ADE 效應主要認為抗體不能中和病毒,反而充當了一個特洛伊木馬,讓病毒更容易感染單核細胞。決定抗體是否引起ADE 的因素主要包括抗體的特異性、滴度、親和力以及抗體的亞型[43]。在猴子模型中,針對SARSCoV 的刺突蛋白的不同表位的抗體,其誘導的反應也各不相同,有些可起到很好的保護作用,有些則容易引起ADE 效應[44]。使用有效的表位設計疫苗可認為是有效避免ADE效應的策略。
4 結論
通過免疫信息學和結構生物學分析,進行基于抗原表位的新冠病毒分子疫苗設計,信息學分析結果初步顯示,具備高保守性、免疫原性的B 細胞表位、MHC 等位基因高人群覆蓋度的IL-4、IFN-γ 誘導型Th 表位及CTL 候選表位構建的疫苗具有良好的平衡體液免疫和細胞免疫應答能力。

