蘋果SHR亞家族基因特征及其器官表達譜分析
于婷婷,許阿飛,張佳林,張瑋瑋,楊洪強
蘋果亞家族基因特征及其器官表達譜分析
于婷婷,許阿飛,張佳林,張瑋瑋,楊洪強*
山東農業大學園藝科學與工程學院, 山東 泰安 271018
SHR(SHORT-ROOT)是植物特異性轉錄因子GRAS的一個亞家族,本研究基于擬南芥SHR序列,從蘋果、白梨、蜜柚、甜橙和掌葉覆盆子全基因組數據庫中分別獲得9、10、5、5和4個亞族成員基因,比較了它們的特征,重點分析了蘋果MdSHR蛋白結構及其基因的器官表達。結果表明,所得SHR亞家族成員編碼蛋白分子質量在29743.72~62251.21Da,全部定位于細胞核。蘋果在染色體上有1對串聯復制基因和1對共線性基因;編碼蛋白含有10個保守基序,其中5個為所獲SHR亞家蛋白共有。MdSHRs和AtSHRs的蛋白二級結構都以無規卷曲和 α 螺旋為主,大部分MdSHRs和AtSHRs的蛋白三維結構相似;9個基因的啟動子上含有脅迫響應及激素應答相關元件。器官表達譜分析顯示,大部分的表達量在蘋果果實和花中較高,在種子和枝條中較低,在根系和幼苗中更低,而在不同器官的表達水平與其他的情況基本相反。
蘋果;亞族基因; 生物信息學分析
轉錄因子能夠通過調控基因轉錄而調控植物生長發育及其對于環境的響應,植物特異性轉錄因子GRAS家族中的SHR(SHORT-ROOT)亞家族參與基本組織(ground tissue)細胞的分裂與分化,調控根的輻射形態(radial patterning)建成,其基因缺失突變體的根尖基本組織不能進行平周分裂,根系變短[1,2]。SHR能夠與SCR(SCARECROW)形成復合體SHR-SCR促進植物根瘤形成[3],也能夠調控植物葉和芽的生長發育[4,5],基因突變后會使番茄果實生長和成熟會延遲[6],而且SHR亞家族成員和參與月季營養生長向生殖生長的轉換以及調控番茄叢枝菌根發育[7,8]等,這些均顯示SHR對植物生長發育具有重要調節作用。
蘋果是世界第二大水果,器官生長發育是其產量產生和品質形成的基礎。本研究基于蘋果等果樹全基因組信息,分析SHR亞家族的基因特征和蘋果SHRs蛋白質結構特征,并進一步分析的啟動子、染色體定位和組織表達等,為揭示功能及深入挖掘蘋果基因組信息提供參考。
1 材料與方法
1.1 SHR亞族基因鑒定
通過TAIR網站(https://www.arabidopsis.org/)獲得4個擬南芥成員SHR蛋白序列。蘋果(Borkh)和掌葉覆盆子(Hu)蛋白數據庫來自GDR(https://www.rosaceae.org)。白梨(Rehd.)蛋白數據庫來自基因組數據來自GIGA(http://gigadb.org/dataset/100083)。甜橙((Linn.) Osbeck)和蜜柚(LOsbeck)蛋白數據庫來自甜橙基因組信息平臺(http://citrus.hzau.edu.cn/)。
以下載的4個擬南芥序列(AT3G13840/AT4G37650/AT3G49950/AT5G67411)為查詢序列,進行本地blastp搜索;通過Pfam數據庫(http://pfam.xfam.org/)獲得GRAS蛋白保守結構域(PF0351)文件,并利用HMM 3.0搜索蛋白數據庫。利用NCBI-CDD[9]和SMART(http://smart.embl-heidelberg. de/)篩選基因,以確定候選基因含有GRAS保守域。并進行系統發育樹分析,獲得SHR基因序列。將所獲得的序列提交于ExPaSy中的ProtParam(http:// web.expasy.org/protparam/),進行理化性質預測。基因名由其在在染色體上的位置決定。亞細胞定位預測通過Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)進行。
1.2 SHR亞家族基因染色體定位
利用TBtools獲得蘋果染色體長度信息,并進一步進行染色體定位。
1.3 SHR亞家族成員系統進化、保守基序及基因結構分析
利用Clustal W軟件對蘋果和擬南芥的蛋白序列進行多重序列比對,利用MEGA 7.0,采用相鄰連接法(Neighbor-Join,NJ),設置參數Bootstrap method為1000,并設置參數Poisson model,Pairwise deletion,生成進化樹。
利用MEME[10]和TBtools[11]進行進化樹、保守基序和基因結構的可視化。
1.4 蘋果SHRs蛋白質結構預測
蛋白質二級結構預測利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html),蛋白質三級結構預測通過SWISS-MODEL(https://swissmodel.expasy.org/)進行[12]。
1.5 蘋果SHRs基因啟動子分析
從蘋果基因組中提取基因翻譯起始位點ATG上游約2000 bp的片段作為啟動子區域,將所有的啟動子序列提交至PlantCARE(http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/),進行啟動子順式作用元件的預測分析。
1.6 MdSHRs在不同器官中的表達譜分析
基于NCBI-GEO(GSE42873)數據庫,利用TBtools繪制熱圖[11],進行蘋果亞族基因表達模式分析。
2 結果與分析
2.1 SHR亞族基因的鑒定
依據擬南芥SHR蛋白序列,基于全基因組信息,通過NCBI-CDD和SMART篩選之后,獲得了9個蘋果亞族基因、10個白梨SHR亞族基因、5個蜜柚亞族基因、5個甜橙亞族基因和4個掌葉覆盆子亞族基因,根據它們在染色體上的位置以及擬南芥中的命名方式,命名如表1。
這些基因編碼蛋白長度在202aa()~561aa(),平均長度463.59aa,分子質量在29743.72~62251.21Da,等電點穩定在4.98~6.19之間。亞細胞定位預測顯示,蘋果、白梨、蜜柚、甜橙、掌葉覆盆子和擬南芥6個物種的亞家族基因編碼蛋白均定位在細胞核中(表1)。

表 1 蘋果中SHR基因的基本信息
2.2 蘋果SHR亞家族染色體分布及共線性分析
蘋果基因染色體定位結果顯示,蘋果的和分別定位于0、2、4、和17號染色體上,和均定位于9號染色體上,、和均定位于15號染色體上。另外,在蘋果染色體上和基因緊靠在一起,出現了串聯復制的現象(圖2)。
圖3顯示,蘋果基因組間有1對共線性基因,即1對節段復制基因,為分別在9號和1號染色體上的和;與擬南芥有4對直系同源基因,即-、-、-和-,它們分別位于擬南芥的3-5號染色體和蘋果除2號、9號、12號和15號染色體上。

圖 1 蘋果SHR基因的染色體定位及串聯復制基因分析
注:相同染色體上基因有紅色線段連接,表示相關基因為串聯重復基因。
Note: The genes on the same chromosome are connected by red lines, indicating that the related genes are tandem repeat genes.

圖2 蘋果SHR基因的共線關系
注:背景中灰色線條表示蘋果基因組內的共線區域,彩色線條突出顯示共線的基因對。
Note: The gray lines in the background indicate the collinear blocks in the apple genome, and the colored line highlights the syntenygene pairs.

圖 3 擬南芥和蘋果SHR基因共線性關系
注:背景中灰色線條表不同基因組中的共線區域,紅色線條突出顯示共線的SHR基因對。Chr00-17為蘋果染色體,AtChr1-5、AtPltd和AtMT為擬南芥染色體。
Note: The gray lines in the scene indicate the collinear blocks within genomes, and the red lines highlight the synteny SHR gene pairs. Chr00-17arechromosomes, AtChr1-5, AtPltd and AtMT arechromosomes.
2.3 蘋果SHR家族進化樹、保守基序及基因結構分析
利用MEGA 7.0程序,構建所獲亞家族成員系統發育進化樹(圖4,A),結果顯示,這些亞家族成員編碼蛋白共分為三組,蘋果MdSHR1、MdSHR4、MdSHR6、MdSHR7、MdSHR8和MdSHR9為第Ⅰ組,與同處第Ⅰ組的擬南芥AtSHR2和AtSHR4以及白梨PbSHR3、PbSHR5、PbSHR7、PbSHR8和PbSHR10親緣關系較近。蘋果MdSHR、MdSHR3和MdSHR8為第Ⅱ組,與擬南芥AtSHR3以及白梨PbSHR1、PbSHR2、PbSHR6和PbSHR9共處同一組。蘋果MdSHR5、白梨PbSHR4和擬南芥AtSHR1共處第Ⅲ組。
利用MEME程序,對蘋果SHR亞家族蛋白序列分析預測到10個保守基序(圖4,B),其中motif 4、motif 7、motif 8、motif 9和 motif 10等5個保守基序為所獲SHR亞家蛋白共有,預示著他們在結構及進化過程中的高度保守性。
基因結構分析顯示(圖4,C),各物種亞家族成員結構同樣高度保守,只有、和含有內含子,其他基因只有外顯子而無內含子。
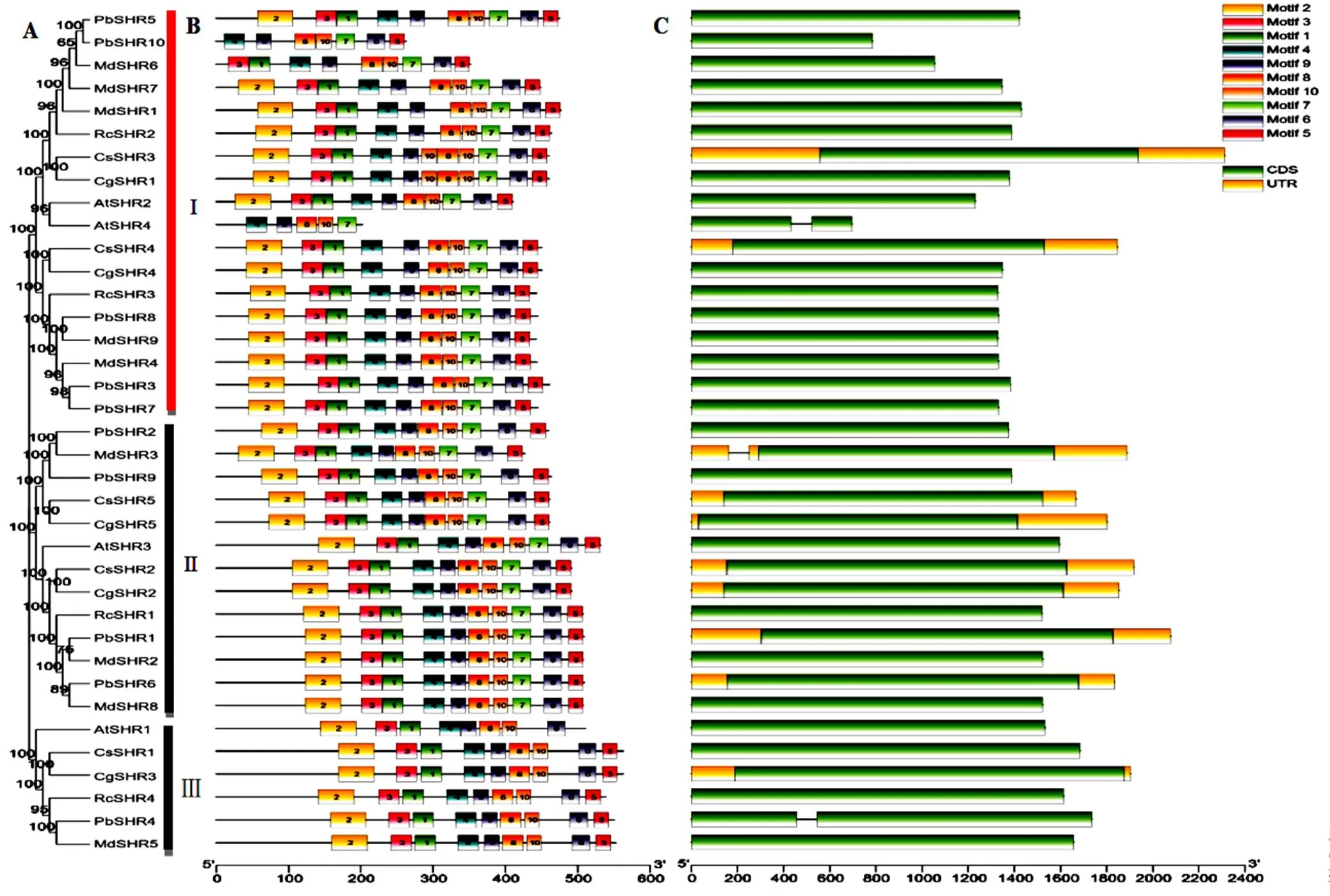
圖4 SHR亞家族進化樹、保守基序及基因結構
2.4 蘋果和擬南芥SHR家族蛋白質結構預測
通過對AtSHRs和MdSHRs蛋白二級結構進行預測(表2),可以發現,13個蛋白質二級結構都以無規卷曲和α螺旋為主,其中,只有AtSHR1和MdSHR5其中無規卷曲占到最大比例。此外,二級結構中還包括β-轉角,其占比例都是最小的。通過同源建模的方法繪制AtSHRs和MdSHRs蛋白質三維結構(圖5),可以發現,大部分MdSHRs蛋白質三維結構相似,與蛋白的二級結構預測結果相符。其中,MdSHR7與AtSHR1和AtSHR3的蛋白質三級結構相似程度較高,MdSHR2、MdSHR4、MdSHR5、MdSHR8、MdSHR9與AtSHR2的蛋白質三級結構相似程度較高,說明這些基因的編碼蛋白具有相似結構,發揮相似功能,也進一步說明進化樹聚類、串聯復制分析和共線性分析的可靠性。

表2 蘋果和擬南芥SHR基因編碼蛋白質二級結構
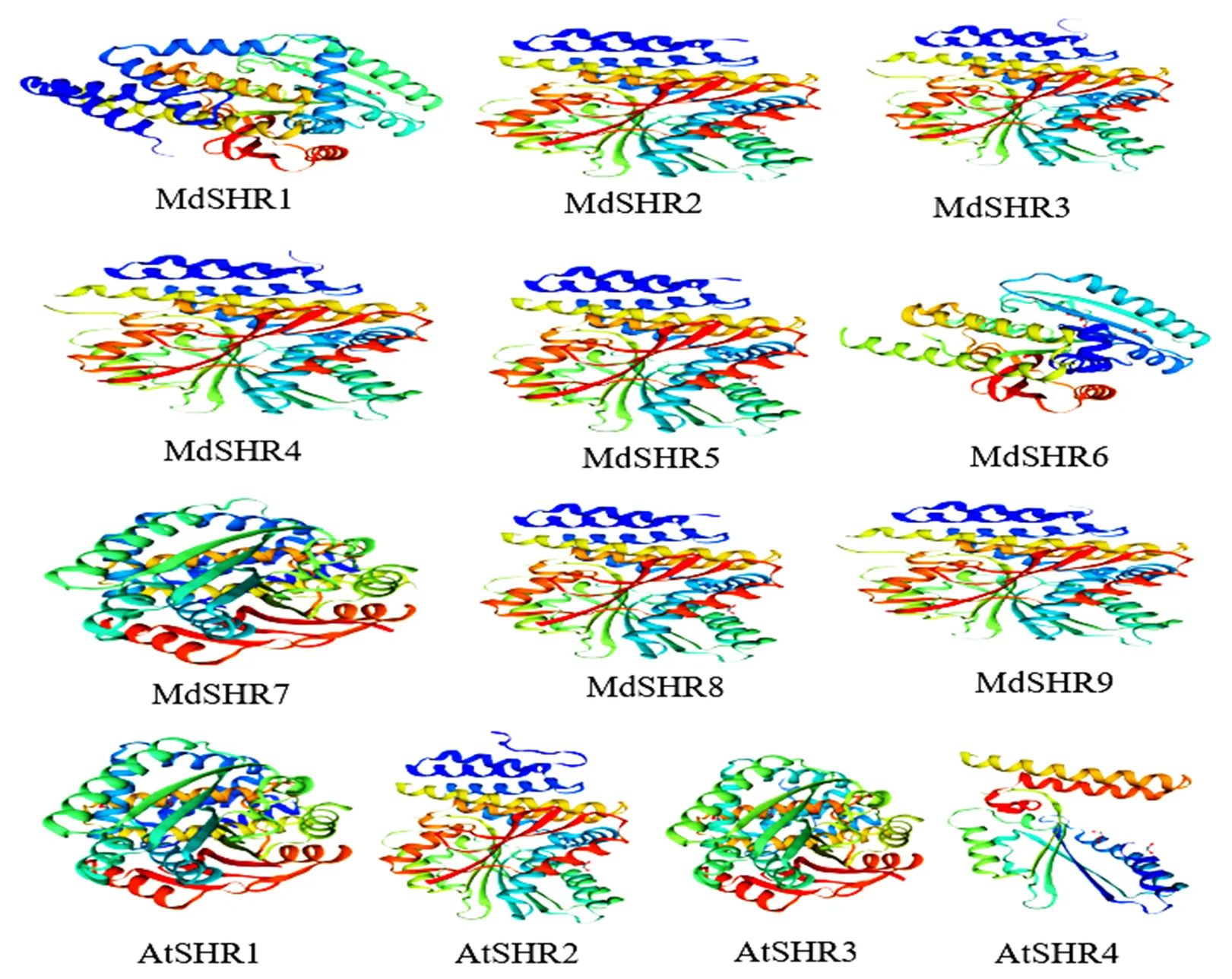
圖5 蘋果和擬南芥SHR蛋白三級結構預測
2.5 蘋果SHR亞家族基因啟動子順式作用元件分析
為預測潛在功能,從蘋果基因組中提取翻譯起始位點 ATG 上游約 2000 bp 的片段作為啟動子區域進行分析(表3),結果顯示,9個均含有與非生物脅迫響應相關的多種作用元件,以及與植物激素反應相關的應答元件,比如9個基因上均含有光響應元件和MYB等脅迫相關的元件。其中,在同一分組的中聚在同一進化分支的和的啟動子上均含有脫落酸響應元件(ABRE)、低氧誘導響應元件(ARE)、赤霉素(GA)響應元件、低溫響應元件(LTR)和茉莉酸(MeJA)響應元件,而與其同在一組的、和啟動子上也都含有茉莉酸(MeJA)響應元件。這說明屬于同一進化分支的基因可能發揮著相似的功能,也進一步說明進化樹聚類的可靠性。

表 3 蘋果MdSHRs亞家族基因啟動子順式作用元件分析
2.6 蘋果SHR亞家族基因器官表達譜分析
基于NCBI-GEO在線分析,從GEO(GSE42873)數據庫,獲到9個分別在GD、M14、M20、M49、M67、M74、X41002、X3069、X9220、X4442和X2596等11蘋果基因型(或品種)不同器官的表達結果,見圖6。該圖顯示,9個在根系和幼苗中表達水平都很低,大部分在果實和花中的表達水平較高、在種子和枝條中的表達水平較低,暗示不同在蘋果器官發育中存在相似功能。但是與其他明顯不同,它在枝條中表達水平較高,在果實中表達水平較低,在種子中下調表達。此外,大部分在不同基因型蘋果的同一器官中的表達量有所不同,比如,在M49的葉中的表達量明顯比在M14中高。

圖6 MdSHR基因在蘋果不同器官的表達譜
注:紅色代表相對較高的表達水平,而白色代表相對較弱的基因表達水平;M14、M20、M49、GD、M67、M74、X41002、X3069、X9220、X4442和X2596代表蘋果品種或基因型
Note: Red represents relatively high expression levels, while white represents relatively weak gene expression levels; M14, M20, M49, GD, M67, M74, X8877, X41002, X4442 and X2596 represent apple varieties or genotype.
3 討 論
本研究鑒定到9個蘋果亞族基因,數量上多于掌葉覆盆子、蜜柚、甜橙、枳()[13]、綠豆()[14]和杜仲([15],低于白梨。Fan[16]等認為基因數量可能與其基因組大小有關,但是,白梨基因組(512 Mb)[17]小于蘋果基因組(881.3 Mb)、杜仲()基因組(725.20 Mb)[18]、甜橙(588.00 Mb)[19]和綠豆基因組(597 Mb)[20],枳基因組(377.30 Mb)[18]也大于掌葉覆盆子基因組(231.21 Mb)[21]。因此,基因組大小與基因成員數量并沒有明確關系[22]。
不同物種中SHRs被分為三組,MdSHR亞家族成員在三組內均有存在,且結構相似,大多數只有一個外顯子,無內含子,于大麻[243、黃瓜[24]等相似,表明,基因在高等植物的進化中具有高度的保守性。而無內含子的原因可能在于植物基因在進化過程中主要是通過水平基因轉移和重復事件起源于原核基因組[25]。通過對其保守基序、蛋白質二級結構和三級結構的分析發現,其蛋白質C末端結構高度保守,這與黃瓜[24]、大麻[23]、枳[13]和杜仲[15]等中的結果相似。通過啟動子預測發現,所有啟動子序列中包含大量的非生物脅迫響應元件以及植物激素響應元件,說明基因在植物生長發育和非生物脅迫響應中起著重要作用。
第Ⅰ組中的擬南芥主要在莖頂表達[26],功能目前研究較少,但其會參與羧酸代謝過程、花發育、芽系統發育和組織發育[27]。第Ⅰ組中的()會促進營養生長向生殖生長轉變[7],還可以促進果實的成熟[28]。蘋果均屬于第Ⅰ組,在花、果和葉中表達水平較高,其中在未成熟的果實中表達水平最高,即其可能在促進果實生長等方面具有重要作用。
第Ⅱ組中的會在莖、葉、花、根等不同器官中表達[29],同一組的蘋果/在不同品種的花、果和葉中具有較高表達量。基因可以調節葉片氣孔的形成[30]和芽的形成[31],還可以調節植物維管束鞘細胞的光合作用[32],會通過NO影響中心碳代謝和內源性植物激素水平促進果實的生長和成熟[6],蘋果/可能在葉和果實中行使相似的功能,調節蘋果的生長發育。而第Ⅱ組中的在莖中具有較高表達量,有可能是在莖中通過調控生長素轉運蛋白LAX3(LIKE AUXIN RESISTANT3)調節生長素含量,進而通過蛋白PINs轉運生長素后調節莖的生長發育[33,34]。
第Ⅲ組中,的表達位點有葉、花、根、萼片、莖,主要在花瓣分化擴張階段表達[28],同樣在花、果實和葉片的表達水平較高。過表達的擬南芥抽苔晚且蓮座葉邊緣鋸齒程度及蓮座葉表面表皮毛的密度均比野生型的高[35];的同源基因還會促進獨腳金內酯的生成[35],而獨腳金內酯調節腋芽生長[36]和葉片衰老[37],可能通過獨腳金內酯影響果樹葉片的生長發育。
4 結 論
基于擬南芥SHR蛋白系列,從蘋果全基因組中鑒定出9個蘋果亞族基因,它們在染色體上有1對串聯復制基因和1對共線性基因,在結構及進化過程中具有高度保守性。蘋果與擬南芥共形成4對直系同源基因,它們編碼蛋白的二級結構和三級結構相似。蘋果含有與非生物脅迫響應相關和與植物激素反應相關的應答元件,大部分表達模式相似,它們在蘋果花和果實中有比較高的表達量,而在根系和枝條中表達水平較低。
[1] Helariutta Y, Fukaki H, Wysocka-Diller J,. The SHORT-ROOT gene controls radial patterning of theroot through radial signaling [J]. Cell, 2000,101:555–567
[2] Hernández-Coronado M, Ortiz-Ramírez C. Root patterning: Tuning SHORT ROOT function creates diversity in form [J]. Front Plant Sci, 2021,12:745861
[3] Dong WT, Zhu YY, Chang HZ,. An SHR–SCR module specifies legume cortical cell fate to enable nodulation [J]. Nature, 2021,589:586-590
[4] Dhondt S, Coppens F, De Winter F,. SHORT-ROOT and SCARECROW regulate leaf growth in Arabidopsis by stimulating S-phase progression of the cell cycle [J]. Plant Physiol, 2010,154:1183-1195
[5] Yoon EK, Dhar S, Lee MH,. Conservation and diversification of the SHR-SCR-SCL23 regulatory network in the development of the functional endodermis inshoots [J]. Mol Plant, 2016,9(8):1197-1209
[6] Bodanapu R, Gupta SK, Basha PO,. Nitric oxide overproduction in tomato hrmutant shifts metabolic profiles and suppresses fruit growth and ripening [J]. Front Plant Sci, 2016,7:1714
[7] Guo X, Yu C, Luo L,. Comparative transcriptome analysis of the floral transition in'Old Blush' andvar.[J]. Sci Rep, 2017,7(1):6068
[8] Ho-Plágaro T, Molinero-Rosales N, Fari?a Flores D,. Identification and expression analysis of GRAS transcription factor genes involved in the control of arbuscular mycorrhizal development in Tomato [J]. Front Plant Sci, 2019,10:268
[9] Marchler-Bauer A, Bo Y, Han L,. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures [J]. Nucleic Acids Research, 2017,45(D1):200-203
[10] Bailey TL, Williams N, Misleh C,. MEME:discovering and analyzing DNA and protein sequence motifs [J]. Nucleic Acids Research, 2006,34(W):369-373
[11] Chen C, Chen H, Zhang Y,. TBtools: An integrative toolkit developed for interactive analyses of big biological data [J]. Mol Plant, 2020,13(8):1194-1202
[12] 諶容,陳敏,楊春賢,等.基于SWISS-MODEL的蛋白質三維結構建模[J].生命的化學,2006,26(1):54-56
[13] 楊杰,陳蓉,胡文娟,等.枳GRAS基因家族鑒定及其對低溫脅迫的響應[J].植物生理學報,2022,58(1):130-140
[14] 張文慧,何光鑫,王子鑫,等.綠豆GRAS基因家族鑒定及其非生物脅迫下的表達模式分析[J/OL].農業生物技術學 報,2022-03-23.861-872.https://kns.cnki.net/kcms/detail/11.3342.S.20220322.1146.012.html
[15] 張傳明,張寶會,姚新轉,等.杜仲全基因組GRAS基因家族的鑒定和系統發育分析[J].特產研究,2021,43(3):65-74
[16] Fan S, Zhang D, Gao C,. Identification, classification, and expression analysis ofgene family in[J]. Front Physiol, 2017,8:253
[17] Wu J, Wang Z, Shi Z,. The genome of the pear (Rehd.) [J]. Genome Res, 2012,23(2):396-408
[18] Kayim M, Ko? NK, Rokka VM. Variation of the nuclear DNA content of species of subtribe Citrinae (Rutaceae) [J]. Hortscience, 1998:1247-1250
[19] Guerra M dos S. Cytogenetics of rutaceae. II. nuclear DNA content [J]. Caryologia, 1984:219-226
[20] Arumuganathan K, Earle ED. Nuclear DNA content of some important plant species [J]. plant Molecular Biology Reporter, 1991,9(3):208-218
[21] Wang L, Lei T, Han G,. The chromosome-scale reference genome of Rubus chingii Hu provides insight into the biosynthetic pathway of hydrolyzable tannins [J]. Plant J, 2021,107(5):1466-1477
[22] 張煥欣,董春娟,尚慶茂.辣椒GRAS家族全基因組鑒定與表達分析[J].園藝學報,2017,44(12):2305-2317
[23] 張苗苗,于江珊,施江,等.大麻GRAS轉錄因子全基因組研究[J].中草藥,2021,52(5):1423-1433
[24] Lu X, Liu W, Xiang C,. Genome-wide characterization of GRAS family and their potential roles in cold tolerance of Cucumber (L.) [J]. International Journal of Molecular Sciences, 2020:3857
[25] Huang W, Xian Z, Kang X,. Genome-wide identification, phylogeny and expression analysis ofS gene family in Tomato [J]. BMC Plant Biology, 2015,15(1):1-18
[26] Obulareddy N, Panchal S, Melotto M. Guard cell purification and RNA isolation suitable for high-throughput transcriptional analysis of cell-type responses to biotic stresses [J]. Mol Plant Microbe Interact, 2013,26(8):844-849
[27] Depuydt T, Vandepoele K. Multi-omics network-based functional annotation of unknowngenes [J]. Plant J, 2021,108(4):1193-1212
[28] Zhao J, Li H, Yin Y,. Fruit ripening inandis associated with distinct gene expression patterns [J]. FEBS Open Bio, 2020,10(8):1550-1567
[29] Schmid M, Davison TS, Henz SR,. A gene expression map ofdevelopment [J]. Nat Genet, 2005,37(5):501-506
[30] Kamiya N, Itoh J, Morikami A,. The SCARECROW gene's role in asymmetric cell divisions in rice plants [J]. Plant J, 2003,36:45-54
[31] Schrader J, Nilsson J, Mellerowicz E,. A high-resolution transcript profile across the wood-forming meristem of poplar identifies potential regulators of cambial stem cell identity [J]. Plant Cell, 2004,16:2278-2292
[32] Cui H, Kong D, Liu X,. SCARECROW, SCR-LIKE 23 and SHORT-ROOT control bundle sheath cell fate and function in[J]. Plant J, 2014,78(2):319-327
[33] Xu J, Hofhuis H, Heidstra R,. A molecular framework for plant regeneration [J]. Science, 2006,311(5759):385-388
[34] Galinha C, Hofhuis H, Luijten M,. PLETHORA proteins as dose-dependent master regulators ofroot development [J]. Nature, 2007,449(7165):1053-1057
[35] 糜利敏.AtSCL26和AtSCL29在氮脅迫下對擬南芥根系發育的功能研究[D].武漢:華中農業大學,2016
[36] 范吳蔚,潘志演,王原秀,等.獨腳金內酯對煙草腋芽生長以及抗氧化酶活性和糖含量的影響[J].植物生理學報,2021,57(4):910-918
[37] Yamada Y, Umehara M. Possible roles of strigolactones during leaf senescence [J]. Plants (Basel), 2015,4(3):664-677
Gene Identification and Organ Expression Characteristics ofSubfamily in
YU Ting-ting, XU A-fei, ZHANG Jia-lin, ZHANG Wei-wei, YANG Hong-qiang*
271018,
SHORT-ROOT (SHR) transcription factors belong to a subfamily of plant-specific GRAS. Based on the SHR sequencein, we obtained nine, ten, five, five and fourgenesfrom the genome-wide databases ofBorkh,Rehd.,(L.)(Linn.)andHu, respectively. The characteristics of these SHR subfamily members were compared and the protein structure and organ-specific expression ofin apple were analyzed. The results showed that the obtained SHR subfamily members encoded proteins with molecular masses ranging from 29743.72 to 62251.21 Da, all of which located in the nucleus. Thehad one pair of tandem duplicating genes and one pair of colinear genes on apple chromosomes;-encoded protein contains 10 conserved motifs, of which five were shared by the obtained SHR subfamily proteins. The protein secondary structures of MdSHRs and AtSHRs were mainly random coils and α-helices, and the three-dimensional structures of most MdSHRs and AtSHRs were similar. The promoters of the nineall contained cis-acting elements related to stress response and hormone response. Organ expression profiling showed that mosthad the highest expressions in flowers and fruits, lower expressions in shoots and seeds, and the lowest expressions in roots and seedling; the expression level ofin different organs of apple was basically opposite to those of other.
Apple; SHR subfamily genes; bioinformatics analysis
S661.1
A
1000-2324(2022)03-0346-09
10.3969/j.issn.1000-2324.2022.03.002
2022-03-05
2022-04-14
國家自然科學基金(32172517 & 31772251);國家重點研發計劃(2019YFD1000103)
于婷婷(1996-),女,碩士研究生,研究方向為蘋果生理與分子生物學. E-mail:1336179602@qq.com
Author for correspondence. E-mail:hqyang@sdau.edu.cn

