蝴蝶蘭PhPP2Aa基因作為低溫脅迫內參基因的研究
梁 芳,許申平,張 燕,王默霏,崔 波
蝴蝶蘭基因作為低溫脅迫內參基因的研究
梁 芳,許申平,張 燕,王默霏,崔 波*
鄭州師范學院生物工程研究中心,河南鄭州 450044
實時熒光定量PCR(qPCR)技術因其具有簡單靈敏、準確高效等諸多優點,成為目的基因表達水平研究最常用的技術手段。而結果的可靠性取決于很多因素,其中使用合適的內參基因是qPCR技術最基本的應用前提。許多研究表明沒有一種內參基因可以在任何條件下都能穩定地表達。目前尚未見到關于蝴蝶蘭低溫生長條件下最佳內參基因選擇的有關報道。蛋白磷酸酶2A(PP2A)是真核生物體內一種主要的細胞內源絲氨酸/蘇氨酸蛋白磷酸酶。本研究根據蝴蝶蘭低溫轉錄組測序結果克隆得到1個PP2A的A亞基基因,其cDNA開放閱讀框(ORF)長度為1764 bp,編碼1個含有587個氨基酸的蛋白,將該基因命名為,GenBank登錄號為MW847782。序列分析結果表明,該基因與其他植物核苷酸序列相似性均在80%以上,氨基酸序列與小蘭嶼蝴蝶蘭PP2A序列相似性為99.66%。基于氨基酸序列進化分析結果表明,蝴蝶蘭PhPP2Aa與小蘭嶼蝴蝶蘭和鐵皮石斛的親緣關系最近。將蝴蝶蘭與其他7種候選內參基因(、、、、、及)進行實時熒光定量PCR,用3種常用內參基因分析軟件對各個基因值進行穩定性分析,結果表明蝴蝶蘭8個候選內參基因在低溫脅迫條件下表達水平最穩定的內參基因為,其次為;最不穩定的基因為,其次為。以蝴蝶蘭作為內參基因探討低溫脅迫響應基因的表達情況,結果顯示蝴蝶蘭的表達模式符合低溫脅迫條件下的表達特性。該結果表明蝴蝶蘭基因可作為低溫脅迫條件下目的基因轉錄水平研究的內參基因。
蝴蝶蘭;蛋白磷酸酶2A;內參基因;低溫脅迫;序列分析
蛋白磷酸酶2A(protein phosphatase 2A,PP2A)是真核生物體內一種主要的細胞內源絲氨酸/蘇氨酸蛋白磷酸酶。PP2A在植物體內參與多種激素信號轉導途徑,由PP2A介導的蛋白去磷酸化在植物生物、非生物脅迫調控中發揮重要作用[1-4]。此外,PP2A還參與植物細胞的油脂代謝[5]和氮代謝[6]、光信號途徑[7]及開花時間調控過程[8]等。PP2A由65kDa結構亞基A、36kDa催化亞基C和多種功能特異的調節亞基B組成,保守的A亞基和C亞基形成二聚體核心酶,再與高度變異的B亞基構成具有生物活性的全酶[9]。每個亞基都有多個基因編碼,在擬南芥中,有3種A亞基、17種B亞基和5種C亞基[10]。A亞基由一系列保守的α螺旋重復序列組成,而且提供與B亞基和C亞基的結合位點[11]。在植物中A亞基(PP2Aa)由一到數個基因編碼,在擬南芥中編碼A亞基的基因有3個,分別為、和,其中在ABA信號轉導的早期起正調控作用,并影響生長素的轉運[12]。有研究表明參與植物脅迫響應[13],當功能缺失時,和才發揮生物學作用[14]。
蝴蝶蘭(spp.)原產于熱帶亞熱帶地區,性喜暖畏寒,在我國北方地區種植時,低溫成為影響其正常生長的重要環境因子。因此,耐冷性資源和基因挖掘是蝴蝶蘭育種工作的重要目標之一。在蝴蝶蘭耐冷性基因篩選過程中,實時熒光定量PCR(qPCR)是較為常用的技術手段,而qPCR技術的應用前提之一是合適內參基因的選擇。目前在蝴蝶蘭內參基因篩選及低溫條件下目的基因表達水平研究中,尚未見到關于低溫生長條件下最佳內參基因選擇的有關報道。傳統的內參基因很多,本研究從低溫轉錄組文庫中,篩選表達相對穩定的3個常用內參基因、、及5個新型內參基因、、、和作為候選內參基因,采用3個常用內參基因分析軟件geNorm[15]、NormFinder[16]和BestKeeper[17]對這8個候選內參基因的表達穩定性進行分析,并用蝴蝶蘭低溫脅迫響應基因作為驗證[18]。該結果為蝴蝶蘭低溫脅迫條件下基因表達分析及功能研究奠定基礎。
1 材料與方法
1.1 材料
以正常生長溫度的葉片為對照1(CK1)取樣,16℃/11℃處理3 d后為對照2(CK2)取樣,于11℃/6℃處理后1 d(T1)、2 d(T2)、3 d(T3)、5 d(T5)和7 d(T7)取樣。
1.2 方法
1.2.1 蝴蝶蘭基因序列分析 根據蝴蝶蘭低溫脅迫條件下葉片轉錄組測序結果,獲得一條蝴蝶蘭基因,在其ORF區兩端設計引物并克隆全長序列,利用DNAMAN軟件比對其推導的氨基端序列的同源性,用Clustal X和MEGA構建該基因的系統進化樹分析進化關系。
1.2.2 候選內參基因的實時熒光定量分析 根據低溫轉錄組測序結果,挑選8個候選內參基因,其readcount值在不同處理時間中相對穩定。分別設計qPCR引物,如表1所示。利用SYBR?Premix Ex TaqTMII對不同高溫脅迫處理時間后各基因表達情況進行qPCR反應檢測,反應總體系為20.0 μL,其中2×SYBR Premix ExII 10.0 μL,引物F和R各0.8 μL,cDNA 模板2.0 μL;ddH2O 6.4 μL;反應條件為:95℃預變性30 s;95℃變性15 s,58℃退火15 s,72℃延伸15 s,共40個循環。qPCR反應在Eppendorf Mastercycler ep realplex2熒光定量PCR儀上進行,每個樣品3次重復,并做陰性對照。PCR反應完成后,經儀器自動分析,查看每個基因的擴增情況,并導出相應的值。

表1 引物序列及用途
1.2.3 候選內參基因表達穩定性分析 根據得到的各個候選內參基因的值,利用分析軟件GeNorm、Bestkeeper和NormFinder對8個候選內參基因的表達穩定性進行分析,篩選蝴蝶蘭低溫生長條件下的最適內參基因。
1.2.4 內參基因標準曲線的制作 取等量不同低溫處理時間葉片的7個cDNA樣品,混合均勻作為標準品,分別稀釋10倍、100倍、1000倍和10 000倍及未稀釋樣品作為5個梯度,分別以5個梯度的cDNA為模板,以表1中設計的引物進行qPCR反應,20.0 μL反應體系配置及反應條件同1.2.2。
1.2.5 蝴蝶蘭基因在低溫條件下的表達 以蝴蝶蘭基因作為內參基因,以為對照,檢測NAC域蛋白基因在蝴蝶蘭低溫脅迫條件下不同處理時間葉片中的表達特性分析。
2 結果與分析
2.1 蝴蝶蘭PhPP2Aa基因的克隆與序列分析
以蝴蝶蘭葉片提取的cDNA第一鏈為模板,克隆得到基因目的片段1859 bp,ORF區全長為1764 bp,編碼587個氨基酸。將蝴蝶蘭基因序列進行BLAST比對分析,結果發現蝴蝶蘭與小蘭嶼蝴蝶蘭(,XM_ 020743358.1)核苷酸序列有31個不同堿基,序列相似性為99.15%;與鐵皮石斛(,XM_020819773.1)序列相似性為93.82%;與油棕(,XM_ 010943973.2)、鳳梨(,XM_0202 44641.1)、海棗(,XM_0087 84816.3)等植物序列相似性均在80%以上。表明該基因為編碼PP2A的A亞基的β基因,因此命名為,GenBank登錄號為MW847782。
將該基因推導的氨基酸序列與NCBI中其他9種相似度較高的植物PP2A蛋白進行多序列比對,結果發現PhPP2Aa與小蘭嶼蝴蝶蘭PP2A氨基酸序列有1個不同,序列相似性為99.66%,在第353位的氨基酸由D(天冬氨酸)變為E(谷氨酸)。與其他8種植物的氨基酸序列有多個位點不同,其中在第55位氨基酸由E變為D,第314位氨基酸由Q(谷氨酰胺)變為E,第322位氨基酸由P(脯氨酸)變為T(蘇氨酸)(圖1)。
在NCBI上下載了其他19種植物基因的氨基酸序列與蝴蝶蘭的序列共同構建系統進化樹,結果見圖2。由圖2可知,這些植物的PP2A氨基酸序列明顯分為2個大分支,蝴蝶蘭PhPP2Aa(hydrid,MW847782)與小蘭嶼蝴蝶蘭(,XM_020743358.1)的PP2A進化關系最近,其次是蘭科植物的鐵皮石斛(,XM_020819771.2),三者處于同一個進化分支;與玉米(,XM_008650108.2)、水稻(,AJ243828.1)、油棕(,XM_010930760.2)等單子葉植物均處于一個大分支;而另一個大分支為罌粟(,XM_026547132.1)、蓮(,XM_010266882.2)、葡萄(,XM_002276144.4)、陸地棉(,XM_016854089.1)等雙子葉植物。表明基因的進化在單雙子葉植物中具有高度的保守性。
以藥物不良反應這一節為例,該章節的重點在于掌握藥物不良反應分類及發生機制,難點在于診斷藥物不良反應的主要依據,在教學中教師需重點突出本章與醫院臨床藥學實踐緊密相結合的特點,爭取將學生的思維引導到如何在實踐中發現、判斷ADR。筆者在該部分內容的教學設計中引入BOPPPS模式,具體設計見表1、表2。

圖1 蝴蝶蘭PhPP2Aa基因編碼的氨基酸序列與其他植物PP2A的多序列比對

圖2 蝴蝶蘭PhPP2Aa與其他PP2A蛋白系統進化樹
2.2 蝴蝶蘭候選內參基因的表達穩定性分析
由圖3可知,蝴蝶蘭的8個候選內參基因PCR結果條帶單一,擴增產物大小位置與預期結果一致,且擴增曲線顯示均為單峰,表明所設計的引物可用于目的基因的qPCR分析。
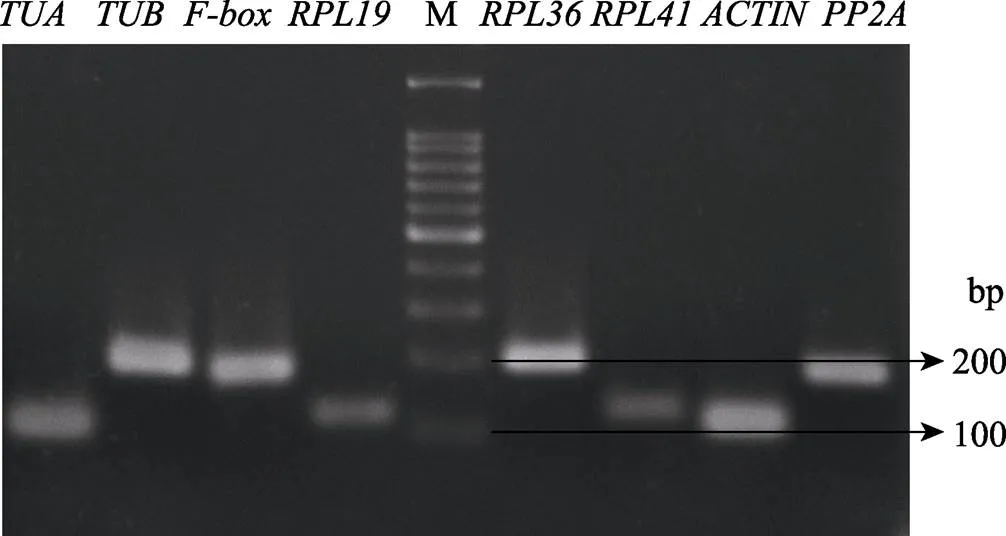
圖3 蝴蝶蘭候選內參基因的qPCR擴增結果
8個候選內參基因在蝴蝶蘭低溫處理不同時間下的表達值在19.11~27.55之間(圖4A),其中以的值最小,表達豐度最高;的值最大,表達豐度最低(圖4B)。
采用3個常用軟件geNorm、NormFinder和BestKeeper對8個候選內參基因的表達穩定性進行分析。geNorm和NormFinder是根據值判斷,值越小,表明基因穩定性越高;值越大,穩定性越低。BestKeeper是根據±值來判斷,值越小,表明該基因穩定性越高。如表2所示,geNorm和NormFinder分析結果均表明,和的值最小,穩定性最高,其次為。BestKeeper分析結果表明,的值最小,穩定性最高,其次為。3個軟件的分析結果均表明最不穩定的內參基因是,其次是。綜合分析表明,蝴蝶蘭8個候選內參基因在低溫脅迫條件下表達水平最穩定的內參基因是,其次是。
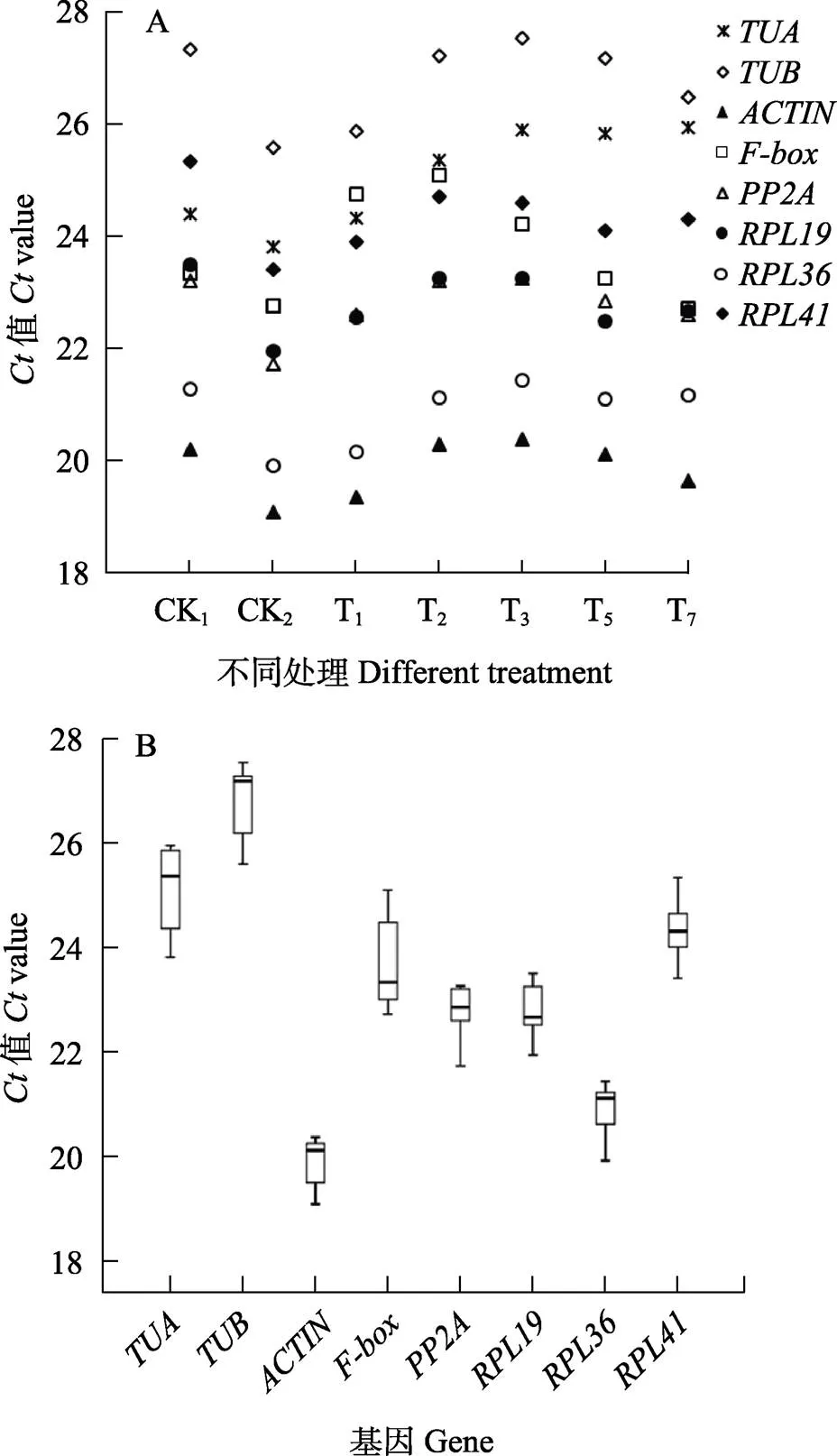
A:Ct平均值分布圖;B:Ct平均值箱式圖。
periods of cold treatment
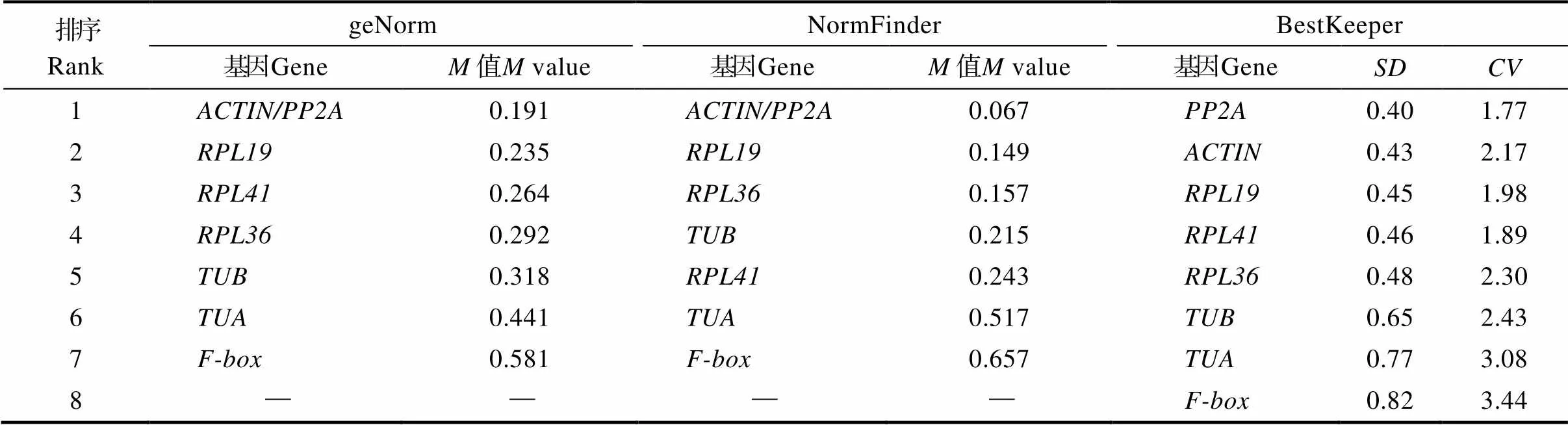
表2 蝴蝶蘭候選內參基因穩定性分析
2.3 蝴蝶蘭PP2A基因標準曲線的制作
由實時熒光定量PCR儀自動生成熔解曲線和標準曲線。結果顯示,基因的qPCR熔解曲線均為單峰,表明所設計的引物具有特異性,產物單一;基因的qPCR標準曲線為= –3.299+20.17,擴增效率為101%,相關系數為0.999。
2.4 蝴蝶蘭PP2A基因作為內參基因檢測低溫下PhNAC1表達量
以蝴蝶蘭為內參基因,檢測基因在低溫處理不同時期葉片中的表達情況,以常用的基因為對照。結果表明,蝴蝶蘭在11℃/6℃低溫處理條件下,基因相對表達量在CK2(低溫馴化階段后)中明顯上升;當逐步降溫過程結束進行11℃/6℃處理1 d后(T1),表達量開始下降,之后又逐漸升高,第5天時(T5)達到最高。基因在2個蝴蝶蘭品種中不同低溫處理時間下的相對表達水平,與以為內參基因時基因表達量變化總體趨勢基本一致(圖5)。表明以為內參基因進行相關基因分析結果是可靠的。
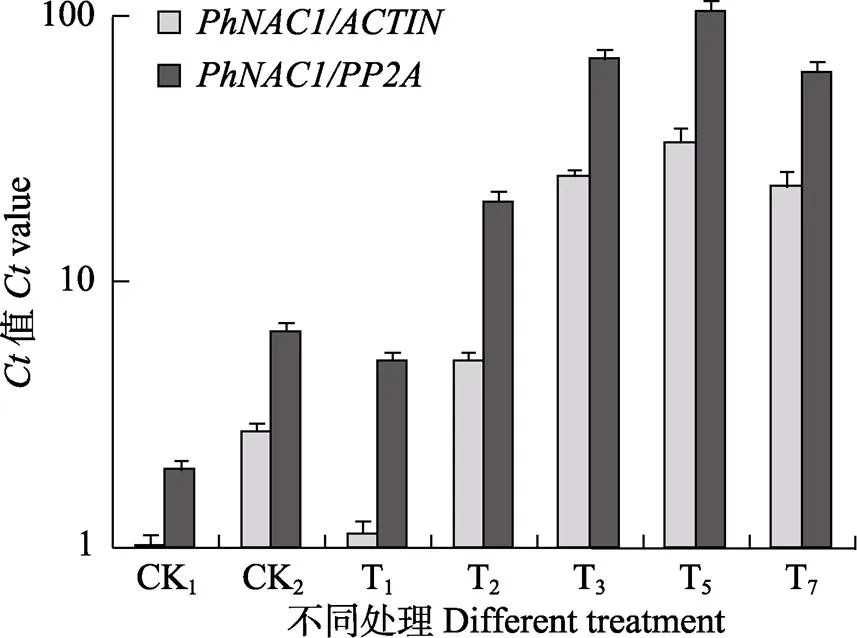
圖5 蝴蝶蘭PhNAC1基因在低溫脅迫條件下的表達特性
3 討論
由于qPCR具有簡單、靈敏度高、準確、快速等優點被廣泛應用于目的基因轉錄水平研究[19]。應用過程中為了保證結果的可重復性和準確性,有很多需要遵循的原則,其中最基本的先決條件就是使用可靠的內參基因[20]。理想的內參基因要求能不受RNA量及各種實驗條件和環境的影響,但是大量研究表明即使是被廣泛運用的內參基因,其表達水平也會隨著物種、組織及處理的不同而不同,沒有一種內參基因被證明可在任何條件下均表達穩定[21]。因此,在進行目的基因表達水平研究時,首先篩選在特定條件下合適的內參基因是一項重要的任務。雖然蝴蝶蘭的內參基因研究已有報道[22],但只研究了蝴蝶蘭在正常溫度下營養生長及生殖生長過程中目的基因研究的最適內參基因,尚未見到溫度脅迫下內參基因研究的相關報道。
較早使用的內參基因主要是看家基因,如、、等,近年來又有很多新的內參基因的相關研究,如、、等。有報道表明在甘草及海州常山干旱條件下[23-24]、板藍根ABA處理條件下[25]及冬油菜冷脅迫條件下的表達均最為穩定[26]。和在香蕉溫度脅迫時表達最穩定[27];在早熟禾低溫脅迫下的根中表達最穩定[28]。在甘薯、黃梁木及海州常山鹽脅迫條件下的研究表明的表達最穩定[24, 29-30],并指出和雖然被廣泛使用,但在甘薯不同品種中的研究表明其表達并不是最穩定的[20]。大豆在鎘脅迫條件下,、及的表達最穩定,表達最不穩定[31];在山胡椒中的研究表明,和在不同組織及果實發育期表達最穩定[32]。作為內參基因的應用已有許多報道,如作為油菜成熟胚中目的基因表達研究的內參基因[33]、橄欖的中果皮[34]、鷹嘴豆不同組織中[35]、生菜生長發育過程中[36]、鼠尾草的生殖階段[37]、狗尾草[38]、白楊的不定根再生階段[39]。由此可見,不同物種、不同條件或組織,最佳內參基因是不同的。因此,本研究從8個候選內參基因中,利用常用的3種內參基因分析軟件,篩選出基因作為研究蝴蝶蘭在低溫條件下目的基因表達功能研究的內參基因,其次為基因。也有相關報道作為低溫脅迫條件下的最佳內參基因,如對葡莖剪股穎在冷脅迫條件下內參基因表達情況的研究表明,最佳內參基因為和的聯合使用[40]。
本研究獲得了蝴蝶蘭基因的全長序列,經BLAST比對發現與其他植物核苷酸序列相似性在80%以上。系統進化分析表明,蝴蝶蘭與小蘭嶼蝴蝶蘭的基因氨基酸序列完全相同,與蘭科植物鐵皮石斛的親緣關系最近,三者處于同一進化分支。以往的研究表明,蝴蝶蘭基因在低溫脅迫條件下表達量會逐漸升高,本研究用基因驗證作為內參基因的穩定性,同時以常用內參基因為對照,結果發現,以和為內參基因時,基因在蝴蝶蘭不同低溫處理時間下的相對表達水平變化總體趨勢基本一致,表明以為內參基因進行相關基因分析結果是可靠的。綜上所述,通過對蝴蝶蘭基因的表達穩定性分析及驗證,表明該基因在蝴蝶蘭正常生長溫度及低溫脅迫條件下,不同時間的葉片中均能穩定表達,說明基因可作為蝴蝶蘭低溫脅迫條件下的內參基因使用,為研究蝴蝶蘭低溫脅迫條件下相關基因的表達特性分析、耐冷性資源及基因挖掘奠定基礎。
[1] THAKORE C U, LIVENGOOD A J, HENDERSHOT III J D, CORUM J W, LATORRE K A, RUNDLE S J. Characterization of the promoter region and expression pattern of threeprotein phosphatase type 2A subunit genes[J]. Plant Science, 1999, 147: 165-176.
[2] DURIAN G, RAHIKAINEN M, ALEGRE S, BROSCHéM, KANGASJ?RVI S. Protein phosphatase 2A in the regulatory network underlying biotic stress resistance in plants[J]. Frontiers in Plant Science, 2016, 7: 812.
[3] XU P, YONG B, SHAO H H, SHEN J B, HE B, MA Q Q, YUAN X H, WANG Y. Cloning and characterization of a serine/threonine protein phosphatase 2A-encoding genefrom(L.) Lam.[J]. Turkish Journal of Biology, 2017, 41: 148-157.
[4] LIU D, LI B, FENG G, MAO X G, LI A, CHANG X P, JING R L. TaPP2AbB??γ, a wheat regulatory subunit of PP2A enhanced abiotic stress tolerance[J]. Plant Growth Regulation, 2019, 89: 345-355.
[5] HEIDARI B, MATRE P, NEMIE-FEYISSA D, MEYER C, ROGNLI O A, MOLLER? S G, LILLO C. Protein phosphatase 2A B55 and a regulatory subunits interact with nitrate reductase and are essential for nitrate reductase activation[J]. Plant Physiology, 2011, 156: 165-172.
[6] KATAYA A R, HEIDARI B, HAGEN L, KOMMEDAL R, SLUPPHAUG G, LILLO C. Protein phosphatase 2A holoenzyme is targeted to peroxisomes by piggybacking and positively affects peroxisomal betaoxidation[J]. Plant Physiology, 2015, 167: 493-506.
[7] TSENG T S, BRIGGS W R. Thercn1-1 mutation impairs dephosphorylation of Phot2, resulting in enhanced blue light responses[J]. Plant Cell, 2010, 22: 392-402.
[8] HEIDARI B, NEMIE-FEYSSSA D, KANGASJ?RVI S, LILLO C. Antagonistic regulation of flowering time through distinct regulatory subunits of protein phosphatase 2A[J]. PLoS One, 2013, 8: e67987.
[9] YU S H, LEI H Y, CHANG W Z,S?LL D, HONG G F. Protein phosphatase 2A: identification in Oryza sativa of the gene encoding the regulatory A subunit[J]. Plant Molecular Biology, 2001, 45: 107-112.
[10] LILLO C, KATAYA A R A, HEIDARI B, CREIGHTON M T, NEMIE-FEYISSA D, GINBOT Z, JONASSEN E M. Protein phosphatases PP2A, PP4 and PP6: mediators and regulators in development and responses to environmental cues[J]. Plant Cell and Environment, 2014, 37(12): 2631-2648.
[11] BOOKER M A, DELONG A. Atypical protein phosphatase 2A gene families do not expand via paleopolyploidization[J]. Plant Physiology, 2017, 173(2): 1283-1300.
[12] KWAK J M, MOON J H, MURATA Y, KAZUYUKI K, LEONHARDT N, DELONG A, SCHROEDER J I. Disruption of a guard cell-expressed protein phosphatase 2A regulatory subunit,, confers abscisic acid insensitivity in[J]. Plant Cell, 2002, 14(11): 2849-2861.
[13] BLAKESLEE J J, ZHOU H W, HEATH J T, SKOTTKE K R, BARRIOS J A R, LIU S Y, DELONG A. Specificity of RCN1-mediated protein phosphatase 2A regulation in meristem organization and stress response in roots[J]. Plant Physiology, 2008, 146(2): 539-553.
[14] ZHOU H W, NUSSBAUMER C, CHAO Y, DELONG A. Disparate roles for the regulatory A subunit isoforms inprotein phosphatase 2A[J]. Plant Cell, 2004, 16(3): 709-722.
[15] VANDESOMPELE J, DE PRETER K, PATTYN F, POPPE B, VAN ROY N, DE PAEPE A, SPELEMAN F. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3: RESEARCH0034.
[16] ANDERSEN C L, JENSEN J L, ?RNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64: 5245-5250.
[17] PFAFFL M W, TICHOPAD A, PRGOMET C, NEUVIANS T P. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26: 509-515.
[18] 梁 芳, 張 燕, 牛蘇燕, 袁秀云, 崔 波. 蝴蝶蘭基因序列分析及對低溫脅迫的響應[J]. 廣西植物, 2020, 40(6): 845-853.
LIANG F, ZHANG Y, NIU S Y, YUAN X Y, CUI B. Sequence analysis ofgene fromand its response to cold stress[J]. Guihaia, 2020, 40(6): 845-853. (in Chinese)
[19] SHUKLA P, REDDY R A, PONNUVEL K M, ROHELAG K, SHABNAM A A, GHOSH M K, MISHRA R K. Selection of suitable reference genes for quantitative real-time PCR gene expression analysis in mulberry (L.) under different abiotic stresses[J]. Molecular Biology Reports, 2019, 46: 1809-1817.
[20] GUTIERREZ L, MAUIAT M, POLLOUX J, BELLINI C, VAN WUYTSWINKEL O. Towards a systematic validation of references in real-time RT-PCR[J]. Plant Cell, 2008, 20: 1734-1735.
[21] ARGYROPOULOS D, PSALLIDA C, SPYROPOULOS C G. Generic normalization method for real-time PCR application for the analysis of the mannanase gene expressed in germinating tomato seed[J]. FEBS Journal, 2006, 273: 770-777.
[22] YUAN X Y, JIANG S H, WANG M F, MA J, ZHANG X Y, CUI B. Evaluation of internal control for gene expression inby quantitative real-time PCR[J]. Applied Biochemistry and Biotechnology, 2014, 173(6): 1431-1445.
[23] MAROUFI A. Selection of reference genes for real-time quantitative PCR analysis of gene expression inunder drought stress[J]. Biologia Plantarum, 2016, 60(4): 645-654.
[24] HUA Y J, YUE Y Z, CHEN G W, YAN T T, DING W J, SHI T T, HU D, WANG L G, YANG X L. Selection of appropriate reference genes for quantitative real-time PCR in[J]. BioRxiv,2019. doi: org/ 10.1101/625145.
[25] QU R, MIAO Y, CUI Y, CAO Y, ZHOU Y, TANG X, YANG J, WANG F. Selection of reference genes for the quantitative real-time PCR normalization of gene expression infortune[J]. BMC Molecular Biology, 2019, 20(1): 9.
[26] MA L, WU J, QI W, COULTER J A, FANG Y, LI X, LIU L, JIN J, NIU Z, YUE J, SUN W. Screening and verification of reference genes for analysis of gene expression in winter rapeseed (L.) under abiotic stress[J]. PLoS One, 2020, 15(9): e0236577.
[27] CHEN L, ZHONG H Y, KUANG J F, LI J G, LU W J, CHEN J Y. Validation of reference genes for RT-qPCR studies of gene expression in banana fruit under different experimental conditions[J]. Planta, 2011, 4(2): 377-390.
[28] NIU K, SHI Y, MA H. Selection of candidate reference genes for gene expression analysis in kentucky bluegrass (L.) under abiotic stress[J]. Frontiers in Plant Science, 2017, 8: 193.
[29] ZHANG D, LI J, LI B, LI C, CHEN X, OUYANG K. Internal reference gene selection under different hormone stresses in multipurpose timber yielding tree[J]. Forests, 2020, 11: 1014.
[30] PARK S C, KIM Y H, JI C Y, PARK S, JEONG J C, LEE H S, KWAK S S. Stable internal reference genes for the normalization of real-time PCR in different sweetpotato cultivars subjected to abiotic stress conditions[J]. PLoS One, 2012, 7(12): e51502.
[31] WANG Y, YU K, POYSA V, CHUN S, ZHOU Y. Selection of reference genes for normalization of qRT-PCR analysis of differentially expressed genes in soybean exposed to cadmium[J]. Molecular Biology Reports, 2012, 39(2): 1585-1594.
[32] LIN L, HAN X, CHEN Y, WU Q, WANG Y. Identification of appropriate reference genes for normalizing transcript expression by quantitative real-time PCR in[J]. Molecular Genetics and Genomics, 2013, 8(12): 727-737.
[33] CHEN X, TRUKSA M, SHAH S, WESELAKE R J. A survey of quantitative real-time polymerase chain reaction internal reference genes for expression studies in[J]. Analytical Biochemistry, 2010, 405(1): 138-140.
[34] RAY D L, JOHNSON J C. Validation of reference genes for gene expression analysis in olive () mesocarp tissue by quantitative real-time RT-PCR[J]. BMC Research Notes, 2014, 7: 304.
[35] REDDY D S, BHATNAGAR-MATHUR P, REDDY P S, SRI CINDHURI K, SIVAJI GANESH A, SHARMA K K. Identification and validation of reference genes and their impact on normalized gene expression studies across cultivated and wildspecies[J]. PLoS One, 2016, 11(2): e0148451.
[36] SGAMMA T, PAPE J, MASSIAH A, JACKSON S. Selection of reference genes for diurnal and developmental time-course real-time PCR expression analyses in lettuce[J]. Plant Methods, 2016, 12: 21.
[37] GOPALAM R, RUPWATE S D, TUMANEY A W. Selection and validation of appropriate reference genes for quantitative real-time PCR analysis in[J]. PLoS One, 2017, 12(11): 0186978.
[38] NGUYEN D Q, EAMENS A L, GROF C P L. Reference gene identification for reliable normalisation of quantitative RT-PCR data in[J]. Plant Methods, 2018, 14: 24.
[39] TANG F, CHU L, SHU W, HE X, WANG L, LU M. Selection and validation of reference genes for quantitative expression analysis of miRNAs and mRNAs in Poplar[J]. Plant Methods, 2019, 15: 35.
[40] CHEN Y, HU B, TAN Z, LIU J, YANG Z, LI Z, HUANG B. Selection of reference genes for quantitative real-time PCR normalization in creeping bentgrass involved in four abiotic stresses[J]. Plant Cell Reports, 2015, 34(10): 1825-1834.
as Reference Gene inunder Low-temperature Stress
LIANG Fang, XU Shenping, ZHANG Yan, WANG Mofei, CUI Bo*
Bioengineering Research Center, Zhengzhou Normal University, Zhengzhou, Henan 450044, China
Quantitative real-time polymerase chain reaction (qPCR) analysis, with the benefits of simplicity, high sensitivity, accuracy and high-throughput characteristics, has been used in many fields to quantify the transcript levels of target genes. There are many rules that must be followed to ensure the reproducible and accurate expression profiles of target genes using qPCR. Among them, the use of a reliable internal control known as a reference gene for data normalization is the elementary prerequisite for valid results and proper analysis. Numerous studies have suggested that no single reference gene is always expressed stably under any condition. There are no reports on the selection of optimal reference genes forunder low temperature conditions. Protein phosphatase 2A (PP2A) is a major intracellular serine / threonine protein phosphatase in eukaryotes.A subunit gene of PP2A was cloned by the results of the transcriptome sequencing ofhybrid under cold stress, which was namedand the GenBank accession number was MW847782. The coding region (ORF) ofwas 1764 bp, encoding 587 amino acids. Homologous alignment showed that it shared over 80% nucleotide sequence similarity within other plants, and that it shared 99.66% amino acid sequence similarity with. The phylogenetic tree analysis based on the amino acid suggested that the relationship of PP2A betweenhybrid,andwas close, which belonged to the same branch. Three conventional software (geNorm, NormFinder, BestKeeper) were used to analyze the expression stability of 8 candidate reference genes (,,,,,,and) from. The results showed that the most stable reference gene was, followed by. And, the most unstable was, followed by. Usingof() as the reference gene to explore transcriptional profile of the target gene, the results demonstrated that the expression pattern ofwas consistent with its characterize under cold stress. Therefore,can be used as the internal reference gene for the analysis of target gene inunder low-temperature stress.
; protein phosphatase 2A; internal reference gene; low-temperature stress; sequence analysis
Q785;Q786
A
10.3969/j.issn.1000-2561.2022.07.004
2022-02-10;
2022-03-19
河南省科技攻關計劃項目(No. 212102110116,No. 222102110470);河南省高校重點科研項目(No. 22A210025)。
梁 芳(1982—),女,博士,講師,研究方向:花卉分子育種。*通信作者(Corresponding author):崔 波(CUI Bo),E-mail:cuibo@zznu.edu.cn。

