基于Csy4與MCP的新型迷你基因組編輯系統(tǒng)的構建
鄧嘉輝 雷建峰 趙燚 劉敏 胡子曜 尤揚子 邵武奎柳建飛 劉曉東
(1. 新疆農業(yè)大學生命科學學院,烏魯木齊 830052;2. 新疆農業(yè)大學農學院 教育部棉花工程研究中心 農業(yè)生物技術重點實驗室,烏魯木齊 830052)
基因組編輯技術是指利用人工構建的特異性核酸酶對生物體核酸序列進行刪除、插入、替換等各種修飾的基因工程技術。該技術的出現為生物體功能基因組學研究提供了便捷的反向遺傳學手段[1-2]。目前,基因編輯技術主要包括鋅指核酸酶技術(zinc finger nucleases, ZFNs)、類轉錄激活因子效應物核酸酶技術(transcription activator?like effector nucleases, TALENs)及CRISPR/Cas(clustered regularly interspaced short palindromic repeat?associated protein)系統(tǒng),其中,CRISPR/Cas系統(tǒng)由于編輯效率高、操作耗費時間少和成本低等優(yōu)點,成為當前廣泛應用的主流基因編輯系統(tǒng)[3-4]。
ZFN由鋅指蛋白(zinc finger protein, ZFP)和限制性內切酶Fok I融合構成。3-5個串聯(lián)的ZFP構成ZFN的DNA結合結構域,可與DNA靶位點特異性結合,然后引導FokI蛋白二聚體發(fā)揮DNA切割功能[5],之后通過引導核酸內切酶切割DNA靶位點序列,形成DNA雙鏈斷裂(DNA double?strand break, DSB),從而誘導細胞進行DNA損傷修復。細胞通過非同源末端連接(non?homologous end joining,NHEJ)或同源重組(homology?directed repair, HDR)兩種不同的DNA修復途徑來實現基因組編輯,包括基因敲除(knockout)、基因定點突變及外源基因的定點整合(knockin)等[6]。TALEN技術則繼承ZFN技術的設計原理,通過類轉錄激活效應因子(transcription activator?like effector, TALE)與DNA靶位點特異結合,誘導FokI蛋白二聚體在靶位點進行切割[7-9]。CRISPR/Cas9技術是通過人工合成的引導RNA(single strand guide RNA, sgRNA)序列與靶位點特異性結合,引導Cas9蛋白進行DNA雙鏈切割[10]。
第一、二代基因組編輯技術ZFNs和TALENs雖然實現了特異性地識別靶位點并對靶標基因進行敲除,但是其構建繁瑣、難度大、時間長、脫靶率高、可操作性不高等問題嚴重限制了它們的應用范圍[11]。CRISPR/Cas技術雖然突變效率、精確度及安全性更高,耗費時間更少,但是其靶向核酸酶分子量大、依然存在脫靶現象和受PAM識別序列的限制等問題,尚需要進一步改進[12]。
MCP蛋白(major capsid protein, MCP)是MS2噬菌體的外殼蛋白,由130個氨基酸組成,它可以精確識別并結合由19或者21個核苷酸組成的MS2小RNA莖環(huán)結構(MS2?SL)[13-14]。目前,MCP蛋白被用來識別MS2?SL莖環(huán)結構并融合轉錄抑制因子或者轉錄激活因子來控制下游基因的表達[15]。Csy4(CRISPR associated protein, Cas6f)是一種參與CRISPR1?F系統(tǒng)crRNA生成的RNA酶,由188個氨基酸組成,其發(fā)揮功能除了需要識別特異性靶序列外,還需要靶序列3′端形成5個堿基配對的莖環(huán)結構。因為同時對堿基序列和二級結構的要求導致Csy4相比傳統(tǒng)內切酶具備較高的特異性[16-17]。由于MCP蛋白具有識別并結合guide?SL的功能,Csy4蛋白也可以識別并結合sgRNA,它們的功能類似于CRISPR/Cas9基因編輯系統(tǒng)內Cas9蛋白,但它們并無切割活性。
本研究基于一代基因編輯技術ZFNs和二代基因編輯技術TALENs的原理,選取分子量小、RNA高結合能力和高特異性的MCP蛋白和Csy4蛋白,與具有核酸內切酶特性的FokI蛋白構建融合蛋白。擬建立MCP?FokI和Csy4?FokI新型雙靶位點編輯系統(tǒng),將FokI蛋白構建至靶向蛋白MCP或Csy4蛋白的C端和N端,讓正反兩個靶向蛋白分別識別目標基因位點左右兩側的DNA序列,通過探究不同的中間間隔區(qū)的間距使FokI在靶位點形成活性二聚體,進而發(fā)揮內切酶活性,造成DSB,細胞通過HDR或NHEJ的機制進行修復進而實現基因編輯(圖1)。希望通過構建MCP?FokI和Csy4?FokI這兩個新型雙靶位點編輯系統(tǒng)來解決CRISPR/Cas基因組編輯技術存在的脫靶現象和受PAM識別序列限制等問題。

圖1 MCP-FokI(FokI-MCP)和Csy4-FokI(FokI-Csy4)基因組編輯體系示意圖Fig. 1 Schematic diagram of MCP-FokI(FokI-MCP)and Csy4-FokI(FokI-Csy4)genome editing system
1 材料與方法
1.1 材料
所用AtU6?26∷sgRNA載體、棉花葉皺縮病毒載體CLCrV?A和CLCrV?B均為新疆農業(yè)大學作物功能基因組學實驗室所保存,載體CLCrV?A和CLCrV?B由浙江大學周雪平教授惠贈。植物基因組DNA提取試劑盒(EasyPure Plant Genomic DNA Kit)、克隆載體(pEASY?B Zero)和感受態(tài)細胞(Trans1?T1)均購于北京全式金生物公司。所用限制性內切酶購于賽默飛生物公司。引物合成及測序由上海生工生物科技有限公司完成,其余試劑及藥品均由新疆本地生物公司代購。
1.2 方法
1.2.1 迷你基因組編輯系統(tǒng)不同中間間隔區(qū)的設計 分別使用2個不同U6啟動子驅動成對的guide?SL左側靶位點和右側靶位點,以減少使用相同啟動子導致基因沉默的概率。已有2個單獨研究團隊研究表明CRISPR/dCas9?FokI對EGFP編輯效率最高的中間間隔區(qū)的距離為14-25 bp[18]、13-17 bp[19]。因為并不知道有效切割所需的間隔距離,因此本研究設計4對針對AtCLA1不同位點的guide?SL和7對不同位點的sgRNA來探究迷你基因組編輯系統(tǒng)的編輯能力。
1.2.2 MCP?FokI(FokI?MCP)和Csy4?FokI(FokI?Cs?y4)基因編輯載體的構建 參照雷建峰[20]方法構建基因編輯載體,首先構建35∷MCP?FokI?Ter?B zero和35∷FokI? MCP?Ter?B zero過渡載體,并經Xho I、Sal I、Xba I和BamH I酶切鑒定。構建4個AtU6∷SL?B zero和4個AtU6?26∷SL?B zero過渡載體,經Kpn I和BamH I雙酶切鑒定。將構建好的靶向敲除AtCLA1的不同AtU6?26∷SL和AtU6∷SL片段與35∷MCP?FokI?Ter和35∷FokI?MCP?Ter片段分別重組為35∷MCP?FokI?Ter?P1300和35∷FokI?MCP?Ter?P1300載體,經Hind III、Sal I、Xba I和Kpn I酶切鑒定,驗證是否成功。
通過Transfer?PCR目的片段替換的方法[21]對35S∷MCP?FokI?Ter和35S∷FokI?MCP?Ter表達載體進行改造,將MCP片段替換為Csy4,經Dpn I酶切3 h,轉化、測序,并經Spe I和Pac I酶切鑒定。分別在35S∷FokI?Csy4載體與35S∷Csy4?FokI載體的5′端添加Spe I酶切位點,3′端添加Pac I酶切位點,經測序比對和Spe I與Pac I雙酶切鑒定,成功構建Spe I?35S∷Csy4?FokI?B zero?Pac I和Spe I?35S∷FokI?Csy4?B zero?Pac I載體。設計7對針對CLA1不同位點的sgRNA,分別對4個AtU6∷SL?B zero和2個AtU6?26∷SL?B zero載體進行改造,通過Tansfer?PCR目的片段替換的方法將SL片段替換為Csy4?sgRNA,構建AtU6∷sgRNA(1-4)?B zero和AtU6?26∷sgRNA(5-6)?B zero 6個過渡載體,分別在這6個載體的5′端添加Pac I酶切位點,3′端添加Avr II酶切位點,之后將4個AtU6∷sgRNA和2個AtU6?26∷sgRNA片段兩兩串聯(lián)連接到SK載體上,經Kpn I、EcoR I和Xba I酶切鑒定。將FokI?Csy4和Csy4?FokI片段與CLCrV?A載體連接,同時,將其分別與7個不同間距的雙靶序列片段與CLCrV?A載體串聯(lián)連接,將FokI?Csy4和Csy4?FokI與CLCrV?A載體連接,同時將其分別與7個不同間距的雙靶序列片段與CLCrV?A載體串聯(lián)連接,經Spe I、Avr II雙酶切鑒定靶向敲除擬南芥AtCLA1的Csy4?FokI和FokI?Csy4編輯載體是否構建成功。
1.2.3 轉化農桿菌感受態(tài) 吸取重組成功的CLCrV?A基因編輯載體與CLCrV?B質粒各20 μL,加入100 μL剛剛解凍的農桿菌GV3101感受態(tài)中,冰浴35 min,液氮速凍2 min,立即放入37℃水浴鍋中熱激3 min,加600 μL無抗LB液體培養(yǎng)基,28℃、220 r/min恒溫搖床中孵育4 h,在LB固體培養(yǎng)基(含50 μg/mL Rif和50 μg/mL Kan)上篩選陽性克隆,酶切鑒定,得到含編輯載體的陽性轉化菌用于注射擬南芥葉片。
1.2.4 浸花法轉化擬南芥 采用浸花法[22]對野生型擬南芥Col?0進行轉化。取農桿菌凍存液10 μL接種于2 mL LB培養(yǎng)基(50 μg/mL Kan和50 μg/mL Rif)中活化。28℃,200 r/min振蕩培養(yǎng)20 h左右。然后按1∶100體積比將活化菌液接種到25 mL LB培養(yǎng)基(50 μg/mL Kan和50 μg/mL Rif)中,于28℃,200 r/min振蕩培養(yǎng),搖至菌液的OD600≈1.6-1.8時,離心收集農桿菌培養(yǎng)物,重懸于新鮮配制的轉化液(1/2 MS液體培養(yǎng)基含5%蔗糖,0.02% Silwet L?77),至終濃度OD600≈0.8-1.0,轉化擬南芥Col?0。在篩選轉化子時,種子用5% NaClO消毒5 min,置于1/2 MS培養(yǎng)基(含30 μg/mL Kan)上萌發(fā)。2周后,將陽性苗移栽到土壤中,在22℃ 16 h光照/8 h黑暗條件下生長。選取生長15 d左右的擬南芥幼苗進行接種轉化試驗。
1.2.5 瞬時轉化擬南芥 將?80℃保存的陽性轉化菌在LB固體培養(yǎng)基(含50 μg/mL Rif和50 μg/mL Kan)上劃線活化,挑取單克隆接種于體積為10 mL LB液體培養(yǎng)基(含50 μg/mL Rif和50 μg/mL Kan)中,28℃、220 r/min恒溫搖床中搖至OD600≈1.5左右,4 000 r/min離心10 min沉淀菌體,接著用含有10 mmol/L MgCl2、10 mmol/L MES和200 μmol/L乙酰丁香酮的懸浮液懸浮至OD600≈1.0,室溫遮光靜置3 h。接種時,將CLCrV?A和CLCrV?B懸浮菌液按1∶1等量混合,使用1 mL注射器注射到擬南芥葉片中進行瞬時表達。
1.2.6 突變檢測 剪取注射農桿菌后的擬南芥葉片100 mg左右,提取基因組DNA。參照Gao等[23]方法檢測AtCLA1是否發(fā)生編輯。利用PCR擴增出含有EcoR V和Xba I酶切位點的目的片段(表1)。用對應的限制性內切酶消化擴增產物,檢查是否存在未被消化的PCR擴增條帶,初步判定是否發(fā)生基因編輯,通過HI?TOM高通量測序,將測序結果與基因序列進行比對,確定AtCLA1的突變類型。

表1 引物及用途Table 1 Primers and applications
1.2.7 融合蛋白結構預測 利用蛋白結構數據庫AlphaFold2(https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/ AlphaFold2.ipynb)對融合蛋白的結構進行預測。
1.2.8 瞬時表達載體檢測 以注射7個不同中間間隔區(qū)距靶序列的Csy4?FokI基因編輯載體14 d后的子葉葉片基因組DNA為模板,分別以未注射農桿菌的擬南芥Col?0和注射未帶靶序列的Csy4?FokI載體為陰性對照。利用CLCrVB病毒載體上特異性引物(表1)進行PCR擴增。
2 結果
2.1 融合蛋白結構分析
由于MCP和Csy4蛋白較小,與FokI融合后是否會影響彼此的三維空間,進而影響各自功能的發(fā)揮。運用AlphaFold2蛋白結構數據庫對MCP?FokI和FokI?MCP融合蛋白進行預測(圖2?A),將FokI蛋白和MCP蛋白的預測結果圖同2個融合蛋白的預測結果圖進行比對,2個功能域融合后,并沒有改變FokI蛋白形成二聚體的功能結構,也沒有改變MCP蛋白識別并結合guide?SL的功能結構。通過對Csy4?FokI和FokI?Csy4融合蛋白進行預測(圖2?B),并將FokI蛋白和Csy4蛋白的預測結構圖同2個融合蛋白的預測結構圖進行比對發(fā)現,沒有改變FokI蛋白形成二聚體的功能結構,也沒有改變Csy4蛋白識別并結合sgRNA的功能結構。

圖2 融合蛋白三級結構預測Fig. 2 Tertiary structure prediction of fusion protein
2.2 MCP?FokI編輯載體構建
本研究設計4對針對CLA1不同位點的guide?SL,中間間隔區(qū)的間距分別為10、14、16和25 bp,中間間隔區(qū)的序列上有EcoR V和Xba I酶切位點,便于突變檢測(圖3?A)。35∷MCP?FokI?Ter?B zero和35∷FokI?MCP? Ter?B zero過渡載體(圖3?C)、4個AtU6∷SL?B zero和4個AtU6?26∷SL?B zero靶序列過渡載體(圖3?D)以及帶有4個不同中間間隔區(qū)的35∷MCP?FokI?Ter?P1300和35∷FokI?MCP?Ter?P1300載體編輯載體(圖3?B, E)經酶切鑒定及測序比對后與預期結果相吻合,說明靶向敲除AtCLA1的MCP?FokI和FokI?MCP編輯載體已構建成功。
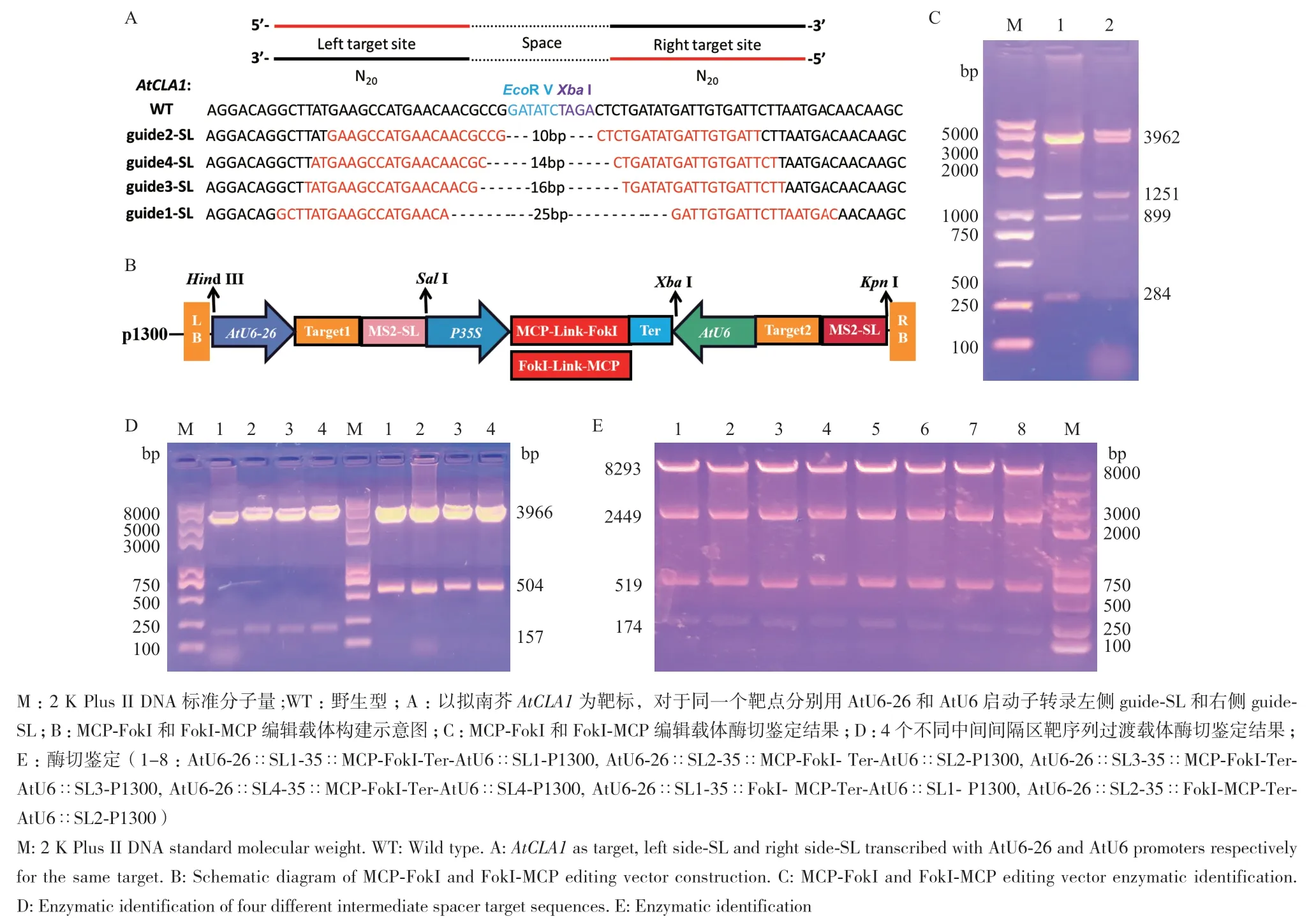
圖3 MCP-FokI和FokI-MCP基因編輯載體構建圖Fig. 3 Construction of MCP-FokI and FokI-MCP gene editing vector
2.3 MCP?FokI基因組編輯技術可行性檢測
運用浸花法將構建的編輯載體分別轉化野生型擬南芥Col?0,潮霉素篩選獲得大量轉基因T1代植株(圖4)。選取轉基因陽性苗基因組DNA為模板進行擴增,PCR擴增覆蓋突變位點的AtCLA1。通過Hi?TOM高通量測序,結果顯示,選取的4對不同中間間隔區(qū)的CLA1?SL、MCP?FokI和FokI?MCP編輯系統(tǒng)均未能實現對靶基因的靶向編輯,與此同時,并沒有觀察到AtCLA1突變植株明顯的表型。

圖4 MCP-FokI和FokI-MCP基因編輯系統(tǒng)靶向敲除AtCLA1Fig. 4 Targeted knockout of AtCLA1 gene by MCP-FokI and FokI-MCP gene editing system
2.4 Csy4?FokI編輯載體的構建
Spe I?35S∷Csy4?FokI?B zero?Pac I和Spe I?35S∷?FokI?Csy4?B zero?Pac I過渡載體(圖5?C)、4個AtU6∷sgRNA?B zero和2個AtU6?26∷sgRNA?B zero過渡載體、7個中間間隔區(qū)的靶序列過渡載體(圖5?D)以及帶有7個中間間隔區(qū)靶序列的FokI?Csy4?CLCrV?A和Csy4?FokI?CLCrV?A載體(圖5?B, E),經酶切鑒定及測序比對后,與預期結果相吻合,表明說明靶向敲除擬南芥CLA1的Csy4?FokI和FokI?Csy4編輯載體已構建成功。

圖5 Csy4-FokI和FokI-Csy4基因編輯載體構建圖Fig. 5 Construction of Csy4-FokI and FokI-Csy4 gene editing vector
2.5 Csy4?FokI基因組編輯技術可行性檢測
通過葉片注射法將構建的編輯載體,分別注射到野生型擬南芥Col?0葉片中進行瞬時表達。接種病毒7-14 d后并未觀察到擬南芥CLA1被編輯后明顯的白化表型。
分別提取野生型Col?0擬南芥基因組DNA與注射Csy4?FokI編輯載體的擬南芥基因組DNA,對CLCrV?B基因組進行PCR擴增。結果顯示,在注射Csy4?FokI編輯載體的擬南芥基葉片中可以檢測到CLCrV?B病毒積累,而在野生型擬南芥未檢測到CLCrV?B病毒的積累(圖6?A, B)。表明編輯載體已成功轉入擬南芥中。

圖6 Csy4-FokI和FokI-Csy4基因編輯系統(tǒng)靶向敲除AtCLA1Fig. 6 Targeted knockout of AtCLA1 gene by Csy4-FokI and FokI-Csy4 gene editing system
通過Hi?TOM高通量測序,結果顯示,在Csy4?FokI基因組編輯體系中,檢測范圍在靶位點sgRNA(1-5)之間64 bp區(qū)域,中間間隔區(qū)為25 bp的植株#5、#6和#7出現了3種類型的堿基替換,平均編輯效率為2.13%;檢測范圍在靶位點sgRNA(2-6)之間48 bp區(qū)域,中間間隔區(qū)為10 bp的植株#15出現了1種類型的堿基替換,編輯效率為0.3%;檢測范圍在靶位點sgRNA(1-6)之間57 bp區(qū)域,中間間隔區(qū)為19 bp的植株#17和#19出現了3種類型的堿基替換,平均編輯效率為1.13%;檢測范圍在靶位點sgRNA(4-5)之間56 bp區(qū)域,中間間隔區(qū)為18 bp的植株#5、#6和#7出現了3種類型的堿基替換,平均編輯效率為1.87%。而在野生型、Csy4?FokI靶位點空載對照和FokI?Csy4基因組編輯體系中未檢測到編輯的發(fā)生。結果表明,在Csy4?FokI基因組編輯體系中選取的7對不同中間間隔區(qū)的sgRNA,CLCrV介導的Csy4?FokI編輯系統(tǒng)中間間隔區(qū)為10、18、19和25 bp能夠實現對靶基因的靶向編輯(圖6?C),但是突變類型均為堿基置換類型且編輯效率很低,而CLCrV介導的FokI?Csy4基因組編輯體系和Csy4?FokI編輯系統(tǒng)中間間隔區(qū)為12、13和16 bp均不能實現基因編輯。
3 討論
基因組編輯技術以準確、高效、簡單地操作而發(fā)展迅速,近年來,以CRISPR/Cas系統(tǒng)為代表的第三代基因組編輯技術在基因功能研究、植物分子育種、疾病治療等方面取得了重要進展,但CRISPR/Cas系列基因組編輯技術的應用領域還存在其靶向核酸酶分子量大、存在脫靶現象和受PAM識別序列的限制等問題,研究者一直致力于尋找解決方法,通過改造修飾Cas9蛋白、使用Cas9直系同源酶等一系列措施,來提高基因編輯效率,降低其脫靶率,減弱PAM位點限制來進一步拓展應用范圍。本研究構建的MCP?FokI和Csy4?FokI融合蛋白可能比Cas9蛋白更具有優(yōu)勢。主要表現在:首先Cas系列基因相對較大,大于4 000 bp[19],而MCP?FokI和Csy4?FokI大小為1 074和1 248 bp,從堿基片段大小來講,兩個融合蛋白小于Cas蛋白,更利于編輯載體的構建。此外,Cas對靶位點的切割依賴于PAM位點,盡管經過人工改造的Cas9可以識別更多的PAM位點[24],但是Cas9蛋白發(fā)揮切割作用仍然存在PAM位點的限制,導致并不是所有基因任意區(qū)段都能被編輯。使用上述融合蛋白靶向目標基因并沒有PAM位點的限制,從而提高了目標位置選擇的靈活性;最后,也就是說CRISPR/Cas9并沒有實質性地解決脫靶問題[25]。若將兩個融合蛋白應用于基因編輯體系必須采用雙切口酶策略,也就是用2個不同的U6啟動子分別驅動guide?SL或sgRNA的轉錄,Fok I分別切割目標DNA片段的正負鏈,產生DNA雙鏈斷裂。只有2個guide?SL或sgRNA同時靶向到同一個基因上,才能發(fā)揮基因編輯的功能,從而提高了基因編輯的特異性進而解決CRISPR/Cas9的脫靶問題。本實驗構建的新型迷你基因組編輯系統(tǒng)雖然具有蛋白小、不受PAM位點限制和脫靶效率低等優(yōu)點,但仍存在一些限制。該系統(tǒng)是一種雙靶位點編輯系統(tǒng),需要在目標基因的兩側設計靶向位點,因此,在設計和載體構建上具有一定的復雜性。此外,迷你基因組編輯系統(tǒng)也會受到靶向蛋白自身性質的影響,例如,與RNA結合能力的強弱、RNA切割活性和靶向蛋白的大小等,這些因素都可能影響該系統(tǒng)的編輯能力。
根據報道[26-27]在CRISPR/dCas9?FokI編輯體系內,FokI在dCas9蛋白的N端,通過設計有效的靶序列才可以對目標基因進行編輯。出現這樣結果的原因可能是dCas9和FokI這兩個蛋白在空間構象上有嚴格的排列順序。為此,本研究構建了4種不同的融合蛋白:FokI核酸酶域與MCP(MCP?FokI)的羧基末端融合和與其氨基末端融合(FokI?MCP),FokI核酸酶域與Csy4(Csy4?FokI)的羧基末端融合和與其氨基末端融合(FokI?Csy4),融合蛋白在結構上與ZFNs和TALENs相似(圖7)。這4種融合蛋白本研究使用帶有SGGSSGGSSGSETPGTSESAT?PESSGGSSGGS序列的32個氨基酸連接子來連接這兩個結構域,同時對MCP蛋白進行了密碼子優(yōu)化,使其能在植物細胞中表達[20]。

圖7 MCP-FokI(FokI-MCP)和Csy4-FokI(FokI-Csy4)融合蛋白示意圖Fig. 7 Schematic diagram of MCP-FokI(FokI-MCP)and Csy4-FokI(FokI-Csy4)fusion protein
病毒誘導的基因編輯(virus?induced genome editing, VIGE)技術能夠改造植物病毒使其能在不同植物中表達外源蛋白和特定長度的RNA,可以通過簡單的轉化方法借助病毒在植物體內系統(tǒng)擴散能力將基因組編輯元件投送到植物細胞內[28],提高了基因編輯的檢出率。棉花葉皺縮病毒(cotton leaf crumple virus, CLCrV)是一種靠煙粉虱傳播的雙生病毒,由CLCrV?A和CLCrV?B兩個環(huán)狀單鏈DNA成分組分構成[29]。Gu等[30]改良了基于CLCrV病毒誘導的基因沉默(virus?induced gene silencing, VIGS)載體,在棉花中實現了高效持久的基因沉默。Lei等[31]基于CLCrV介導的VIGE體系,在擬南芥中利用FT?sgRNA策略成功獲得了可遺傳的基因編輯后代。趙燚等[32]采用CLCrV介導VIGE體系,成功編輯了棉花內源基因。利用病毒載體進行基因編輯已在多種植物中得到成功應用,但是受病毒基因組承載外源DNA片段的限制阻礙了其承載大片段DNA進行基因編輯的應用。然而也有研究報道少數病毒可承載完整的基因編輯載體進行基因編輯,如Ma等[33]采用苦苣菜黃網病毒(sonchus yellow net virus, SYNV)將整個CRISPR/Cas9系統(tǒng)投遞到煙草體內的,并實現了基因編輯;Ariga等[34]利用馬鈴薯病毒X(potato virus X, PVX)病毒載體表達Cas9和sgRNA成功在本氏煙草中實現了高效編輯;Liu等[35]基于一種廣寄主范圍的番茄斑萎病毒(tomato spotted wilt virus, TSWV)建立了高效的遺傳操作系統(tǒng)。研究者通過建立瞬時遞送系統(tǒng)向多種作物不同品種穩(wěn)定遞送一系列大的CRISPR/Cas核酸酶分子,展現出SYNV、PVX和TSWV具有較好的承載力。CLCrV能否像以上病毒一樣能夠承載完整的編輯系統(tǒng),進而快速驗證Csy4?FokI基因組編輯系統(tǒng)的編輯能力。為此,本研究嘗試將完整的編輯體系構建到CLCrV病毒載體上,利用CLCrV病毒載體投送到擬南芥葉片中快速驗證Csy4?FokI基因編輯體系的可行性。
本研究構建的MCP?FokI基因組編輯系統(tǒng)通過穩(wěn)定遺傳轉化擬南芥的方式并未檢測到基因編輯的發(fā)生,推測其原因可能有:設計的sgRNA不同中間間隔區(qū)的設計是否有效、蛋白融合方式、與RNA結合能力的強弱。其中需要解決的是通過蛋白質與RNA互作檢測手段來明確MCP蛋白與MS2?guide?SL的結合能力。如果結合能力較弱,可以通過串聯(lián)更多的MS2?SL以提高MCP蛋白與MS2?guide?SL的結合能力,進而提高編輯效率。
對于Csy4?FokI編輯系統(tǒng),利用CLCrV病毒載體投送到擬南芥葉片中來快速驗證Csy4?FokI基因編輯體系。經過Hi?TOM高通量檢測結果的分析發(fā)現,CLCrV介導的Csy4?FokI編輯系統(tǒng)的編輯效率較低。推測造成這一問題的原因可能包括以下幾點:首先在該系統(tǒng)中,構建的融合蛋白的基因片段相對較大(3 316 bp)。然而,CLCrV病毒載體在承載了3 000 bp外源基因片段后,可能會導致其復制能力和擴散侵染能力大幅下降,不能侵入更多的組織細胞。由于病毒載體需要有效感染目標細胞才能實現基因編輯,因此低細胞感染率可能是導致編輯效率低的主要原因之一。其次,Csy4?FokI編輯系統(tǒng)的靶向蛋白除具有和sgRNA高識別與結合特異性外,還存在RNA切割活性。這可能導致sgRNA在與Csy4靶向蛋白結合后被切割,從而降低了結合的能力。此外,使用Csy4蛋白進行植物基因組編輯存在一定的風險,因為研究發(fā)現在植物中引入Csy4蛋白加工sgRNA或pegRNA時可能產生毒性[36-37]。這可能是因為Csy4蛋白影響了植物mRNA的功能,對植物細胞產生了毒害作用,進而影響了編輯效率。后續(xù)研究將對Csy4蛋白進行改造,使其不具有RNA切割活性,而只是作為RNA結合蛋白來進一步驗證該迷你基因組編輯系統(tǒng)的編輯效率。未來的研究也可以嘗試通過驗證更多基因和設計不同的中間間隔區(qū),并采用浸花法等穩(wěn)定轉化的方式來驗證其編輯能力。
綜上所述,本研究采用病毒誘導的基因編輯(VIGE)技術注射擬南芥葉片,初步探索了Csy4?FokI與MCP?FokI小型基因組編輯體系的編輯能力,為今后優(yōu)化并利用該技術體系進行植物分子育種奠定前期基礎。
4 結論
初步建立了Csy4?FokI的迷你的、無PAM位點限制的新型基因組編輯系統(tǒng)。CLCrV介導的Csy4?FokI迷你基因組編輯系統(tǒng)能夠實現對目標基因進行靶向編輯,但是檢測到的突變類型只有堿基置換,且編輯效率很低。

