秈型血緣滲入對北方粳稻產量和品質的影響
徐海,李秀坤,2,蘆佳浩,姜愷,馬玥,徐正進,徐銓
秈型血緣滲入對北方粳稻產量和品質的影響
徐海1,李秀坤1,2,蘆佳浩1,姜愷1,馬玥1,徐正進1,徐銓1
1沈陽農業大學水稻研究所,沈陽 110866;2河北農業大學農學院/華北作物改良與調控國家重點實驗室/河北省種質資源實驗室,河北保定 071000
【目的】分析秈型血緣滲入對粳稻品種產量和品質的影響,為優化秈型血緣利用的北方粳稻育種方案提供理論基礎和基因組資源。【方法】以秈粳交重組自交系(RIL)及從黑龍江省、遼寧省、山東省和江蘇省收集的74份不同年代粳稻主栽品種為試材,結合基于Illumina HiSeq2500平臺的全基因組高通量測序和多地表型調查,分析秈型血緣對北方粳稻產量和品質的影響,并使用CRISPR/Cas9基因編輯技術對秈型血緣滲入所引入的不利基因進行基因敲除。【結果】分析RIL發現秈型血緣與穗長、粒長呈顯著正相關,與碾磨品質呈負相關,其中,與整精米率達到顯著水平。秈型血緣在江蘇省與直鏈淀粉含量呈顯著負相關,與粗蛋白含量呈顯著正相關。秈型頻率與粒形的相關性隨著緯度的增加而增強,而與穗長和整精米率的相關性隨著緯度的增加而減弱。通過調查粳稻主要稻區不同年代主栽品種,發現秈型血緣滲入位置和比例并不是均勻分布在12條染色體上,在第1、10、11和12染色體上,秈型血緣分布較多。江蘇省和遼寧省主栽品種的秈型血緣顯著高于黑龍江省和山東省,而且2000年后育成的品種秈型血緣顯著高于2000年之前的品種。各粳稻區不同年代主栽品種秈型血緣滲入顯著增加了每穗粒數,秈型血緣滲入區域中包含多個抗性和育性相關基因。在鹽豐47的第5染色體上有一段秈型血緣滲入片段,包含秈型粒型調控基因和秈型堊白調控基因,增加了鹽豐47的千粒重,但影響了其堊白性狀。應用CRISPR/Cas9技術敲除鹽豐47的,純合基因編輯植株的粒形與鹽豐47相似,其堊白性狀得到了顯著改善。【結論】秈型血緣滲入主要通過增加每穗粒數增加粳稻產量潛力,但對碾磨品質有負面影響。通過全基因組高通量測序挖掘品種不良等位基因,結合CRISPR/Cas9基因編輯打破秈粳雜交育種中的遺傳累贅是一種高效的育種輔助手段,可以針對目標性狀進行快速準確的改良。
水稻;粳稻育種;秈型血緣滲入;產量;品質;分子設計育種
0 引言
【研究意義】秈粳稻雜交育種是中國特別是北方粳型超級稻育種的總體技術路線,分析秈型血緣滲入在粳稻育種中的作用,可以最大限度地避開亞種間雜交產生的負面影響,將為進一步聚合亞種間優勢性狀、擴寬粳型栽培稻的遺傳多樣性、在更高水平上平衡粳稻產量和品質及突破粳稻育種瓶頸提供理論基礎和種質資源。【前人研究進展】水稻是世界重要糧食作物,為全球約一半人口提供主食,其中90%以上集中在亞洲。亞洲栽培稻分為秈(Xian,XI)和粳(Geng,GJ)2個亞種,在長期自然選擇和人工選擇過程中,秈粳稻基因組不斷分化,生態適應性等生物學特性產生顯著差異,農藝性狀各有利弊[1-4]。秈稻約占亞洲栽培稻的90%,廣泛分布在印度、中國、東南亞,粳稻主要在中國、日本、韓國等國家種植[5]。我國秈粳并重,其中2/3為秈稻,主要在緯度和海拔較低地區種植,粳稻主要在緯度和海拔較高地區種植,中部地區秈粳交錯。我國是世界上唯一同時大面積種植秈稻和粳稻的國家,促使相關遺傳基礎研究成為得天獨厚且成果卓著的優勢研究領域[6-8]。早在1959年楊守仁先生率先提出秈粳稻雜交育種新思路[9],在此基礎上,陳溫福院士團隊構建了理想株型與綜合秈粳亞種有利性狀相結合的育種理論體系,成為我國北方粳型超級稻育種的總體技術路線[10]。通過對20世紀80年代以來我國北方稻區推廣的優良品種進行系譜分析,發現其中絕大部分都是由秈粳稻雜交所育成。秈粳雜交所引入的秈型血緣顯著增加了我國粳稻產量潛力,但是這些品種一般不是直接通過秈稻與粳稻品種雜交,而是以秈粳交育成品種(系)或衍生后代雜交育成,由于親本繁多,系譜和育種過程復雜,育種家并不清楚究竟利用了另一亞種的哪些優異基因。總之,隨著秈型血緣的引入,北方粳稻的品質有所下降,但是課題組前期發現一些秈型血緣較高的粳型超級稻品種,不但產量潛力顯著高于傳統日本粳稻品種,外觀品質和加工品質也毫不遜色,只是食味值與傳統日本粳稻品種相比還有待提高[11]【本研究切入點】秈型血緣的引入在提高產量潛力的同時是否對品質性狀有直接負面影響,迄今研究還不能圓滿回答這個問題,相關基因組層面研究滯后是限制秈粳稻雜交育種效率的主要瓶頸。【擬解決的關鍵問題】本研究圍繞秈型血緣在粳稻育種中的作用這一關鍵科學問題,對秈粳交重組自交系和我國不同年代育成粳稻品種進行全基因組測序,明確秈型血緣對我國粳稻重要農藝性狀的影響,為通過秈粳雜交聚合亞種有利基因實現精準育種提供新思路。
1 材料與方法
1.1 試驗材料
以秈型恢復系材料瀘恢99(R99)和粳型超級稻品種沈農265(SN265)雜交后自交10代,隨機選取151個株系構建秈粳交重組自交系(recombinant inbred lines,RIL)。于2021年將試驗材料分別種植于廣東省(GD)中國農業科學院深圳農業基因組研究所試驗田(22°N,114°E)、江蘇省(JS)淮安市盱眙縣古桑鄉原種場試驗田(32°N,120°E)、四川省(SC)農業科學院水稻高粱研究所試驗基地(32°N,104°E)和遼寧省(LN)沈陽農業大學水稻研究所試驗田(41°N,123°E)。試驗選擇完全隨機區組設計,田間種植3次生物學重復。每個株系種植4行,每行10株,行株距為30.0 cm×15.0 cm,單苗移植,4個生態環境下均在植株抽穗后45 d進行收獲。
試驗進行大范圍的粳稻品種收集,其中,包括黑龍江省(HLJ)、遼寧省(LN)、山東省(SD)、江蘇省(JS)不同年代的74份粳稻主栽品種,品種按育成年代可分為1980年之前育成、1980—1990年育成、1990—2000年育成和2000年后育成4個時間段。于2021年將74份主栽品種分別種植于江蘇省(JS)淮安市盱眙縣古桑鄉原種場試驗田(32°N,120°E)、山東省(SD)青島農業大學水稻試驗田(37°N,119°E)、遼寧省(LN)沈陽農業大學水稻研究所試驗田(41°N,123°E)、黑龍江省(HLJ)農業科學院耕作栽培研究所(45°N,126°E),4個試驗地點基本包含了主要粳稻種植區的緯度跨度,種植方式與RIL相同。
1.2 產量和品質性狀調查
植株抽穗后45 d,在各小區中部隨機選取長勢中等的5株調查有效穗數和株高,取長勢均勻的10穗調查每穗粒數、結實率和千粒重。稻谷在收獲曬干后存放3個月,待理化特性穩定后進行品質測定。檢測指標有糙米率、精米率、整精米率、堊白度、堊白粒率、蛋白質含量、膠稠度、堿消值、直鏈淀粉含量和蛋白含量,具體測定方法參照Li等[12]。
使用Microsoft Excel 2016整理試驗數據,采用SPSS 20.0進行方差分析和顯著性分析,LSD(<0.05)方法用于檢驗品種間差異的顯著性,并用Graphpad 8.0進行作圖。
1.3 高通量測序
在人工氣候箱中播種SN265、R99和151個RIL,以及74份主栽品種。播種四周后取嫩葉,采用CTAB法提取基因組DNA,用于構建文庫,隨后在Illumina HiSeq2500平臺進行全基因組高通量測序。經過質量控制和過濾后,R99和SN265分別得到13.04和13.38 Gb的Clean data;151個子代共得到414.37 Gb的Clean data,平均每個材料2.74 Gb的Clean data。74份主栽品種共獲得約1 638.72 Gb的Clean data,Q30平均達到92.51%。SN265和R99的測序深度分別為30×和32×;子代的平均測序深度為6.25×。利用SOAP2軟件將序列與參考基因組日本晴(Nipponbare,http:// rapdb.dna.affrc.go.jp/download/irgsp1.html/)進行比對,利用GATK軟件進行SNP檢測,共得到1 456 445個SNP。74份主栽品種的平均測序深度為53.34×,篩選后得到11 952 151個SNP。
1.4 秈型血緣計算
秈粳特異性SNP數據來自517份水稻種質資源的測序結果[2],將517份水稻種質資源中粳型種質資源基因型一致,且秈型種質資源為另一種基因型的SNP定義為秈粳特異SNP,共得到100 528個秈粳特異SNP,其中,第1染色體有15 961個;第2染色體有23 282個;第3染色體有12 450個;第4染色體有3 637個;第5染色體有5 333個;第6染色體有4 741個;第7染色體有4 107個;第8染色體有7 347個;第9染色體有8 443個;第10染色體有7 701個;第11染色體有3 944個;第12染色體有3 582個。RIL和主栽品種中秈型SNP數值與全部秈粳特異SNP的比值定義為秈型血緣滲入比例。
1.5 CRISPR/Cas9基因編輯
以鹽豐47為遺傳背景材料進行CRISPR基因編輯。使用華南農業大學亞熱帶農業生物資源保護與利用國家重點實驗室劉耀光院士團隊開發的基因編輯工具包CRISPR-GE(http://skl.scau.edu.cn/)進行靶位點的設計,應用BLAST比對日本晴參考基因組確認靶點的特異性(https://rapdb.dna.affrc.go.jp/tools/ blast)。基因編輯靶點序列和引物合成,以及測序服務均由華大基因完成。參照Li等[12]方法進行基因編輯載體構建及基因編輯植株的遺傳轉化和篩選。
2 結果
2.1 RIL群體的產量和品質性狀
將SN265和R99雜交構建的RIL群體分別種植于遼寧省(LN)、四川省(SC)、江蘇省(JS)和廣東省(GD),并對主要產量和品質性狀展開調查。結果顯示,JS和LN的穗數顯著高于SC和GD(圖1-a),SC和JS的每穗粒數顯著高于LN和GD(圖1-b),千粒重在4個地區沒有顯著差異(圖1-c),LN和SC的結實率顯著高于JS和GD(圖1-d)。SC和JS的穗長顯著長于LN和GD(圖1-e),著粒密度為SC>JS>LN>GD,且地區間差異均達到顯著水平(圖1-f),SC和JS的株高顯著高于LN和GD(圖1-g),抽穗期在4個地區間差異顯著,依次為LN>SC>JS>GD(圖1-h)。RIL在SC展現出較好的碾磨品質,其糙米率、精米率和整精米率都顯著高于其他3個地區(圖1-i—k)。RIL的外觀品質在4個地區展現出較大差異,LN堊白粒率顯著高于其他3個地區(圖1-i),LN和SC的堊白度顯著高于JS(圖1-m)。粒形一度被認為主要受遺傳因素調控,受環境因素影響較小,發現RIL的粒形在不同地區也體現出顯著性差異,JS和GD的籽粒顯著長于LN和SC(圖1-n),其中,LN和SC的粒寬顯著大于JS和GD(圖1-o),而GD的粒長顯著長于其他3個地區(圖1-p)。進一步調查RIL在各地區的食味相關品質,發現LN和GD的堿消值顯著高于SC和JS(圖1-q)。LN、SC和GD之間膠稠度沒有顯著性差異,均顯著高于JS(圖1-r),LN的直鏈淀粉含量顯著高于SC和JS,SC和JS顯著高于GD(圖1-s)。粗蛋白含量展現出隨著維度降低而增加的趨勢,且4個地區間差異均達到顯著水平(圖1-t)。
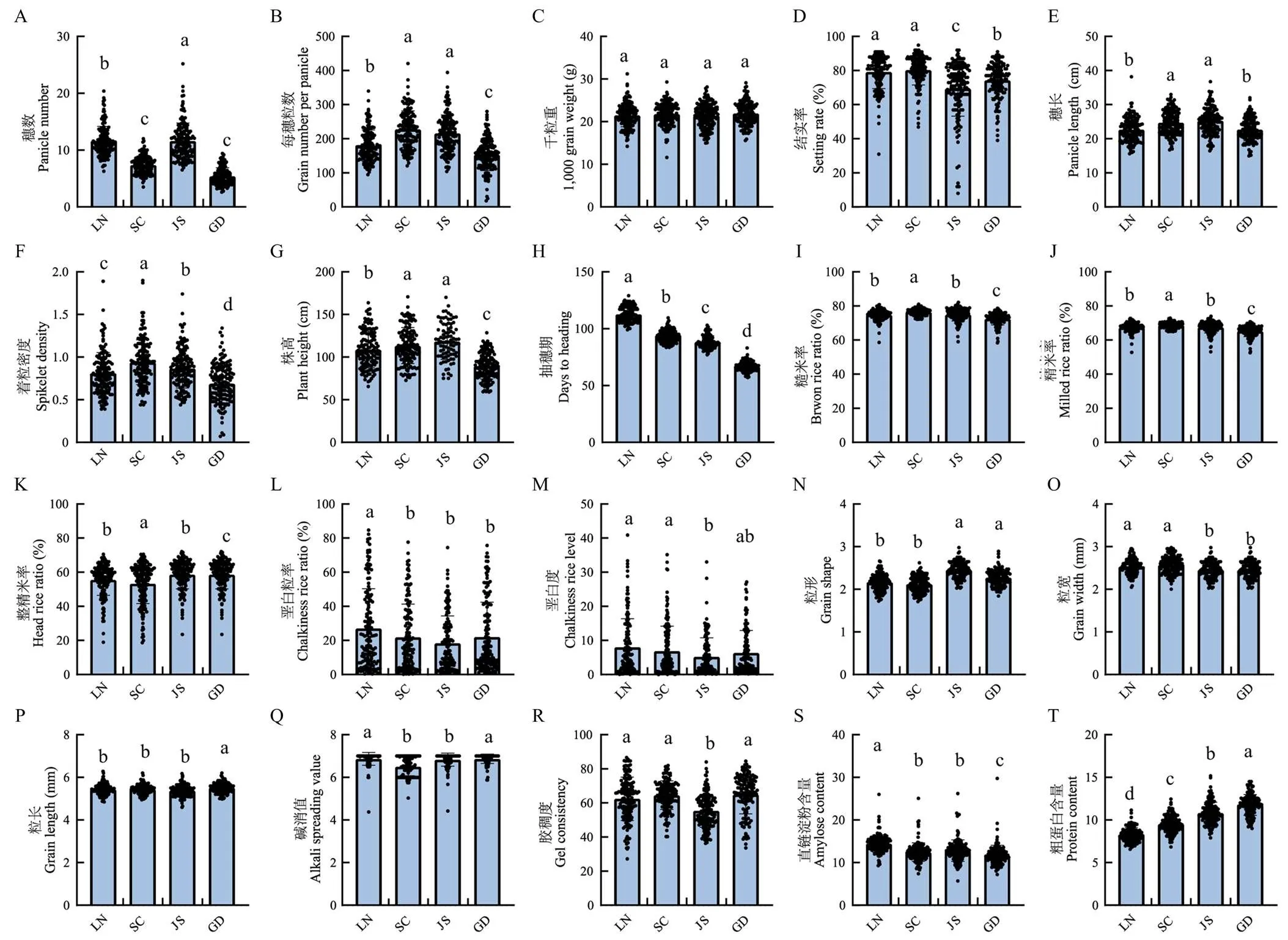
LN:遼寧省;SC:四川省;JS:江蘇省;GD:廣東省。不同小寫字母表示在5%水平上差異顯著。下同
2.2 RIL群體中秈型血緣對產量和品質的影響
利用高通量測序數據和秈粳特異SNP確定了每個RIL自交系的秈型血緣比例,結果顯示,RIL群體的秈型血緣比例呈正態分布(圖2-a),說明RIL的構建理想,滿足后續試驗分析需求。分別在4個地區對秈型血緣與重要農藝性狀的相關性進行分析。結果顯示,秈型血緣在4個地區都與穗長呈正相關,在LN、JS和GD達到顯著水平,秈型血緣與每穗粒數為正相關,其中,在SC、LN和JS達到顯著水平。秈型血緣與結實率和著粒密度呈正相關,與千粒重和穗數呈負相關,4個地區均未達到顯著水平。秈型血緣與理論產量呈正相關,在GD達到顯著水平。秈型血緣與株高呈負相關,其中,在SC和LN達到顯著水平。秈型血緣與糙米率、精米率和整精米率呈負相關,其中,與糙米率和精米率在GD和JS達到顯著水平,與整精米率在4個地區均為顯著負相關。秈型血緣與堊白粒率和堊白度為正相關,在4個地區均未達到顯著水平。秈型血緣與粒長和籽粒長寬比在4個地區均顯著正相關,與粒寬顯著負相關。秈型血緣與堿消值和膠稠度呈正相關,在4個地區均未達到顯著水平。秈型血緣與直鏈淀粉含量呈負相關,在SC和JS達到顯著水平。秈型血緣與粗蛋白含量呈正相關,在JS達到顯著水平(圖2-b)。
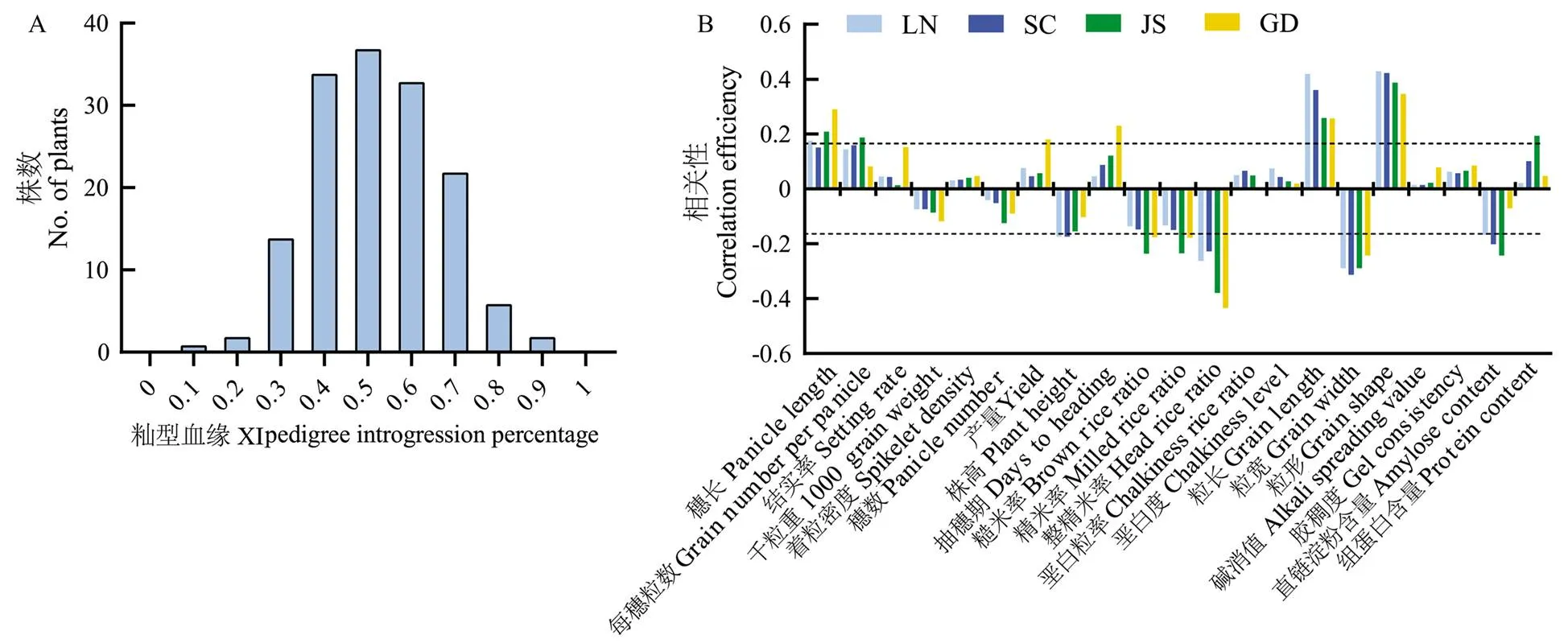
a:RIL群體的秈型血緣比例分布;b:RIL群體中秈型血緣和產量與品質性狀的相關性分析。虛線范圍外表示在0.05水平差異顯著。下同
2.3 主栽品種的產量相關性狀
為了分析秈型血緣對粳稻主栽品種產量性狀的影響,從黑龍江省(HLJ)、遼寧省(LN)、山東省(SD)和江蘇省(JS)4個粳稻稻區收集了74份主栽品種,收集品種的育成年代可分為1980年之前、1980—1990年、1990—2000年和2000年之后4組。將74份品種分別種植于HLJ、LN、SD和JS,并在這4個地區對74份品種的產量相關性狀進行調查,結果顯示,74份品種在JS穗數顯著高于HLJ、LN和SD,HLJ穗數顯著高于LN,HLJ和SD之間以及LN和SD之間均沒有顯著性差異(圖3-a)。4個地區的每穗粒數和結實率相近,地區間沒有顯著性差異(圖3-b—c)。JS和LN的千粒重顯著高于HLJ,HLJ顯著高于SD(圖3-d)。74份品種在LN株高顯著高于HLJ、SD和JS,SD株高顯著高于HLJ,HLJ和JS及SD和JS之間株高沒有顯著性差異(圖3-e)。進一步分析不同育成年代株高和產量構成因素的特點和規律發現穗數和每穗粒數這一對負相關的性狀體現出年代變化規律,其中,2000年以后育成品種穗數顯著少于之前3個時期的育成品種。相反,2000年后育成品種的每穗粒數顯著多于之前3個時期的育成品種(圖3-f—g)。結實率也表現出改善的趨勢,其中,2000年后育成品種結實率顯著高于1980年前育成品種(圖3-h)。千粒重和株高在不同年代間沒有顯著性差異(圖3-i—j)。
2.4 秈型血緣對主栽品種產量性狀的影響
為了進一步分析秈型血緣對重要農藝性狀的影響,繪制了74份品種全基因組秈型血緣滲入比例和位置示意圖(圖4-a—b)。不同品種的秈型血緣滲入比例和位置展現出豐富的多態性,且秈型血緣滲入位置和比例并不是均勻分布在12條染色體上,在第1、10、11和12染色體上,秈型血緣分布較多(圖4-a)。統計不同育成年代品種的秈型血緣滲入比例,發現2000年之前育成的品種秈型血緣較低,普遍低于2.0%,其中1980年以前育成品種的秈型血緣滲入比例小于0.5%。2000年后育成品種的秈型血緣滲入比例顯著增加,超過5.0%(圖4-c)。進一步分析發現秈型血緣滲入比例在不同地區間存在差異,其中,HLJ和SD秈型血緣滲入比例較低,SD品種秈型血緣滲入比例小于1.0%,HLJ品種秈型血緣滲入比例在1.1%左右。LN和JS品種的秈型血緣滲入比例較高,其中,遼寧省品種達到3.0%,JS品種的秈型血緣滲入比例超過4.5%(圖4-d)。結合高通量測序獲得的秈型血緣滲入比例和4個地區種植獲得的產量構成因素數據,分析秈型血緣對主栽品種產量的影響。結果顯示,秈型血緣滲入比例與株高、千粒重和結實率呈正相關,與穗數均呈負相關,在4個地區均未達到顯著水平。秈型血緣滲入比例與每穗粒數在4個地區均呈顯著正相關(圖4-e)。
綜上所述,秈型血緣滲入主要增加了粳稻品種的每穗粒數。發現很多品種的特定染色體區域有大量秈型血緣滲入,其中,第1染色體短臂端是每穗粒數基因所在區域,在秈粳之間存在差異,秈型第1個外顯子處6 bp的缺失和2個SNP(C/G和G/A),以及第4個外顯子處的1個SNP(G/T),導致其蛋白產物與粳稻品種產生差異,引起花序分裂組織中細胞分裂素的積累,進而增加每穗粒數[13]。通過分析74份粳稻主栽品種中的基因型,發現絕大部分粳稻主栽品種的基因型均與日本晴和SN265相同,暗示秈型等位基因還沒有廣泛應用到粳稻育種中。但是黑龍江省大面積推廣的粳稻品種龍粳31的位點基因型與R99相同,均為典型秈型等位基因(圖4-f)。進一步比對目前已經公布的基于長讀長測序的155份水稻基因組[14-16],發現秈型等位基因廣泛分布于熱帶粳稻種質資源當中,溫帶粳稻中僅有來自韓國的MAEKJO、來自馬來西亞的MR19和中國的安農晚粳B與龍粳31攜帶秈型基因型。
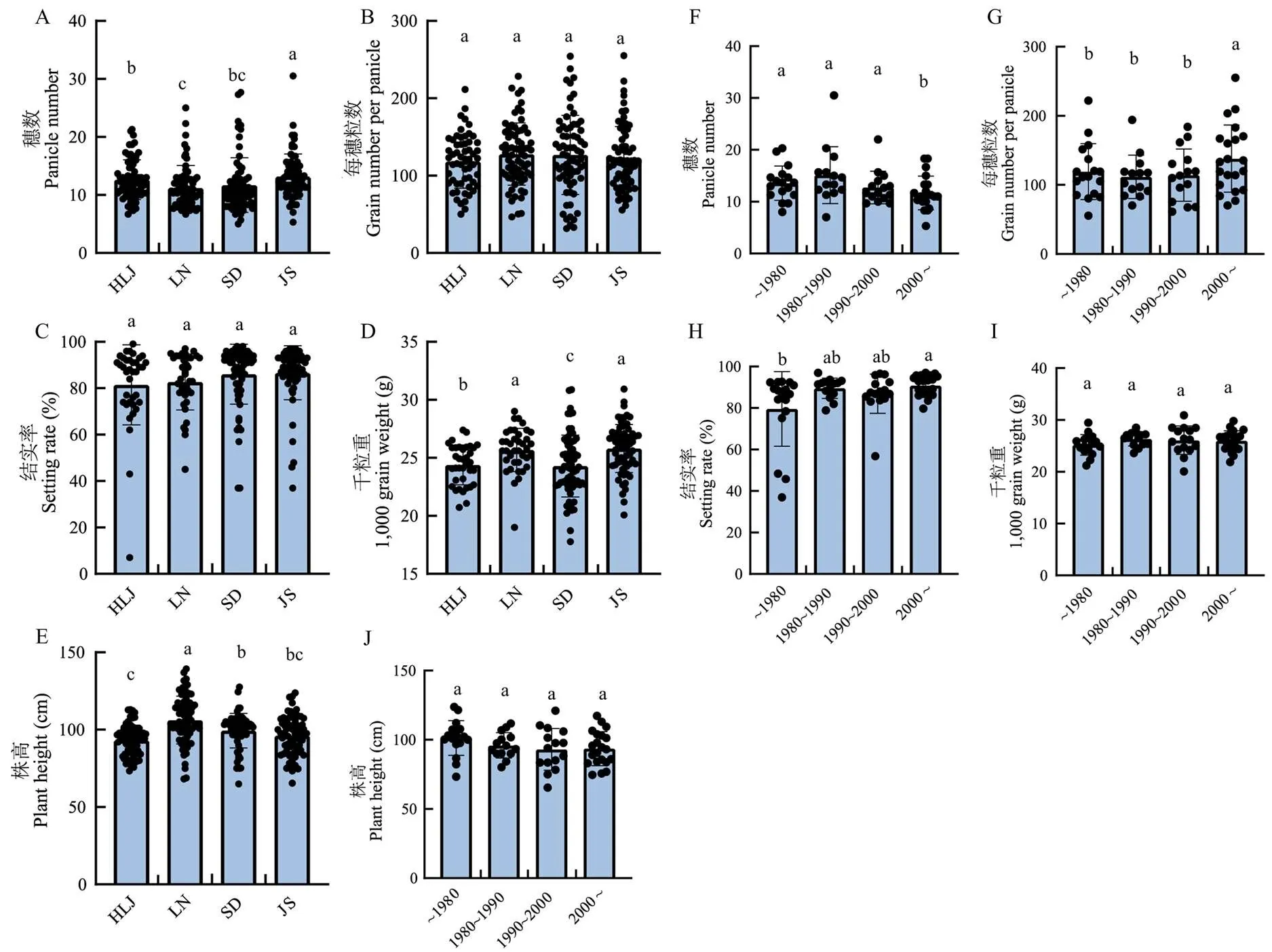
a—e:四地區主栽品種;f—j:不同時期育成品種。HLJ:黑龍江省;SD:山東省。下同
為了進一步揭示秈型血緣滲入所引入的關鍵農藝性狀調控基因,篩選了在74份粳稻品種中超過20%的品種為秈型的SNP,并分析與這些高頻秈型SNP位置重合的已知功能基因,共發現11個已知功能基因(圖4-g),其中4個為抗逆性基因[17]、[18]、[19]和[20],2個抗病性基因[21]和[22],3個育性基因[23]、[24]和[25],1個水稻株型調控基因[26]和1個水稻抽穗期基因[27],暗示粳稻品種中的秈型血緣滲入主要引入了抗性和育性相關基因。

a:主栽品種中的秈型血緣滲入位置;b:主栽品種中秈型血緣滲入比例;c:不同年代育成品種中的秈型血緣滲入;d:不同地區主栽品種中的秈型血緣;e:秈型血緣與產量性狀的相關性分析;f:沈農265(SN265)、R99、日本晴(Nipponbare)和龍粳31(LG31)Gn1a位點的基因型對比;g:秈型血緣滲入所引入的重要功能基因
2.5 基因編輯打破秈粳雜交育種中的遺傳累贅
調控重要農藝性狀的功能位點在基因組上的分布并不均勻,許多功能位點集中分布在2 cM區間內,這種現象可能導致引入優異等位基因的育種過程中,因為遺傳累贅(genetic drag)也引入了不良性狀調控基因[6]。分析主栽品種中秈型血緣滲入區域內的重要農藝性狀調控基因。以鹽豐47為例,鹽豐47為遼寧省、山東省等地區的主栽品種,其產量潛力高,千粒重達到26.2 g,高于大部分粳稻品種,但是堊白性狀有待提高。發現鹽豐47的第5染色體短臂端有一段秈型血緣滲入,該區間內包含了重要粒形調控基因和堊白調控基因(圖5-a)。編碼一個絲氨酸羧肽酶,其優勢等位基因(ZS97型)顯著增加水稻籽粒大小[28]。在上游的為液泡膜質子轉運焦磷酸酶,具有無機焦磷酸水解活性和質子轉運活性,影響水稻籽粒堊白的形成和精米率等品質性狀,其ZS97型等位基因顯著增加籽粒堊白度和堊白粒率[29]。通過序列比對,發現鹽豐47的和均與秈稻品種ZS97相同(圖5-b)。
綜上所述,推測鹽豐47在育種過程中為了追求高產而引入了秈型等位基因以期增加千粒重,與相鄰的秈型也隨之滲入到鹽豐47基因組,導致堊白性狀惡化。為了保留鹽豐47的千粒重優勢,改良其堊白性狀,對鹽豐47的位點進行定點基因敲除。在T2代篩選到2個無Cas9的純合突變系CR-1和CR-2,其中CR-1缺失了一個堿基(A),CR-2缺失2個堿基(TA),二者都造成移碼突變,提前產生終止子(圖5-c)。突變體CR-1和CR-2與鹽豐47在粒形上無顯著差異(圖5-d),其堊白性狀顯著改善(圖5-e—f)。
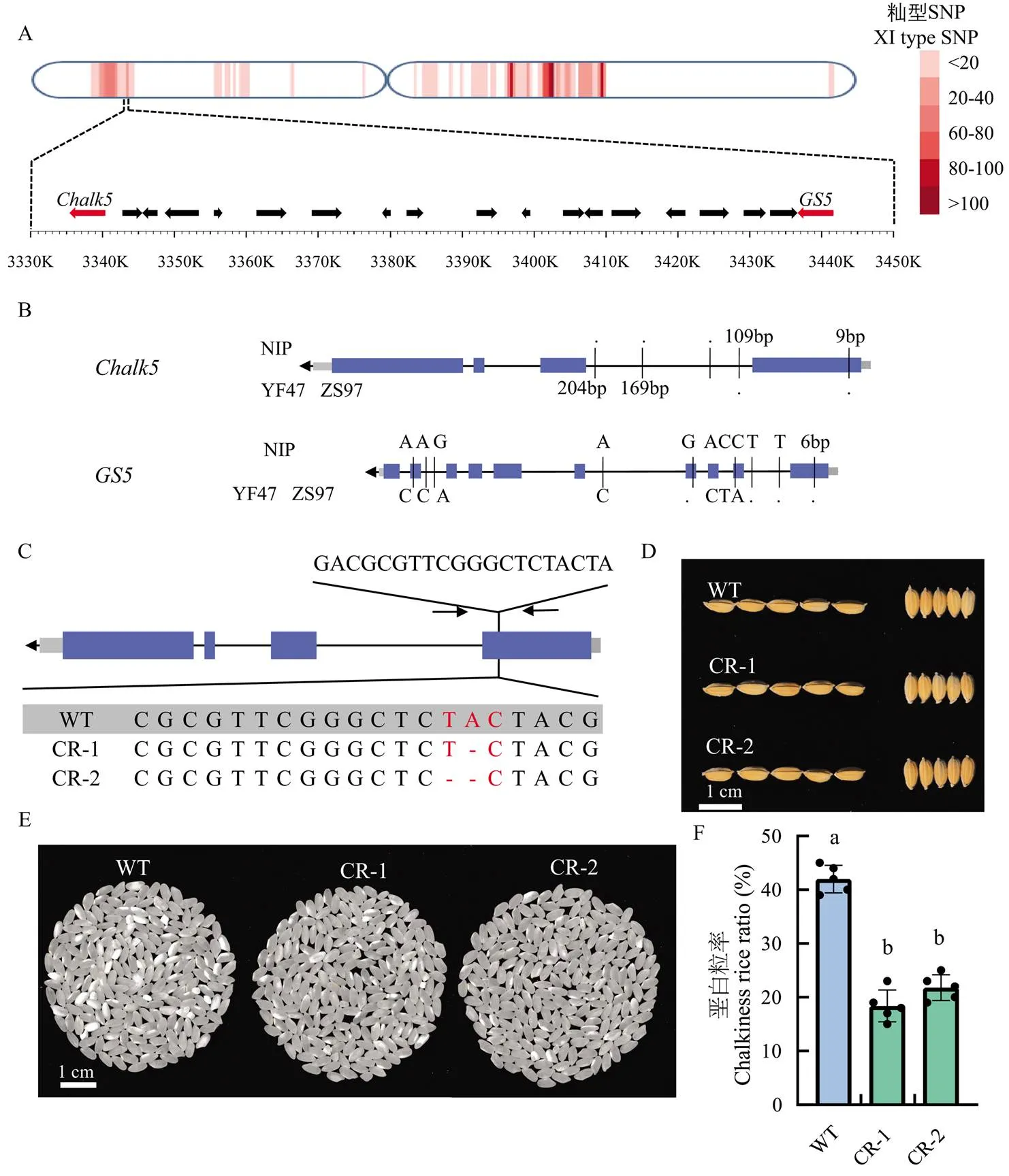
a:Chalk5和GS5的位置;b:鹽豐47中Chalk5和GS5的基因型;c:CRISPR/Cas9基因編輯的靶點設計和突變體序列比對;d:野生型和突變體的粒形比較;e:野生型和突變體的堊白性狀比較;f:野生型和突變體的堊白粒率
3 討論
3.1 秈粳雜交育種顯著提高北方粳稻產量潛力
目前,全球粳稻種植面積超過1 478萬hm2,主要分布在中國東北、日本和韓國。早在1959年,楊守仁先生率先提出秈粳稻雜交育種新思路,在此基礎上,陳溫福院士團隊構建了理想株型與秈粳優勢互補相結合的理論體系,即“利用秈粳稻雜交創造新株型和強優勢,通過復交優化性狀組配聚合有利基因,選育理想株型與秈粳優勢互補相結合的超級稻”,成為中國特別是北方粳型超級稻育種的總體技術路線,在此路線的指導下東北粳稻產量水平顯著提高,為中國乃至世界糧食安全作出了巨大貢獻。截至2014年,東北三省育成超級稻品種22個,覆蓋率達到60%以上,促進東北水稻單產由21世紀初平均的6.60 t·hm-2(2000—2004年)提高到近年的7.56 t·hm-2(2010— 2014年),增產幅度達到12.62%,顯著高于同期全國平均增產水平(6.20 t·hm-2、6.71 t·hm-2、6.42%)[10]。本研究發現2000年后育成品種秈型血緣顯著增加,與我國東北水稻21世紀初開始單產增幅顯著提高相吻合,進一步驗證秈粳雜交引入秈型血緣可以增加我國粳稻產量潛力,因此,從基因組層面揭示秈型血緣在東北水稻育種中的作用是未來在更高水平上突破粳稻育種瓶頸的關鍵之一。
3.2 全基因組高通量測序加速粳稻基因組相關研究
高通量測序技術的出現為解決粳稻基因組研究瓶頸提供了有效方法,應用高通量測序技術獲得的高密度標記,成功追蹤了亞洲栽培稻的馴化起源和水稻育種過程中的基因組印記[30-31]。幾項大規模水稻群體的深度測序和表型觀測為水稻研究和育種提供了寶貴的基因組信息和種質資源[3, 32]。3 000份水稻(3K-RGP)項目廣泛收集和測序了世界主要水稻產區的種質資源,成為水稻群體遺傳研究的一個里程碑[5]。對我國不同時期代表性的816份和546份粳稻品種進行二代重測序發現,20世紀80年代以來粳稻品種的秈型血緣持續增加,生育期、低溫結實率、稻瘟病抗性和粒形上表現出顯著的年代差異,使粳稻的重要農藝性狀發生了顯著變化[33-34]。雖然基于高通量測序技術的水稻基因組研究已經廣泛開展,但是對于秈型血緣在我國粳稻育種中的作用還有待探討。本研究通過對秈粳交重組自交系和我國不同年代粳稻主栽品種的分析發現,秈型血緣的引入主要增加了粒長和每穗粒數,而且2000年后推廣的品種秈型血緣顯著高于2000年之前。這與基于2004—2018年全國水稻區試試驗的結果相吻合,我國北方粳稻2004—2018年的增產主要得益于每穗粒數的增加[35]。2000年后育成品種秈型血緣增加顯著提高了北方粳稻的每穗粒數,進而提升了北方粳稻的產量潛力。本研究還發現,隨著秈型血緣滲入,我國北方粳稻品種中引入了秈型的抗性、育性、株型和抽穗期相關基因。
3.3 基因編輯技術改善秈粳雜交育種的遺傳累贅現象
CRISPR基因編輯技術自2013年首次應用于水稻之后發展迅速[36],應用CRISPR技術成功鑒定了大量水稻基因功能[37-38],并構建了大量CRISPR基因敲除突變體庫[39]。華南農業大學劉耀光院士團隊利用CRISPR/Cas9基因編輯技術進行轉錄水平和轉錄后水平調控,對水稻Wx等位基因的啟動子區域、非編碼區和內含子剪接點進行基因編輯,成功得到了多個新等位基因,新等位基因可產生不同直鏈淀粉含量的秈稻新種質資源[40]。揚州大學劉巧泉團隊通過CRISPR/Cas9技術編輯水稻啟動子上的關鍵順式作用元件,得到了一個溫和調節表達的啟動子靶位點,獲得了6種農藝性狀良好的新等位基因及其新種質資源,有潛力應用于稻米品質改良育種[41]。李家洋院士團隊通過基于平鋪刪除策略的CRISPR/ Cas9技術,創制了在啟動子中含有54 bp缺失的等位基因,該區域可以與結合以抑制穗中的表達,導致次級和小穗分生組織受到抑制,進而同時增加穗數和每穗粒數,從而提高水稻產量,說明平鋪缺失篩選方法可以用來平衡植物育種中的基因多效性[42]。本研究應用CRISPR/Cas9基因編輯技術對鹽豐47的位點進行定點基因敲除,在不改變鹽豐47其他農藝性狀的同時改善其堊白性狀。
4 結論
秈粳稻雜交育種中,秈型血緣滲入主要通過增加每穗粒數增加粳稻產量潛力,同時對粳稻碾磨品質有負面影響。通過全基因組高通量測序挖掘秈粳雜交育種過程中因遺傳累贅所引入的不良等位基因,應用CRISPR/Cas9基因編輯技術對不良等位基因進行快速準確的改良,是一種高效的育種輔助手段。
[1] Garris A J, Tai T H, Coburn J,KRESOVICH S, MCCOUCH S. Genetic structure and diversity inL.. Genetics, 2005, 169(3): 1631-1638.
[2] Huang XH, WEI X H, SANG T, ZHAO Q, FENG Q, ZHAO Y, LI C Y, ZHU C R, LU T T, ZHANG Z W, LI M, FAN D L, GUO Y L, WANG A H, WANG L, DENG L W, LI W J, LU Y Q, WENG Q J, LIU K Y, HUANG T, ZHOU T Y, JING Y F, LI W, LIN Z, BUCKLER E S, QIAN Q, ZHANG Q F, LI J Y, HAN B. Genome-wide association studies of 14 agronomic traits in rice landraces. Nature Genetics, 2010, 42(11): 961-967.
[3] Huang X H, ZHAO Y, WEI X H, LI C Y, WANG A H, ZHAO Q, LI W J, GUO Y L, DENG L W, ZHU C R, FAN D L, LU Y Q, WENG Q J, LIU K Y, ZHOU T Y, JING Y F, SI L Z, DONG G J, HUANG T, LU T T, FENG Q, QIAN Q, LI J Y, HAN B. Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm. Nature Genetics, 2012, 44(1): 32-39.
[4] Yu H, Lin T, Meng X B, DU H L, ZHANG J K, LIU G F, CHEN M J, JING Y H, KOU L Q, LI X X, GAO Q, LIANG Y, LIU X D, FAN Z L, LIANG Y T, CHENG Z K, CHEN M S, TIAN Z X, WANG Y H, CHU C C, LI J Y. A route todomestication of wild allotetraploid rice. Cell, 2021, 184(5): 1156-1170. e14.
[5] Wang W S, MAULEON R, HU Z Q, CHEBOTAROV D, TAI S S, WU Z C, LI M, ZHENG T Q, FUENTES R R, ZHANG F, MANSUETO L, COPETTI D, SANCIANGCO M, PALIS K C, XU J L, SUN C, FU B Y, ZHANG H L, GAO Y M, ZHAO X Q, SHEN F, CUI X, YU H, LI Z C, CHEN M L, DETRAS J, ZHOU Y L, ZHANG X Y, ZHAO Y, KUDRNA D, WANG C C, LI R, JIA B, LU J Y, HE X C, DONG Z T, XU J B, LI Y H, WANG M, SHI J X, LI J, ZHANG D B, LEE S, HU W S, POLIAKOV A, DUBCHAK I, ULAT V J, BORJA F N, MENDOZA J R, ALI J, LI J, GAO Q, NIU Y C, YUE Z, NAREDO M E B, TALAG J, WANG X Q, LI J J, FANG X D, YIN Y, GLASZMANN J C, ZHANG J W, LI J Y, HAMILTON R S, WING R A, RUAN J, ZHANG G Y, WEI C C, ALEXANDROV N, MCNALLY K L, LI Z K, LEUNG H. Genomic variation in 3,010 diverse accessions of Asian cultivated rice. Nature, 2018, 557(7703): 43-49.
[6] Wei X, Qiu J, Yong K C, FAN J J, ZHANG Q, HUA H, LIU J, WANG Q, OLSEN K M, HAN B, HUANG X H. A quantitative genomics map of rice provides genetic insights and guides breeding. Nature Genetics, 2021, 53(2): 243-253.
[7] Liu Y Q, WANG H R, JIANG Z M, WANG W, XU R N, WANG Q H, ZHANG Z H, LI A F, LIANG Y, OU S J, LIU X J, CAO S Y, TONG H N, WANG Y H, ZHOU F, LIAO H, HU B, CHU C C. Genomic basis of geographical adaptation to soil nitrogen in rice. Nature, 2021, 590(7847): 600-605.
[8] Zhou G, Chen Y, Yao W, ZHANG C J, XIE W B, HUA J P, XING Y Z, XIAO J H, ZHANG Q F. Genetic composition of yield heterosis in an elite rice hybrid. Proceedings of the National Academy of Sciencesof the United States of America, 2012, 109(39): 15847-15852.
[9] 楊守仁, 趙紀書. 秈粳稻雜交問題之研究. 農業學報, 1959, 10 (4): 256-268.
Yang S R, Zhao J S. A study on the problem of cross between XI and GJ rice. Acta Agriculture Sinica, 1959, 10(4): 256-268. (in Chinese)
[10] 徐正進, 陳溫福. 中國北方粳型超級稻研究進展. 中國農業科學, 2016, 49(2): 239-250.
Xu Z j, Chen W f. Research progress and related problems on japonica super rice in northern China. Scientia Agricultura Sinica, 2016, 49(2): 239-250. (in Chinese)
[11] 徐銓, 唐亮, 徐凡, 福嶌陽, 黃瑞冬, 陳溫福, 徐正進. 粳稻食味品質改良研究現狀與展望. 作物學報, 2013, 39(6): 961-968.
Xu Q, Tang L, Xu F, fu d y, HUANG R D, CHEN W F, XU Z J. Research advances and prospects of eating and quality improvement inrice (L.) Acta Agronomica Sinica, 2013, 39(6): 961-968. (in Chinese)
[12] Li X k, Wu L, Wang J H, SUN J, XIA X H, GENG X, WANG X H, XU Z J, XU Q. Genome sequencing of rice subspecies and genetic analysis of recombinant lines reveals regional yield- and quality- associated loci. BMC Biology, 2018, 16(1): 102.
[13] Ashikari M, Sakakibara H, Lin S Y, YAMAMOTO T, TAKASHI T, NISHIMURA A, ANGELES E R, QIAN Q, KITANO H, MATSUOKA M. Cytokinin oxidase regulates rice grain production. Science, 2005, 309(5735): 741-745.
[14] Wang Y, Li F c, Zhang F,WU L, XU N, SUN Q, CHEN H, YU Z W, LU J H, JIANG K, WANG X C, WEN S Y, ZHOU Y, ZHAO H, JIANG Q, WANG J H, JIA R Z, SUN J, TANG L, XU H, HU W, XU Z J, CHEN W F, GUO A P, XU Q. Time-ordering japonica/geng genomes analysis indicates the importance of large structural variants in rice breeding. Plant Biotechnology Journal, 2023, 21(1): 202-218.
[15] Zhang F, Xue H z, Dong X r, LI M, ZHENG X M, LI Z K, XU J L, WANG W S, WEI C C. Long-read sequencing of 111 rice genomes reveals significantly larger pan-genomes. Genome Research, 2022, 32(5): 853-863.
[16] Qin P, Lu H W, DU H L, WANG H, CHEN W L, CHEN Z, HE Q, OU S J, ZHANG H Y, LI X Z, LI X X, LI Y, LIAO Y, GAO Q, TU B, YUAN H, MA B T, WANG Y P, QIAN Y W, FAN S J, LI S G. Pan-genome analysis of 33 genetically diverse rice accessions reveals hidden genomic variations. Cell, 2021, 184(13): 3542-3558. e16.
[17] Ghosh A, Pareek A, Sopory S K, SINGLA-PAREEK S L. A glutathione responsive rice glyoxalase II, OsGLYII-2, functions in salinity adaptation by maintaining better photosynthesis efficiency and anti-oxidant pool. The Plant Journal, 2014, 80(1): 93-105.
[18] Du H, Wang N L, CUI F, LI X H, XIAO J H, XIONG L Z. Characterization of the beta-carotene hydroxylase gene DSM2 conferring drought and oxidative stress resistance by increasing xanthophylls and abscisic acid synthesis in rice. Plant Physiology, 2010, 154(3): 1304-1318.
[19] Zou J, Liu AL, CHEN X B, ZHOU X Y, GAO G F, WANG W F, ZHANG X W. Expression analysis of nine rice heat shock protein genes under abiotic stresses and ABA treatment. Journal of Plant Physiology, 2009, 166(8): 851-861.
[20] Zhou L Y, NI E D, YANG J W, ZHOU H, LIANG H, LI J, JIANG D G, WANG Z H, LIU Z L, ZHUANG C X. Rice OsGL1-6 is involved in leaf cuticular wax accumulation and drought resistance. PlosOne, 2013, 8(5): e65139.
[21] Liu Y q, Wu H, Chen H, LIU Y L, HE J, KANG H Y, SUN Z G, PAN G, WANG Q, HU J L, ZHOU F, ZHOU K N, ZHENG X M, REN Y L, CHEN L M, WANG Y H, ZHAO Z G, LIN Q B, WU F Q, ZHANG X, GUO X P, CHENG X N, JIANG L, WU C Y, WANG H Y, WAN J M. A gene cluster encoding lectin receptor kinases confers broad-spectrum and durable insect resistance in rice. Nature Biotechnology, 2015, 33(3): 301-305.
[22] Zhao H J, WANG X Y, JIA Y L, MINKENBERG B, WHEATLEY M, FAN J B, JIA M H, FAMOSO A, EDWARDS J D, WAMISHE Y, VALENT B, WANG G L, YANG Y N. The rice blast resistance gene Ptr encodes an atypical protein required for broad-spectrum disease resistance. Nature Communications, 2018, 9: 2039.
[23] Zhou H, Zhou M, Yang Y Z, LI J, ZHU L Y, JIANG D G, DONG J F, LIU Q J, GU L F, ZHOU L Y, FENG M J, QIN P, HU X C, SONG C L, SHI J F, SONG X W, NI E D, WU X J, DENG Q Y, LIU Z L, CHEN M S, LIU Y G, CAO X F, ZHUANG C X. RNaseZS1processesUbmRNAs and controls thermosensitive genic male sterility in rice. Nature Communications, 2014, 5: 4884.
[24] Zhang P P, ZHANG Y X, SUN L P, SINUMPORN S, YANG Z F, SUN B, XUAN D D, LI Z H, YU P, WU W X, WANG K J, CAO L Y, CHENG S H. The rice AAA-ATPase OsFIGNL1 is essential for male meiosis. Frontiers in Plant Science, 2017, 8: 1639.
[25] Song S y, Chen Y, Liu L, SEE Y H B, MAO C Z, GAN Y B, YU H. OsFTIP7 determines auxin-mediated anther dehiscence in rice. Nature Plants, 2018, 4(7): 495-504.
[26] Zhou L J, Xiao L T, Xue H W. Dynamic cytology and transcriptional regulation of ricejoint development. Plant Physiology, 2017, 174(3): 1728-1746.
[27] Zhang J, Fan X w, Hu Y, ZHOU X C, HE Q, LIANG L W, XING Y Z. Global analysis of CCT family knockout mutants identifies four genes involved in regulating heading date in rice. Journal of Integrative Plant Biology, 2021, 63(5): 913-923.
[28] Li Y b, Fan C c, Xing YZ, JIANG Y H, LUO L J, SUN L, SHAO D, XU C J, LI X H, XIAO J H, HE Y Q, ZHANG Q F. Natural variation in GS5 plays an important role in regulating grain size and yield in rice. Nature Genetics, 2011, 43(12): 1266-1269.
[29] Li Y b, Fan C c, Xing Y Z, YUN P, LUO L J, YAN B, PENG B, XIE W B, WANG G W, LI X H, XIAO J H, XU C G, HE Y Q. Chalk5 encodes a vacuolar H+-translocating pyrophosphatase influencing grain chalkiness in rice. Nature Genetics, 2014, 46(4): 398-404.
[30] Huang X h, Kurata N, Wei X H, WANG Z X, WANG A H, ZHAO Q, ZHAO Y, LIU K Y, LU H Y, LI W J, GUO Y L, LU Y Q, ZHOU C C, FAN D L, WENG Q J, ZHU C R, HUANG T, ZHANG L, WANG Y C, FENG L, FURUUMI H, KUBO T, MIYABAYASHI T, YUAN X P, XU Q, DONG G J, ZHAN Q L, LI C Y, FUJIYAMA A, TOYODA A, LU T T, FENG Q, QIAN Q, LI J Y, HAN B. A map of rice genome variation reveals the origin of cultivated rice. Nature, 2012, 490(7421): 497-501.
[31] Xie W B, WANG G W, YUAN M, YAO W, LYU K, ZHAO H, YANG M, LI P B, ZHANG X, YUAN J, WANG Q X, LIU F, DONG H X, ZHANG L J, LI X L, MENG X Z, ZHANG W, XIONG L Z, HE Y Q, WANG S P, YU S B, XU C G, LUO J, LI X H, XIAO J H, LIAN X M, ZHANG Q F. Breeding signatures of rice improvement revealed by a genomic variation map from a large germplasm collection. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(39): E5411-E5419.
[32] Zhao K Y, TUNG C W, EIZENGA G C, WRIGHT M H, ALI M L, PRICE A H, NORTON G J, ISLAM M R, REYNOLDS A, MEZEY J, MCCLUNG A M, BUSTAMANTE C D, MCCOUCH S R. Genome- wide association mapping reveals a rich genetic architecture of complex traits in. Nature Communications, 2011, 2: 467.
[33] Cui D, Zhou H, Ma X D, LIN Z C, SUN L H, HAN B, LI M M, SUN J C, LIU J, JIN G X, WANG X J, CAO G L, DENG X W, HE H, HAN L Z. Genomic insights on the contribution of introgressions from Xian/to the genetic improvement of Geng/rice cultivars. Plant Communication, 2022, 3(3): 100325.
[34] Chen Z, Bu Q Y, LIU G F, WANG M Q, WANG H R, LIU H Z, LI X F, LI H, FANG J, LIANG Y, TENG Z F, KANG S, YU H, CHENG Z K, XUE Y B, LIANG C Z, TANG J Y, LI J Y, CHU C C. Genomic decoding of breeding history to guide breeding-by-design in rice. National Science Review, 2023, 10(5): nwad029.
[35] Fei C, Xu Q, Xu Z J, CHEN W F. Effect of rice breeding process on improvement of yield and quality in China. Rice Science, 2020, 27(5): 363-367.
[36] Shan Q W, Wang Y P, Li J, ZHANG Y, CHEN K L, LIANG Z, ZHANG K, LIU J X, XI J J, QIU J L, GAO C X. Targeted genome modification of crop plants using a CRISPR-Cas system. Nature biotechnology, 2013, 31(8): 686-688.
[37] Cui Y, Zhu M M, Xu Z J, XU Q. Assessment of the effect of ten heading time genes on reproductive transition and yield components in rice using a CRISPR/Cas9 system. Theoretical and Applied Genetics, 2019, 132(6): 1887-1896.
[38] Cui Y, Jiang N, Xu Z J, XU Q. Heterotrimeric G protein are involved in the regulation of multiple agronomic traits and stress tolerance in rice. BMC Plant Biology, 2020, 20(1): 90.
[39] Lu Y M, YE X, GUO R M, HUANG J, WANG W, TANG J Y, TAN L T, ZHU J K, CHU C C, QIAN Y W. Genome-wide targeted mutagenesis in rice using the CRISPR/Cas9 system. Molecular Plant, 2017, 10(9): 1242-1245.
[40] Zeng D C, LIU T L, MA X L, WANG B, ZHENG Z Y, ZHANG Y L, XIE X R, YANG B W, ZHAO Z, ZHU Q L, LIU Y G. Quantitative regulation of Waxy expression by CRISPR/Cas9-based promoter and 5'UTR-intron editing improves grain quality in rice. Plant Biotechnology Journal, 2020, 18(12): 2385-2387.
[41] Huang L C, LI Q F, ZHANG C Q, CHU R, GU Z W, TAN H Y, ZHAO D S, FAN X L, LIU Q Q. Creating novel Wx alleles with fine-tuned amylose levels and improved grain quality in rice by promoter editing using CRISPR/Cas9 system. Plant Biotechnology Journal, 2020, 18(11): 2164-2166.
[42] Song X G, MENG X B, GUO H Y, CHENG Q, JING Y H, CHEN M J, LIU G F, WANG B, WANG Y H, LI J Y, YU H. Targeting a gene regulatory element enhances rice grain yield by decoupling panicle number and size. Nature Biotechnology, 2022, 40(9): 1403-1411.
The effect of/Xian pedigree introgression in/Geng rice breeding in China
Xu Hai1, Li Xiukun1,2, Lu Jiahao1, Jiang Kai1, Ma Yue1, Xu Zhengjin1, Xu Quan1
1Rice Research Institute, Shenyang Agricultural University, Shenyang 110866;2College of Agronomy, Hebei Agricultural University/State Key Laboratory of North China Crop Improvement and Regulation/Key Laboratory for Crop Germplasm Resources of Hebei, Baoding 071001, Hebei
【Objective】To demonstrate the impact of(XI) pedigree introgression on the yield and quality of(GJ) rice varieties, providing a theoretical basis and genomic resources for optimizing XI pedigree introgression breeding programs in northern GJ rice.【Method】In this study, the whole genome sequence on Illumina platform was employed to elucidate the effects of XI pedigree introgression on the yield and quality of rice in Northeast China were analyzed using recombinant inbred lines (RIL) derived from the cross between XI and GJ varieties, and 74 major GJ varieties grown from Heilongjiang, Liaoning, Shandong, and Jiangsu provinces as test materials. Using CRISPR/Cas9 gene editing technology to knock out the unfavorable genes introduced by XI pedigree introgression. 【Result】Analysis of RIL revealed a significant positive correlation between XI pedigree introgression and panicle length, grain length, and a negative correlation with head rice ratio. XI pedigree introgression was significantly negatively correlated with Amylose content, and significantly positively correlated with protein content in Jiangsu. With the increase of latitude, the correlation efficiency between XI pedigree introgression and grain shape increased, while the correlation between XI pedigree introgression and panicle length and head rice ratio decreased. The genomic fragments of XI pedigree introgression are unevenly distributed across different chromosomes and are more abundantly present on chromosomes 1, 10, 11, and 12. The XI pedigree introgression of the major cultivars in Jiangsu and Liaoning provinces is significantly higher than that in Heilongjiang and Shandong provinces, and the XI pedigree introgression of the cultivars after 2000 is significantly higher than that before 2000. The XI pedigree introgression includes multiple resistance and fertility-related genes. The project identified an XI pedigree introgression fragment on chromosome 5 of YF47, including the XI type grain regulatory geneand XI type chalkiness regulatory gene, which increased the 1000 grain weight of YF47 but affected its chalkiness-related traits. The project uses CRISPR/Cas9 technology to knock out thegene of YF47. The grain shape of the homozygous gene editing plants is similar to those of YF47, and its chalkiness character has been significantly improved. 【Conclusion】The XI pedigree introgression mainly increases the yield potential of GJ rice by increasing the number of grains per panicle, but has a negative impact on milling quality. Exploring the unfavorable alleles in varieties through high-throughput genome sequencing, combined with CRISPR/Cas9 gene editing, to break the genetic drag in breeding using the cross between XI and GJ, is an efficient breeding strategy that can quickly and accurately improve target traits.
rice;/Geng breeding;/Xian pedigree introgression; yield; quality; molecular design breeding

10.3864/j.issn.0578-1752.2023.22.001
2023-05-19;
2023-06-16
國家自然科學基金(32071982)
徐海,E-mail:chinaxuhai@163.com。通信作者徐銓,E-mail:kobexu34@syau.edu.cn
(責任編輯 李莉)

