牛冠狀病毒纖突蛋白的結構與功能分析





摘"要:【目的】""研究新疆牛冠狀病毒(Bovine coronavirus,BCoV)流行株的遺傳變異規律,分析纖突(Spike,S)蛋白結構與功能特征。
【方法】""對新疆巴楚縣某牛場BCoV陽性的犢牛糞便,提取病毒RNA,采用RT-PCR方法擴增S基因,通過序列測定和拼接,獲得BCoV S基因全長序列,對其開展生物信息學分析和遺傳進化分析。
【結果】""獲得BCoV S基因核苷酸序列(GenBank索引號:OR136878.1)。S基因全長4 092核苷酸(nucleotide,nt),編碼1 363氨基酸(amino acid,aa)。BCoV S蛋白分子量為150.67 Ku,等電點為5.29,有1個跨膜螺旋區,親水性較弱,疏水性略強。S蛋白主要分布于宿主細胞的內質網膜和高爾基體,具有信號肽的概率為0.984 2,是分泌型蛋白,含有14個潛在的N-糖基化位點和132個磷酸化位點。S蛋白具有3個結構域,其中受體結合結構域(Receptor binding domain,RBD)共含有215 aa,無規則卷曲(Coiled coil, Cc)占比最高(62.79%),其次為延伸鏈(Extended strand, Es)(20.47%)和α-螺旋(α-helix, Hh)(12.56%),β-轉角(β-turn, Tt)占比最少(4.19%)。篩選出S蛋白15個優勢B細胞表位和11個T細胞表位。S蛋白能與SWISS-MODEL數據庫中模板(SMTL ID:7sbw.1.C)同源建模,二者序列相似性為92.00%,模型GMQE值為0.76,QMEAN值為0.82,符合率較高,拉氏圖(Ramachandran plots)的Ramachandran favored值為96.14%,表明空間構象合理,模型準確可靠。遺傳進化試驗擴增的BCoVS基因序列,與2016年我國新疆的BCV-Aks-01株處于同一小分支,兩者S基因核苷酸序列同源性為99.1%。
【結論】""BCoV S蛋白存在多個抗原表位。RBD在病毒進入宿主細胞時具有重要作用,可針對RBD序列設計疫苗靶點,阻止病毒與宿主受體結合。采用生物信息學方法首次分析BCoV S蛋白理化性質、信號肽和亞細胞定位、磷酸化位點、糖基化位點、結構域、抗原表位、三級結構和RBD二級結構等特征,為BCoV S基因遺傳進化、結構與功能研究、疫苗研發、靶向藥物設計提供參考。
關鍵詞:""牛冠狀病毒;纖突蛋白;生物信息學分析
中圖分類號:"S858.23""""文獻標志碼:"A""""文章編號:"1001-4330(2024)11-2844-09
0"引 言
【研究意義】犢牛腹瀉是由多因素引起的以腹瀉為主要特征的癥候群。腹瀉可引起低血容量和酸中毒,對犢牛甚至是致命的,腹瀉還可導致厭食癥和共濟失調[1]。BCoV是引起腹瀉的病原之一,可導致犢牛腹瀉、成年牛冬痢和牛呼吸道疾病,還可引起犢牛死亡率升高、牛生產性能及產奶量下降,常造成經濟損失[2-5]。分析BCoV S蛋白生物信息學特征,為BCoV疫苗研發提供理論依據。【前人研究進展】近年來對BCoV的研究,包括檢測、遺傳進化分析和流行病學調查。Lotfollahzadeh S等[6]擴增了BCoV S基因,對其進行遺傳進化分析。Qinghe Zhu等[7]對我國東北1 016頭腹瀉牛的BCoV流行病學調查顯示,糞便陽性率為12.20%(124/1 016),鼻拭子陽性率21.53%(79/367)。寇美玲等[8]通過RT-PCR擴增,獲得了BCoV的全基因組序列,并分析了遺傳進化關系。張瑩鈺等[9]分離到BCV-Aks-01株,且進行了遺傳進化分析。張坤[10]對新疆北疆7個奶牛場的BCoV開展檢測,陽性率為0%~40%(ELISA)和0%~60%(RT-PCR)。【本研究切入點】BCoV屬于套式病毒目(Nidovirales)冠狀病毒科(Coronaviridae)冠狀病毒屬(Coronavirus)2a亞群,基因組全長約31 000 nt,編碼5種結構蛋白:纖突蛋白(S)、核衣殼蛋白(N)、跨膜蛋白(M)、血凝素酯酶蛋白(HE)、小衣殼蛋白(E)[11-14]。其中S蛋白的主要負責病毒與易感細胞的結合,介導膜融合及誘導機體產生中和抗體[15]。目前,對BCoV S蛋白遺傳進化分析的報道較多[6,8-9],但尚無生物信息學分析方面較全面的報道。需研究新疆牛冠狀病毒流行株的遺傳變異規律。【擬解決的關鍵問題】從腹瀉犢牛糞便中提取BCoV RNA,通過RT-PCR擴增S基因,采用生物信息學方法分析S蛋白結構與功能,并分析遺傳進化關系,為病毒-宿主相互作用、S蛋白基因工程疫苗研究奠定基礎。
1"材料與方法
1.1"材 料
1.1.1"樣 品
新疆巴楚縣某牛場腹瀉犢牛糞便,干冰運輸,-80℃凍存備用。
1.1.2"主要試劑
體液病毒DNA/RNA小量制備試劑盒AP-MN-BF-VNA-250、DNA凝膠回收試劑盒AP-GX-250,購自Axygen;RT-PCR試劑盒購自TaKaRa公司;GeneRuler 1 kb Plus DNA Ladder購自Thermo Fisher。
1.1.3"主要儀器、器皿
Eppendorf低溫高速離心機、德國耶拿Biometra PCR儀、美國Bio-Rad核酸電泳系統和凝膠成像系統,錐形瓶、50 mL離心管。
1.1.4"引物設計與合成
以GenBank中BCoV S基因序列(GenBank索引號:KU886219.1)為參考序列,應用Oligo6.0軟件,將S基因分為相互重疊的6段,分別設計6對特異性引物,由通用生物(安徽)股份有限公司合成。表1
1.2"方 法
1.2.1"BCoV S基因的擴增
將腹瀉犢牛糞便用1∶"PBS稀釋5倍,5 000 r/min離心5 min,取上清200 μL,提取病毒RNA,具體方法按體液病毒DNA/RNA小量制備試劑盒說明書操作。以RNA為模板,用RT-PCR試劑盒擴增BCoV S基因的6個片段。采用反轉錄PCR(RT-PCR)方法擴增S基因,反應條件:50℃ 30 min;94℃ 2 min,預變性;再以94℃ 30 s,54℃ 20 s,72℃ 50 s,30個循環擴增;最后72℃ 10 min,延伸。RT-PCR產物經1%瓊脂糖凝膠電泳,DNA凝膠回收試劑盒純化后,送通用生物(安徽)股份有限公司測序,將測序結果拼接后得到S基因全長序列。
1.2.2"S蛋白理化性質、生物信息學及遺傳進化
利用多種在線網站和相關軟件對BCoV S蛋白開展理化性質與生物信息學分析。將S基因序列在NCBI中用BLAST搜索同源序列,根據序列一致性分值的排序,依次下載得分高的部分序列,再下載國內外具有代表性的部分序列,運用分子生物學軟件Mega 5.0,選擇連接近鄰法構建系統發生樹。表2
2"結果與分析
2.1"BCoV S基因擴增
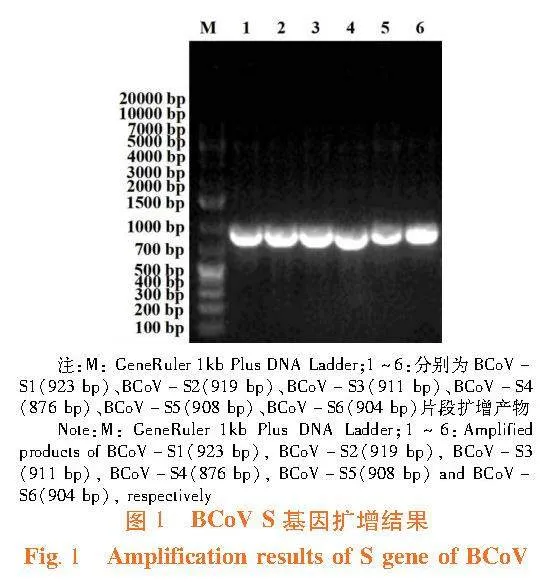
研究表明,獲得BCoV S基因的6個片段,每個片段的大小與預期相符。經序列測定和拼接,獲得4092 nt的BCoV S基因全長核苷酸序列,已將該序列發布于GenBank(索引號:OR136878.1)。圖1
2.2"S蛋白的理化性質
研究表明,S蛋白編碼1363 aa,S蛋白分子量為150.67 kDa,分子式為C6745H10311N1729O2038S76,等電點理論值為5.29,負電荷(Asp+Glu)殘基總數為112個,正電荷(Arg + Lys + His) 殘基總數為89個,脂肪指數:85.30。波長280 nm條件下,在水溶液中的消光系數為197 750 L/(mol·cm)。在體外培養的哺乳動物網織紅細胞內半衰期為30 h,在酵母體內半衰期gt;20 h,在大腸埃希氏桿菌體內的半衰期gt;10 h。不穩定系數為33.37,該蛋白屬于穩定蛋白。
2.3"S蛋白跨膜結構預測
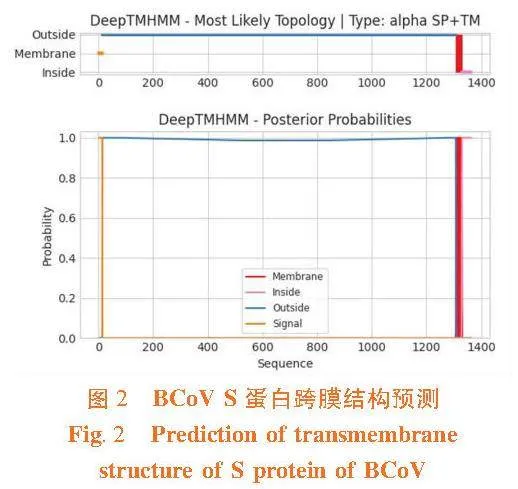
研究表明,S蛋白具有1個跨膜螺旋區,處于W1308~I1328位置,依此確定S蛋白為跨膜蛋白。同時,該蛋白細胞膜外的肽段處于V15~P1307,細胞膜內的肽段處于C1329~D1363。圖2"
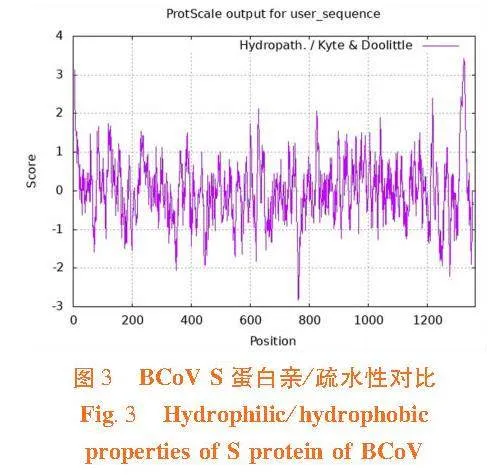
2.4"S蛋白親/疏水性對比
研究表明,S蛋白親水性最強的殘基是R764,Score=-2.833,而疏水性最強的是F1326,Score=3.411。比較親水氨基酸與疏水氨基酸的數目,二者相差不大。S蛋白親水性較弱,疏水性略強。圖3
2.5"S蛋白亞細胞定位和信號肽預測
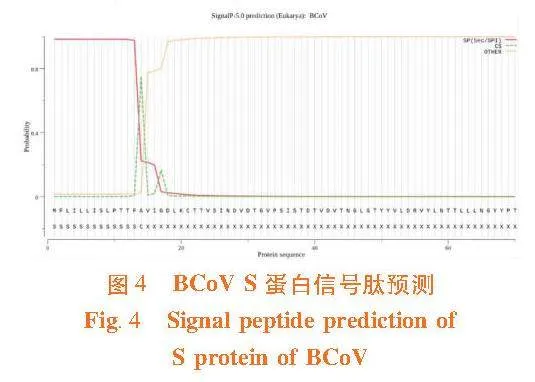
研究表明,內質網膜占44.4%、高爾基體占33.3%、細胞膜占22.2%。S蛋白具有信號肽的概率為0.984 2,遠高于閾值0.5,說明該蛋白是分泌型蛋白。此外,信號肽切割位點位于A14和V15之間(TFA-VI),概率為0.752 1。圖4
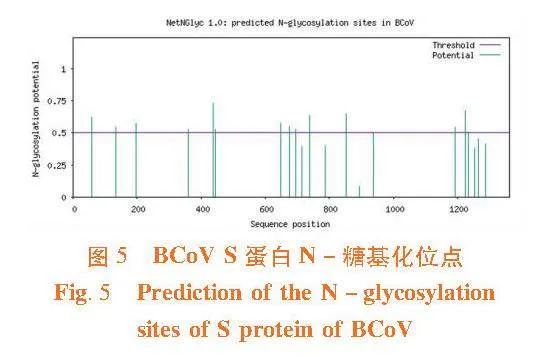
2.6"S蛋白糖基化位點預測
研究表明,采用在線軟件對糖基化位點預測,發現S蛋白含有14個潛在的N糖基化位點,其位置分別為59、133、198、359、437、444、649、676、696、739、852、1 194、1 224和1 234。圖5
2.7"S蛋白磷酸化位點預測
研究表明,S蛋白共有132個磷酸化位點,各類氨基酸位點所占數量為:絲氨酸(Serine,S)63個,蘇氨酸(Threonine,T)46個,酪氨酸(Tyrosine,Y)23個。圖6
2.8"S蛋白結構域
研究表明,共有3個保守的功能結構域,分別是N端結構域(N-terminal Domain,NTD)、纖突RBD和S2糖蛋白。NTD位于I16~S292,纖突RBD(S1亞基)位于P326~T540,S2糖蛋白(S2亞基)位于E780~S1360。S蛋白在761~772 aa(序列為:STKRRSRRSITT)區域存在一個低復雜度區域(玫紅色位點處)。768~769 aa之間存在一個蛋白酶切割位點。圖7
2.9"S蛋白RBD二級結構
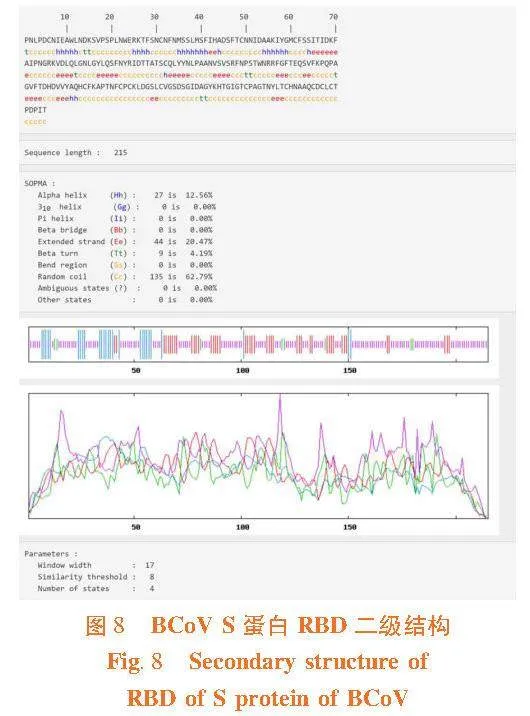
研究表明,RBD共有215個aa,其中α-螺旋(α-helix, Hh)27個(12.56%),延伸鏈(Extended strand, Es)44個(20.47%),β-轉角(β-turn, Tt)9個(4.19%),無規則卷曲(Coiled coil, Cc)135個(62.79%)。圖8
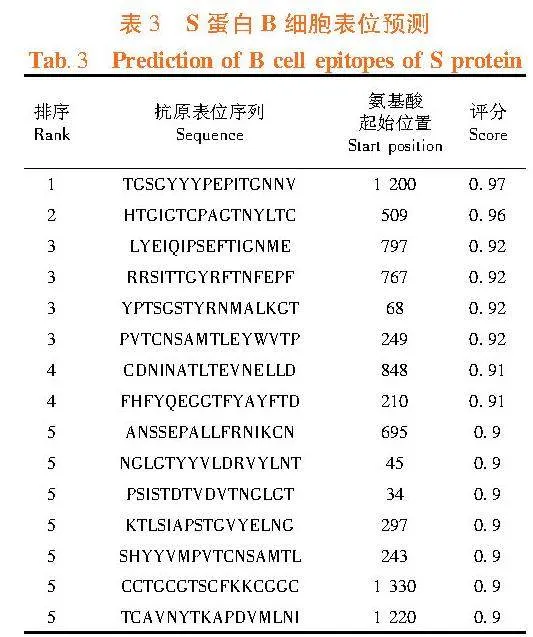
2.10"BCoV S蛋白B細胞和T細胞表位預測
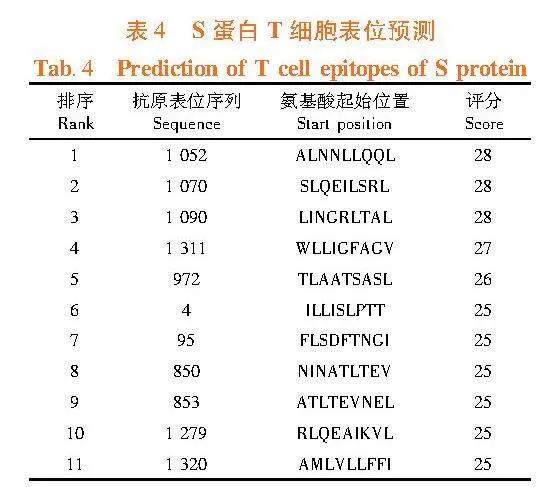
研究表明,采用ABCpred軟件預測可能的B細胞表位,Scoregt;0.80的B細胞表位數量為64個,Scoregt;0.90的B細胞表位數量為15個。在SYFPEITHI網站中HLA-A*0201條件下,篩選出11個細胞毒性T細胞(CLT)表位(Score≥25)。表3,表4
2.11"BCoV S蛋白三級結構同源建模
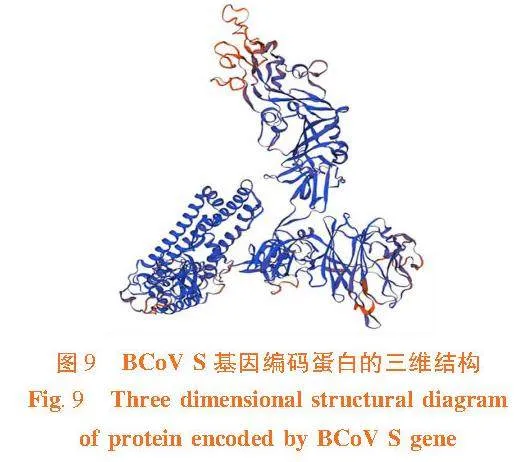
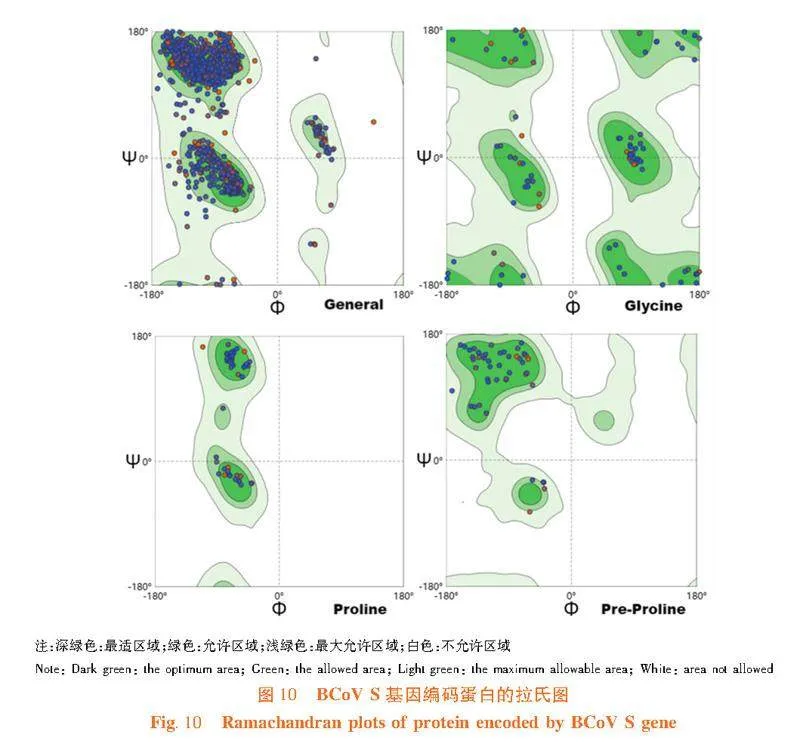
研究表明,在SWISS-MODEL數據庫中選擇模板(SMTL ID:7sbw.1.C),對BCoV S蛋白的氨基酸序列同源建模,獲得BCoV S蛋白三維結構模型,兩者序列一致性為92.00%,模型GMQE值為0.76,QMEAND值為0.82。利用拉氏圖評價所建模型,Ramachandran favored值為96.14%,空間構象合理,模型準確可靠。圖9,圖10
2.12"BCoV S基因系統發生樹的構建
研究表明,試驗擴增的BCoV S基因序列,與2016年我國新疆南疆的BCV-AKS-01毒株(GenBank索引號:KU886219.1)處于同一小分支。利用DANMAN軟件將兩者S基因核苷酸序列比對,序列一致性為99.07%。圖11,圖12
3"討 論
BCoV在牛群中廣泛存在[16-17]。BCoV與新冠病毒(SARS-CoV-2)同屬于β冠狀病毒屬,對SARS-CoV-2研究表明,S蛋白在病毒表面呈三聚體狀態排列,對病毒入侵宿主細胞至關重要[20],是疫苗、治療性抗體和診斷的關鍵靶點[18-19]。SARS-CoV-2的生物信息學已有報道[20],但未見對BCoV S蛋白生物信息學分析較全面的報道。我國尚無商品化BCoV疫苗,因此,有必要對BCoV S蛋白進行生物信息學分析,為疫苗研發提供參考。
BCoV S蛋白包含2個疏水區:一個位于蛋白質的N端作為分泌信號,另一個位于C端作為膜錨定區[21-22]。研究結果顯示,S蛋白親水性較弱,疏水性略強,推測與上述報道的2個疏水區有關。糖基化和磷酸化修飾是生物體內最重要的翻譯后修飾方式。病毒的糖基化過程對病毒蛋白的折疊與穩定、病毒的感染和入侵、宿主細胞受體識別和病毒免疫逃逸等起著重要作用[23]。磷酸化修飾的病毒蛋白參與調控病毒復制、病毒增殖和病毒粒子裝配等一系列代謝活動,在調控病毒與宿主的代謝中起著重要作用[24]。研究分析顯示,BCoV S有14個潛在的N-糖基化位點,132個磷酸化位點;3類氨基酸在該蛋白中的磷酸化位點數量分別是:S63個、T46個、Y23個。這些位點為S蛋白糖基化和磷酸化的修飾提供了基礎。
Parker等[21]認為,病毒感染細胞時BCoV S的初始切割發生在763~769 aa之間的某個位點,其中764~768 aa序列為RRSRR。Abraham S等[15]認為S蛋白在768~769 aa之間裂解為S1和S2兩個小亞基。試驗中BCoV S蛋白761~772 aa(序列為:STKRRSRRSITT)區域存在一個低復雜度區域(圖7中玫紅色位點處),其中764~768 aa(序列為:RRSRR)與上述報道相同,因此,推測其切割位點也在768~769 aa之間。BCoV S蛋白主要負責細胞的粘附、血凝、膜融合及誘導中和抗體,S1負責病毒-宿主細胞的識別與結合,S2與病毒-細胞間的融合有關[15]。冠狀病毒進入細胞,依賴于病毒粒子表面的S蛋白三聚體與宿主細胞受體之間的特異性相互作用[25]。S1亞基的NTD在病毒附著到宿主細胞表面聚糖時起著關鍵作用,RBD負責特異性結合宿主蛋白受體;S1的 C端結構域(C-terminal domain,CTD)保持著無活性的“平放”構象,一旦S蛋白三聚體與宿主受體結合,CTD就會擴展為有活性的“直立”構象[18]。與宿主受體結合后,S蛋白三聚體被組織蛋白酶、TMPRRS2或其它蛋白酶切割,病毒與細胞膜融合,病毒進入宿主細胞[25]。上述過程是冠狀病毒感染機制的關鍵步驟,因此,可針對RBD序列設計疫苗靶點,阻止病毒與受體的結合,從而阻止病毒進入宿主細胞。
S蛋白結構復雜,多種二級結構混合存在,為其功能提供了基礎。通過表位預測,篩選出S蛋白優勢B細胞表位15個,優勢T細胞表位11個,可為疫苗靶點的設計提供參考。研究的BCoV S蛋白氨基酸序列,與SWISS-MODEL數據庫模板(SMTL ID:7sbw.1.C)同源建模,GMQE值(0.76)和QMEAN值(0.82)較高,序列一致性為92.00%,說明二者相似度較高,經拉氏圖評價,表明所建模型準確可靠。從預測圖看出,S蛋白由三個單體結合而成。蛋白質序列一級結構的同源性高,相應結構的可靠性也高,這為S蛋白設計和改造提供了較為堅實的結構基礎。遺傳進化分析顯示,研究擴增的序列與2016年我國新疆南疆的BCV-Aks-01毒株處于同一小分支,兩者核苷酸序列同源性很高(99.1%),表明在局部地區,BCoV的遺傳距離相對較近,毒株之間交叉保護的可能性會較高。
4"結 論
研究的BCoV S基因核苷酸序列與2016年我國新疆BCV-Aks-01株序列在同一分支,親緣關系最近。S蛋白存在多個抗原表位,推測其免疫原性較強。RBD在病毒進入宿主細胞時具有重要作用,可針對RBD序列設計疫苗靶點,阻止病毒與宿主受體結合。BCoV S蛋白同源建模結果準確可靠。研究采用生物信息學方法,首次全面分析了BCoV S蛋白理化性質、信號肽和亞細胞定位、磷酸化位點、糖基化位點、結構域、抗原表位、三級結構和RBD二級結構等特征,并對S基因進行遺傳進化分析。
參考文獻"(References)
[1]"Maier G U, Breitenbuecher J, Gomez J P, et al.Vaccination for the prevention of neonatal calf diarrhea in cow-calf operations: a scoping review[J].Veterinary and Animal Science, 2022, 15: 100238.
[2] Woode G N, Bridger J C, Meyling A.Significance of bovine coronavirus infection[J].The Veterinary Record, 1978, 102(1): 15-16.
[3] Benfield D A, Saif L J.Cell culture propagation of a coronavirus isolated from cows with winter dysentery[J].Journal of Clinical Microbiology, 1990, 28(6): 1454-1457.
[4] Geng HL, Meng XZ, Yan WL.et al.Prevalence of bovine coronavirus in cattle in China: A systematic review
and meta-analysis, Microbial Pathogenesis [J].Microb Pathog, 2023, 176:106009.
[5] Saif L J.Bovine respiratory coronavirus[J].The Veterinary Clinics of North America Food Animal Practice, 2010, 26(2): 349-364.
[6] Lotfollahzadeh S, Madadgar O, Reza Mohebbi M, et al.Bovine coronavirus in neonatal calf diarrhoea in Iran[J].Veterinary Medicine and Science, 2020, 6(4): 686-694.
[7] Zhu Q H, Su M J, Li Z J, et al.Epidemiological survey and genetic diversity of bovine coronavirus in Northeast China[J].Virus Research, 2022, 308: 198632.
[8] 寇美玲, 謝佳芮, 楊佳萍, 等.牛冠狀病毒的全基因組測序及遺傳進化分析[J].動物醫學進展, 2022, 43(10): 1-7.
KOU Meiling, XIE Jiarui, YANG Jiaping, et al.Whole genome sequencing and Genetic Evolution Analysis of Bovine Coronavirus[J].Progress in Veterinary Medicine, 2022, 43(10): 1-7.
[9] 張瑩鈺, 張迎春, 王青青, 等.新疆南疆牛冠狀病毒BCV-Aks-01株分離及基因型鑒定[J].中國獸醫雜志, 2018, 54(2): 12-14, 18, 2.
ZHANG Yingyu, ZHANG Yingchun, WANG Qingqing, et al.Isolation and genotype identification of bovine coronavirus BCV-aks-01 strain in southern Xinjiang[J].Chinese Journal of Veterinary Medicine, 2018, 54(2): 12-14, 18, 2.
[10] 張坤.新疆北疆地區規模化奶牛場犢牛病毒性腹瀉相關病原的調查研究[D].石河子: 石河子大學, 2016.
ZHANG Kun.Investigation of Calves Viral Diarrhea Related Pathogen of Large-scale Dairy Farm in Northern Xin Jiang Region[D].Shihezi: Shihezi University, 2016.
[11] Masters P S.The molecular biology of coronaviruses[J].Advances in Virus Research, 2006, 66: 193-292.
[12] Gunn L, Collins P J, O’Connell M J, et al.Phylogenetic investigation of enteric bovine coronavirus in Ireland reveals partitioning between European and global strains[J].Irish Veterinary Journal, 2015, 68: 31.
[13] Franzo G, Drigo M, Legnardi M, et al.Bovine coronavirus: variability, evolution, and dispersal patterns of a No longer neglected betacoronavirus[J].Viruses, 2020, 12(11): 1285.
[14] Singh S, Singh R, Singh K P, et al.Immunohistochemical and molecular detection of natural cases of bovine rotavirus and coronavirus infection causing enteritis in dairy calves[J].Microbial Pathogenesis, 2020, 138: 103814.
[15] Abraham S, Kienzle T E, Lapps W, et al.Deduced sequence of the bovine coronavirus spike protein and identification of the internal proteolytic cleavage site[J].Virology, 1990, 176(1): 296-301.
[16] Boileau M J, Kapil S.Bovine coronavirus associated syndromes[J].The Veterinary Clinics of North America Food Animal Practice, 2010, 26(1): 123-146.
[17] Toftaker I, Holmy I, Ndtvedt A, et al.A cohort study of the effect of winter dysentery on herd-level milk production[J].Journal of Dairy Science, 2017, 100(8): 6483-6493.
[18] Yoshizawa N, Ishihara R, Omiya D, et al.Application of a photocatalyst as an inactivator of bovine coronavirus[J].Viruses, 2020, 12(12): 1372.
[19] Salem E, Dhanasekaran V, Cassard H, et al.Global transmission, spatial segregation, and recombination determine the long-term evolution and epidemiology of bovine coronaviruses[J].Viruses, 2020, 12(5): 534.
[20] Wrapp D, Wang N S, Corbett K S, et al.Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation[J].Science, 2020, 367(6483): 1260-1263.
[21] Yuan M, Wu N C, Zhu X Y, et al.A highly conserved cryptic epitope in the receptor binding domains of SARS-CoV-2 and SARS-CoV[J].Science, 2020, 368(6491): 630-633.
[22] 閆靜靜, 遲曉妍, 盧佳琪, 等.SARS-CoV-2結構蛋白S和N的生物信息學比較分析及應用研究[J].中國病原生物學雜志, 2023, 18(4): 377-384.
YAN Jingjing, CHI Xiaoyan, LU Jiaqi, et al.Comparative bioinformatics analysis of structural proteins s and N of SARS-CoV-2 and their application[J].Journal of Pathogen Biology, 2023, 18(4): 377-384.
[23] Parker M D, Yoo D, Cox G J, et al.Primary structure of the S peplomer gene of bovine coronavirus and surface expression in insect cells[J].The Journal of General Virology, 1990, 71 ( Pt 2): 263-270.
[24] St Cyr-Coats K S, Storz J, Hussain K A, et al.Structural proteins of bovine coronavirus strain L 9: effects of the host cell and trypsin treatment[J].Archives of Virology, 1988, 103(1/2): 35-45.
[25] 向田, 章曉聯.病毒與宿主細胞的糖基化修飾及相關功能[J].生物化學與生物物理進展, 2017, 44(10): 898-907.
XIANG Tian, ZHANG Xiaolian.Glycosylation modification and related functions of virus and host cells[J].Progress in Biochemistry and Biophysics, 2017, 44(10): 898-907.
Structure and function analysis of spike protein ""of bovine coronavirus
LU Guili, MIAO Shukui, "WEI Jie, WEI Yurong, MI Xiaoyun, Hailiqiemu Maimaitiyiming
(Xinjiang Key Laboratory of Animal Infectious Diseases/ Institute of Veterinary Medicine, Xinjiang Academy of Animal Sciences, Urumqi 830013, China)
Abstract:【Objective】 ""The purpose of this paper is to grasp the genetic variation patterns of the prevalent strains of bovine coronavirus (BCoV) in Xinjiang, and analyze the characteristics of structural and functional of spike (S) protein.Providing data support for the genetic evolution, structure and function research, vaccine research and development, and targeted drug design of S gene of BCoV.
【Methods】 """The viral genomic RNA was extracted from the feces of BCoV positive calves in a cattle farm in Bachu County, Xinjiang, and the S gene was amplified by RT-PCR.The full length of the S gene was obtained through sequencing and splicing, and after that, the bioinformatics analysis and genetic evolution analysis were carried out.
【Results】 """The BCoV S gene nucleotide sequence (Published on GenBank.Accession number: OR136878.1) was successfully obtained, with a total length of 4 092 nucleotides (nt).The S gene was encoded by 1 363 amino acids (aa).The molecular weight of S protein was 150.67 Ku, and its isoelectric point was 5.29.It had a transmembrane helical region with weak hydrophilicity and slightly stronger hydrophobicity.It has a transmembrane spiral region, with weak hydrophilicity and slightly strong hydrophobicity.S protein was mainly located in the endoplasmic reticulum membrane and golgi apparatus of the host cell, and the probability of signal peptide in N-terminal was 0.9842.The S protein contained 14 potential N-glycosylation sites and 132 phosphorylation sites, and had 3 structural domains, among which the receptor binding domain (RBD) had a total of 215 aa.The proportion of random curls in RBD was the highest (62.79%), followed by extended chains (20.47%) and α-helices (12.56%), the lowest proportion comprised β-turns (4.19%).15 dominant B cell epitopes and 11 T cell epitopes were picked out by software ABCpred and SYFPEITHI.The S protein could be homologously modeled with the template (MTL ID: 7sbw.1.C) in the SWISS-MODEL database, with a sequence identity of 92.00%.The GMQE score of the model was 0.76, and the QMEAND score was 0.82, which indicated a high coincidence rate between them.The Ramachandran favored value of the ramachandran plot is 96.14%, indicating that the spatial conformation was reasonable and the model was accurate and reliable.Evolutionary analysis showed that the BCoV S gene amplified in this experiment was located in the same branch as BCV-AKS-01 strain in southern Xinjiang of China in 2016, and the nucleotide sequence identity of those S gene was 99.07%.
【Conclusion】 """The S protein of BCoV has multiple antigenic epitopes and immunogenicity.RBD plays an important role in the entry of viruses into host cells, and vaccine targets can be designed based on the RBD sequence to prevent the virus from binding to host receptors.The results of homologous modeling of the S protein of BCoV are accurate and reliable.In this study, the physicochemical properties, signal peptides and subcellular localization, phosphorylation sites, glycosylation sites, domains, antigenic epitopes, tertiary structures, and RBD secondary structures of the S protein of BCoV are analyzed using bioinformatics methods.
Key words:""bovine coronavirus; spike protein; bioinformatics analysis
Fund projects:"nbsp;Natural Science Foundation of Xinjiang Uygur Autonomous Region(2021D01A60)
Correspondence author:"""MIAO Shukui(1983- ), male,from Gansu,master, senior experimentalist, research direction:etiology and immunology of animal viruses, (E-mail) 903076760@qq.com
收稿日期(Received):
2024-04-11
基金項目:
新疆維吾爾自治區自然科學基金項目(2021D01A60)
作者簡介:
陸桂麗(1977- ),女,江蘇人,研究員,博士,研究方向為動物傳染病防控,(E-mail)66498744@qq.com
通訊作者:
苗書魁(1983- ),男,甘肅人,正高級實驗師,碩士,研究方向為動物病毒病原學及免疫學,(E-mail)903076760@qq.com

