高中生物學PCR技術中的引物設計
陳鐵鑫

[摘 要]聚合酶鏈式反應(PCR)是江蘇高考生物試題中的常考內容,PCR技術的用途并不局限于最初、最基本的獲取目的基因。PCR技術的使用場景極其靈活,通過引物設計,可以達到融合基因、定點誘變等效果。文章對PCR技術中的引物設計思路進行分類,介紹其衍生的各種應用,為教師教學提供參考。
[關鍵詞]PCR;引物設計;高中生物學
[中圖分類號]??? G633.91??????? [文獻標識碼]??? A??????? [文章編號]??? 1674-6058(2024)14-0084-04
核心素養是高考命題和各類考試命題的指導方向和原則。為加深學生對基因工程大概念的理解,提升學生的生物學科核心素養,《普通高中生物學課程標準(2017年版2020年修訂)》要求教師在基因工程的教學過程中開展“利用聚合酶鏈式反應(PCR)擴增DNA片段并完成電泳鑒定,或運用軟件進行虛擬PCR實驗”活動[1]。引物直接決定PCR的特異性與成功與否。人教版高中生物學選擇性必修三中通過圖片簡單演示了PCR的基本過程,并未涉及引物設計,而近幾年各地的高考試題為了響應新課標要求,基于核酸檢測、DNA測序等真實情境,增加了許多與引物設計相關的技術困境內容。為跨越教材與實際應用的鴻溝,筆者對不同應用場景中的引物設計進行分析,以促進學生對PCR的理解,為教師教學提供參考。
一、以基因定位為目的的反向引物設計
以真核生物為受體細胞時,目的基因一般需整合至受體細胞的染色體DNA中,以實現穩定存在與表達。在上述情境中,為進行目的基因定位,需要在已知目的基因的附近逐步搜索并測序相鄰的DNA區域,這就需要用到反向PCR。
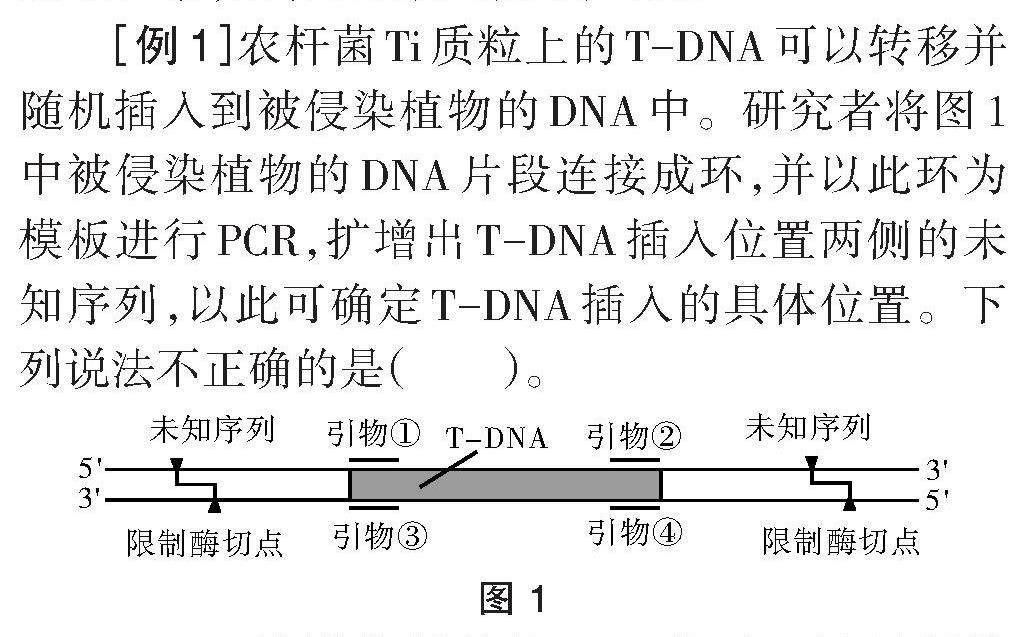
[例1]農桿菌Ti質粒上的T-DNA可以轉移并隨機插入到被侵染植物的DNA中。研究者將圖1中被侵染植物的DNA片段連接成環,并以此環為模板進行PCR,擴增出T-DNA插入位置兩側的未知序列,以此可確定T-DNA插入的具體位置。下列說法不正確的是()。
圖 1
A.PCR擴增依據的是DNA分子雙鏈復制的原理
B.進行PCR擴增需要熱穩定DNA聚合酶
C.利用圖中的引物②、③組合可擴增出兩側的未知序列
D.通過與受體細胞的基因組序列比對,可確定T-DNA的插入位置
解析:對含目的基因的T-DNA中兩側未知序列通過同種限制酶或是同尾酶進行酶切(目的基因中不能存在該酶的酶切位點),可取得相同的黏性末端。通過DNA連接酶令圖 1含T-DNA的片段自身環化[2]成圖2甲的環形DNA分子。
礙于DNA聚合酶的特性,子鏈總是從5'端往3'端延伸。若目的為擴增T-DNA,應選擇指向已知擴增片段的內側的引物②、③。選擇引物①、④,子鏈向外側未知序列進行延伸。由于PCR體系中沒有DNA連接酶,因此子代DNA仍是線性(如圖2乙)。擴增后,原本在兩側的未知序列被轉移到已知序列的內部(如圖2丙)。隨后,我們可以對未知序列進行測序,比對受體細胞的基因組文庫,即可確定T-DNA插入的位置。
圖 2
參考答案:C
二、鑒定目的基因連接方式的反向引物設計
構建基因表達載體時,若使用單酶切可能出現目的基因的反向連接。目的基因的篩選與檢測是基因工程的重要環節,可以通過設計引物進行PCR,篩選目的基因與載體的正向連接產物。
[例2]為鑒定篩選出的菌落中是否含有正確插入目的基因的重組質粒,擬設計引物進行PCR鑒定。圖3所示為甲、乙、丙3條引物在正確重組質粒中的相應位置。
(1)PCR鑒定時應選擇的一對引物是????????????? 。
(2)某學生嘗試用圖中另外一對引物從某一菌落的質粒中擴增出了400 bp片段,原因是?????????????????????????????????????????????????????????????????????????????????????????????? 。
解析:如圖 4所示,黑色區域為目的基因,我們在目的基因內、外各設計一引物,圖中為正向連接時各引物序列的位置。使用引物1、引物3可擴增出對應片段,當目的基因反向連接時,引物1與引物3方向相同,無法有效擴增。同理,選擇引物2、引物4,也可以進行上述驗證。與預期相符的DNA長度就可以用于確認目的基因的正向連接。
圖3為正向連接時的引物分布。引物丙、引物甲位于目的基因的上游和下游,無論連接方向是否正確,均可擴增出片段且長度相同,無法作為排除的依據。
若使用引物丙、引物乙進行PCR,考慮反向連接時,引物乙的方向與引物丙相同,無法擴增片段,正向連接時引物丙、引物乙可以擴增出350 bp長度的片段。
同理,若選擇引物甲、引物乙進行PCR,反向連接時能擴增出400 bp片段,正向連接時無擴增產物,但空質粒也會帶來相同的結果,無法確定實現了正向連接。
因此,PCR鑒定時應選擇引物乙、引物丙。學生擴增出400 bp片段的原因是目的基因反向連接。
參考答案:(1)乙、丙 (2)目的基因反向連接
三、PCR體系中的引物序列改造
目的基因想要導入表達載體,兩側需要有合適的酶切位點。實驗室用到的限制酶價格高昂,種類有限,為了不被基因自身序列限制,就需要對引物序列進行改造。引物設計的依據是目的基因的兩側序列,但這并不意味著引物需要與模板完全互補。我們既可以在5'端進行添加限制酶識別序列,也可以在中間進行少量堿基的增刪或者替換。引物改造的部分序列不能和原對應模板配對,但不影響引物和模板的整體配對,因此,其擴增產物的對應位置就會引入識別序列,或是發生堿基對的增刪或者替換[3]。
(一)引入序列與連接DNA
引物與模板的結合并不需要完全一一對應。引物的3'端是DNA聚合酶添加游離脫氧核苷酸的位點,如果引入的核苷酸序列添加在3'端,會導致擴增產物的特異性下降。因此,我們常常只在5'端添加所需要的酶切位點。若在兩個DNA片段對應引物的5'端分別構建相同或者互補序列,對獲得的新DNA進行變性與雜交,還可以達到連接DNA的效果。
[例3]科研人員通過敲除里氏木霉基因組中去乙酰化酶基因(HDAC),探究組蛋白乙酰化對纖維素酶基因表達的影響,其主要過程如圖5。請據圖回答:引物設計是PCR的關鍵,本研究中引物R1的5'端添加與引物FG互補的序列,其目的是???????????????????? 。在PCR5中加入的引物是?????????????????????? 。
圖 5
解析:通過PCR1、PCR2,可獲得新霉素抗性基因GR與H1的構造(如圖 6)。
圖 6
以GR 基因、H1為模板,F1和RG為引物進行PCR4擴增,在高溫作用下雙鏈會打開。由題可知,引物R1的5'端添加了與引物FG互補的序列,因此其延伸產物H1基因的b鏈5'端也存在引物FG的互補序列,b鏈與c鏈就能夠部分配對(如圖 7)。Taq? DNA聚合酶只能從5'端向3'端延伸,這兩條鏈的3'端都缺乏模板,無法繼續延伸。
圖 7
a鏈的3'端與R1序列互補,d鏈的3'端與FG互補。由于R1與FG互補,因此a鏈的3'端和d鏈的3'端也是互補的(如圖 8)。
圖 8
在Taq? DNA聚合酶的作用下,可將重疊的兩條鏈補齊(如圖9)。
圖 9
兩個基因就這樣融合成功。隨后a鏈、d鏈分別結合引物RG、F1進行PCR4,就得到了大量的H1-GR融合基因。這種借助引入序列連接DNA的方法我們稱為重疊延伸PCR。按照相同的邏輯,可以將基因H2和GR也進行連接。
圖10
如圖10,通過類比,不難想到應令c鏈和f鏈發生互補配對、延伸。隨后c鏈、f鏈分別以R2、FG為對應引物,進行大量擴增。為確保這兩條鏈的堿基互補配對,可在F2的5'端通過引入序列,添加與RG互補的序列。
綜上,本研究中在引物R1的5'端添加與引物FG互補的序列,其目的是便于PCR4時H1和GR基因的連接。在PCR5中加入的引物是R2和FG。
參考答案:便于H1和GR的拼接 R2和FG
(二)定點誘變
引物是PCR產物5'端的一部分,引物長度一般為15~27 bp,因此最初建立的PCR定點誘變技術只適用于DNA的末端突變。突變位點在基因中部時,我們就得設計兩對引物,基因中部的兩個誘變引物分別與兩側常規引物將目的基因分段擴增,再以上文中連接DNA的方式將兩段誘變DNA融合。
[例4]重疊延伸PCR技術是采用具有互補末端的引物,使PCR產物形成重疊鏈,從而在隨后的擴增反應中通過重疊鏈的延伸,獲得想要的目的基因。某科研團隊運用重疊延伸PCR技術在水蛭素基因的特定位點引入特定突變,以實現基因的定點突變,原理如圖11。下列說法錯誤的是()。
圖11
A. 過程②需要含Mg2+的緩沖液、DNA模板、引物、四種脫氧核苷酸、耐高溫的DNA聚合酶等
B. 若引物1、引物2組成的反應系統和引物3、引物4組成的反應系統中均進行一次DNA分子的復制后,一共會產生3種DNA分子
C. 經過過程④獲得的雜交DNA有2種,其中只有一種可以經過過程⑤獲得目的基因
D. 過程⑤使用耐高溫的DNA聚合酶延伸,需要引物
解析:過程④獲得的雜交DNA具有游離的3'端,也具有模板,可以直接使用耐高溫的DNA聚合酶延伸,不需要引物。其連接機制與例3相同。
參考答案:D
四、特異性擴增的嵌套引物設計
引物的長度一般在20個堿基左右,因此從概率上講,待擴增片段上出現與引物配對序列的概率是[1420],約萬億分之一,因此一段長度為20 bp的序列在人類基因組約31.6億堿基對的排列組合中理論上重復的概率很低。但堿基對的排列順序并不是完全隨機的,引物與模板鏈的結合也并不需要完全互補,因此總有非特異性擴增條帶的出現。巢式PCR為了解決上述困境,對PCR體系中的引物數量進行了設計。
[例5]普通PCR在擴增DNA的過程中存在一定概率的堿基錯誤配對,得到部分的錯誤產物。巢式PCR是一種在普通PCR基礎上改良的新型PCR技術,擴增的精確性比普通PCR更高。巢式PCR對目標DNA進行擴增時需要用到兩對不同的引物,首先使用第一對引物(外引物)對目標DNA進行第一次擴增得到中間產物,然后使用第二對引物(內引物)對中間產物進行第二次擴增得到目標產物,過程如圖12所示。請回答相關問題:
圖12
巢式PCR通常在第一支試管中進行第一次擴增,然后把得到的中間產物分離出來轉移到另外一支試管中進行第二次擴增反應,兩次擴增不在同一支試管中擴增的原因是??? ???????????????????????????????????????。
解析:針對已知的目的基因兩側部分序列,設計外引物1、4,內引物2、3。
圖13
如圖13,首先通過外引物1、4擴增序列,此時依然存在這樣的可能性:引物與其他未知片段結合。經過第一次擴增后,提取反應產物作為下一輪模板,使用內引物2、3進行第二次擴增。若第一次擴增出非特異性條帶,第二次擴增因缺乏模板無法正常進行。第二次擴增的成功也是對第一次擴增反應正常進行的鑒定。因此,若兩次擴增在同一個反應體系中進行,精確性就無法得到驗證。
參考答案:防止內引物直接參與第一次擴增,無法實現巢式PCR高精確性擴增
五、利用濃度不對稱的引物制備DNA單鏈
基因組DNA分析經常用到southern印跡,依賴單鏈DNA探針與固定的DNA雜交。為了制備擴增特異長度的單鏈DNA,我們采用不同濃度的引物。高濃度引物的量是低濃度引物的50~100倍。在一個PCR體系中,低濃度引物被快速消耗完后,高濃度引物主導之后的PCR進程,從而產生大量單鏈DNA。
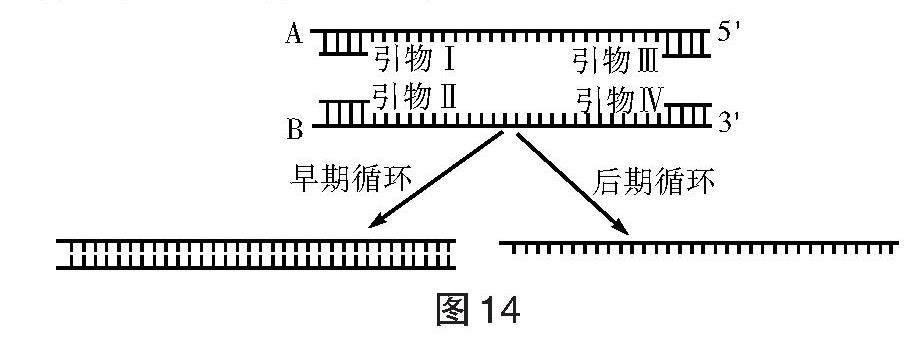
[例6]DNA分子雜交時,常需要大量的單鏈DNA探針。不對稱PCR是一種常見的單鏈DNA探針制備方法,其原理是反應體系中加入一對數量不相等的引物。在PCR反應的早期10~15個循環中,擴增產物主要是雙鏈DNA,當后期數量較少的限制性引物耗盡后,數量較多的非限制性引物將引導合成大量目標DNA單鏈。
圖14
若圖14中A鏈為所需的DNA分子探針,則非限制性引物應選用引物???????????????? 。
解析:若選擇引物Ⅱ、Ⅲ,限于子鏈延伸方向向外,擴增的是無效片段。引物Ⅳ與B鏈互補,A鏈與B鏈互補,故引物Ⅳ是合成大量A鏈所必不可少的一部分。為合成大量A鏈,就需要大量引物Ⅳ作為非限制性引物。通過最初10~15個循環,獲得大量模板B鏈?。后15次循環只以大量B鏈為模板產生大量A鏈作探針。單鏈的分子量較小,通過電泳可以將這些單鏈DNA探針分離出來。
參考答案:Ⅳ
六、檢測特定基因的存在或缺失的多重PCR
一個抗原可能具備多個抗原決定簇,對應多個基因位點。有時需要檢測一份樣本中是否含有多種病原體,或者對具有多個位點的病毒進行探究,如SARS-CoV-2的檢測診斷至少需要兩個基因靶標[4]。這種情況下,為節約人力物力,并且高效快速檢測,可以在同一反應體系中加入兩對以上的引物。這種方式稱為多重PCR,其優點是可以通過一次PCR同時對多個目的序列進行擴增,還可以檢測特定基因序列的存在或缺失。
[例7]人類γ基因啟動子上游的調控序列中含有BCL11A蛋白結合位點,該位點結合BCL11A蛋白后,γ基因的表達被抑制。通過改變該結合位點的序列,解除對γ基因表達的抑制,可對某種地中海貧血癥進行基因治療。科研人員擴增了γ基因上游不同長度的片段,將這些片段分別插入表達載體中進行轉化和熒光檢測,以確定BCL11A蛋白結合位點的具體位置。F1~F7,R均為引物。相關信息如圖15所示。
圖15
含F1~F4與R擴增產物的受體細胞不再有熒光,而含F5~F7與R擴增產物的受體細胞仍有熒光。若γ基因上游調控序列上與引物序列所對應的位置不含有BCL11A蛋白的結合位點序列,據此結果可推測,BCL11A蛋白結合位點位于???????????? 。
解析:熒光蛋白終止子在DNA左側,為使熒光蛋白基因表達,應將調控序列及啟動子反向連接在熒光蛋白基因與啟動子P之間。參考圖16。
圖16
若擴增產物含有BCL11A蛋白結合位點,該位點與BCL11A蛋白的結合將抑制熒光蛋白的表達,進而使得受體細胞不能發出熒光。使用F5、R擴增的片段仍有熒光,因此無結合位點。使用F4、R擴增的片段熒光消失,因此結合位點在F4~F5對應序列之間的區段。
參考答案:F4~F5對應序列之間的區段
[?? 參?? 考?? 文?? 獻?? ]
[1]? 中華人民共和國教育部.普通高中生物學課程標準:2017年版2020年修訂[M].北京:人民教育出版社,2020.
[2]? 屈伸,劉志國.分子生物學實驗技術[M].北京:化學工業出版社,2008.
[3]? 任桂杰,王志玉.PCR介導的定點突變與隨機突變的應用[J].山東大學學報(醫學版),2005(9):865-867.
[4]? 李慶超,房學迅,徐小潔.多重PCR檢測新型冠狀病毒核酸的研究進展[J].軍事醫學,2022(10):798-801.
(責任編輯 羅 艷)

