多基因編輯技術的發展及其在畜牧種質創新中的應用
摘 要: CRISPR基因編輯技術可以更精準、高效地更改基因組DNA序列,近年來在動植物育種中被廣泛應用。實踐中對農業生物多性狀協同改良的需求越來越大,僅靠對單一基因或位點的改變不能滿足上述需求,所以亟需建立一套多基因同步編輯體系,對多個基因進行協同修飾。多個sgRNA同時表達是多基因同步編輯的關鍵,常見表達策略包括建立多個單順反子sgRNA并聯表達和多順反子sgRNA串聯表達。常用串聯表達工具有核酸酶Csy4、tRNA系統以及自裂核酶等。本文對以上多基因編輯技術的優劣進行了分析和總結,探討了下一步的發展方向,并指出了它的其重要意義和應用前景。
關鍵詞: CRISPR;多基因編輯;種質創新
中圖分類號:S813.2
文獻標志碼:A
文章編號:0366-6964(2024)08-3267-09
收稿日期:2024-01-08
基金項目:豬無痕跡基因編輯技術(NK2022010207);湖北省中央引導地方科技發展資金項目(2022BGE231);科技援疆項目(2022E02138)
作者簡介:劉雯雯(1998-),女,河南鄭州人,碩士,主要從事臨床獸醫研究,E-mail:1512513865@qq.com
通信作者:董發明,主要從事臨床獸醫研究,E-mail:756146646@qq.com;畢延震,主要從事畜禽生物育種研究,E-mail:sukerbyz@126.com
The Development of Multi-Gene Editing Technology and Its Application in Agricultural
Biological Germplasm Innovation
LIU Wenwen1, 2, DONG Faming1*, BI Yanzhen2, 3*
(1.College of Animal Science and Technology, Henan University of Science and Technology, Luoyang
471023," China;
2.Hubei Provincial Key Laboratory of Animal Embryo Engineering and Molecular Breeding,
Institute of Animal Husbandry and Veterinary Medicine, Hubei Academy of Agricultural Sciences, Wuhan
430064," China;
3.Hubei Hongshan Laboratory, Wuhan 430070," China)
Abstract:" CRISPR gene editing technology can accurately and efficiently change the DNA source code and has been widely used in animal and plant breeding in recent years.In practice, the demand for multi-trait parallel improvement of agricultural organisms is increasing. The change of a single gene or locus alone cannot meet the above requirements.Therefore,it is desirable to establish a multi-gene simultaneous editing system to modify multiple genes. Simultaneous expression of multiple sgRNAs is critical to simultaneous editing of multiple genes. The commonly multi-sgRNA expression strategies include multiple single-cistron sgRNA parallel expression and multi-cistron sgRNA tandem expression. The frequently-commonly used tandem expression tools include nuclease Csy4, tRNA system and self-cleaving ribozyme. This review analyzes and summarizes the advantages and disadvantages of the above multi-gene editing technology, discusses the future direction, and stresses its significance and application prospects.
Key words: CRISPR; multi-gene editing; germplasm innovation
*Corresponding authors:DONG Faming, E-mail:756146646@qq.com ; BI Yanzhen, E-mail:sukerbyz@126.com
1 多基因編輯的意義
基因編輯是一種通過DNA序列敲除、插入和堿基編輯等手段來改變生物體特定基因組DNA從而改變其特性和功能的基因工程活動[1]。主要基因編輯技術包括鋅指核酸酶(zinc finger nucleases,ZFNs)[2]、轉錄激活因子樣效應核酸酶(transcription activator-like effector nucleases TALENs)[3-4]和成簇的規律間隔短回文重復序列(clustered regularly spaced short palindromic repeats, CRISPR)/Cas9基因編輯技術[5]。其中應用最廣泛的是CRISPR/Cas9基因編輯技術[6],CRISPR/Cas9基因編輯技術通過一條單一向導與靶序列互補配對的RNA(single guide RNA,sgRNA)定位編輯位點,進而引導Cas9蛋白對目標靶序列進行切割,具有成本低廉、操作簡便和精確高效的特點,且相較另外兩種基因編輯方法克服了試驗周期長和應用范圍小的問題[7]。在過去的三十年中,基因編輯技術取得了廣為人知的巨大進步,除了應用于生命科學基礎研究和臨床上應用外,它在畜牧業中的應用也越來越廣泛[8]。
基因編輯技術發展迅速,但是這種技術仍局限于編輯單個基因、改良單一性狀,已經無法滿足現階段的育種需求。除此之外在育種工作中,一個性狀可能由多個基因控制,僅對單個基因編輯很難達到改變性狀的目的。傳統育種方法生產具備多種優良性狀的農業生物,往往需要經過反復克隆和篩選[9-11],該方法步驟繁復、培育周期長,生產效率低[12]。為擺脫這一困境,建立一套簡單高效的多基因編輯體系,利用基因編輯技術對多位點同時進行編輯或轉錄調控成為大勢所趨,對未來農業、畜牧業種質改良具有重要意義。
2 多基因編輯技術的類型
多基因編輯技術需要一起同時引入兩個及以上sgRNA[13],Cas9蛋白在多個sgRNA引導下,可以有效地識別多個靶位點[14],只需要改變20 bp的序列就能改變Cas9的靶向特異性[15]。目前,不同研究者基于高效釋放多個sgRNA開發了不同的方法[16]。較為常用的方法主要分為兩類:一是建立含有多個單順反子sgRNA表達盒,所有的sgRNA被特定啟動子轉錄,經過加工被釋放,同時對多個基因進行修飾[17],常見的方法有串聯多個重復的PolⅢ型啟動子啟動的sgRNA表達框[18];二是構建多順反子sgRNA 表達盒,在所有sgRNA兩側安裝可切割的位點,如核酸酶Csy4介導的多順反子sgRNA加工成熟[19]、基于內源性轉運RNA(tRNA)多順反子sgRNA加工成熟[20] 以及利用自裂核酶介導的多順反子sgRNA加工成熟。經過加工或者切割釋放多個sgRNA,識別不同的靶位點,從而實現多個基因的同步編輯[21]。
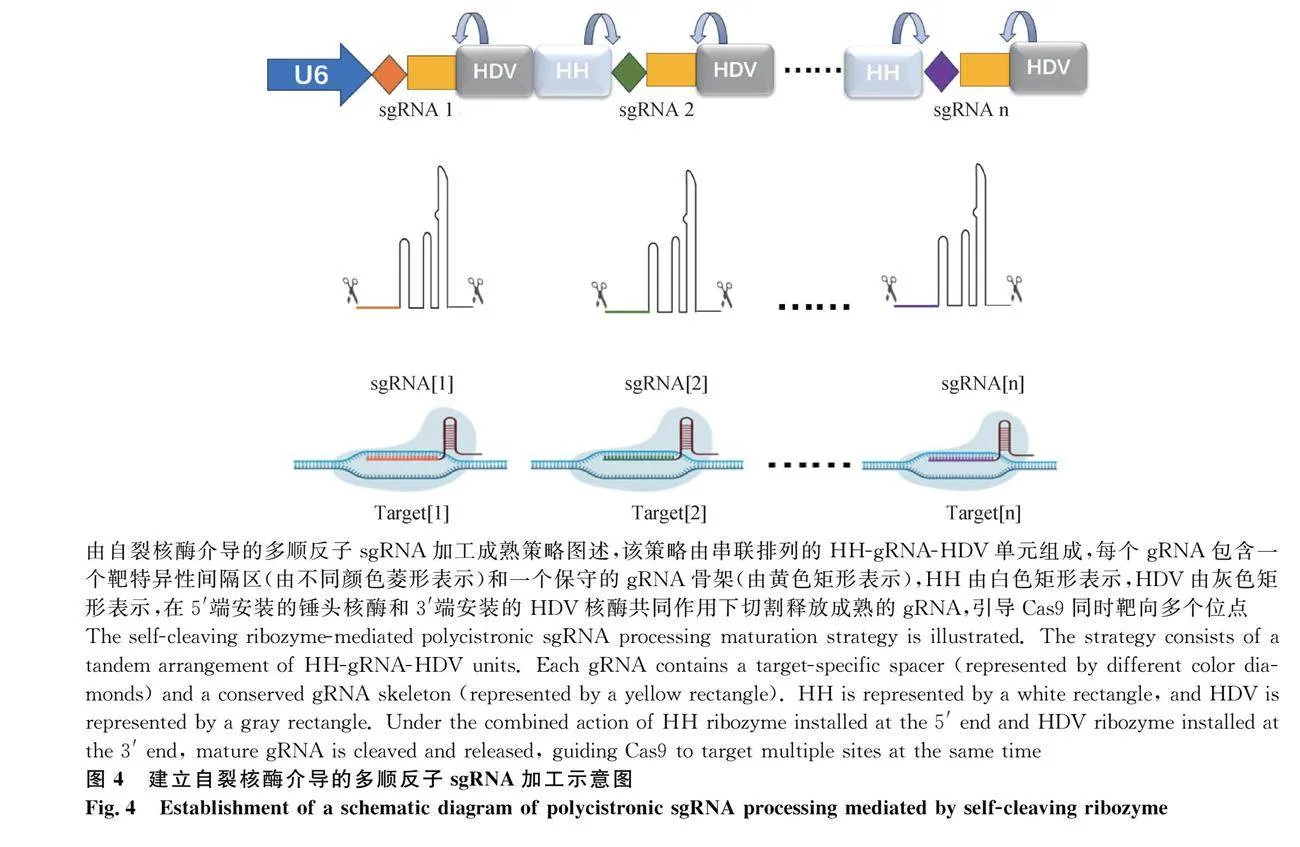
2.1 建立多個單順反子sgRNA表達盒
早期對于多重sgRNA的表達是通過多個單獨的sgRNA表達盒產生,每個盒都由單獨的RNA 聚合酶Ⅲ(PolⅢ)啟動子轉錄[21]。常見為U3和U6基因啟動子。這些PolⅢ啟動子轉錄起始于特定核苷酸。如U3啟動子從“A”開始轉錄,U6啟動子從“G”開始轉錄[22],轉錄的 RNA 會在細胞核內保留。一個sgRNA盒由PolⅢ啟動子、sgRNA和PolⅢ終止子組成,大小為300~500 bp,以啟動子-sgRNA-終止子為最小重復單元[23](圖1)。但是,該系統的設計必須分別組裝啟動子、終止子、sgRNA骨架(sgRNA scaffold),采用同源重組或Golden Gate等方式組裝。有研究人員在擬南芥中使用3種不同的PolⅢ啟動子成功表達6個sgRNA,同時靶向脫落酸受體PYL蛋白家族中的6個基因,在T3后代中鑒定出純合六元突變體[24];利用該系統同時靶向豬CD163和肌肉生長抑制素(MSTN)基因,成功培育出瘦肉率高且對藍耳病不易感的仔豬[25]。但多個PolⅢ啟動子可能會產生啟動子串聯干擾效應的“不良反應”,兩個近端排列的轉錄單元可能會因為啟動子串聯影響彼此的轉錄活性[26-27]。除此之外,由于重復序列和較大片段合成比較繁瑣,構建成功并不容易,需要更換靶序列時又必須重復上述過程,不僅繁瑣更增加了時間成本[28]。
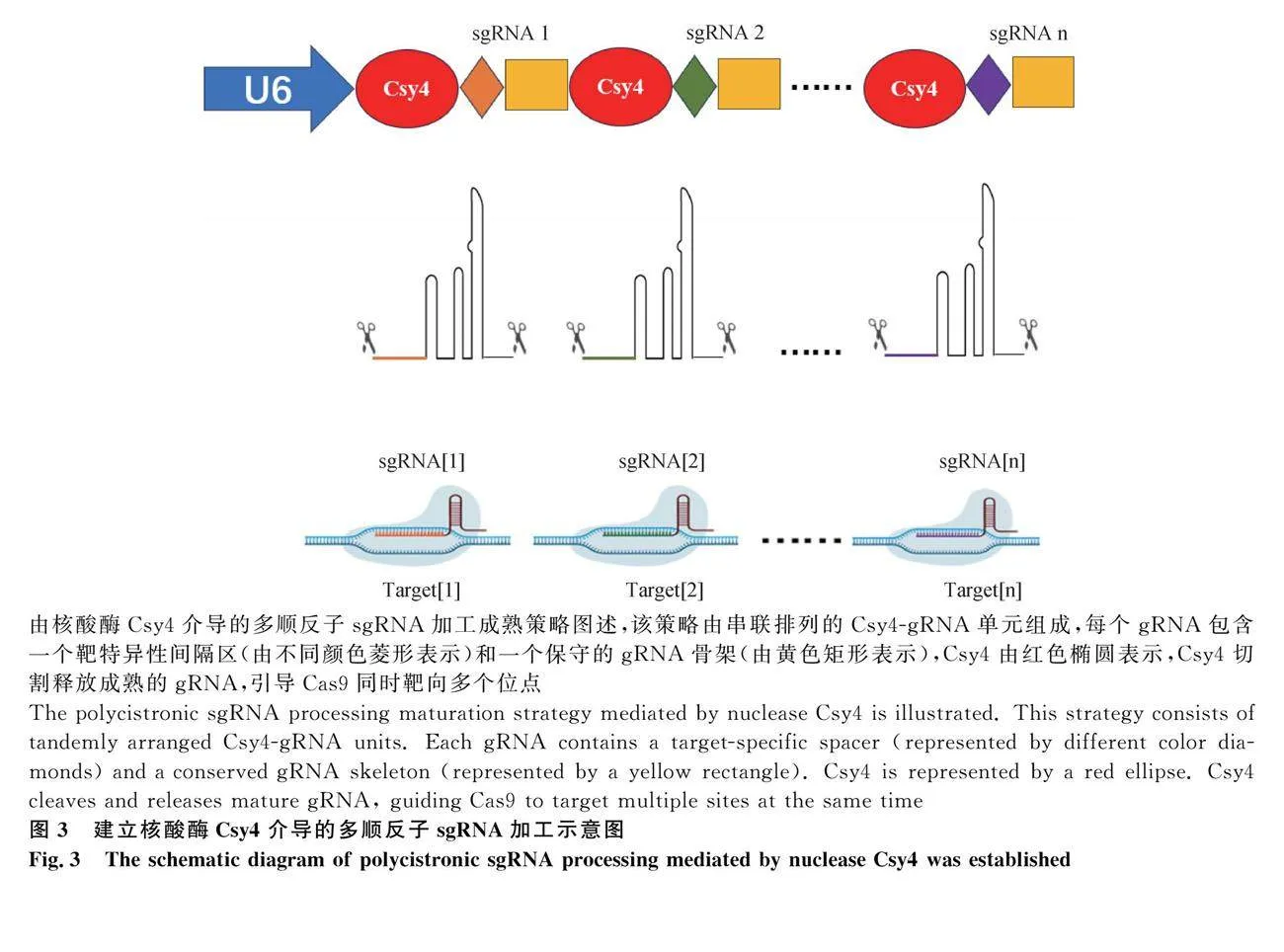
2.2 tRNA介導的多順反子sgRNA加工成熟
tRNA是在蛋白質合成期間將氨基酸運送到核糖體的載體,存在于所有生物體內[29]。前體tRNA 5′端和3′端序列能夠被細胞內源性RNaseP和RNaseZ酶特異性識別并切割[13],釋放每個獨立的tRNA和成熟的sgRNA[30],引導Cas9蛋白切割靶位點。人工多順反子-tRNA-gRNA(PTG策略)基因串聯表達系統以tRNA-sgRNA-sgRNA骨架(sgRNA scaffold)為最小重復單元,PTG策略盒串聯兩個及以上重復單元,僅使用一個PolⅢ啟動子就能高效表達多個sgRNA[31],提高了Cas9蛋白的多重靶向能力(圖2)。只需更換20 bp的sgRNA序列,就能改變Cas9蛋白的特異靶向性,很大程度地簡化了質粒的構建。PTG策略不需要引入外源蛋白,重復單元序列相對較短,降低了合成的難度,在簡化質粒結構的同時,實現了對質粒快速高效的構建。此外,tRNA可作為增強子并提高PolⅢ轉錄效率,是該系統的另一個優勢,目前已在一些真核生物中廣泛應用[32-33]。在植物育種領域中,該方法首先被應用于水稻的基因組多位點編輯以及染色體片段刪除[34],編輯效率可高達100%。通過設計多個tRNA-sgRNA單元用于多基因編輯,把tRNA系統首次應用于玉米,有效準確地缺失了染色體片段[33]。盡管tRNA是多基因組編輯的最佳工具,但存在一定的局限性,該系統不能很好地處理數十個以上的sgRNA,當sgRNA種類過多時彼此之間相互競爭導致編輯效率低下[35]。
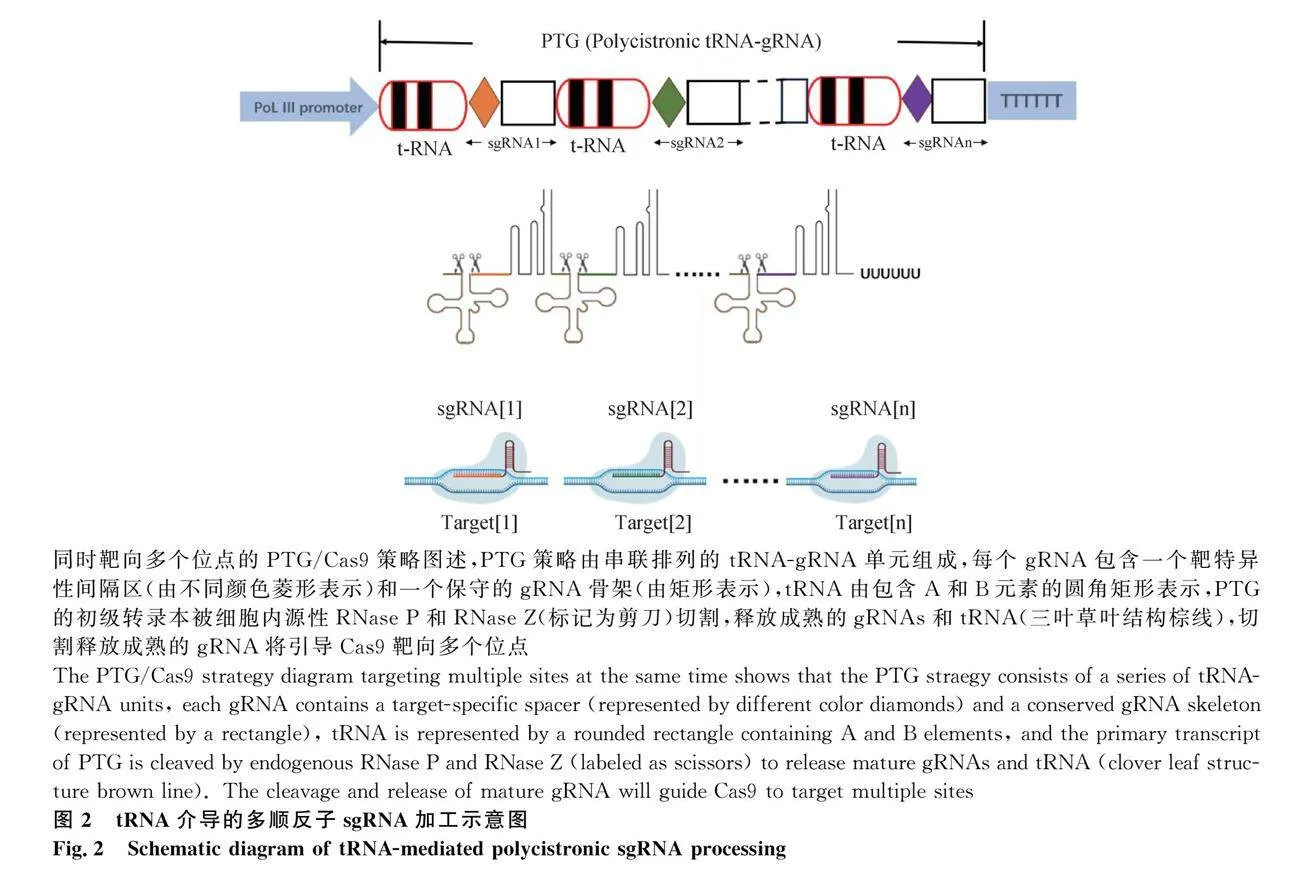
2.3 核酸酶Csy4介導的多順反子sgRNA加工成熟
Csy4是來自銅綠假單胞菌中用于 crRNA 加工的一種蛋白,是特異性核糖核酸內切酶(endo-RNase )。Csy4對發夾RNA有很高的親和力[36],用于可切割帶有發夾序列的RNA,已被用于在各種生物體中被用于產生多個sgRNA。基于Csy4的特性,有研究者制作了人工sgRNA陣列,每個sgRNA兩側安裝有Csy4切除位點[37],構建具有串聯結構的Cys4-processed-sgRNAs系統,Csy4可以對其中的每個重復序列進行切割[38],Csy4介導的gRNAs陣列能夠同時高效釋放大量的sgRNA[39](圖3)。該系統最早是在酵母中發展起來的,在釀酒酵母測試菌落中進行了四重缺失,Csy4系統效率可達96%,并成功有效地轉錄調控了3個基因[40];在番茄中應用Csy4系統缺失4個基因、使用多達12個sgRNA同時在雙子葉植物和單子葉植物中創建多重靶向基因缺失,并監測sgRNA在陣列中位置的影響,證實了Csy4可以快速有效地進行多重基因組編輯[41]。但該系統的缺點是需要轉化和表達外源Csy4核糖核酸內切酶,且Csy4具有細胞毒性,會對生物體造成危害,所以該策略適用于基礎研究,不適合農業生物育種。
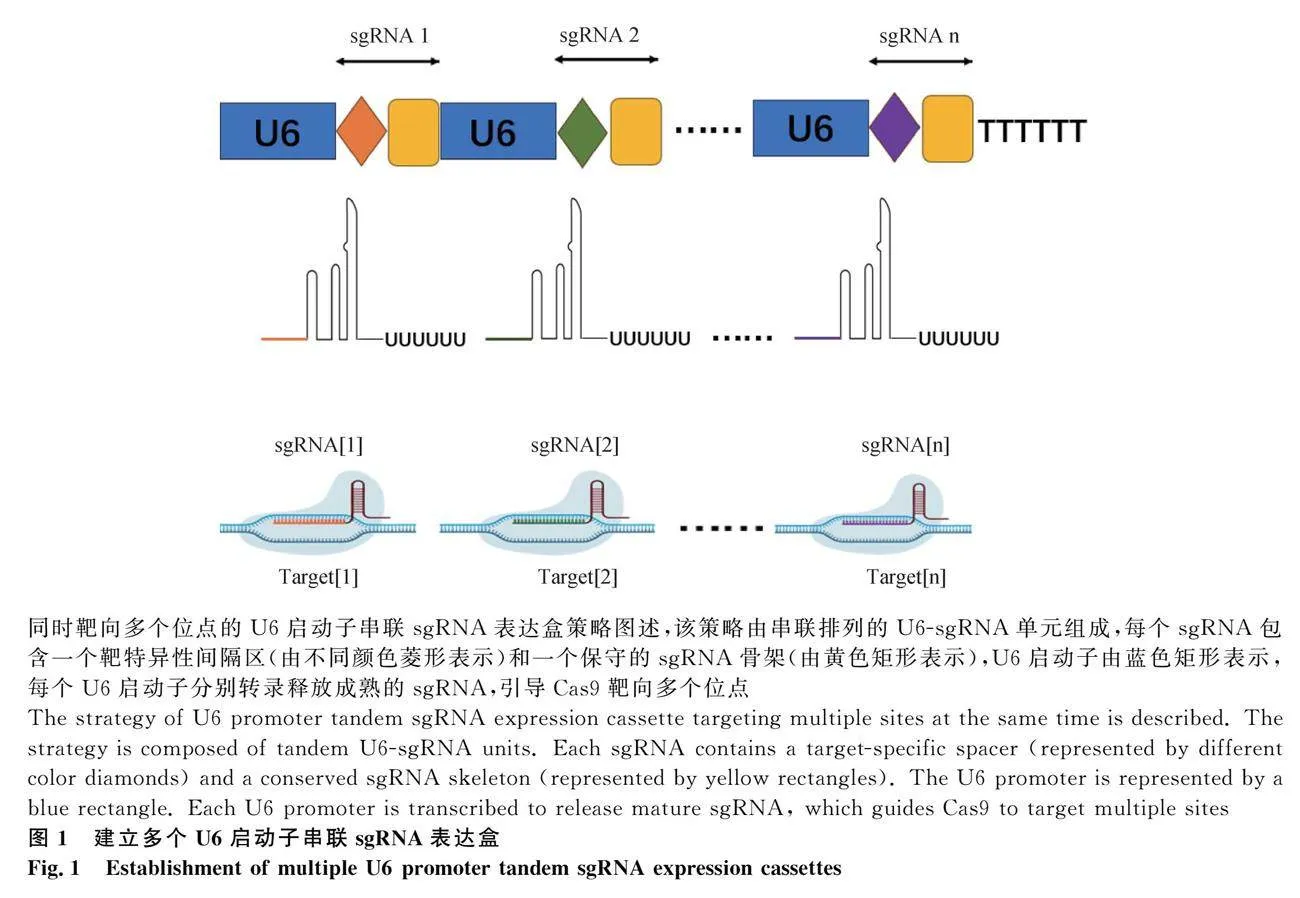
2.4 利用自裂核酶介導的多順反子sgRNA加工成熟
錘頭(hammerhead ribozyme,HH)核酶是一種存在于小型植物致病RNA中,在類病毒基因組復制過程中將多聚體轉錄物加工成單體的自裂解的RNA結構,其體積小,只有約50個核苷酸[42]。它可以作為催化劑,通過轉脂反應剪切自身磷酸二脂鍵,將裂解位點活性中心與其余部分分離。因此,錘頭核酶可以被用于在體內切割特定的病毒和細胞序列。丁型肝炎病毒(hepatitis D virus, HDV)核酶是一種RNA衛星病毒,與目標序列不會形成二級結構,一般安裝在目標序列的3′端。這種利用具有自裂解功能的核酶進行多重基因編輯是在酵母中最先被發展起來的一種方法[43],在sgRNA兩側安裝具有核酶活性的RNA序列,在5′端安裝錘頭(hammer head, HH)核酶,3′端安裝HDV核酶,這種方法不僅能釋放正確的sgRNA,也不會影響蛋白質的表達(圖4)。這種方法已經在多種生物中被證明[44],在人類細胞中實現通過RNAP II啟動子成功釋放sgRNA進行基因調控,且沒有產生三重序列,并能通過殘留的3′核糖酶序列保護目的基因轉錄物[45] 。在多諾瓦尼利什曼原蟲中利用錘頭(HH核酶和HDV核酶,開發了一種雙sgRNA表達載體精準缺失3 kb基因組片段,更清晰地了解了利什曼原蟲感染和發病機制[46]。該系統相較于Csy4系統無需額外表達外源Csy4核糖核酸內切酶,但目前仍未見在動植物上利用自剪切裂解功能表達兩個及以上sgRNA的報道。
3 多基因編輯技術在農業生物育種中的應用
基因編輯技術已成為農業生物種質改良的重要工具,合理運用該技術可以實現同時對多個基因進行修飾,改良多個基因控制的重要經濟性狀,同時也能實現對多種農業動物傳染病的抵抗和預防作用。
隨著基因編輯技術的發展,sgRNA引導Cas9蛋白最早成功應用于植物基因編輯中成功應用,目前該技術已被應用于誘導多種植物物種基因組突變。如在玉米中設計5條sgRNA同時靶向4個基因獲得了高達23.1%的靶向效率[47];在棉花中以外源性基因DsRed2和內源性基因GhCLA1作為靶標,成功利用CRISPR/Cas9系統在同種異體四倍體棉花中完成了多位點基因組編輯[48];利用馬鈴薯病毒X構建了一種在成年茄科植物中表達多種sgRNA的載體,有效地靶向本氏煙草基因,產生了近80%的插入缺失[49];在小麥中利用3種工具包括自裂核酶、tRNA以及TRSP系統(tandem repeats of separate U3 or U6 promoters)在內的3種工具靶向小麥8個靶位點同時進行基因組編輯,結果證實tRNA和核酶系統在多重基因組編輯中更有效[47]。
禽白血病是造成養禽業重大經濟損失的重要原因之一,但缺乏有效的防治手段。它是由禽白血病病毒(ALV)引起的在禽類中垂直傳播的腫瘤疾病,發病率、死亡率高,由于沒有特效藥,只能通過病雞淘汰達到種群凈化的目的[50]。禽類對ALV的抵抗力由TVA、TVB、TVC三個位點決定,TVB 基因上TVBSI和TVBS3等位基因決定著禽類禽白血病病毒ALV的B、D和E亞型的易感性,在TVB基因第二、第三外顯子分別設計sgRNA,利用t-RNA構建多順反子表達盒,成功對TVB基因造成大片段刪除,為抗禽白血病奠定了基礎[51]。
我國是全世界畜禽養殖規模最大的國家。豬病頻發是我國養豬業目前面臨的最大挑戰,造成了巨大的經濟損失[52]。通過多基因編輯可獲得抗三種重大疫病的豬[53]。豬繁殖與呼吸綜合征是我國重點防治的一類傳染病,又名藍耳病,由豬繁殖與呼吸綜合征病毒(PRRSV)引起的妊娠母豬流產、仔豬和育肥豬肺炎的急性傳染病。CD163分子是PRRSV生命周期中最重要和最關鍵的分子[54],它由9個富半胱氨酸清道夫受體(SRCR 1~9)組成[55],蛋白結構域SRCR 5(對應第七外顯子)是PRRSV感染所必需的[56]。通過基因編輯敲除第七外顯子所包含的功能域,能有效地抵御對PRRSV的感染[57-59]。傳染性胃腸炎病毒(TGEV)和豬德爾塔冠狀病毒(PDCoV)會導致仔豬致命性腹瀉[60]。豬氨基肽酶(pAPN)是TGEV感染的主要受體[61-62]。病毒糖蛋白與小腸上皮細胞表面pAPN受體結合并介導膜融合,使得病毒進入上皮細胞[62]。對pAPN基因第二外顯子進行編輯,使病毒受體蛋白失活可以顯著抵抗TGEV感染[63],同時對PDCoV的易感性降低。通過多基因編輯技術對豬CD163基因第七外顯子和pAPN基因第二外顯子進行同步編輯,獲得能同時抵御繁殖與呼吸綜合征、傳染性胃腸炎和豬德爾塔冠狀病毒侵襲的綜合性能優良的種豬。
豬肉是人類重要的動物蛋白質來源,國內豬地方品種多、肉質好但瘦肉率較低,隨著人們營養需求的變化,目前人們更偏向于低脂、高蛋白的畜產品。培育具有瘦肉率高且抗病等多種優良性狀的豬既是當務之急也是長久之計。有學者采用三種sgRNA表達策略成功培育出對CD163和MSTN同步編輯的豬[25]。MSTN是由動物肌細胞產生的一種蛋白,對肌細胞的生長能產生抑制作用,它不僅抑制肌肉細胞的增殖和分化,而且決定了肌肉纖維的數量,是肌肉生長速度和瘦肉率的負調節因子[64]。在MSTN信號肽區域引入突變,可顯著下調MSTN基因的表達,增加肌纖維數量,促進肌肉生長[65]。敲除豬肌肉抑制因子MSTN基因和CD163基因,獲得表現出更高瘦肉率且對PRRSV不易感的優質豬種,更有助于提高豬的抗病、屠宰和繁殖等性能。
多基因同步編輯是從遺傳角度利用CRISPR基因編輯技術研究培育新品種的關鍵突破口,該技術可以同時對多個基因進行精準修飾。但是CRISPR/Cas9介導的多基因編輯技術存在一個亟待解決的問題:串聯的sgRNA數量越多敲除效率越低,是因為當sgRNA種類過多,會競爭細胞內有限的核酸酶,這種效應導致改變這一現象使得原本sgRNA的編輯效率被改變。這個問題對于獲得多種優質性狀的種質資源是個嚴峻的挑戰,通過改進基因編輯工具或研究編輯策略或許能給這個問題帶來新的解決思路,這對未來農業、畜牧業生產領域具有重要意義。
參考文獻(References):
[1] VAN DER OOST J,PATINIOS C.The genome editing revolution[J].Trends Biotechnol,2023,41(3):396-409.
[2] KIM Y G,CHA J,CHANDRASEGARAN S.Hybrid restriction enzymes:zinc finger fusions to Fok I cleavage domain[J].Proc Natl Acad Sci U S A,1996,93(3):1156-1160.
[3] CHRISTIAN M,CERMAK T,DOYLE E L,et al.Targeting DNA double-strand breaks with TAL effector nucleases[J]. Genetics, 2010, 186(2):757-761.
[4] MILLER J C,TAN S Y,QIAO G J,et al.A TALE nuclease architecture for efficient genome editing[J].Nat Biotechnol,2011, 29(2):143-148.
[5] CONG L,RAN F A,COX D,et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science, 2013, 339(6121):819-823.
[6] DONOHOUE P D,BARRANGOU R,MAY A P.Advances in industrial biotechnology using CRISPR-cas systems[J].Trends Biotechnol,2018,36(2):134-146.
[7] 張佳珊,譚 韜.CRISPR-Cas9系統編輯DNA誘導基因敲除的發展及優缺點[J].中國免疫學雜志,2019,35(6):767-770.
ZHANG J S,TAN T.Development of CRISPR-Cas9 system edit DNA and induce targeted knockout as well advantages and disadvantages[J].Chinese Journal of Immunology,2019,35(6):767-770.(in Chinese)
[8] 于海穎,路永強,張 魯,等.CRISPR/Cas9系統在基因編輯豬生產中的應用[J].黑龍江動物繁殖,2022,30(1):34-40.
YU H Y,LU Y Q,ZHANG L,et al.Application of the CRISPR/Cas9 system to produce gene-edited pigs[J].Heilongjiang Journal of Animal Reproduction,2022,30(1):34-40.(in Chinese)
[9] MIYAGAWA S,MATSUNARI H,WATANABE M,et al.Generation of α1,3-galactosyltransferase and cytidine monophospho-N-acetylneuraminic acid hydroxylase gene double-knockout pigs[J].J Reprod Dev,2015,61(5):449-457.
[10] RICHTER A,KUROME M,KESSLER B,et al.Potential of primary kidney cells for somatic cell nuclear transfer mediated transgenesis in pig[J].BMC Biotechnol,2012,12:84.
[11] SUZUKI S,IWAMOTO M,SAITO Y,et al.Il2rg gene-targeted severe combined immunodeficiency pigs[J].Cell Stem Cell,2012,10(6):753-758.
[12] GUO X C,GENG L S,JIANG C Q,et al.Multiplexed genome engineering for porcine fetal fibroblasts with gRNA-tRNA arrays based on CRISPR/Cas9[J].Anim Biotechnol,2023,34(9):4703-4712.
[13] DONG F P,XIE K B,CHEN Y Y,et al.Polycistronic tRNA and CRISPR guide-RNA enables highly efficient multiplexed genome engineering in human cells[J].Biochem Biophys Res Commun,2017,482(4):889-895.
[14] 樊祥瑞,王俊燕,梁麗亞,等.基于CRISPR/Cas系統的多重基因編輯與調控技術[J].生物工程學報,2023,39(6):2449-2464.
FAN X R,WANG J Y,LIANG L Y,et al.Multiplex gene editing and regulation techniques based on CRISPR/Cas system[J].Chinese Journal of Biotechnology,2023,39(6):2449-2464.(in Chinese)
[15] 郎 楠,梁洛瑜,汪軍麗,等.CRISPR-Cas9多基因編輯技術在植物研究中的應用[J].分子植物育種,2023,21(8):2665-2670.
LANG N,LIANG L Y,WANG J L,et al.Application of CRISPR-Cas9 enabled multiplex gene editing in plant research[J]. Molecular Plant Breeding,2023,21(8):2665-2670.(in Chinese)
[16] FENG X,ZHAO D D,ZHANG X L,et al.CRISPR/Cas9 assisted multiplex genome editing technique in Escherichia coli[J].Biotechnol J,2018,13(9):e1700604.
[17] GUO X C,GENG L S,JIANG C Q,et al.Multiplexed genome engineering for porcine fetal fibroblasts with gRNA-tRNA arrays based on CRISPR/Cas9[J].Anim Biotechnol,2023,34(9):4703-4712.
[18] SAKUMA T,NISHIKAWA A,KUME S,et al.Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system[J].Sci Rep,2014,4:5400.
[19] ALOK A,CHAUHAN H,UPADHYAY S K,et al.Compendium of plant-specific CRISPR vectors and their technical advantages[J].Life (Basel),2021,11(10):1021.
[20] 王秉政,張 超,張佳麗,等.利用單轉錄本表達Cas9和sgRNA條件性編輯果蠅基因組[J].遺傳,2023,45(7):593-601.
WANG B Z,ZHANG C,ZHANG J L,et al.Conditional editing of the Drosophila melanogaster genome using single transcripts expressing Cas9 and sgRNA[J].Hereditas (Beijing),2023,45(7):593-601.(in Chinese)
[21] 徐 磊,趙育蓉,胡悅旻,等.基于CRISPR/Cas9系統的多基因敲除載體的構建及其敲除效率檢測[J].農業生物技術學報,2022,30(5):1023-1030.
XU L,ZHAO Y R,HU Y M,et al.Construction and knockout efficiency detection of multiple knockout vector based on the CRISPR/Cas9 system[J].Journal of Agricultural Biotechnology,2022,30(5):1023-1030.(in Chinese)
[22] KOR S D,CHOWDHURY N,KEOT A K,et al.RNA PolⅢ promoters-key players in precisely targeted plant genome editing[J].Front Genet,2023,13:989199.
[23] REIS A C,HALPER S M,VEZEAU G E,et al.Simultaneous repression of multiple bacterial genes using nonrepetitive extra-long sgRNA arrays[J].Nat Biotechnol,2019,37(11):1294-1301.
[24] ZHANG Z J,MAO Y F,HA S,et al.A multiplex CRISPR/Cas9 platform for fast and efficient editing of multiple genes in Arabidopsis[J].Plant Cell Rep,2016,35(7):1519-1533.
[25] ZHANG J Q,GUO J X,WU X J,et al.Optimization of sgRNA expression strategy to generate multiplex gene-edited pigs[J].Zool Res,2022,43(6):1005-1008.
[26] XING H L,DONG L,WANG Z P,et al.A CRISPR/Cas9 toolkit for multiplex genome editing in plants[J].BMC Plant Biol,2014,14:327.
[27] NIE L H,DAS THAKUR M,WANG Y M,et al.Regulation of U6 promoter activity by transcriptional interference in viral vector-based RNAi[J].Genomics Proteomics Bioinformatics,2010,8(3):170-179.
[28] 盧 揮,張 啟,于思禮,等.谷氨酸棒桿菌中基于CRISPR/Cas9的多位點堿基編輯系統的優化[J].生物工程學報,2022,38(2):780-795.
LU H,ZHANG Q,YU S L,et al.Optimization of CRISPR/Cas9-based multiplex base editing in Corynebacterium glutamicum[J]. Chinese Journal of Biotechnology,2022,38(2):780-795.
[29] GU H Q,LIAN B,YUAN Y X,et al.A 5′ tRNA-Ala-derived small RNA regulates anti-fungal defense in plants[J].Sci China Life Sci,2022,65(1):1-15.
[30] SINGH J,SHARMA D,BRAR G S,et al.CRISPR/Cas tool designs for multiplex genome editing and its applications in developing biotic and abiotic stress-resistant crop plants[J].Mol Biol Rep,2022,49(12):11443-11467.
[31] ZALATAN J G,LEE M E,ALMEIDA R,et al.Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds[J].Cell,2015,160(1-2):339-350.
[32] PORT F,BULLOCK S L.Augmenting CRISPR applications in Drosophila with tRNA-flanked sgRNAs[J].Nat Methods, 2016, 13(10):852-854.
[33] QI W W,ZHU T,TIAN Z R,et al.High-efficiency CRISPR/Cas9 multiplex gene editing using the glycine tRNA-processing system-based strategy in maize[J].BMC Biotechnol,2016,16(1):58.
[34] MINKENBERG B,XIE K B,YANG Y N.Discovery of rice essential genes by characterizing a CRISPR-edited mutation of closely related rice MAP kinase genes[J].Plant J,2017,89(3):636-648.
[35] XIE K B,MINKENBERG B,YANG Y N.Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system[J].Proc Natl Acad Sci U S A,2015,112(11):3570-3575.
[36] HUANG S S,ZHANG Z W,TAO W Y,et al.Broadening prime editing toolkits using RNA-Pol-II-driven engineered pegRNA[J].Mol Ther,2022,30(9):2923-2932.
[37] MUSHTAQ M,AHMAD DAR A,SKALICKY M,et al.CRISPR-based genome editing tools:insights into technological breakthroughs and future challenges[J].Genes (Basel),2021,12(6):797.
[38] KISHIMOTO T,NISHIMURA K,MORISHITA K,et al.An engineered ligand-responsive Csy4 endoribonuclease controls transgene expression from Sendai virus vectors[J].J Biol Eng,2024,18(1):9.
[39] HAURWITZ R E,STERNBERG S H,DOUDNA J A.Csy4 relies on an unusual catalytic dyad to position and cleave CRISPR RNA[J].EMBO J,2012,31(12):2824-2832.
[40] FERREIRA R,SKREKAS C,NIELSEN J,et al.Multiplexed CRISPR/Cas9 genome editing and gene regulation using csy4 in Saccharomyces cerevisiae[J].ACS Synth Biol,2018,7(1):10-15.
[41] CACˇGERMáK T,CURTIN S J,GIL-HUMANES J,et al.A multipurpose toolkit to enable advanced genome engineering in plants[J].Plant Cell,2017,29(6):1196-1217.
[42] DOUDNA J A.Ribozymes:the hammerhead swings into action[J].Curr Biol,1998,8(14):R495-R497.
[43] GAO Y B,ZHAO Y D.Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing[J].J Integr Plant Biol,2014,56(4):343-349.
[44] DEANER M,MEJIA J,ALPER H S.Enabling graded and large-scale multiplex of desired genes using a dual-mode dCas9 activator in Saccharomyces cerevisiae[J].ACS Synth Biol,2017,6(10):1931-1943.
[45] NISSIM L,PERLI S D,FRIDKIN A,et al.Multiplexed and programmable regulation of gene networks with an integrated RNA and CRISPR/Cas toolkit in human cells[J].Mol Cell,2014,54(4):698-710.
[46] ZHANG W W,MATLASHEWSKI G.CRISPR-Cas9-mediated genome editing in Leishmania donovani[J].mBio,2015, 6(4):e00861.
[47] LI J H,ZHANG S J,ZHANG R Z,et al.Efficient multiplex genome editing by CRISPR/Cas9 in common wheat[J].Plant Biotechnol J,2021,19(3):427-429.
[48] WANG P C,ZHANG J,SUN L,et al.High efficient multisites genome editing in allotetraploid cotton (Gossypium hirsutum) using CRISPR/Cas9 system[J].Plant Biotechnol J,2018,16(1):137-150.
[49] URANGA M,ARAGONéS V,SELMA S,et al.Efficient Cas9 multiplex editing using unspaced sgRNA arrays engineering in a Potato virus X vector[J].Plant J,2021,106(2):555-565.
[50] 魏 杰,劉勝宇.禽白血病凈化防控技術探討[J].山東畜牧獸醫,2023,44(11):47-49.
WEI J,LIU S Y.Discussion on purification and prevention technology of avian leukosis[J].Shandong Journal of Animal Science and Veterinary Medicine,2023,44(11):47-49.(in Chinese)
[51] 梁晶晶.CRISPR多重基因編輯技術敲除雞TVB基因及新型多重基因編輯體系的建立[D].南寧:廣西大學,2022.
LIANG J J.Knockout of chicken TVB gene by multiplexed crispr technologies and establishment of a new multiple gene editing[D].Nanning:Guangxi University,2022.(in Chinese)
[52] 付婷婷,葉 莉,范君文,等.近年來我國動物傳染病研究現狀分析及展望[J].中國比較醫學雜志,2021,31(2):107-113.
FU T T,YE L,FAN J W,et al.The research status of infectious diseases in domestic animals[J].Chinese Journal of Comparative Medicine,2021,31(2):107-113.(in Chinese)
[53] XU K,ZHOU Y R,MU Y L,et al.CD163 and pAPN double-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance[J].eLife,2020,9:e57132.
[54] ZHANG X X,GUO C H.Recent advances in inhibition of porcine reproductive and respiratory syndrome virus through targeting CD163[J].Front Microbiol,2022,13:1006464.
[55] ZHU J Q,HE X,BERNARD D,et al.Identification of new compounds against PRRSV infection by directly targeting CD163[J].J Virol,2023,97(5):e0005423.
[56] BURKARD C,OPRIESSNIG T,MILEHAM A J,et al.Pigs lacking the scavenger receptor cysteine-rich domain 5 of CD163 are resistant to porcine reproductive and respiratory syndrome virus 1 infection[J].J Virol,2018,92(16):e00415-18.
[57] BURKARD C,LILLICO S G,REID E,et al.Precision engineering for PRRSV resistance in pigs:macrophages from genome edited pigs lacking CD163 SRCR5 domain are fully resistant to both PRRSV genotypes while maintaining biological function[J].PLoS Pathog,2017,13(2):e1006206.
[58] WELCH S K W,CALVERT J G.A brief review of CD163 and its role in PRRSV infection[J].Virus Res,2010,154(1-2):98-103.
[59] PRATHER R S,WELLS K D,WHITWORTH K M,et al.Knockout of maternal CD163 protects fetuses from infection with porcine reproductive and respiratory syndrome virus (PRRSV)[J].Sci Rep,2017,7(1):13371.
[60] CHENG S P,WU H G,CHEN Z H.Evolution of transmissible gastroenteritis virus (TGEV):a codon usage perspective[J].Int J Mol Sci,2020,21(21):7898.
[61] CHEN J W,PAN K Y,CHEN Z,et al.Production of porcine aminopeptidase N (pAPN) site-specific edited pigs[J].Anim Sci J,2019,90(3):366-371.
[62] 李寶賢,馬廣鵬,葛俊偉,等.豬流行性腹瀉病毒功能性受體的鑒定[J].病毒學報,2009,25(3):220-225.
LI B X,MA G P,GE J W,et al.Porcine aminopeptidase N is a functional receptor for the PEDV coronavirus[J].Chinese Journal of Virology,2009,25(3):220-225.(in Chinese)
[63] JI C M,WANG B,ZHOU J Y,et al.Aminopeptidase-N-independent entry of porcine epidemic diarrhea virus into Vero or porcine small intestine epithelial cells[J].Virology,2018,517:16-23.
[64] LEE S J.Targeting the myostatin signaling pathway to treat muscle loss and metabolic dysfunction[J].J Clin Invest,2021,131(9):e148372.
[65] LI R Q,ZENG W,MA M,et al.Precise editing of myostatin signal peptide by CRISPR/Cas9 increases the muscle mass of Liang Guang Small Spotted pigs[J].Transgenic Res,2020,29(1):149-163.
(編輯 郭云雁)

