豬MKRN3基因的印記表達和DNA甲基化狀態分析


摘 要: 旨在探討MKRN3基因在野生型豬和克隆豬中的印記狀態及其DNA甲基化水平。本研究利用西方豬種(杜洛克豬)與本地豬種(巴馬豬或榮昌豬)中存在的種間單核苷酸多態性(single nucleotide polymorphism,SNP),檢測MKRN3基因在3只足月出生0天的野生型雜交豬中的印記狀態。利用MethPrimer網站預測MKRN3啟動子區附近的CpG島,并在卵母細胞和精子基因組中驗證CpG島是否為差異甲基化區域(differentially methylated region,DMR)。對3只野生型雜交豬和兩只克隆豬基因組DNA進行亞硫酸鹽轉化后測序,檢測MKRN3-DMR在野生型豬和克隆豬的甲基化狀態,并分析其甲基化狀態與基因表達的關系。結果表明,在MKRN3基因的編碼區域,西方豬種(杜洛克豬)與本地豬種(巴馬豬和榮昌豬)之間存在一個SNP: A/G;RT-PCR測序結果顯示,MKRN3在其中1只野生型個體(杜洛克豬與巴馬豬雜交后代:DB2)的心臟組織呈雙等位基因表達,在肝臟、脾臟、肺等器官和腦組織中均為單等位基因表達,且均表達父本來源的等位基因。MKRN3-DMR在精子中表現為低甲基化(0%),在卵母細胞中表現為高甲基化(70.9%)。雜交豬6個組織中的MKRN3-DMR平均甲基化水平分別為:心臟(43.1%)、肝臟(46.2%)、脾臟(48.4%)、肺(45.6%)、腎臟(48.5%)、腦(44.3%);在新生克隆豬的大部分組織中,MKRN3表達父本來源等位基因,MKRN3-DMR甲基化水平與野生型雜交豬相近。而在NT201和NT207脾臟中,其甲基化水平分別為79.0%和80.1%,SNP測序結果也表明在兩個克隆豬脾臟中,MKRN3不再維持印記狀態,提示克隆豬脾臟組織中MKRN3印記異常。綜上所述,MKRN3在野生型豬肝臟、脾臟、肺、腎和腦組織中呈母本印記、父本表達,在心臟為非印記狀態;克隆豬部分組織中,MKRN3印記表達和甲基化狀態均為異常;啟動子的DMR調控MKRN3表達。
關鍵詞: DNA甲基化;豬;DMR;基因組印記
中圖分類號: S828.2
文獻標志碼:A
文章編號: 0366-6964(2024)09-3853-11
Analysis of Imprinted Expression and DNA Methylation Status of the Porcine MKRN3
Gene
CHEN" Nanzhu, LI" Junliang, YU" Dawei, ZHOU" Xinyi, WANG" Jing, ZOU" Huiying*, DU" Weihua*
(Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China)
Abstract:" The aim of this study was to investigate the imprinting status of the MKRN3 gene and its DNA methylation level in wild type and cloned pigs. The imprinting status of the MKRN3 gene in the 3 wild-type crossbred pigs, which were born at full term, were examined using interspecies single nucleotide polymorphism(SNP) present in Western (Duroc) and local (Bama or Rongchang) pig breeds. CpG islands near the MKRN3 promoter region were predicted using the MethPrimer website, and CpGs were verified as differentially methylated region (DMR) in oocyte and sperm genomes. The genomes of the 3 wild-type hybrid and 2 cloned pigs were sequenced after bisulfite transformation to detect the methylation status of MKRN3-DMR in wild-type and cloned pigs, and to analyze the relationship between methylation status and expression. The results showed that a single nucleotide polymorphism (SNP) existed in the coding region of the MKRN3 gene between Western (Duroc) and local (Bama and Rongchang) pig breeds: A/G; RT-PCR sequencing showed that MKRN3 was bi-allelic expressed in the heart tissue of one of the wild-type individual (offspring of a cross between Duroc pig and Bama pig: DB2), and monoallelic expressed in organs such as liver, spleen, lungs, and brain tissues, and all of them expressed the allele from paternal origin. MKRN3-DMR showed hypomethylation (0%) in spermatozoa and showed hypermethylation (70.9%) in oocytes. The average methylation levels of MKRN3-DMR in the six tissues of hybrid pigs were: heart (43.1%), liver (46.2%), spleen (48.4%), lung (45.6%), kidney (48.5%), and brain (44.3%), respectively. In most tissues of the newborn cloned pigs, MKRN3 was expressed as the allele of paternal origin, and the level of MKRN3-DMR methylation was similar to that of the wild-type crossbred pigs. While MKRN3-DMR methylation assay results showed that the methylation levels were 79.0% and 80.1% in spleen and spleen of NT201 and NT207, respectively, SNP sequencing results also indicated that the imprinted state was no longer maintained in the two cloned pig spleens. It is suggested that MKRN3 imprinting status was abnormal in cloned pig spleen tissues. In summary, MKRN3 was maternally imprinted and paternally expressed in liver, spleen, lung, kidney and brain tissues of wild-type pigs, and was in a non-imprinted state in the heart; MKRN3 imprinted expression and methylation status were abnormal in some tissues of the cloned pigs. The expression of MKRN3 was regulated by the DMR of the promoter.
Key words: DNA methylation; porcine; DMR; genomic imprinting
*Corresponding authors:DU Weihua, E-mail:duweihua@caas.cn; ZOU Huiying, E-mail:zouhuiying@caas.cn
基因組印記是一種獨特的遺傳現象,具體表現為基因轉錄活性僅局限于來自單親的等位基因,而對應另一個親本來源的等位基因則處于轉錄沉默的狀態,這類基因被定義為印記基因[1]。印記基因不僅在胚胎發育階段發揮重要作用,其表達異常還會對個體出生后的代謝功能、神經系統發育等方面產生深遠影響[2-3]。印記基因偏好成簇存在,并且接受順式作用元件的調控[4-5]。印記基因表達的調控區域受到多種表觀遺傳修飾的影響,包括DNA甲基化、組蛋白修飾以及各類轉錄因子的相互作用;其中,DNA甲基化起主導作用[6-10]。與印記基因表達的親本特異性一致,調控區域的甲基化狀態同樣具有親本來源特異性,即父源或母源染色體上調控區域的甲基化狀態不同,因此被稱為差異甲基化區域(differentially methylated region,DMR)[11]。在受精后發生的表觀遺傳重編程進程中,DMR甲基化的動態建立與擦除可分為兩個階段性過程。受精后,早期胚胎的父源基因組發生主動去甲基化,而母源基因組則發生被動去甲基化;然而,印記基因DMR區域的甲基化狀態保持穩定,不受去甲基化過程的影響。其次,在多能性細胞分化成原始生殖細胞(primordial germ cell,PGC)后,PGC中印記基因的DMR和轉座子等區域發生甲基化的擦除和重建[12]。
MKRN3基因位于調控普拉德維利綜合征(Prader Willi syndrome,PWS)的印記簇上,已在多個物種如小鼠、人、牛和羊中被證明是母源印記、父源表達[13-15]。該基因編碼的MKRN3蛋白含有鋅指結構域,能夠將甲基結合蛋白3(methyl-DNA binding protein 3, MBD3)泛素化。MDB3蛋白能夠與促性腺激素釋放激素(gonadotropin releasing hormone,GnRH)啟動子結合,并募集去甲基化酶(ten-eleven-translocation protein,TET)擦除GNRH啟動子區域的甲基化修飾,從而調節GNRH的表達。在人生長發育過程中,MKRN3基因的過度表達會導致泛素化的MBD3蛋白水平升高,進而引發GNRH表達量減少,這種生理變化與兒童身材矮小、智力發育遲滯的呆小癥癥狀密切相關[16-17]。此外,MKRN3基因與人中樞性早熟(central precocious puberty,CPP)的發病機制相關。小鼠MKRN3蛋白的過表達會致使雌性小鼠的青春期延遲,而牛MKRN3基因在青春期前后表達水平存在顯著差異[18]。
在體細胞核移植后,供體細胞核會經歷一次表觀重編程。體細胞基因組特有的甲基化、組蛋白修飾會被重編程,X染色體重新激活;而在這個過程中,印記基因需要維持原有的表觀修飾狀態[19-20]。諸多研究表明,克隆個體中印記基因的表達模式常發生偏差,且這種偏差與DMR的異常甲基化水平相關[21]。在猴體細胞核移植胚胎中,全基因組的DNA甲基化水平下降,母源印記基因的印記狀態丟失[22]。在克隆豬中,調控DLK1-DIO3區域的DMR表現高甲基化,同時該區域的基因如GTL2等表達異常[23]。本研究利用不同豬種間存在的單核苷酸多態性(single nucleotide polymorphism,SNP),對野生型豬和克隆豬不同組織中的MKRN3進行印記狀態分析,并探究MKRN3 DMR甲基化狀態,揭示MKRN3在野生型豬和克隆豬中的印記表達規律,為提高克隆豬效率提供理論基礎。
1 材料與方法
1.1 試驗材料
本試驗使用的3只野生型雜交豬(DB1、DB2和DB3)是由杜洛克豬(公豬)和巴馬豬(母豬)雜交獲得的。克隆豬NT201和NT207的體細胞為妊娠30 d的杜洛克豬(公豬)與榮昌豬(母豬)雜交后代(RD)的胎兒成纖維細胞。試驗用到的組織樣本包括心臟、肝臟、脾臟、肺、腎臟等器官和腦組織,分別來自出生0 d的野生型雜交仔豬和新生克隆仔豬。
1.2 試驗方法
1.2.1 不同組織基因組提取
獲得的野生型豬和克隆豬不同組織保存于液氮中,基因組提取步驟參照QIAamp DNA Mini Kit(QIAGEN)制造商提供的標準操作程序進行處理,提取后取1 μL檢測濃度并記錄。
1.2.2 不同組織RNA提取和RT-PCR
在組織樣本中加入1 mL Trizol(Invitrogen)和適量研磨磁珠,使用超低溫研磨儀研磨8 min。隨后加入200 μL氯仿進行相分離,收集上層水相,加入600 μL異丙醇進行沉淀。沉淀物用1 mL乙醇洗滌后,溶解于20 μL DEPC處理過的水中,并儲存在-80℃條件下。RT-PCR反應試劑來自PrimeScript RT reagent Kit(TaKaRa),按照制造商的說明進行操作。
1.2.3 SNP檢測
在ENSEMBL數據庫檢索Sus scrofa 11.1基因組,獲取MKRN3基因外顯子區域的序列。設計特異性引物(表1),對不同品種豬MKRN3外顯子進行全長的分段PCR擴增,擴增產物送往北京華大六合進行測序。分析測序結果中峰圖的雙峰位置:在雜交豬測序中表現為雙峰,但是父母本中為單峰的即為候選SNP。擴增體系(25 μL):KOD OneTM PCR Master Mix 12.5 μL,H2O 9.5 μL,上、下游引物(10 μmol·L-1)各1 μL,模板1 μL。擴增程序:98℃預變性3 min;98℃變性,55℃退火15 s,68℃延伸15 s,循環35次;68℃延伸5 min,儲存于4℃或-20℃。
1.2.4 印記狀態分析
在MKRN3基因的蛋白編碼區(coding sequence,CDS)重新設計特異性引物(表1),以野生型雜交豬和克隆豬的不同組織cDNA為模板,以25 μL體系進行擴增,將擴增產物送北京六合華大測序,對測序結果中SNP的峰圖進行分析,表現為父源堿基則為父源表達,母源印記,否則為父源印記。
1.2.5 亞硫酸鹽轉化
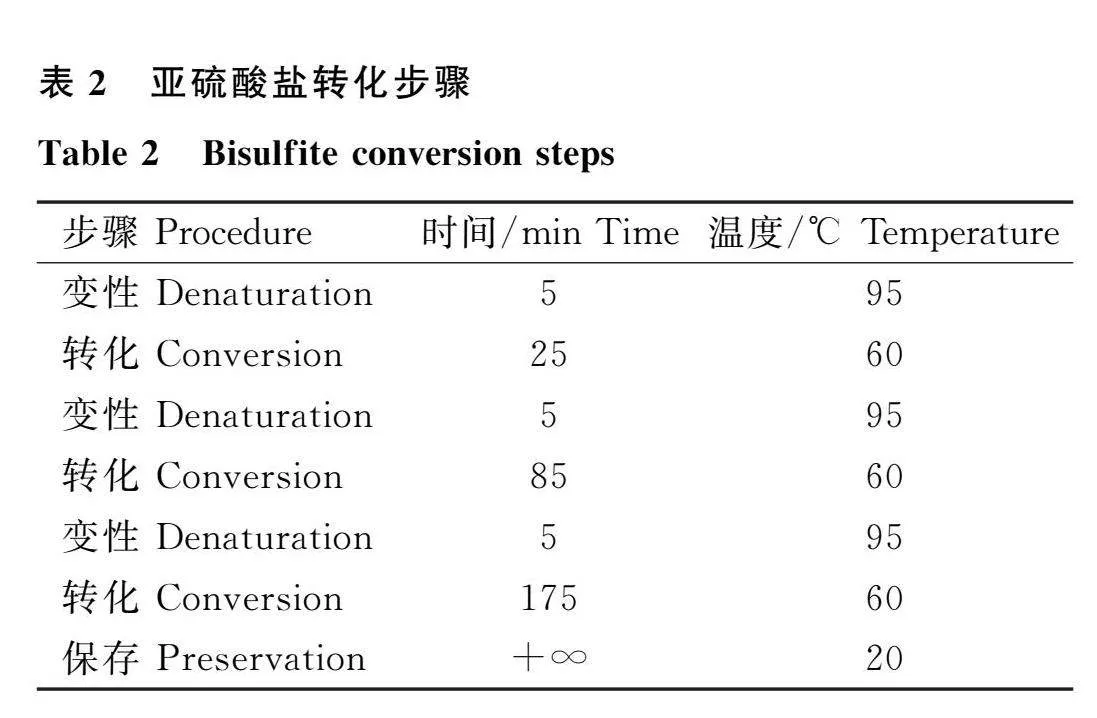
取1 μg的各組織基因組DNA,加入85 μL亞硫酸氫鹽混合物和35 μL DNA保護緩沖液,按照表2中的程序進行處理。后續洗脫步驟參照EpiTect Bisulfite Kit(QIAGEN)進行。
1.2.6 甲基化水平分析
使用特異性甲基化測序引物(表1)對MKRN3-DMR進行巢式擴增,擴增產物使用E.Z.N.A Gel Extraction Kit(Omega Bio-tek)純化。純化后的產物與TOPO-Blunt Cloning Mix中載體連接:37℃反應5 min后,置于冰上。取33 μL DH5α感受態細胞加入以上反應液中,置于冰上30 min,42℃熱激30 s,迅速置于冰上2 min。將大腸桿菌混合物均勻涂到帶有氨芐抗性的培養基上,待其吸收完后倒置于37℃培養箱中過夜生長。次日選取至少8個單克隆進行菌落PCR,將PCR產物送北京六合華大測序。測序結果使用"" 甲基化分析量化工具(QUMA; http://quma.cdb.rikenjp/)分析甲基化狀態。
2 結 果
2.1 豬MKRN3基因印記表達分析
2.1.1 豬MKRN3基因的SNP檢測
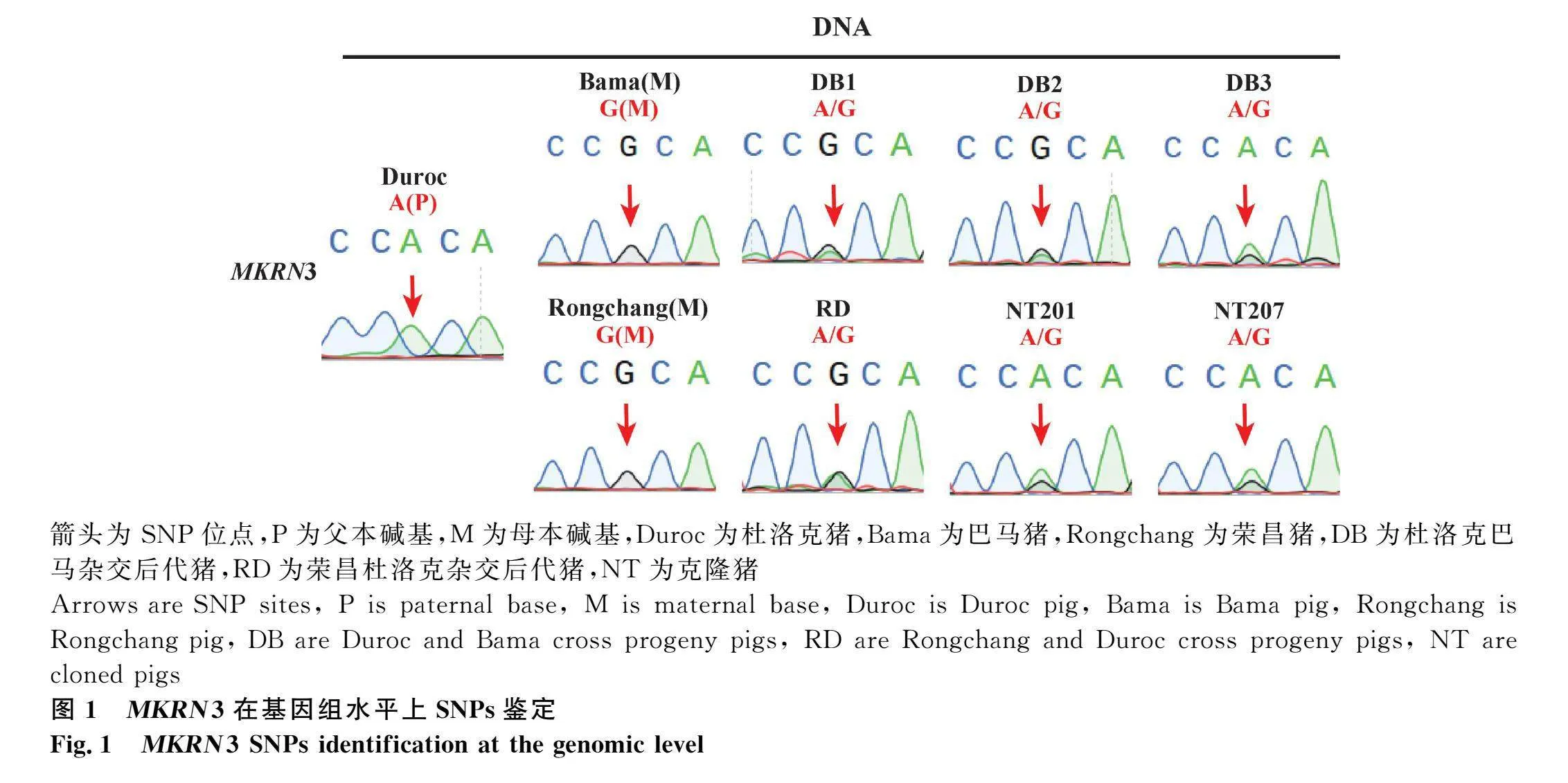
為探究MKRN3基因在3種豬種間是否存在SNP,針對MKRN3外顯子DNA序列設計引物(表1),使得擴增產物能夠完整覆蓋CDS序列。以杜洛克(Duroc)、巴馬豬(Bama)、榮昌豬(Rongchang)及其雜交豬胎兒成纖維細胞(RD)、克隆豬(NT)和雜交豬腦組織(DB)的基因組DNA為模板,進行擴增測序,檢測MKRN3基因是否存在SNP。結果顯示,在杜洛克豬與巴馬豬、榮昌豬的基因組中鑒別出一個SNP位點(圖1)。該SNP位于1∶14249695(A/G變異),位于轉錄起始位點的第764 bp。
2.1.2 豬MKRN3基因在不同組織的印記狀態分析
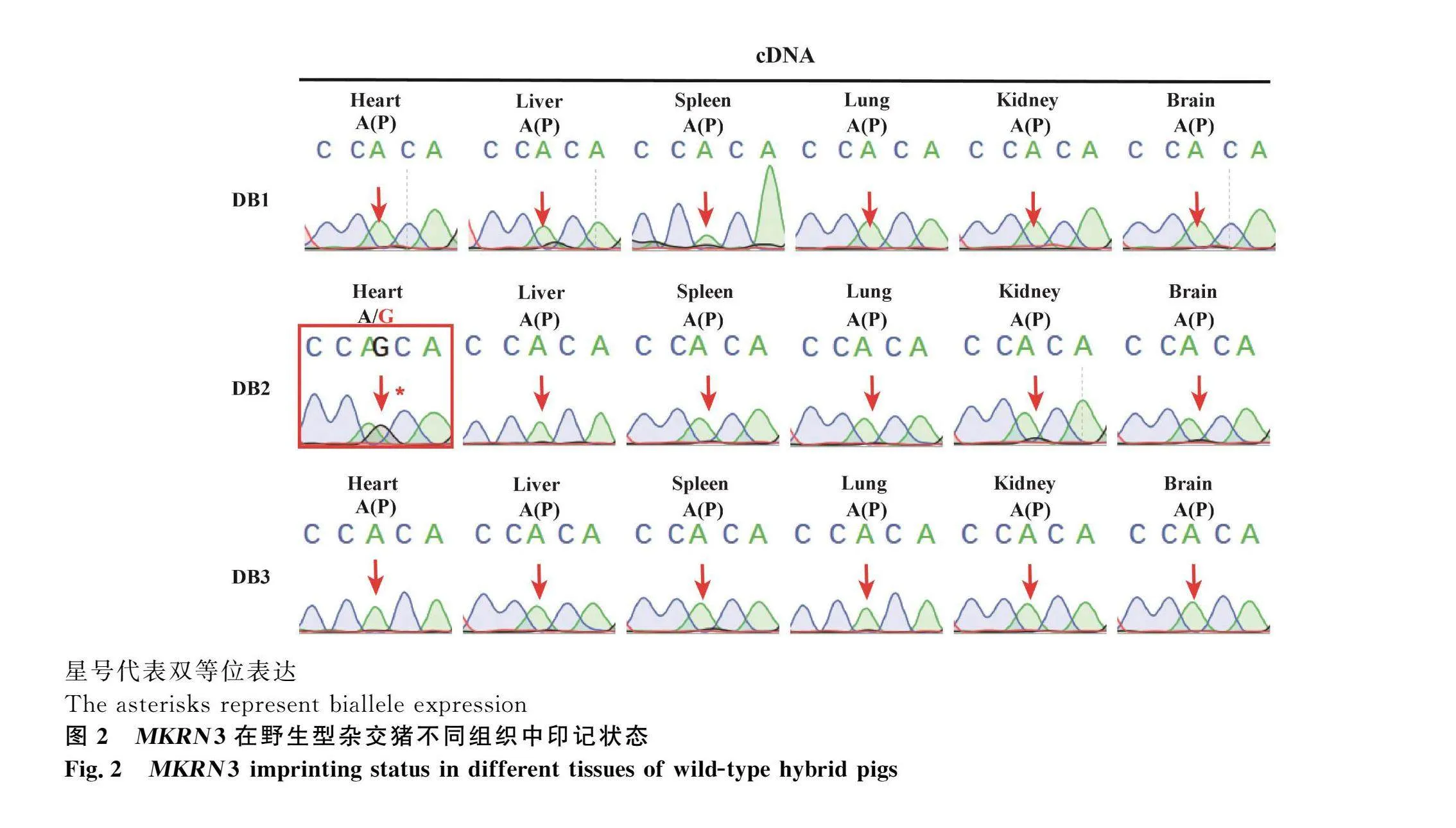
在對雜交豬MKRN3基因印記狀態的研究中,本試驗重新在候選SNP附近設計引物(cDNA-SNP1F和cDNA-SNP1R),對野生型雜交豬DB1、DB2和DB3的不同組織樣本進行深入分析。首先,從各組織中提取總RNA,并通過逆轉錄反應生成cDNA,以此作為模板進行擴增包含候選SNP位點的169 bp片段。隨后,對PCR產物進行Sanger測序以獲取序列信息。測序結果表明,在肝臟、脾臟、肺、腎臟以及腦組織中,MKRN3基因的SNP位點均呈現單一峰,表明這些組織中表達的是父本來源的等位基因(A)。然而,MKRN3基因在雜交豬DB2的心臟中表現為雙峰(A/G),表明存在母本等位基因的表達;而DB1和DB3個體的心臟組織中,SNP位點則顯示單一峰,即父本等位基因表達(A)(圖2)。可見,在不同個體的心臟組織中,MKRN3基因的表達模式可能存在差異。綜上所述,在野生型雜交豬的多數組織中,MKRN3基因的表達模式為父本特異性印記;而在心臟組織中,該基因的印記模式存在個體差異。
2.2 豬MKRN3基因甲基化狀態分析
2.2.1 豬MKRN3基因的CpG預測
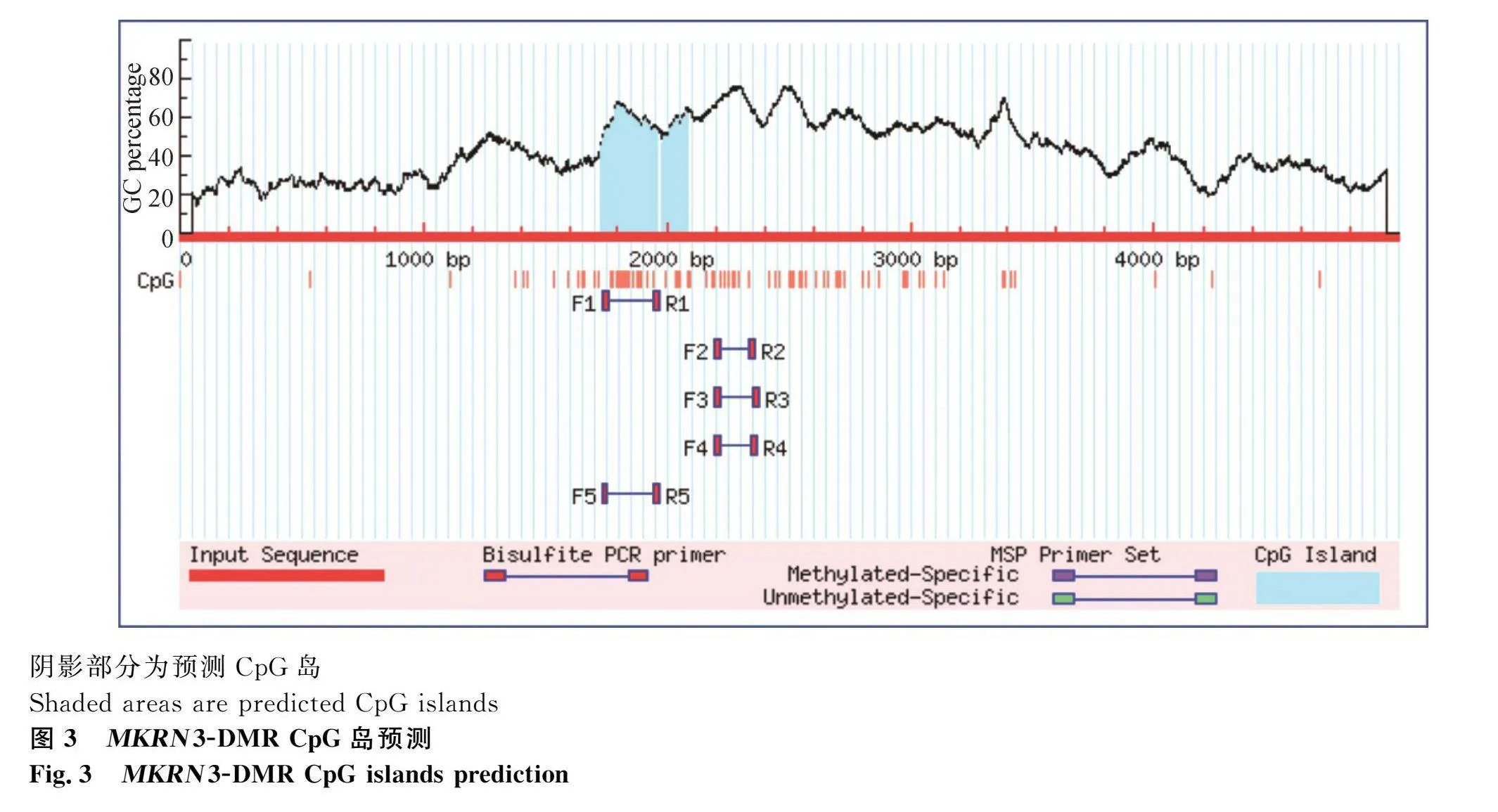
利用Methprimer網站(https://www.urogene.org/methprimer/)的在線工具,對第一外顯子上游2 000 bp以及下游3 000 bp,總計5 000 bp的區域進行生物信息學分析。結果發現,MKRN3啟動子附近存在兩個CpG島,分別命名為CpG島1(-274 bp~-35 bp,239 bp)和CpG島2(-17 bp~+84 bp,102 bp)。由于兩個CpG島在基因組中的相對位置較為接近,因此合并兩個CpG島作為一個潛在的DMR進行試驗驗證(圖3)。
2.2.2 豬MKRN3基因的甲基化狀態分析
對合并后的DMR設計兩對特異性引物(MKRN3-WF1、 MKRN3-WR1, MKRN3-NF1、 MKRN3-NR1),用以分析豬MKRN3基因的甲基化狀態。在對精子、卵母細胞以及野生型雜交豬的不同組織器官進行基因組甲基化處理之后進行聚合酶鏈式反應,對擴增反應產物測序。研究結果揭示,在精子中MKRN3 DMR區域呈現低甲基化狀態(0%),而在卵母細胞中則觀察到高甲基化狀態(70.9%),這一現象與差異甲基化的特征相符合(圖4)。進一步地,在野生型雜交豬的不同組織中,MKRN3-DMR區域表現出差異性的甲基化模式:一半的鏈表現為高甲基化的狀態,另一半的鏈表現為低甲基化的狀態,總體呈現50%左右的甲基化水平。其中,心臟組織的平均甲基化水平約為43.1%,肝臟為46.2%,脾臟為48.4%,肺為45.6%,腎臟為48.5%,腦組織為44.3%(圖4)。
精子和卵母細胞中觀察到的差異甲基化模式表明,所鑒定的CpG富集區域為豬MKRN3基因的DMR。同時,在各種組織中DMR區域維持中等水平的甲基化狀態,這也進一步證實該區域作為MKRN3基因調控的關鍵DMR區域。
2.3 克隆豬MKRN3基因印記異常
2.3.1 克隆豬MKRN3基因的印記狀態分析
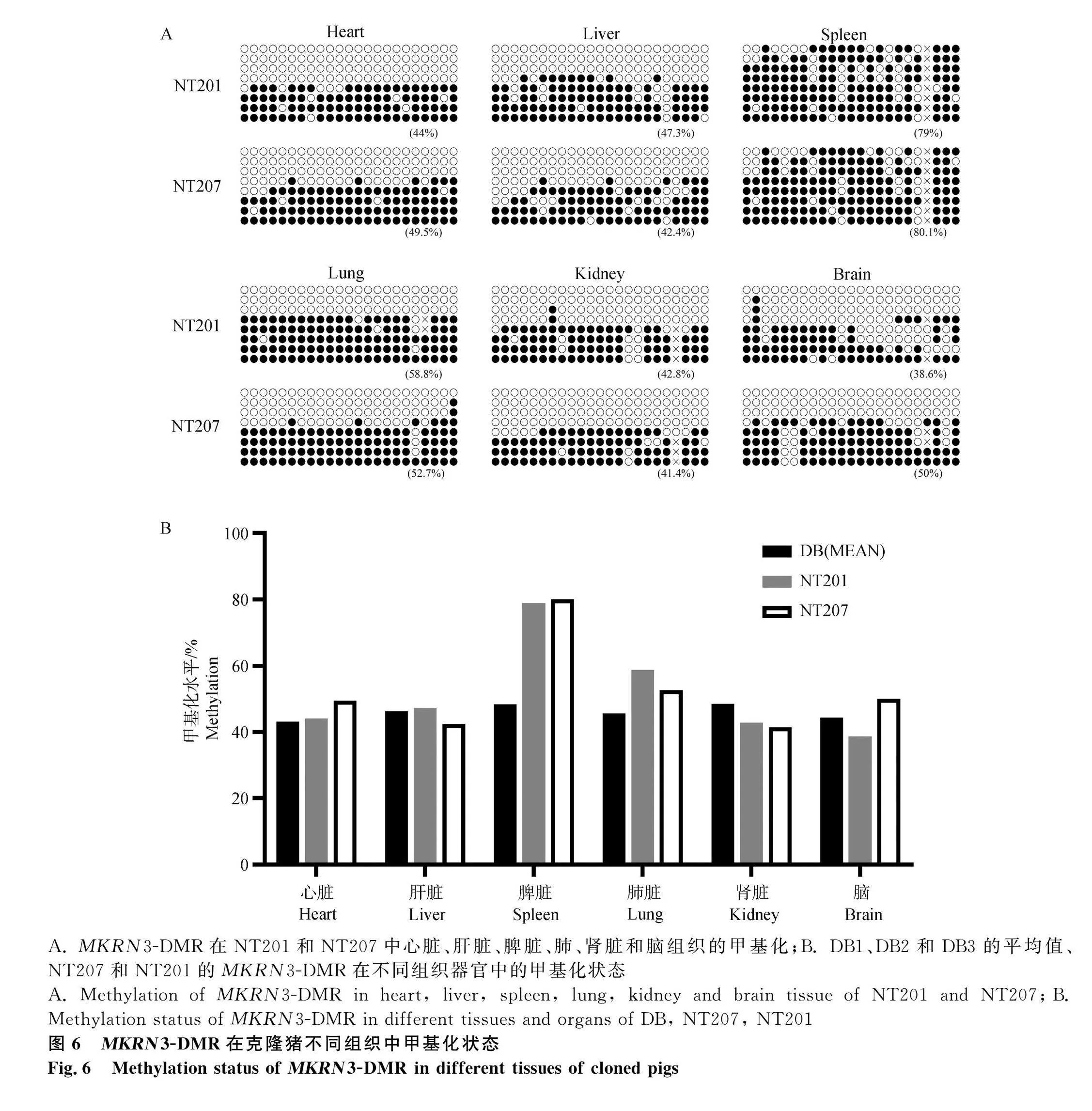
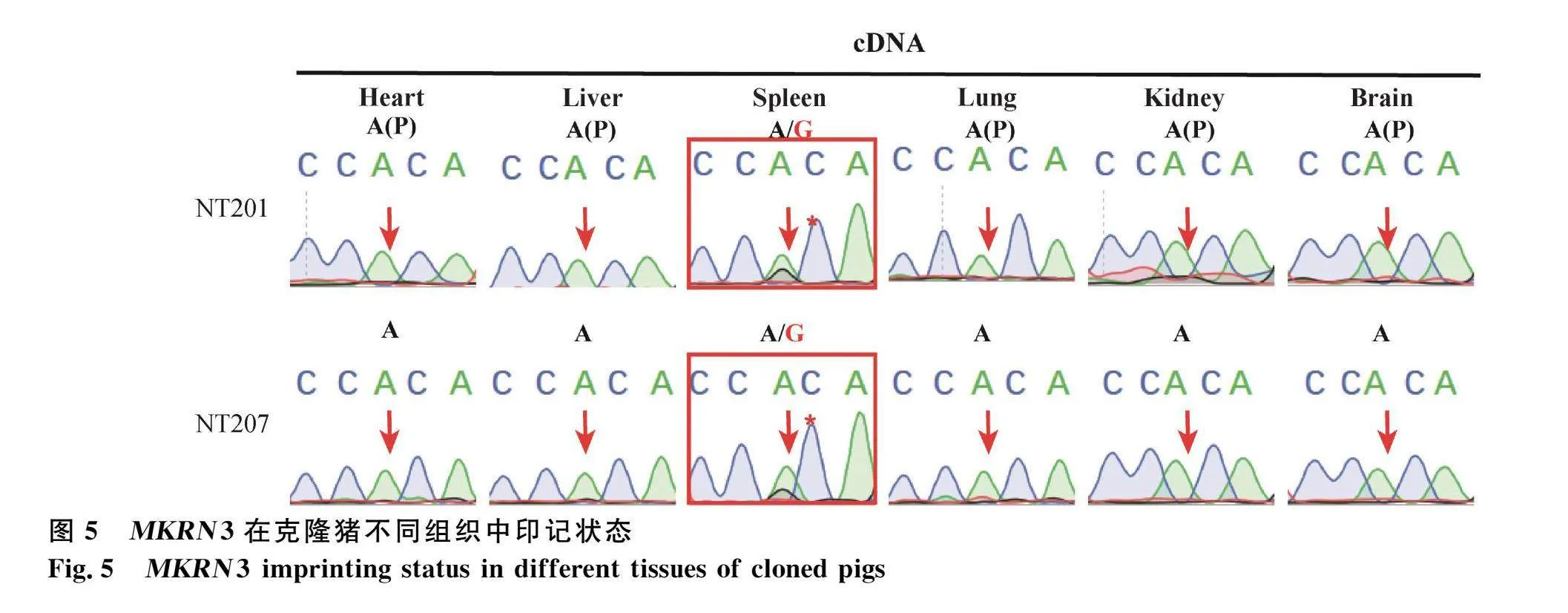
為了探究克隆豬MKRN3基因的表達印記狀態,本研究從兩只克隆豬的不同組織中提取總RNA。將這些RNA通過逆轉錄反應轉化為cDNA,并以此作為模板,使用特定的引物(表1)針對MKRN3基因上的SNP位點進行PCR,并對擴增產物進行測序分析。測序結果表明,在肝臟、肺、腎臟和腦組織中,MKRN3基因的SNP位點均表現為單一峰(圖5),且所表達的堿基均為父本來源(A),這與野生型雜交豬中的印記模式相一致。然而,在脾臟組織中,MKRN3基因的SNP位點則呈現出雙峰(A/G),表明在克隆豬的脾臟組織中,MKRN3基因的印記狀態不同于野生型雜交豬(圖5)。因此,在克隆豬的肝臟、肺、腎臟和腦組織中,MKRN3基因的表達印記狀態得到保持,但在某些組織如脾臟中,印記狀態丟失。
2.3.2 克隆豬MKRN3基因的甲基化狀態分析
在探究印記基因表達調控機制的過程中,DNA甲基化被廣泛認為是關鍵的表觀遺傳修飾方式。因此,本研究旨在在克隆豬心臟、肝臟、脾臟、腎臟、肺和腦組織中對MKRN3基因的DMR甲基化水平進行系統檢測。試驗結果顯示,在表達異常的兩只克隆豬脾臟中,MKRN3的DMR區域出現異常的高甲基化現象(圖6A),甲基化水平分別為79%和80.1%,明顯高于野生型雜交豬脾臟組織的甲基化水平(圖6B)。然而,在心臟、肝臟、腎臟、肺和腦組織中MKRN3 DMR的甲基化水平與野生型雜交豬的相應組織水平相當(圖6B)。因此,克隆豬脾臟組織中MKRN3基因表達異常與DMR區域異常高甲基化相關,而其他組織中該基因的DMR甲基化狀態正常,并維持印記表達的模式。
3 討 論
印記基因的表達模式在不同物種間通常具有較高的保守性。例如,在人類、小鼠、牛和羊等物種中,MKRN3基因在心臟、肝臟和腦等關鍵組織器官中均表現為印記狀態。然而,本研究發現MKRN3基因在豬的不同組織中的印記狀態并不一致,特別是在心臟組織中MKNR3表現為非印記狀態,這種差異性可能與印記基因所具有的組織特異性表達模式相關,同時也突顯了不同個體中印記基因表達調控的復雜性。小鼠Slc38a4基因在胎盤中表現出印記狀態,轉錄的蛋白質承擔轉運氨基酸的功能;然而牛Slc38a4基因并非印記基因[24-27]。因此,即使印記基因在進化過程中具有一定的保守性,但其表達模式和功能在不同物種間仍存在顯著差異。對印記基因的跨物種比較研究有助于深化對其保守性和多樣性的理解。
經典印記基因的調控機制中,差異甲基化區域扮演著至關重要的角色。小鼠和人Nespas基因的DMR被認為是Gnas印記簇的調控中心,負責調節兩個反義基因Gnasxl和Gnas的表達,類似于一個“開關”[28]。在不同組織中DMR的甲基化水平可能普遍維持在中等程度,但大多數印記基因的表達具有明顯的組織特異性,表明可能有別的機制參與印記基因組織特異性的調控,如組蛋白乙酰化參與人NDN基因的組織特異的表達[29]。
本研究中,克隆豬脾臟的MKRN3 DMR甲基化出現異常,該基因也呈現出雙重表達的異常現象,表明DNA甲基化在MKRN3基因表達調控中的關鍵作用。在野生型豬中,MKRN3 DMR的差異甲基化狀態與其單等位基因表達的印記狀態一致,說明DMR甲基化水平是維持正常印記狀態的關鍵因素。這些結果揭示了在印記基因表達調控機制中,DMR的甲基化狀態作為調控中心,精確控制父母源等位基因的表達模式[30-31]。克隆豬和野生型豬中的發現提示,DMR甲基化水平的改變可能是導致印記狀態異常的關鍵因素,這對于理解克隆動物的發育異常和疾病發生至關重要。研究顯示,多能性基因POU5F1 DMR在克隆牛中出現異常高甲基化,導致POU5F1表達下降[32]。在克隆牛胎兒的肌肉、心臟和肝臟等組織中,IGF2R基因表達的降低引起胎兒異常增大,類似于Beckwith-Wiedemann綜合征中由IGF2基因印記異常引起的胎兒過度生長[33]。克隆豬DLK1-DIO3的IG-DMR甲基化水平異常升高,導致其出生死亡[23]。另一方面,卵母細胞在去核化前已具備完整的雌性配子表觀遺傳修飾,這可能對其重編程能力產生影響[34]。
由于試驗樣本有限,本研究僅能對6個組織的印記狀態進行分析。胎盤組織是一個依賴印記基因調控生長的器官,本試驗缺少胎盤組織,因此也不能對MKRN3在胎盤的印記狀態進行分析[35-36]。此外,除了DNA甲基化參與印記基因表達的調控之外,各種組蛋白修飾以及染色質開放程度等因素也是未來進一步研究的重點。
4 結 論
在豬的肝臟、脾臟、肺、腎臟以及腦組織中,MKRN3基因表現為母源印記基因,即主要表達父本等位基因;然而,在心臟組織中,該基因的印記表達模式在不同個體之間不一致,并非嚴格遵循典型的母源印記模式。位于啟動子附近的CpG島為負責調控MKRN3表達的DMR;克隆豬部分組織中MKRN3的DMR甲基化異常,進而導致基因的異常表達。本研究揭示了野生型豬和克隆豬中MKRN3的印記狀態,為通過修復異常的印記基因提高克隆效率提供理論基礎。
參考文獻(References):
[1] LIAO J,SONG S M,GUSSCOTT S,et al.Establishment of paternal methylation imprint at the H19/Igf2 imprinting control region[J].Sci Adv,2023,9(36):eadi2050.
[2] PLASSCHAERT R N,BARTOLOMEI M S.Genomic imprinting in development,growth,behavior and stem cells[J]. Development,2014,141(9):1805-1813.
[3] CARRION S A,MICHAL J J,JIANG Z H.Imprinted genes:genomic conservation,transcriptomic dynamics and phenomic significance in health and diseases[J].Int J Biol Sci,2023,19(10):3128-3142.
[4] MONK D,MACKAY D J G,EGGERMANN T,et al.Genomic imprinting disorders:lessons on how genome, epigenome and environment interact[J].Nat Rev Genet,2019,20(4):235-248.
[5] PENG X Q,LUO H Z,KONG X Y,et al.Metrics for evaluating differentially methylated region sets predicted from BS-seq data[J].Brief Bioinform,2022,23(1):bbab475.
[6] MCEWEN K R,FERGUSON-SMITH A C.Distinguishing epigenetic marks of developmental and imprinting regulation[J].Epigenetics Chromatin,2010,3(1):2.
[7] ARNAUD P.Genomic imprinting in germ cells:imprints are under control[J].Reproduction,2010,140(3):411-423.
[8] ZALETAEV D V,NEMTSOVA M V,STRELNIKOV V V.Epigenetic regulation disturbances on gene expression in imprinting diseases[J].Mol Biol (Mosk),2022,56(1):1-28.
[9] LUO X M,ZHANG T J,ZHAI Y X,et al.Effects of DNA methylation on TFs in human embryonic stem cells[J]. Front Genet, 2021,12:639461.
[10] STEINHOFF C,PAULSEN M,KIELBASA S,et al.Expression profile and transcription factor binding site exploration of imprinted genes in human and mouse[J].BMC Genomics,2009,10:144.
[11] ECKHARDT F,LEWIN J,CORTESE R,et al.DNA methylation profiling of human chromosomes 6,20 and 22[J].Nat Genet,2006,38(12):1378-1385.
[12] SINGH A,RAPPOLEE D A,RUDEN D M.Epigenetic reprogramming in Mice and Humans:from fertilization to primordial germ cell development[J].Cells,2023,12(14):1874.
[13] FANIS P,MORROU M,TOMAZOU M,et al.Methylation status of hypothalamic Mkrn3 promoter across puberty[J]. Front Endocrinol (Lausanne),2023,13:1075341.
[14] LI J L,CHEN W N,LI D J,et al.Conservation of imprinting and methylation of MKRN3,MAGEL2 and NDN genes in cattle[J].Animals (Basel),2021,11(7):1985.
[15] 張永杰.多浪羊MKRN3、NDN、MAGEL2基因的印記和甲基化狀態分析[D].阿拉爾市:塔里木大學,2023.
ZHANG Y J.Analysis of imprinting and methylation status of MKRN3,NDN and MAGEL2 genes in Dolomite sheep[D].Alaer:Tarim University,2023.(in Chinese)
[16] LI C Y,LU W L,YANG L G,et al.MKRN3 regulates the epigenetic switch of mammalian puberty via ubiquitination of MBD3[J].Natl Sci Rev,2020,7(3):671-685.
[17] LI C Y,HAN T T,LI Q R,et al.MKRN3-mediated ubiquitination of Poly(A)-binding proteins modulates the stability and translation of GNRH1 mRNA in mammalian puberty[J].Nucleic Acids Res,2021,49(7):3796-3813.
[18] KARAMI K,ZEREHDARAN S,JAVADMANESH A.Differential expression of RNAseq imprinted genes from bovine females before and after puberty[J].Biochem Genet,2023,61(6):2633-2649.
[19] NIEMANN H,TIAN X C,KING W A,et al.Epigenetic reprogramming in embryonic and foetal development upon somatic cell nuclear transfer cloning[J].Reproduction,2008,135(2):151-163.
[20] RUAN D G,PENG J Y,WANG X S,et al.XIST derepression in active X chromosome hinders pig somatic cell nuclear transfer[J].Stem Cell Rep,2018,10(2):494-508.
[21] OKAE H,MATOBA S,NAGASHIMA T,et al.RNA sequencing-based identification of aberrant imprinting in cloned mice[J].Hum Mol Genet,2014,23(4):992-1001.
[22] LIAO Z D,ZHANG J X,SUN S Y,et al.Reprogramming mechanism dissection and trophoblast replacement application in monkey somatic cell nuclear transfer[J].Nat Commun,2024,15(1):5.
[23] LI J L,YU D W,WANG J,et al.Identification of the porcine IG-DMR and abnormal imprinting of DLK1-DIO3 in cloned pigs[J].Front Cell Dev Biol,2022,10:964045.
[24] ZAITOUN I,KHATIB H.Assessment of genomic imprinting of SLC38A4,NNAT,NAP1L5,and H19 in cattle[J].BMC Genet,2006,7:49.
[25] KADIFE E,HARPER A,DE ALWIS N,et al.SLC38A4 amino acid transporter expression is significantly lower in early preterm intrauterine growth restriction complicated placentas[J].Int J Mol Sci,2022,24(1):403.
[26] XIE Z,ZHANG W,ZHANG Y.Loss of Slc38a4 imprinting is a major cause of mouse placenta hyperplasia in somatic cell nuclear transferred embryos at late gestation[J].Cell Rep,2022,38(8):110407.
[27] HALUKOV "J,HOLE ACˇG KOV "B,STANI ACˇG OV "J.DNA methylation studies in cattle[J].J Appl Genet,2021,62(1):121-136.
[28] IWASAKI Y,AKSU C,REYES M,et al.The long-range interaction between two GNAS imprinting control regions delineates pseudohypoparathyroidism type 1B pathogenesis[J].J Clin Invest,2023,133(8):e167953.
[29] LAU J C,HANEL M L,WEVRICK R.Tissue-specific and imprinted epigenetic modifications of the human NDN gene[J].Nucleic Acids Res,2004,32(11):3376-3382.
[30] BARLOW D P,BARTOLOMEI M S.Genomic imprinting in mammals[J].Cold Spring Harb Perspect Biol,2014,6(2):a018382.
[31] LIANG D,AYGN N,MATOBA N,et al.Inference of putative cell-type-specific imprinted regulatory elements and genes during human neuronal differentiation[J].Hum Mol Genet,2023,32(3):402-416.
[32] JOZI M,JAFARPOUR F,MORADI R,et al.Induced DNA hypomethylation by folic acid deprivation in bovine fibroblast donor cells improves reprogramming of somatic cell nuclear transfer embryos[J].Sci Rep,2020,10(1):5076.
[33] GOLDKAMP A K,LI Y H,RIVERA R M,et al.Differentially expressed tRNA-derived fragments in bovine fetuses with assisted reproduction induced congenital overgrowth syndrome[J].Front Genet,2022,13:1055343.
[34] VARGAS L N,CAIXETA F M C,DODE M A N,et al.DNA methylation profile of single in vitro matured bovine oocytes[J].Mol Reprod Dev,2023,90(4):227-235.
[35] INOUE K,OGONUKI N,KAMIMURA S,et al.Loss of H3K27me3 imprinting in the Sfmbt2 miRNA cluster causes enlargement of cloned mouse placentas[J].Nat Commun,2020,11(1):2150.
[36] CHU C,ZHANG W H,KANG Y,et al.Analysis of developmental imprinting dynamics in primates using SNP-free methods to identify imprinting defects in cloned placenta[J].Dev Cell,2021,56(20):2826-2840.e7.
(編輯 郭云雁)

